水稻矮化小穗基因DSP2的鉴定与克隆
2022-03-17裘霖琳刘窍付亚萍刘文真胡国成翟玉凤庞礴汪得凯
裘霖琳 刘窍 付亚萍 刘文真 胡国成 翟玉凤 庞礴 汪得凯,*
水稻矮化小穗基因的鉴定与克隆
裘霖琳1刘窍1付亚萍2刘文真2胡国成2翟玉凤1庞礴1汪得凯1,*
(1浙江理工大学 生命科学与医药学院,杭州 310018;2中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;*通信联系人,E-mail: kay77@163.com)
【】水稻株高和穗型在产量形成中发挥着重要作用。鉴定与克隆水稻株高和穗型发育相关基因,可以丰富水稻株高穗型发育调控的分子机理,为分子设计育种奠定理论基础和提供基因资源。【】在粳稻日本晴T-DNA插入群体中筛选到矮化小穗突变体,对其主要农艺性状进行了分析;采用图位克隆法结合T-DNA标签分离法进行了基因定位和克隆;利用半定量PCR和qRT-PCR进一步确定的候选基因;遗传转化实验验证了的功能。【】与野生型日本晴相比,突变体表现为半矮化、穗轴和枝梗明显缩短、穗型直立、千粒重降低等特征;遗传分析表明该突变体受一对不完全显性单基因控制;利用图位克隆将定位于第2染色体标记RM208和RM7337之间,与RM3850共分离;随后遗传分析发现,T-DNA插入位点与表型共分离,利用TAIL-PCR分离T-DNA插入序列,显示T-DNA插入到上述RM208和RM7337之间的两个基因之间。RT-PCR检测发现,位于T-DNA插入位点下游的一个编码LOB家族转录因子基因的表达量明显增加,而其他5个基因的表达量变化不明显,表明该基因可能为的候选基因;在野生型日本晴中过量表达基因,转化植株出现半矮化、小穗的表型,与表型类似,从而验证了的功能。【】基因的过量表达是产生突变表型的原因;基因对水稻株高和穗长发育具有负向调控作用;为进一步丰富株高和穗型的遗传调控网络打下了基础。
水稻;矮化;穗长;突变体;基因克隆
水稻产量是由株高、茎粗、分蘖数、每穗粒数、粒形等多种农艺性状共同决定的[1-3]。矮化是水稻育种中一个有价值的农艺性状,可以减少倒伏,提高产量。20世纪60年代,以降低农作物株高、半矮化育种为特征的第一次“绿色革命”,使得全世界水稻和小麦产量水平急剧提升,为解决温饱问题和粮食安全作出了重大贡献[4, 5]。
目前,水稻中已定位的与株高相关的位点已达上百个(http://www.ricedata.cn)。水稻株高调控途径主要有激素途径和非激素途径,其中激素主要有赤霉素(gibberellin,GA)、油菜素内酯(brassinosteroid,BR)、独脚金内酯(strigolactone,SL),吲哚乙酸(3-indoleacetic acid,IAA),脱落酸(abscisic acid,ABA)和乙烯(ethylene,ETH)等,主要涉及激素的合成、代谢和信号传导。赤霉素合成和代谢途径研究较为深入,如水稻“绿色革命”基因编码GA20氧化酶2[4]。和分别编码OsGA3ox2和OsKO2,是赤霉素合成中的关键酶,突变体表型为植株半矮化,外源赤霉素可以恢复其突变表型[6, 7]。编码一个能失活GA的P450蛋白酶,调控赤霉素代谢[8]。是GA受体,突变体表型为GA不敏感[9]。、和编码油菜素内酯合成过程中的关键酶,其突变体共同特征是植株半矮化、叶色深绿、叶夹角变小[10-12]。BR信号途径中的多个基因已经被鉴定,如编码BR受体蛋白激酶,其突变体对外施BR不敏感[13]。编码GRAS家族基因,在BR信号中起正调控作用[14]。独脚金内酯(SL)是一种重要植物激素,在植株分枝和生长发育中发挥作用,如、CCD7和CCD8分别编码β-胡萝卜素异构酶、类胡萝卜素裂解双加氧酶7和类胡萝卜素裂解双加氧酶8,在独脚金内酯合成步骤中发挥重要作用,其突变体、、表现为独脚金内酯缺乏、植株矮化、分蘖增加等表型[15-17]。其中,和基因在水稻“绿色革命”中被共选择,对水稻的稳产和广适发挥了重要作用[18]。关于SLs受体及其感知机制取得了突破性进展,编码一个α/β折叠水解酶,在水解SLs过程中发生构象变化,形成D14-D3复合体,招募D53并介导其泛素化降解,触发SLs信号传导,诱导D53/SMXL2,6,7,8发生泛素化修饰介导的蛋白降解,解除对下游基因的转录抑制,调控植物株型以及作物产量[19-22]。是IAA合成过程中的关键基因,其缺失突变表现为胚致死,再生植株表现为矮化、窄叶、花器官异常等表型[23]。是Aux/IAA家族成员,受IAA诱导,过量表达表现为植株矮化、株型松散的表型,表明在IAA信号响应中发挥重要作用[24]。编码乙烯响应因子,过量表达出现矮化表型,显示茎秆长度受GA和ETH交叉调控[25]。水稻株高除了受激素调控外,还受非激素途径调控,如细胞壁发育、细胞质谷氨酰胺合成途径、RNA编辑、细胞分裂、泛素-蛋白酶体途径和脂肪酸代谢等。编码类纤维素合成酶,突变体表现为矮化,纤维素和木糖含量显著降低[26]。编码胞质谷氨酰胺合成酶,其插入突变体生长缓慢、穗变小、籽粒灌浆受阻[27]。编码knotted1同源框家族转录因子,影响细胞分裂和伸长,其缺失突变表现为矮化,表皮和维管束细胞排列不规则[28]。随着众多水稻矮化基因位点的发掘,对控制水稻株高性状的遗传基础和分子调控机理研究也将会不断深入。
本实验室从粳稻品种日本晴T-DNA插入突变体库中筛选到一系列株高和穗部性状有关的突变体,其中一个突变体表现为半矮化、穗轴和枝梗缩短,穗型直立、着粒减少对进行了表型鉴定和遗传学分析,并通过图位克隆结合T-DNA标签的方法克隆了基因。实时荧光定量PCR分析T-DNA插入位点上下游基因在野生型和突变体中的表达差异,确定了候选基因,通过遗传转化实验过量表达基因,从而验证了基因的功能,为丰富水稻穗型种质资源和穗型基因调控网络打下了基础。
1 材料与方法
1.1 实验材料
从粳稻品种日本晴T-DNA插入突变体库中[29],筛选到一个半矮化,穗轴、一次枝梗和二次枝梗明显缩短的突变材料,经连续种植,发现该性状可以稳定遗传,将其命名为,供试材料分别种植于浙江杭州和海南陵水实验基地。
1.2 突变体的表型鉴定
抽穗后随机选取野生型日本晴、杂合体和纯合分别对其株高、穗型、着粒数、千粒重等农艺性状进行考查,每个材料10个重复。
1.3 遗传分析和定位群体构建
突变体的遗传分析从T1代开始,统计正常植株、杂合体和纯合植株的数量,单株收获T1代种子,种植T2代。自交后代均单株收获,按株系种植,观察植株的表型,调查后代有无分离。
以作母本与籼稻品种龙特甫B杂交,获得/龙特甫B的F1杂交种子,将F1种子收获后单本种植,去除假杂种后套袋自交,单株收获F2种子,F2种子按株系进行单本种植,从F2分离群体中选取极端隐性表型(野生型正常表型)的纯合表型单株用于初步定位。
1.4 基因定位
采用CTAB法[29]提取各单株叶片总DNA,并参照Michelmore等[30]的BSA法,根据F2分离群体植株表型,随机等量取10株野生型和10株极端表型植株的DNA,分别构建野生型DNA池和突变型DNA池。利用本实验室保存的183对在上述两亲本间有多态性的SSR标记对两个近等基因池进行BSA分析,然后选取有偏态扩增的SSR标记对隐性单株进行分析,初步确定突变基因的位置。PCR扩增参照Panaud等[31]的方法进行,本研究中使用15 μL的PCR扩增体系:1 μL模板DNA,7.5 mL PCR Mix(2×),正反向引物各1 μL,4.5 μL ddH2O。PCR扩增程序:94℃下变性5 min;94℃下预变性30 s,55℃下退火30 s,72℃下延伸1 min,72℃下重复延伸5 min,16℃下保存,共计35个循环。反应产物用4%琼脂糖凝胶电泳检测,用Alpha Imager EP凝胶成像仪观察并拍照。

表1 PCR鉴定及定量PCR所用的引物
1.5 连锁分析
将的SSR扩增带型记为“1”,龙特甫B的带型记为“2”,杂合体带型记为“3”,用MapMaker/EXP 3.0软件进行连锁分析[33],结合SSR标记在染色体上的位置,对基因进行定位。
T-DNA插入位点连锁分析,选取T2代表型分离的群体,分别选取野生型、杂合体和纯合突变型三种表型单株,取叶片提取DNA,利用T-DNA区的基因设计PCR扩增引物进行扩增,所用反应程序:94℃下5 min;94℃下30 s,55℃下30 s,72℃下1 min,30个循环;72℃下5 min,16℃保存。PCR产物用1%琼脂糖凝胶电泳检测。
1.6 候选基因表达分析
采用Trizol法提取野生型和突变体幼穗发育第Ⅲ期的总RNA,野生型和突变体各取3个生物学重复。使用反转录试剂盒(TOYOBO公司的ReverTra Ace®qPCR RT Master Mix with gDNA Remover)进行逆转录第1链cDNA合成,步骤参照试剂盒说明书。以水稻为内参基因,用NOVO protein公司的2×PCR Master Mix试剂进行半定量PCR检测。反应程序如下:94℃下5 min;94℃下30 s,55℃下30 s,72℃下1 min,25个循环;72℃下5 min,16℃保存。PCR产物取8 μL,用1%的琼脂糖凝胶电泳,Alpha Imager EP凝胶成像仪拍照。
利用前述方法获得的cDNA,采用SYBR荧光定量PCR试剂盒进行反应,依照产品说明书完成操作,20 μL反应体系含1 μL反转录产物。反应在Lightcycler 2.0荧光定量PCR仪(Roche瑞士)上进行,所用反应程序如下:95℃下10 s;95℃下5 s,55℃下10 s,72℃下10 s,40个循环。采用2法对基因相对表达量进行比较[34]。
1.7 转基因过量表达
以基因序列为模板,设计扩增基因全长ORF的引物,用TaKaRa公司的PrimeSTAR®HS DNA聚合酶结合2×GC缓冲液进行对cDNA进行PCR扩增。反应程序如下:98℃下10 s,60℃下5 s,72℃下2 min,35个循环;72℃下5 min,16℃保存。取5 μL PCR产物电泳检测,确认扩增成功后用无水乙醇沉淀,然后溶于适量的ddH2O,用Ⅰ和Ⅰ对获得的目的基因片段进行酶切、回收,连接到用同样酶切后回收的线性化载体上,形成载体,测序验证。将测序正确的载体导入农杆菌,采用农杆菌介导法转化野生型日本晴,以空载体为对照,转化方法参照文献[35]。利用G418筛选获得抗性愈伤,将抗性愈伤组织转移到含有150 mg/L G418的预分化培养基上预分化10 d左右,然后转移至分化培养基上培养1个月左右,获得转基因植株,用引物进行扩增,所用反应程序为:94℃下5 min;94℃下30 s,55℃下30 s,72℃下1 min,30个循环;72℃下5 min,16℃下保存。PCR产物用1%琼脂糖凝胶电泳检测。检测阳性的植株移栽到田间,于抽穗期观察植株表型并取样进行qRT-PCR验证。
2 结果与分析
2.1 dsp2-D突变体的形态特征与性状分析
自然条件下(杭州夏季,6月1日前后播种),突变体表现为株高显著降低,野生型、杂合体和纯合突变型的株高分别为91.7、80.6和69.9 cm,杂合体和纯合突变分别是野生型的87.9%和76.2%。穗轴、一次枝梗和二次枝梗显著缩短,其中野生型、杂合体和纯合突变型的穗长分别为21.5、11.7和10.0 cm,杂合体和纯合突变分别是野生型的54.4%和46.5%。穗粒数显著减少,野生型、杂合体和纯合突变型的每穗粒数分别为122.5、49.6和11.6,杂合体和纯合突变型分别是野生型的40.5%和9.5%。而野生型、杂合体和纯合突变型的结实率分别为85.3%、35.5%和14.5%,杂合体和纯合突变型分别是野生型的41.6%和17.0%(图1)。穗轴、一次和二次枝梗缩短,导致穗型直立,着粒密度变大(图1)。此外,分蘖数显著增加,杂合体和纯合突变型分别比野生型增加26.4%和52.9%。
2.2 dsp2-D突变体的遗传分析
的T1代突变株系中可见两种突变表型,其中一种为极端突变体,表现为半矮化、穗颈极度缩短,另外一种突变表型,株高、穗长等介于野生型和极端型之间,野生型∶中间型∶纯合体植株数量分别8∶16∶6(χ2=0.18,<0.05)。将两类突变体单株收获,通过后代株系的表型分离确定其遗传特性。分别调查分析了分离群体中,极端表型和中间表型各4个单株后代株系的表型,其中极端突变体自交后代全部出现极端表型,不再分离,而4个中间型表型的植株后代均再次出现野生型、中间型和极端型3种表型分离,且3种表型的数量比例符合1∶2∶1的分离关系(结果未显示)。以上结果表明,突变体表型受一对不完全显性基因控制。
AA、Aa、aa分别代表野生型日本晴植株、杂合体和dsp2-D纯合体。不同字母代表差异极显著(P<0.01,Tukey检验),n=10。
AA, Wild-type ‘Nipponbare’ plants; Aa, dsp2-D heterozygous plants; aa, dsp2-D homozygous plants. Values are mean ± SD of ten biological replicates. Samples with different letters are significantly different (P < 0.01; Tukey’s test).
图1 突变体dsp2-D的表型
Fig. 1. Phenotypes of the dsp2-D mutant.
2.3 dsp2-D的基因定位和T-DNA插入位点分析
利用在日本晴和龙特甫B两亲本间有多态性的、均匀分布于水稻12条染色体的183对SSR引物对两个近等基因池进行筛选,发现位于第2染色体相邻的3对SSR引物(RM240,RM166和RM266)在两基因池具有明显的偏态扩增。利用这3对引物对93个隐性单株进行扩增,结果显示出明显的偏分离,表明该突变基因位于第2染色体上的SSR标记RM240和RM266之间。进一步在RM240和RM266之间发展SSR标记,其中3对标记(RM208、RM3850和RM7337)在两亲本间有多态性,利用这3对有多态性的引物继续检测93个单株的扩增产物,将突变基因定位在SSR标记RM208和RM7337之间,与RM3850共分离,与两个标记RM208和RM7337的遗传距离分别约为3.5 cM和2.3 cM(图2-A)。
利用T-DNA中的潮霉素抗性基因,对F2分离群体中的基因进行PCR扩增,发现杂合体和纯合体均可以扩增出目的条带,而野生型表型不能扩增(图2-B),表明突变表型与T-DNA插入共分离。
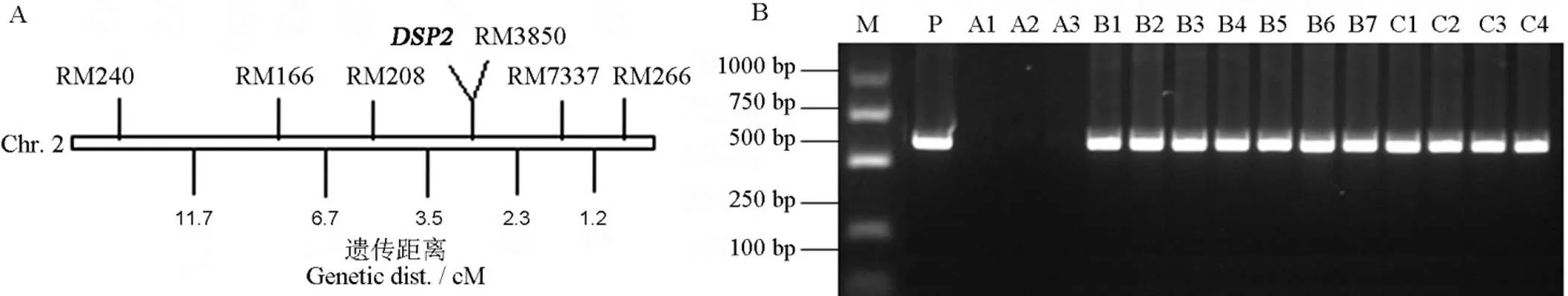
A―DSP2基因的初步定位;B―T-DNA插入位点共分离分析。M, 1 kb ladder; P为质粒阳性对照; A1~A3为野生型;B1~B7为杂合体;C1~C4为纯合体。
Fig. 2. Location ofon chromosome 2 and co-segregation analysis of T-DNA.
2.4 候选基因DSP2表达分析
利用TAIL-PCR分离了T-DNA插入位点的旁侧序列,通过NCBI网站进行BLAST比对分析,发现T-DNA插入到水稻第2染色体克隆P0474F11上两个基因之间,位于所在的SSR标记RM208和RM7337的区间,提示表型是T-DNA插入引起(图3-A)。
为了确定候选基因DSP2,分别对T-DNA插入位点上下游各3个基因进行了RT-PCR半定量分析(图3-A)。对野生型和突变体中6个基因进行表达分析的结果显示,这5个基因在突变体中的表达量和野生型相比没有显著差异,位于T-DNA插入位点下游的基因在野生型中表达量很低(T-DNA位于ORF1启动子方向的上游),但是在突变体中其表达水平显著上调(图3-B)。实时定量PCR结果也与前述半定量结果相符。T-DNA插入位点下游基因在突变体中其表达水平显著上调,而其他几个分析的基因表达量无显著差异(图3-C)。结果显示,可能是的候选基因。
2.5 转基因功能验证
利用载体构建了水稻启动子驱动的过量表达载体,命名为-OE,通过农杆菌介导法转化水稻品种日本晴。随机选取3株-过量表达载体转化植株的幼穗提取RNA,反转录后进行qRT-PCR分析,基因表达量比空载体转化对照植株显著增加(图4-A)。至抽穗期,基因的过量表达植株,其株高均出现显著降低,OE#1, OE#2和OE#3三个阳性植株株高分别为75.6、72.4和69.8 cm,株高与基因的表达量负相关,稻穗变短,穗直立,表型与突变体类似(图4-B、4-C)。上述研究结果显示,突变体表型确实是由基因的过量表达引起的。
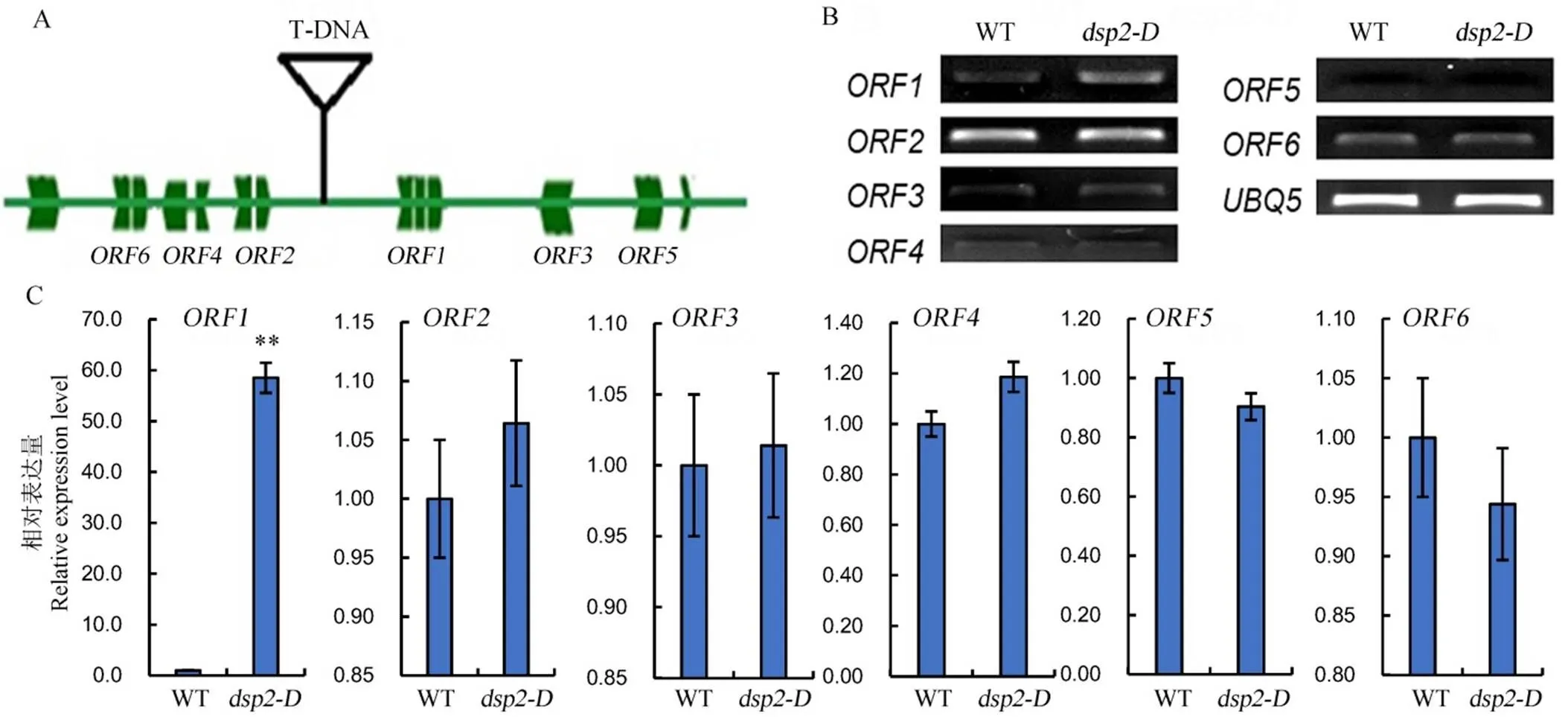
A―T-DNA插入位点;B, C―6个基因在突变体dsp2-D和野生型中的表达水平,水稻OsUBQ5为内参基因。Bar值表示3个生物学重复的平均值±SD;**表示0.01极显著水平。
Fig. 3. Expression analysis of candidate gene
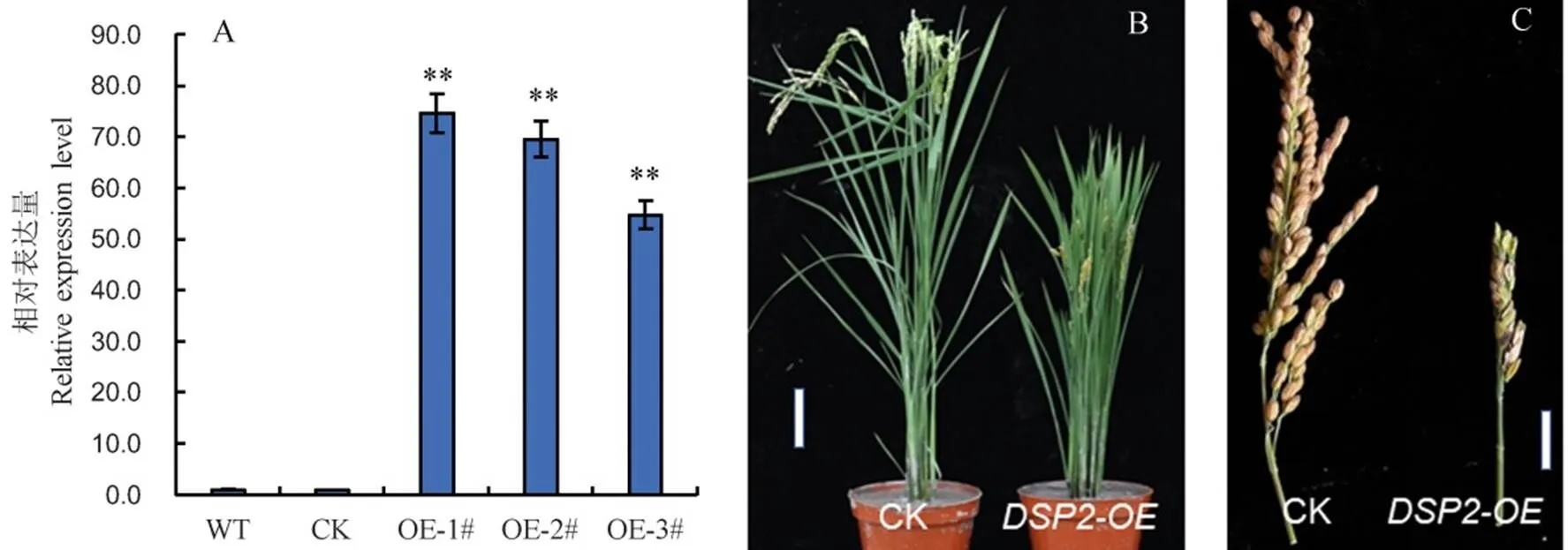
A―DSP2基因在野生型和转基因植株中的表达水平,其中WT代表野生型日本晴,CK代表空载体转化植株,OE-1#~OE-3#代表DSP2-OE转化植株;Bar值表示3个生物学重复的平均值±SD;**表示0.01极显著水平。B, C―DSP2过表达转基因植株的表型,CK为空载体转化植株。B图中标尺表示10 cm; C图中标尺表示5 cm。
Fig. 4. Overexpression ofgene and functional identification.
3 讨论
水稻是我国乃至世界上的重要粮食作物,全世界超过一半人口以稻米为主食。水稻生产在我国的粮食安全中占有极为重要的地位,水稻株高、穗型会直接影响最终的产量和品质,是水稻育种和生产中最受关注的性状之一[36-38]。对水稻穗型突变体及其调控的基因进行研究,有助于阐明穗发育的遗传基础和分子调控机理,对水稻穗型和产量的遗传改良研究具有重要的指导意义。
本研究中,突变体出现半矮化,穗轴和枝梗缩短,着粒密度变大等表型,遗传分析显示,突变是由一对单基因控制的不完全显性基因控制。利用分子标记定位结合T-DNA标签分离,确定了是的候选基因,进一步通过转基因过量表达实验验证了基因的功能,表明基因对水稻穗型具有重要的负向调控作用,是一个调控水稻株高和穗型的新基因,深入研究的功能和分子调控机理,可以丰富水稻穗型发育基因调控网络。后续需要对基因进行基因编辑或抑制基因表达,进一步深入研究对水稻株高和穗型的调控机理,并筛选基因的自然等位变异,获得具有增产效应的优异等位基因,为育种利用提供支持。
基因编码LOB家族转录因子,基因家族是一类植物特异的转录因子家族,主要作用于分生组织边界,影响叶片近-远轴极性的建立[39]。基因家族在侧生器官的发育和形成过程中发挥重要功能,此外,基因功能还与植物的生长发育、光形态建成、叶枕和叶柄发育、抗病性、激素反应、代谢调节有关,其功能对植物的生长非常重要[40]。水稻中有35个基因,绝大部分基因功能未知[41]。其中编码LOB蛋白,其缺失突变体表现出不定根缺失,根向地性丢失,对生长素、乙烯等不敏感,但是在NAA处理条件下,在根系中表达量上升,表明该基因参与生长素应答途径而影响细胞分化途径[42-43]。编码另外一个LOB蛋白,其T-DNA插入缺失突变体颖壳退化、雌雄蕊数目异常、部分雄性不育[44]。编码LOB蛋白,是玉米的同源基因,OsIG1-RNAi植株表现为颖花、颖壳发育异常[45]。为玉米的同源基因,异位表达枝梗缩短,表达量降低,枝梗伸长[46]。OsLBD37和OsLBD38通过抑制下游开花基因和的表达,调控抽穗期[47]。本研究中,编码LOB蛋白,其过量表达后导致矮化,穗轴、一次枝梗和二次枝梗缩短,结实率下降,为一调控株高和穗型的新基因,与基因具有类似的表型。对功能的研究,有助于进一步阐明LOB家族的生物学功能。
水稻是重要的粮食作物,也是禾本科植物研究的模式植物,深入发掘基因的功能和分子机理,可以为分子设计育种提供基因资源和研究思路。水稻基因的发掘和成功利用使得水稻产量取得重大突破,引发了“第一次绿色革命”,近年来,水稻生产中利用有利功能基因及其弱等位突变取得了重要突破,如东北和长江中下游晚粳稻区的直立穗和半直立穗品种中普遍利用的直立穗基因[48-49],籼粳杂交稻甬优系列品种中利用的粗大穗理想株型基因[50-51],以及华占品种中利用的多蘖矮秆基因[18]。因此,为实现在现有产量水平上实现单产水平的进一步突破,充分挖掘作物产量遗传潜力,对株型、穗型形成的分子机理进行遗传解析具有重要的理论意义和应用价值。
谢辞:本研究所用水稻材料由原中国水稻研究所孙宗修研究员提供,特此致谢。
[1] 刘坚, 陶红剑, 施思, 叶卫军, 钱前, 郭龙彪. 水稻穗型的遗传和育种改良[J]. 中国水稻科学, 2012, 26(2): 227-234.
Liu J, Tao H J, Shi S, Ye W J, Qian Q, Guo L B. Genetics and breeding improvement for panicle type in rice[J]., 2012, 26(2): 227-234. (in Chinese with English abstract).
[2] 冷语佳, 钱前, 曾大力. 水稻理想株型的遗传基础研究[J]. 中国稻米, 2014, 20(2): 1-6.
Leng Y J, Qian Q, Zeng D L. Progress on genetic basis of rice ideal plant type[J]., 2014, 20(2): 1-6. (in Chinese with English abstract).
[3] 淳雁, 李学勇. 水稻穗型的遗传调控研究进展[J]. 植物学报, 2017, 52(1): 19-29.
Chun Y, Li X Y. Research progress in genetic regulation of rice panicle architecture[J]., 2017, 52(1): 19-29. (in Chinese with English abstract)
[4] Sasaki A, Ashikari M, Ueguchi-Tanaka M, Itoh H, Nishimura A, Swapan D, Ishiyama K, Saito T, Kobayashi M, Khush G S, Kitano H, Matsuoka M. A mutant gibberellin-synthesis gene in rice[J]., 2002, 416(6882): 701-702.
[5] Peng J, Richards D E, Hartley N M, Murphy G P, Devos K M, Flintham J E, Beales J, Fish L J, Worland A J, Pelica F, Sudhakar D, Christou P, Snape J W, Gale M D, Harberd N P. “Green revolution” genes encode mutant gibberellin response modulators[J]., 1999, 400(6741): 256-261.
[6] Ogawa S, Toyomasu T, Yamane H, Murofushi N, Ikeda R, Morimoto Y, Nishimura Y, Omori T. A step in the biosynthesis of gibberellins that is controlled by the mutation in the semi-dwarf rice cultivar Tan-Ginbozu[J]., 1996, 37(3): 363-368.
[7] Sakamoto T, Morinaka Y, Ishiyama K, Kobayashi M, Itoh H, Kayano T, Iwahori S, Matsuoka M, Tanaka H. Genetic manipulation of gibberellin metabolism in transgenic rice[J]., 2003, 21(8): 909-913.
[8] Zhu Y, Nomura T, Xu Y, Zhang Y, Peng Y, Mao B, Hanada A, Zhou H, Wang R, Li P, Zhu X, Mander L N, Kamiya Y, Yamaguchi S, He Z.encodes a cytochrome P450 monooxygenase that epoxidizes gibberellins in a novel deactivation reaction in rice[J]., 2006, 18(2): 442-456.
[9] Ueguchi-Tanaka M, Ashikari M, Nakajima M, Itoh H, Katoh E, Kobayashi M, Chow T, Hsing Y, Kitano H, Yamaguchi I, Matsuoka M.encodes a soluble receptor for gibberellin[J]., 2005, 437(7059): 693-698.
[10] Hong Z, Ueguchi-Tanaka M, Umemura K, Uozu S, Fujioka S, Takatsuto S, Yoshida S, Ashikari M, Kitano H, Matsuoka M. A rice brassinosteroid-deficient mutant,, is caused by a loss of function of a new member of cytochrome P450[J]., 2003, 15(12): 2900-2910.
[11] Sakamoto T, Morinaka Y, Ohnishi T, Sunohara H, Fujioka S, Ueguchi-Tanaka M, Mizutani M, Sakata K, Takatsuto S, Yoshida S, Tanaka H, Kitano H, Matsuoka M. Erect leaves caused by brassinosteroid deficiency increase biomass production and grain yield in rice[J]., 2006, 24(1): 105-109.
[12] Hong Z, Ueguchi-Tanaka M, Fujioka S, Takatsuto S, Yoshida S,Hasegawa Y, Ashikari M, Kitano H, Matsuoka M. The rice brassinosteroid-deficientmutant, defective in the rice homolog of/, is rescued by the endogenously accumulated alternative bioactive brassinosteroid, dolichosterone[J]., 2005, 17(8): 2243-2254.
[13] Yamamuro C, Ihara Y, Wu X, Noguchi T, Fujioka S, Takatsuto S, Ashikari M, Kitano H, Matsuoka M. Loss of function of a ricehomolog prevents internode elongation and bending of the lamina joint[J]., 2000, 12(9): 1591-1605.
[14] Tong H, Jin Y, Liu W, Li F, Fang J, Yin Y, Qian Q, Zhu L, Chu C. DWARF AND LOW-TILLERING, a new member of the GRAS family, plays positive roles in brassinosteroid signaling in rice[J]., 2009, 58(5): 803-816.
[15] Lin H, Wang R, Qian Q, Yan M, Meng X, Fu Z, Yan C, Jiang B, Su Z, Li J, Wang Y. DWARF27, an iron-containing protein required for the biosynthesis of strigolactones, regulates rice tiller bud outgrowth[J]., 2009, 21(5): 1512-1525.
[16] Zou J, Chen Z, Zhang S, Zhang W, Jiang G, Zhao X, Zhai W, Pan X, Zhu L. Characterizations and fine mapping of a mutant gene for high tillering and dwarf in rice (L.)[J]., 2005, 222(4): 604-612.
[17] Arite T, Iwata H, Ohshima K, Maekawa M, Nakajima M, Kojima M, Sakakibara H, Kyozuka J., an//ortholog, controls lateral bud outgrowth in rice[J]., 2007, 51(6): 1019–1029.
[18] Wang Y, Shang L, Yu H, Zeng L, Hu J, Ni S, Rao Y, Li S, Chu J, Meng X, Wang L, Hu P, Yan J, Kang S, Qu M, Lin H, Wang T, Wang Q, Hu X, Chen H, Wang B, Gao Z, Guo L, Zeng D, Zhu X, Xiong G, Li J, Qian Q. A strigolactone biosynthesis gene contributed to the green revolution in rice[J]., 2020, 13(6): 923-932.
[19] Jiang L, Liu X, Xiong G, Liu H, Chen F, Wang L, Meng X, Liu G, Yu H, Yuan Y, Yi W, Zhao L, Ma H, He Y, Wu Z, Melcher K, Qian Q, Xu H E, Wang Y, Li J. DWARF 53 acts as a repressor of strigolactone signaling in rice[J]., 2013, 504(7480): 401-405.
[20] Zhou F, Lin Q, Zhu L, Ren Y, Zhou K, Shabek N, Wu F, Mao H, Dong W, Gan L, Ma W, Gao H, Chen J, Yang C, Wang D, Tan J, Zhang X, Guo X, Wang J, Jiang L, Liu X, Chen W, Chu J, Yan C, Ueno K, Ito S, Asami T, Cheng Z, Wang J, Lei C, Zhai H, Wu C, Wang H, Zheng N, Wan J. D14-SCF(D3)-dependent degradation of D53 regulates strigolactone signalling[J]., 2013, 504(7480): 406-410.
[21] Yao R, Ming Z, Yan L, Li S, Wang F, Ma S, Yu C, Yang M, Chen L, Li Y, Yan C, Miao D, Sun Z, Yan J, Sun Y, Wang L, Chu J, Fan S, He W, Deng H, Nan F, Li J, Rao Z, Lou Z, Xie D. DWARF14 is a non-canonical hormone receptor for strigolactone[J]., 2016, 536(7617): 469-473.
[22] Wang L, Wang B, Yu H, Guo H, Lin T, Kou L, Wang A, Shao N, Ma H, Xiong G, Li X, Yang J, Chu J, Li J. Transcriptional regulation of strigolactone signalling in Ara[J]., 2020, 583(7815): 277-281.
[23] Sazuka T, Kamiya N, Nishimura T, Ohmae K, Sato Y, Imamura K, Nagato Y, Koshiba T, Nagamura Y, Ashikari M, Kitano H, Matsuoka M. A ricemutant,, contains a reduced level of indole acetic acid and develops abnormal flowers and organless embryos[J]., 2009, 60(2): 227-241.
[24] Song Y, You J, Xiong L. Characterization ofgene, a member of rice Aux/IAAfamily involved in auxin and brassinosteroid hormone responses and plant morphogenesis[J]., 2009, 70(3): 297-309.
[25] Qi W, Sun F, Wang Q, Chen M, Huang Y, Feng Y Q, Luo X, Yang J. Rice ethylene-response AP2/ERF factorrestricts internode elongation by down-regulating a gibberellin biosynthetic gene[J]., 2011, 157(1): 216-228.
[26] Luan W, Liu Y, Zhang F, Song Y, Wang Z, Peng Y, Sun Z.encodes a putative member of the cellulose synthase-like D sub-family and is essential for rice plant architecture and growth[J]., 2011, 9(4): 513-524.
[27] Tabuchi M, Sugiyama K, Ishiyama K, Inoue E, Sato T, Takahashi H, Yamaya T. Severe reduction in growth rate and grain filling of rice mutants lacking OsGS1;1, a cytosolic glutamine synthetase1;1[J]., 2005, 42(5): 641-651.
[28] Sato Y, Sentoku N, Miura Y, Hirochika H, Kitano H, Matsuoka M. Loss-of-function mutations in the rice homeobox geneaffect the architecture of internodes resulting in dwarf plants[J]., 1999, 18(4): 992-1002.
[29] Zhu Z Z, Fu Y P, Xiao H, Hu G C, Si H M, Yu Y H, Sun Z X.Transposition activity in transgenic rice population and DNA flanking sequence of Ds insertion sites [J]., 2003(1): 102-107.
[30] Rogers S O, Bendich A J. Extraction of DNA from milligram amounts of fresh, herbarium and mummified plant tissues[J]., 1985, 5(2): 69-76.
[31] Michelmore R W, Kesseli I. Identification of markers linked to disease-resistance genes by bulked segregant analysis: A rapid method to detect markers in specific genomic regions by using segregating populations[J]., 1991, 88(21): 9828-9832.
[32] Panaud O, Chen X, McCouch S R. Development of microsatellite markers and characterization of simple sequence length polymorphism (SSLP) in rice (L.)[J]., 1996, 259(5): 597-607.
[33] Lander E S, Green P, Abrahamson J, Barlow A, Daly M J, Lincoln S E, Newburg L. Mapmaker: An interactive computer package for constructing primary genetic linkage maps of experimental and natural populations[J].1987, 1(2):174-181.
[34] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2method[J]., 2001, 25(4): 402-408.
[35] Hiei Y, Ohta S, Komari T, Kumashiro T. Efficient transformation of rice (L.) mediated byand sequence analysis of the boundaries of the T-DNA[J]., 1994, 6(2): 271-282.
[36] 郭龙彪, 程式华, 钱前. 水稻基因设计育种的研究进展与展望[J]. 中国水稻科学, 2008, 22(6): 650-657.
Guo L B, Chen S H, Qian Q. Progress and prospects of breeding by gene design in rice[J]., 2008, 22(6): 650-657. (in Chinese with English abstract)
[37] Wang Y H, Li J Y. Molecular basis of plant architecture[J]., 2008, 59(1): 253-279.
[38] Gurdv K. Productivity improvements in rice[J]., 2010, 61 (6): S114-116.
[39] Majer C, Hochholdinger F. Defining the boundaries: structure and function of LOB domain proteins[J]., 2011, 16(1): 47-52.
[40] Zhang Y, Li Z, Ma B, Hou Q, Wan X. Phylogeny and functions of LOB domain proteins in plants[J]., 2020, 21(7): 2278.
[41] Yang Y, Yu X, Wu P. Comparison and evolution analysis of two rice subspeciesdomain gene family and their evolutionary characterization from[J]., 2006, 39(1): 248-262.
[42] Liu H, Wang S, Yu X, Yu J, He X, Zhang S, Shou H, Wu P. ARL1, a LOB-domain protein required for adventitious root formation in rice[J]., 2005, 43(1): 47-56.
[43] Inukai Y, Sakamoto T, Ueguchi-Tanaka M, Shibata Y, Gomi K, Umemura I, Hasegawa Y, Ashikari M, Kitano H, Matsuoka M., which is essential for crown root formation in rice, is a target of an Auxin Response Factor in auxin signaling[J]., 2005, 17(5): 1387-1396.
[44] Li A, Zhang Y, Wu X, Tang W, Wu R, Dai Z, Liu G, Zhang H, Chen G, Pan X. DH1, a LOB domain-like protein required for glume formation in rice[J]., 2008, 66(5): 491-502.
[45] Zhang J, Tang W, Huang Y, Niu X, Zhao Y, Han Y, Liu Y. Down-regulation of a-like gene,, leads to occurrence of unusual double ovules and developmental abnormalities of various floral organs and megagametophyte in rice[J]., 2015, 66(1): 99-112.
[46] Lu H, Dai Z, Li L, Wang J, Miao X, Shi Z.shapes panicle architecture through regulating pedicel length[J]., 2017, 8(12): 1538.
[47] Li C, Zhu S, Zhang H, Chen L, Cai M, Wang J, Chai J, Wu F, Cheng Z, Guo X, Zhang X, Wan J. OsLBD37 and OsLBD38, two class II type LBD proteins, are involved in the regulation of heading date by controlling the expression ofin rice[J]., 2017, 486(3): 720-725.
[48] Huang X Z, Qian Q, Liu Z, Sun H, He S, Luo D, Xia G, Chu C, Li J, Fu X. Natural variation at thelocus enhances grain yield in rice[J]., 2009, 41(4): 494-497.
[49] Zhou Y, Zhu J, Li Z, Yi C, Liu J, Zhang H, Tang S, Gu M, Liang G. Deletion in a quantitative trait geneassociated with panicle erectness improves plant architecture during rice domestication[J]., 2009, 183(1): 315-324.
[50] Miura K, Ikeda M, Matsubara A, Song X J, Ito M, Asano K J, Matsuoka M, Kitano H, Ashikari M.promotes panicle branching and higher grain productivity in rice[J]., 2010, 42(6): 545-549.
[51] Jiao Y, Wang Y, Xue D, Wang J, Yan M, Liu G, Dong G, Zeng D, Lu Z, Zhu X, Qian Q, Li J. Regulation ofby OsmiR156defines ideal plant architecture in rice[J]., 2010, 42(6): 541-544.
Identification and Gene Cloning ofin Rice (L.)
QIU Linlin1, LIU Qiao1, FU Yaping2, LIU Wenzhen2, HU Guocheng2, ZHAI Yufeng1, PANG Bo1, WANG Dekai1,*
(1College of Life Sciences and Medicine, Zhejiang Sci-Tech University, Hangzhou 310018, China;2State Key Laboratory of Rice Biology,China National Rice Research Institute,Hangzhou 310006, China;*Corresponding author, E-mail: kay77@163.com)
【】Plant height and panicle length play important roles in rice production. Identifying and cloning genes related to rice plant height and panicle architecture will enrich the molecular mechanism of the regulation of rice plant height and panicle development, and will provide a theoretical foundation and genetic resources for molecular design breeding.【】 A dwarf and small panicle mutant() was obtained from T-DNA insertion population of Nipponbare. The main agronomic traits ofand the wild-type were characterized under conventional planting conditions infield. An F2mapping population was generated by crossing themutant with Longtefu B (anrice variety) andwas identified by map-based cloning and T-DNA tag method. The candidate gene was determined by the relative expression analysis of three genes located upstream and downstream of the T-DNA insertion site, respectively, which was carried out by real-time RT-PCR. The function ofwas confirmed by overexpressing() in wild-type Nipponbare.【】 The plant height, panicle length, the length of primary and secondary inflorescence branches ofwas dramatically reduced compared with the wild type. Genetic analysis indicated that the phenotypes ofwere regulated by an incomplete dominant gene. Thegene was mapped to a region between RM208 and RM7337 on chromosome 2, and co-segregated with RM3850. Subsequently, T-DNA insertion site was found to be co-segregated withphenotype, and T-DNA insertion sequence was separated by TAIL-PCR, which showed that T-DNA was inserted into the two genes between RM208 and RM7337. Real-time PCR analysis indicated that the expression level ofwhich located down-stream of T-DNA site, was dramatically up-regulated. While the expression levels of the rest five detected geneswere not significantly changed in.() overexpressing transgenic plants showed semi-dwarf and small panicle,which were similar with the phenotypes inThe results confirmed thatgene was the target gene controlling【】The overexpression ofcaused the mutant phenotypes of. And we found thatencodes a LOB family transcript factor and negatively regulates plant height and panicle length in rice. Our findings will open new perspectives on-based improvement of plant height and panicle type of rice.
rice;dwarf; panicle length; mutant; gene cloning
2021-06-17;
2021-09-14。
国家重点研发专项(2016YFD0100401);国家自然科学基金资助项目(31571742);浙江理工大学科研启动基金资助项目(19042142-Y)。
