基于生物信息学探讨植物拟南芥的UV-B辐射保护机制
2022-03-11付苏宏郝豆豆施静张勇群
万 莎,付苏宏,郝豆豆,施静,张勇群
(西藏自治区人民政府驻成都办事处医院,分子医学实验室,四川 成都 610041)
太阳光对于植物来说是非常重要的物质,不仅提供其光合作用的能量来源,更是调节其生长发育环境信号,例如调节种子发芽和开花等.紫外线(ultraviolet, UV)是太阳光的组成部分,根据其波长主要可分为UV-A (315-400 nm)、UV-B (280-315 nm)和UV-C (<280 nm).UV-C和大部分的UV-B在经过地球的臭氧层时被吸收,只有UV-A和少部分的UV-B能到达地球表面.虽然只有< 0.5%的UV-B到达地面,但是其在日光光谱中的能量却最高,对地球上的生物圈产生的影响最大,过量的UV-B射线会损害植物的光合作用过程[1],以及引起DNA光解产物的生成从而造成细胞损伤,甚至会直接损伤蛋白质、脂质和RNA[2-3].此外,UV-B的照射还会引起植物形态学上的改变,Strømme等发现UV-B会影响白杨芽的定型以及延迟花蕾的定植[4],Mark等曾报道增加UV-B照射会显著降低欧洲玉米的高度[5],Johanson等曾报道落叶性越橘属植物的叶片厚度随着UV-B照射的增加减小[6],Jenkins等曾总结发现UV-B照射通常引起植物叶腋分支增多[7].
臭氧是UV-B主要的吸收物质,随着地球臭氧层被破坏,越来越多的UV-B到达地面,对地球上的天然植物系统造成影响[8].因此,探讨UV-B对植物基因表达的影响从而了解植物对UV-B的应激保护机制则变得十分重要.GEO (gene expression omnibus)数据库包含有多个物种的基因表达数据,可从中检索出与研究相关的表达芯片数据,通过数据分析从而挖掘出数据背后的深层含义.拟南芥是模式植物,挖掘拟南芥的相关数据可获取植物UV-B保护机制的参考.本研究从GEO数据库中选取GSE3533和GESE22951表达芯片数据,通过差异基因筛选找出UV-B照射后表达改变的DEGs(differentially expressed genes),并进一步对DEGs进行GO(Gene Ontology)功能和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路富集分析,并进行DEGs编码蛋白的PPI网络分析(protein-protein network),从中找出关键的DEGs编码蛋白,进而揭示植物受UV-B照射后的应激保护机制.本研究基于生物信息学的方法对公共数据库进行数据挖掘,为植物的UV-B应激分子机制奠定了科学的研究基础.
1 材料和方法
1.1 数据来源
选取模式生物拟南芥(Arabidopsisthaliana)为研究物种,从公共数据库美国生物技术中心GEO数据库中下载UV-B辐射相关的基因表达芯片数据,登录号为GSE3533[9]和GSE22951,测试平台为GPL198,白光照射作为对照组,UV-B照射作为实验组,表达芯片数据的具体信息如表1所示.

表1 表达芯片数据详细信息
1.2 差异表达基因(DEGs)筛选
采用在线软件GEO2R( http://www.ncbi.nlm.nih.gov/geo/geo2r/ ) 对GSE3533和GSE22951表达芯片数据进行差异表达分析,将基因表达值倍数变化(fold change, FC)和错误检测率 (False discovery rate, FDR)作为评定指标,使用 Benjamini - Hochberg 错误检测率 (FDR) 方法校正 P 值,校正后的 P 值 <0.05 和 |log2FC| ≥1作为筛选条件[10].利用R软件分别绘制GSE3533和GSE22951表达芯片的表达火山图.利用在线软件Venny 2.1.0 (http://bioinfogp.cnb.csic.es/tools/venny/)进行GSE3533和GSE22951表达芯片的交叉基因筛选,并绘制Venny图.
1.3 GO功能注释和KEGG通路富集分析
GO可提供基因产物的功能信息以助于对基因能有更全面的认识[11],KEGG能系统地分析基因功能,揭示基因功能之间的生物联系[12],而DAVID (Database for Annotation, Visualization and Integration Discovery, https://david.ncifcrf.gov/)则可对一系列基因进行功能解释,实现基因列表的GO功能富集分析[13].利用DAVID在线软件对筛选得到的DEGs做GO功能注释富集分析,设定筛选条件为P<0.05,获取DEGs富集的GO条目,利用R语言的ggplot2包对富集结果进行可视化绘图.DEGs的KEGG通路富集分析与可视化通过Cytoscape中的ClueGO插件实现.
1.4 PPI网络分析
将上述分析得到的上调的交叉DEGs和下调的交叉DEGs分别导入在线数据库STRING (https://string-db.org/)中,构建其编码蛋白的相互作用关系,并利用本地软件Cytoscape 3.6.1进行PPI网络图的可视化,根据degree值筛选出中心度较高的DEGs.
2 结果
2.1 DEGs鉴定
数据来源于GSE3533和GSE22951两个表达芯片,分别选取其中6例样本,3例经白光照射作为对照组;另3例经UV-B照射作为实验组.GSE3533和GSE22951所有检测到的基因分别如图1A和图1B所示,分别筛选出2 741个和1 079个DEGs,其中上调基因分别为1 535个和443个(a区域),下调基因1 183个和630个(b区域).按表达上调和下调分别取GSE3533和GSE22951的交集,共鉴定到251个上调DEGs和246个下调DEGs (图1C和D).

图1 差异表达基因筛选
2.2 GO功能与KEGG通路富集分析
利用DAVID分别对251个上调的交叉DEGs和246个下调的交叉DEGs进行GO功能富集分析.上调的DEGs共注释到58个GO条目,前20的GO条目如图2A所示,其中显著性较高的有karrikin反应、UV-B反应、热反应、UDP-糖基转移酶活性和类黄酮生物合成过程;下调的DEGs共注释到46个GO条目,前20的GO条目如图2B所示,其中显著性较高的有胞外区、3-氧-花生四烯酸基-CoA合酶活性、3-氧-二十四烷酰基-CoA合酶活性、3-氧-蜡基-CoA合酶活性、羧酸酯水解酶活性、细胞壁.
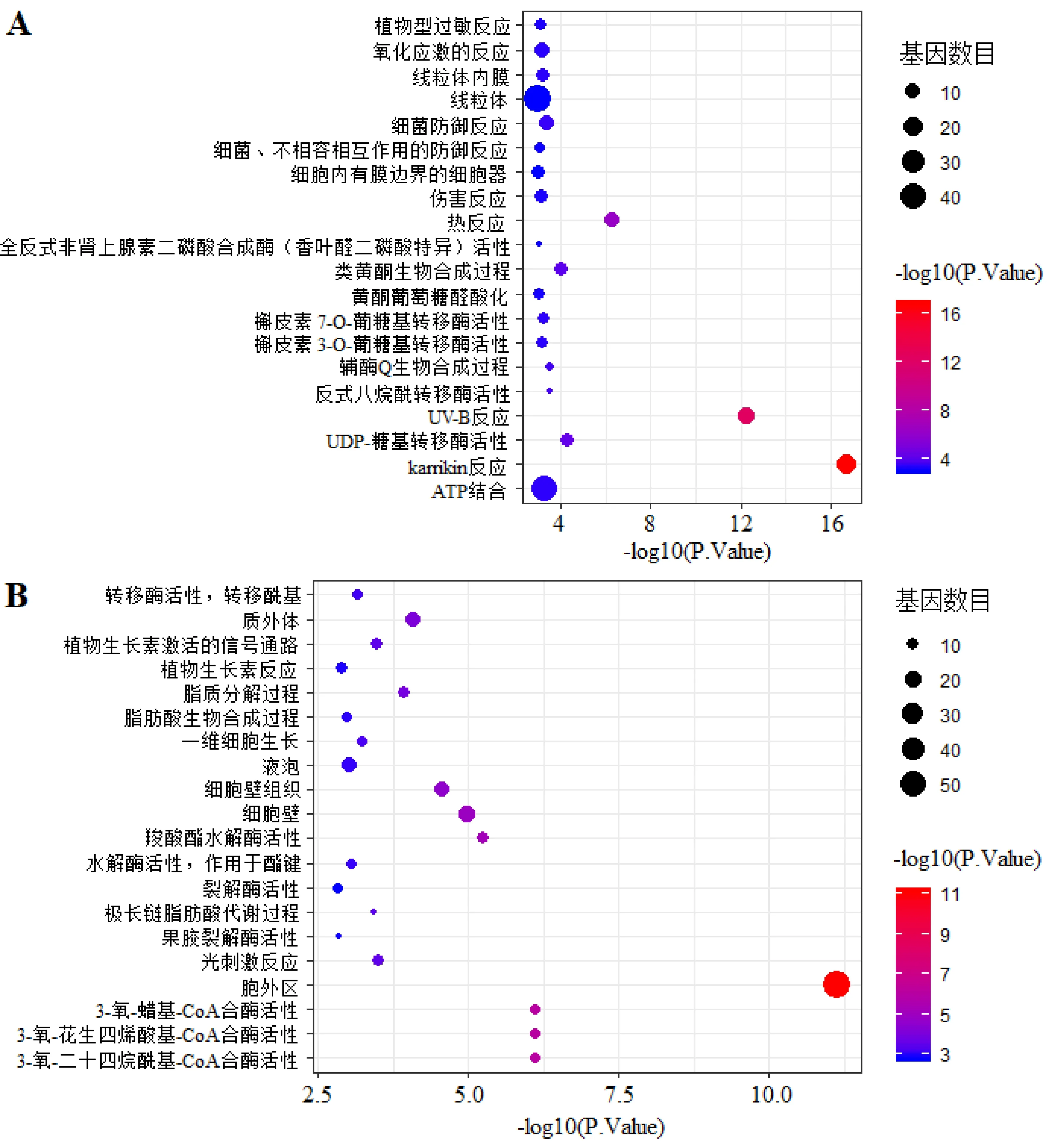
图2 GO功能富集分析
采用Cytoscape的ClueGO插件对交叉DEGs进行KEGG通路富集分析,结果显示上调的交叉DEGs主要注释到苯丙氨酸代谢、类黄酮生物合成、内质网蛋白加工、植物-病原体相互作用和昼夜节律通路中(图3A);下调的交叉DEGs主要注释到戊糖和葡萄糖醛酸互变异构、脂肪酸延长和吞噬体通路中(图3B).
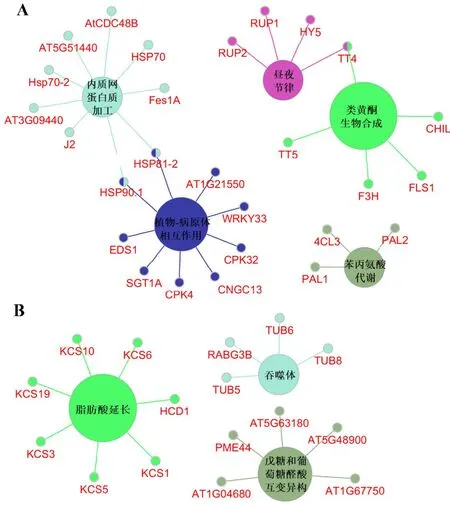
图3 KEGG通路富集分析
2.3 PPI网络分析与关键基因筛选
为获取DEGs编码蛋白的相互作用关系,我们采用STRING数据库和Cytoscape软件构建交叉DEGs编码蛋白的PPI网络图,每一条边表示蛋白之间的相互作用,每一个节点表示一个蛋白,节点越大对应其degree值越大,degree值越大的节点表明其对应的蛋白在PPI网络图中的中心度较高,能与多个蛋白发生相互作用(图4).上调的DEGs编码蛋白的PPI网络图中总共包括219个节点,1 080条边,根据网络拓扑学性质,degree值前10%的关键基因为AT5G25930、BCS1、BRL3、TOC64-V、MTHSC70-1、AT1G19020、SIGE、WRKY33、HSP70、MTHSC70-2、HSP90.1、NUDT7、AT5G24810、ATRPAC42、AT3G09440、HSP70-2、WRKY46、NRPA2、HSP81-2、F3H、CRK11,其中WRKY33注释到植物-病原体相互作用通路中,HSP70、HSP90.1、AT3G09440、HSP70-2、HSP81-2注释到内质网蛋白加工通路中,F3H注释到类黄酮生物合成通路中.下调的DEGs编码蛋白的PPI网络图中总共包括153个节点,306条边,degree值排前10%的关键基因为AT3G49670、HTH、AT1G09750、AT4G23820、AT3G16370、CYP86A2、DRT100、PME44、SAUR68、GAE1、CER2、KCS10、AT3G20820、FLA8、SHY2,其中PME44注释到戊糖和葡萄糖醛酸互变异构通路中,KCS10注释到脂肪酸延长通路中.
3 讨论
本研究基于生物信息学的分析手段,下载GEO数据库中拟南芥的GSE3533和GSE22951两个表达芯片数据,旨在从中挖掘出与植物UV-B保护机制相关的基因表达信息.通过对GSE3533和GSE22951进行差异表达基因筛选并整合,总共得到251个上调DEGs和246个下调DEGs,对这些DEGs分别进行GO功能和KEGG通路富集分析发现,上调的DEGs注释到karrikin反应、UV-B反应、热反应、UDP-糖基转移酶活性和类黄酮生物合成过程等GO功能,以及苯丙氨酸代谢、类黄酮生物合成、内质网蛋白加工、植物-病原体相互作用和昼夜节律KEGG通路中;下调的DEGs注释到胞外区、3-氧-花生四烯酸基-CoA合酶活性、3-氧-二十四烷酰基-CoA合酶活性、3-氧-蜡基-CoA合酶活性、羧酸酯水解酶活性、细胞壁等GO功能,以及戊糖和葡萄糖醛酸互变异构、脂肪酸延长和吞噬体KEGG通路中.此外,PPI网络分析表明上调DEGs中关键基因主要与植物-病原体相互作用/内质网蛋白加工和类黄酮生物合成相关,下调DEGs中的关键基因则主要与戊糖和葡萄糖醛酸互变异构和脂肪酸延长通路相关.
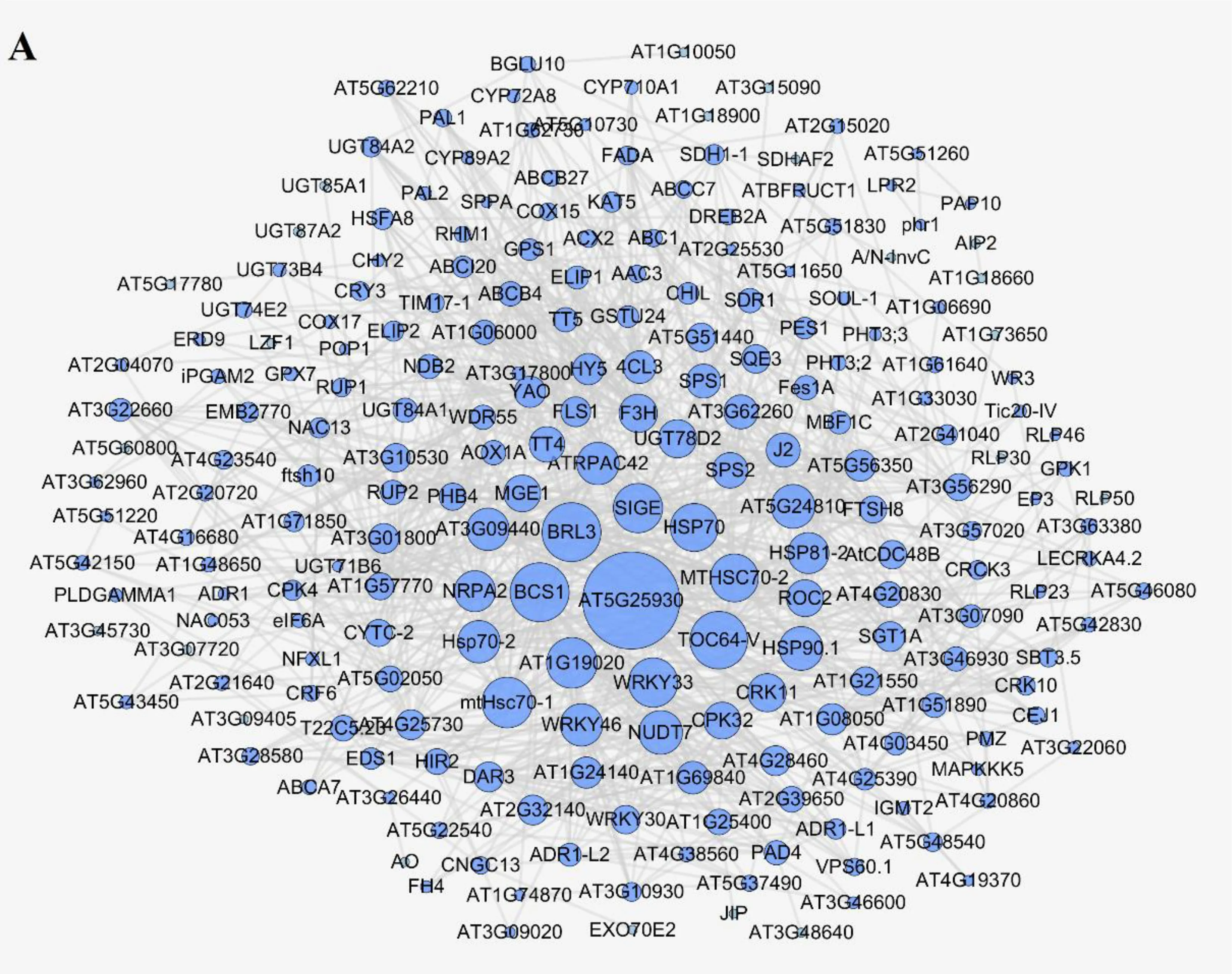

图4 交叉DEGs的PPI网络图Fig.4 PPI network diagram of cross DEGs
太阳光中的UV-B作为非生物压力信号会对植物造成多种影响,总的而言,植物对UV-B的反应主要分为两类:应激反应和光形态建成反应,而反应的类型主要取决于曝光率和植物是否已适应所照射的UV-B[2].高UV-B曝光率照射引起植物体内压力相关的生理过程,例如DNA损伤、ROS (reactive oxygen species)生成、细胞过程损害等,然而调节这些反应的信号通路在其他压力中也存在,并不是UV-B所特异性[14];低UV-B曝光率照射则引起植物体内的光形态建成反应,主要引起植物下胚轴生长和子叶扩张抑制、黄酮类和花青素类化合物生物合成、气孔打开等,是UV-B照射所特异的植物反应[15-16].研究表明,高海拔地区生长的植物相较于低海拔地区生长的植物来说拥有更强的UV-B耐受性,其主要原因在于高海拔植物体内的类黄酮含量更高且具有更强的染色质重构能力[8, 17-18].F3H全名为黄烷酮3-羟化酶,催化黄烷酮羟化生成黄酮醇,进一步合成花青素、原花色素以及各类黄酮醇衍生物[19-20],F3H基因在UV组中表达上调,并且具有较高的degree值,即F3H在上调基因的PPI网络中占据有重要的位置,说明F3H是拟南芥UV-B保护重要的分子机制之一.AT5G25930是一类富含亮氨酸重复序列型受体蛋白激酶,在上调基因的PPI网络中位于中心度最高的位置,该类蛋白在抗逆性反应、介导植物激素信号转导过程中发挥着重要的作用,与植物对应答逆境胁迫以及形态相关[21-23].而在下调基因的PPI网络中AT3G49670占据着最中心的位置,调控多数DEGs的表达,AT3G49670对植物的发育进行调控[24-25].
通过对表达芯片数据GSE3533和GSE22951的挖掘与分析,结果发现,在应对UV-B胁迫时,拟南芥上调AT5G25930和F3H的表达,进而调控植物形态的改变以及类黄酮类化合物的生物合成,可能与植物叶片变厚、叶柄变短、茎变短、腋窝分枝增多和根芽比改变有关[26];另一方面,受到UV-B胁迫时拟南芥抑制AT3G49670的表达,引起拟南芥的生长发育受到抑制.本研究通过挖掘公共数据库得到3个关键基因:AT5G25930、F3H和AT3G49670,它们在植物的UV-B紫外线保护机制中扮演着重要的角色,为今后进行深入的分子机制研究奠定了基础.
