草地早熟禾蛋白激酶OSK4基因的克隆及对非生物胁迫响应分析
2022-03-08高岩松李洪影赵清峰曹业萍邸浩洋金一锋
陈 阳, 高岩松, 李洪影, 赵清峰, 曹业萍, 邸浩洋, 金一锋*
(1.齐齐哈尔大学生命科学与农林学院, 黑龙江 齐齐哈尔 161006; 2.抗性基因工程与寒地生物多样性保护黑龙江省重点实验室,黑龙江 齐齐哈尔 161006; 3.齐齐哈尔大学理学院, 黑龙江 齐齐哈尔 161006)
草坪草在自然界中面临着干旱、盐分、氮、磷胁迫等多种非生物逆境胁迫,植物可启动自身的防御机制,以适应及抵抗逆境胁迫,蛋白质的磷酸化和去磷酸化过程是植物响应环境胁迫信号的重要机制[1-2]。蛋白激酶(Protein kinase)是植物中一种重要的调节因子,可通过膜受体蛋白感知环境信号,激活不同的蛋白磷酸化途径,调节下游抗逆基因的表达,进而抵抗逆境胁迫所带来的伤害。近年来,许多研究报道了与抗性相关的蛋白激酶,即受体蛋白激酶(Receptor-like protein kinases,RLK)、丝裂原活化蛋白激酶(Mitogen-activated protein kinase,MAPK)、钙依赖性蛋白激酶(Calcium-dependent protein kinases,CDPK)和蔗糖非发酵蛋白1(Sucrose non-fermenting 1,SNF1)激酶家族[3-6]。蔗糖非发酵1相关蛋白激酶SnRKs(Sucrose non-fermenting-1 related protein kinases)属于丝氨酸/苏氨酸Ser/Thr蛋白激酶(Serine/ threonine protein kinase)。
植物中SnRK家族已经扩展并分化为三个亚科:SnRK1(结构和功能上与SNF1和AMPK最相似)、SnRK2和SnRK3[7]。SnRK1蛋白激酶与酵母SNF1和哺乳动物中AMPK蛋白激酶高度同源,主要参与碳氮反应代谢和能量感应[6],SnRK1在调节生物和非生物胁迫反应以及植物生长发育也起着关键作用[8]。而SnRK2和SnRK3家族基因在信号通路和基因表达调控中起着重要作用[9],例如SnRK2s主要在ABA信号通路中发挥作用,SnRK3s在Ca2+信号作用下与CBL蛋白相互作用,调节各种应激反应[10-11]。
SnRK1基因家族是一个相对较小的亚家族,SnRK1基因已经从许多植物中分离出来,SnRK1是由一个催化亚基和两个调节亚基组成的异源三聚体复合物。研究证明,SnRK1可以调节蔗糖合成酶和ADP葡萄糖焦磷酸化酶基因表达,有助于马铃薯(SolanumtuberosumL.)块茎中的淀粉生物合成[12],SnRK1在水稻(Oryzasativa)、高粱(Sorghumbicolor)籽粒以及大麦(Hordeumvulgare)的花粉中起着类似的作用[13-14]。水稻包含4个SnRK1基因家族,根据氨基酸序列的相似性和表达模式,将其分为SnRK1a 和SnRK1b两个亚家族,其中OSK4和OSK35属于SnRK1b亚家族[15]。研究证明,水稻萌发和幼苗生长过程中SnRK1a蛋白激酶在糖信号转导中起着关键作用[16]。Thelander等研究表明,SnRK1在植物应对光周期生长的代谢过程中起到一定作用[17]。研究发现,水稻OSK4激酶可以与调节因子HDR1联合控制光周期开花[18],OSK35是水稻抗病性的正调控因子[19]。番茄(Lycopersiconesculentum)冷诱导基因ShCIGT可通过与SnRK1的相互作用提高番茄的非生物胁迫耐受性[20]。植物生长过程中SnRK1蛋白激酶和脱落酸信号传导途径关键基因之间相互作用显著[21]。综上可见,SnRK1家族基因在植物生长发育、抗逆性中起着重要作用。
草地早熟禾(PoapratensisL.)是多年生冷季型草坪草,其具有叶色浓绿、坪观质量良好、耐低温等优点,被广泛应用于我国北方地区的园林建设[22]。针对禾本科草坪草的SnRK家族基因功能的研究甚少,研究草地早熟禾SnRK1b亚家族OSK4基因的非生物胁迫表达模式,将丰富草坪草SnRK1抗逆机制的相关研究。本研究克隆得到草地早熟禾OSK4基因,进行了典型结构域、同源进化关系、理化性质等生物信息学分析,利用实时荧光定量PCR方法观测不同组织特异性及非生物胁迫下的表达机制,为进一步研究其功能奠定基础。
1 材料与方法
1.1 试验材料及处理方法
试验于2021年1—6月在齐齐哈尔大学生命科学与农林学院园林遗传育种研究室进行,试验供试材料为草地早熟禾‘午夜2号’品种。挑选饱满的种子将其置于培养土中(壤土∶细沙∶蛭石=3∶1∶1),播种量为12 g·m-2,将其放置光照培养箱中,培养条件参数设置为:昼夜温度为25℃/15℃,相对湿度60%,光/暗为14 h/10 h,光照强度为600 μmol·m-2·s-1,待培养3个月后进行取样及诱导处理。
选取生长健康、长势一致的草地早熟禾植株,将其根部洗净后置于沙子中,每日向直径为19.5 cm,高度为14 cm的盆中浇灌1/2 Hoagland水培营养液,待培养14 d后进行不同干旱、氮素、磷浓度溶液处理。具体处理分组为:(1)将10% PEG6000(Polyethylene glycol)加入1/2 Hoagland营养液中,对植株进行模拟干旱处理,干旱诱导时间分别为:0 h,2 h,16 h,对其叶部及根部进行取样,置于—80℃保存;(2)采用NaNO3为氮源,配置3种不同浓度(0 mM,1.5 mM,15 mM)NaNO3的1/2 Hoagland营养液(见表1),每天每盆一次性浇灌400 mL培养液,处理21 d后取样,置于-80℃保存。(3)采用磷酸二氢钾KH2PO4为磷源,配置3种不同浓度的KH2PO4的1/2 Hoagland营养液(见表2),KH2PO4浓度分别为0 mM,0.01 mM,1 mM,每天浇灌1次400 mL培养液,处理21 d后取叶样,置于-80℃保存。
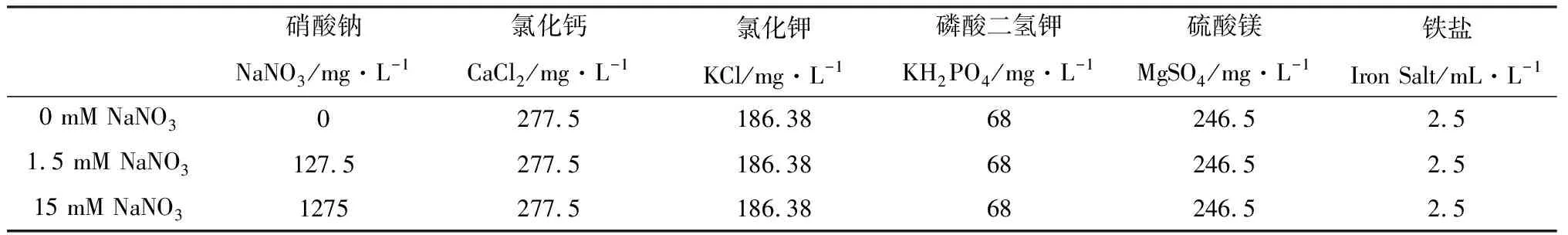
表1 不同氮浓度的营养液Table 1 Nutrient solutions with different nitrogen concentration
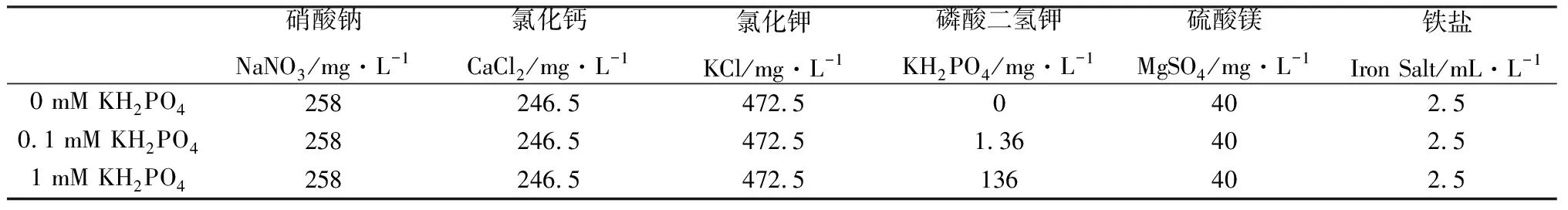
表2 不同磷浓度的营养液Table 2 Nutrient solutions with different phosphorus concentration
1.2 草地早熟禾OSK4基因的克隆
选取健康的草地早熟禾植株进行总RNA的提取,具体方法参照植物总RNA提取试剂盒(TianGen,Beijing),采用超微量荧光全功能分光光度计(DeNovix,USA)进行总RNA浓度、纯度的检测,利用2%琼脂糖凝胶电泳鉴定总RNA完整性。待草地早熟禾总RNA检测合格后,以其为模板,采用PrimeScriptTMII 1 st Strand cDNA Synthesis Kit (TaKaRa,Japan) 获得草地早熟禾cDNA,用于后续OSK4基因克隆。根据前期转录组测序得到的草地早熟禾相关基因序列为基础,并结合Genebank公布的二穗短柄草OSK4(OX_024312805.1)、节节麦OSK4(XP_02018300.1),设计特异性引物pPOSK4-F,pPOSK4-R引物序列见表3。草地早熟禾cDNA为模板,利用特异性引物pPOSK4-F/R,2xEs Taq MasterMix(CWBIO,CW0690)进行RT-PCR,反应程序为:94℃预变性2 min后,94℃变性30 s、退火56℃ 30 s、延伸72℃ 30 s,共32个循环,最终延伸72℃ 2 min,获得草地早熟禾OSK4基因序列。特异性引物的合成和PCR产物的测序均由生工生物工程股份有限公司(上海)完成。
1.3 草地早熟禾OSK4基因生物信息学分析
利用NCBI-CDD在线工具分析草地早熟禾OSK4蛋白保守结构功能域;利用MEGA7.0构建系统进化树,比对其同源性;利用ClustalW进行多物种OSK4编码氨基酸同源性比对分析;利用ProtParam分析OSK4编码氨基酸的基本理化性质;利用SOPMA在线预测蛋白质二级、三级结构。
1.4 实时荧光定量检测草地早熟禾OSK4基因
本研究分别提取草地早熟禾植株不同部位的RNA,选取部位分别为:根部、茎部、叶部、穗。提取PEG6000模拟干旱处理的植株叶部、根部的RNA,提取氮、磷胁迫后的草地早熟禾叶部RNA。待RNA纯度、浓度检测合格后反转录成cDNA,作为qRT-PCR模板。采用qRT-PCR相对定量方法,参照TB GreenTMPremix Ex TaqTMII进行试验。qRT-PCR反应体系为50 μL,包含:4 μL cDNA,2 μL Q-OSK4-F,2 μL Q-OSK4-R,17 μL ddH2O,25 μL TB Green Premix Ex Taq II。PCR条件为:PCR反应条件为:95℃预变性30 s,95℃ 5 s,60℃ 30 s,共45个循环。选择UBQ为内参基因,Q-UBQ-F,Q-UBQ-R引物见表3。该试验生物学重复3次,试验重复3次,利用2-ΔΔCt法处理数据。
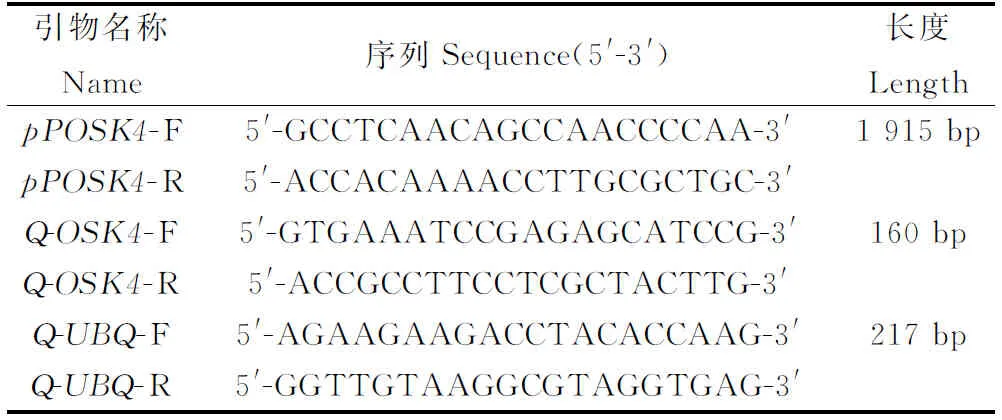
表3 所需RT-PCR及qRT-PCR引物Table 3 RT-PCR and QRT PCR primers
2 结果与分析
2.1 草地早熟禾OSK4基因编码区克隆及生物信息学分析
RNA凝胶电泳图结果显示28S和18S两条条带清晰且比值大于1(图1),RNA吸光值OD260/280为1.95,可见,此RNA符合后续试验要求,保存到—80℃冰箱中备用。利用设计的特异性引物OSK4-F/R及草地早熟禾cDNA为模板进行PCR扩增,图2为PCR产物琼脂糖凝胶电泳图。
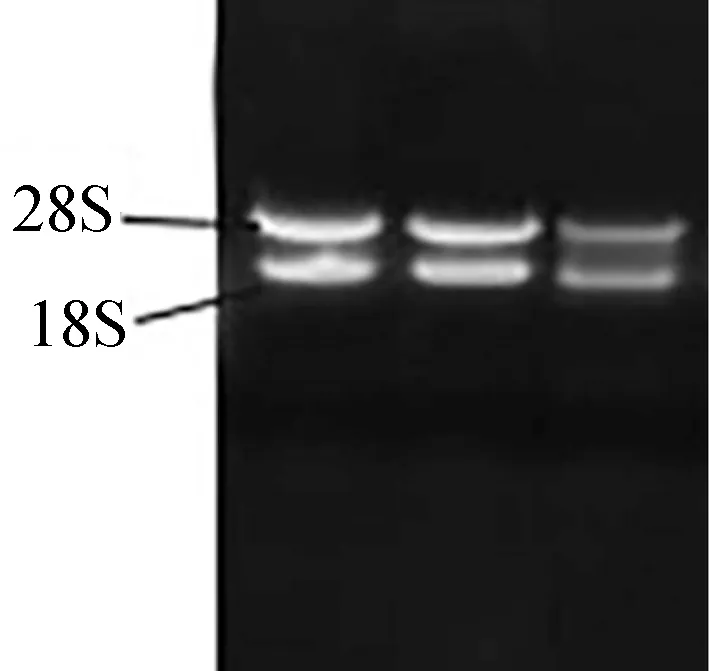
图1 草地早熟禾总RNA琼脂糖凝胶的电泳图Fig.1 Total RNA agarose gel electrophoresis picture of kentucky bluegrass
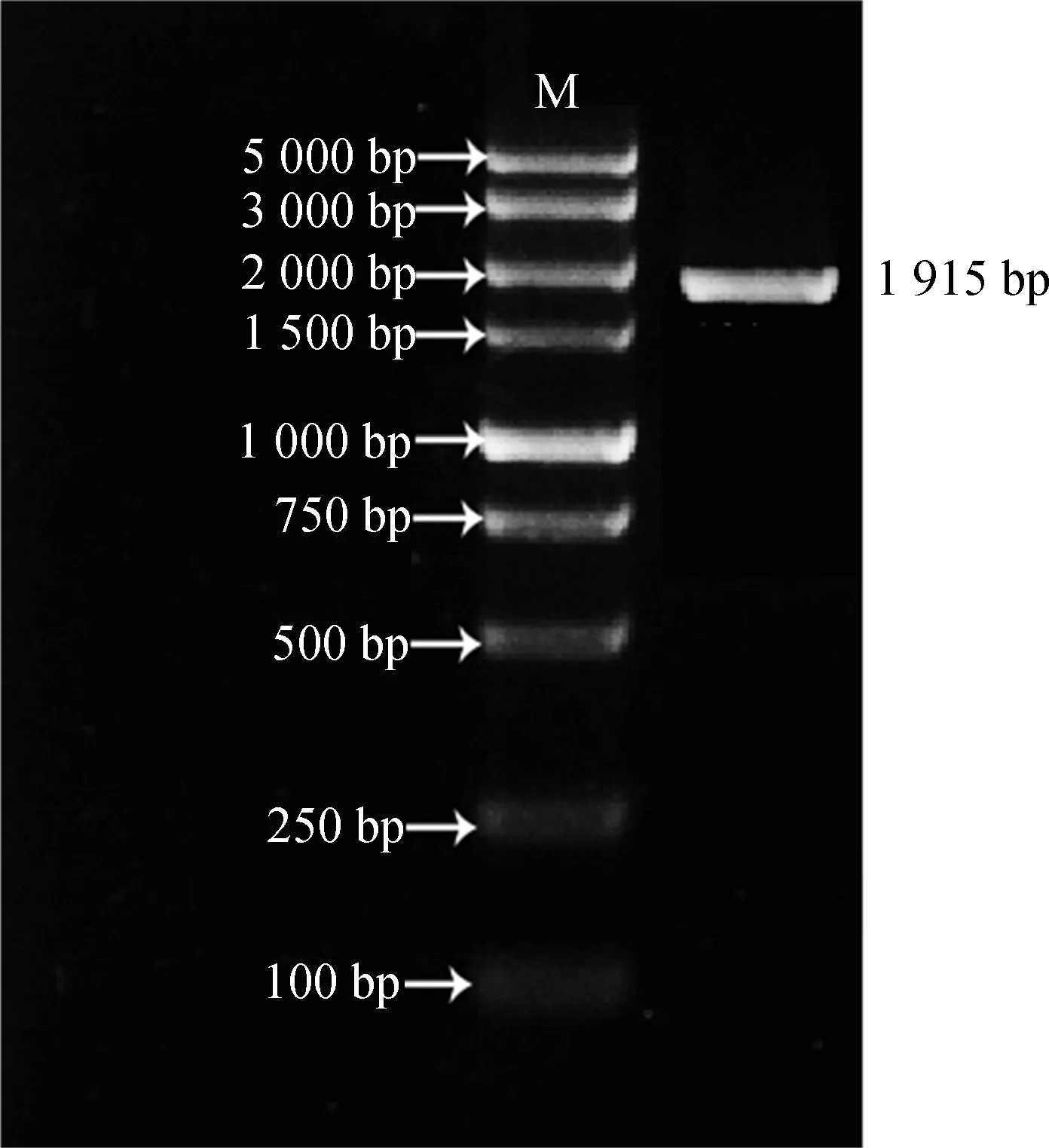
图2 草地早熟禾OSK4基因PCR电泳图Fig.2 PCR electrophoresis of OSK4 gene in kentucky bluegrass
利用NCBI在线分析,草地早熟禾OSK4基因序列长度为1 915 bp,ORF包含1 530 bp,共编码509个氨基酸。NCBI-CDD在线分析结果表明,该蛋白为STKc_AMPK_alpha and AMPKA_C典型结构域蛋白,包含典型的结构域STKc_ AMPK_alpha,UBA_SnRK1_plant和AMPKA_C(图3)。利用MEGA7.0对多物种OSK4编码氨基酸进行进化树分析,发现草地早熟禾OSK4编码氨基酸与二穗短柄草、小麦、节节麦OSK4编码氨基酸同源性最高,分别为94.5%,94.11%,93.91%(图4)。利用在线软件MEME进行OSK4保守序列区域分析,MEME设置成15个保守基序进行比较,由图4可知出草地早熟禾与水稻、二穗短柄草、节节麦等禾本科植物的OSK4蛋白结构域非常相似,同时可以看到禾本科植物的OSK4与OSK3高度相似。草地早熟禾OSK4与小麦、节节麦、二穗短柄草OSK4氨基酸序列进行多序列比较,发现其包含ATP binding site,Active site,Activation loop结合位点,及heterotrimer interface,beta subunit interface,gamma subunit interface三种典型的多肽位结合位点等位置(图5)。
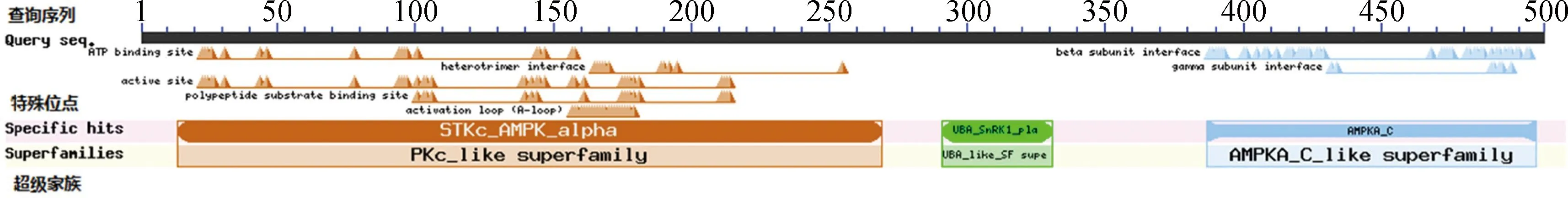
图3 草地早熟禾OSK4的CDD结构域Fig.3 Conserved Domain Database of OSK4 in kentucky nluegrass
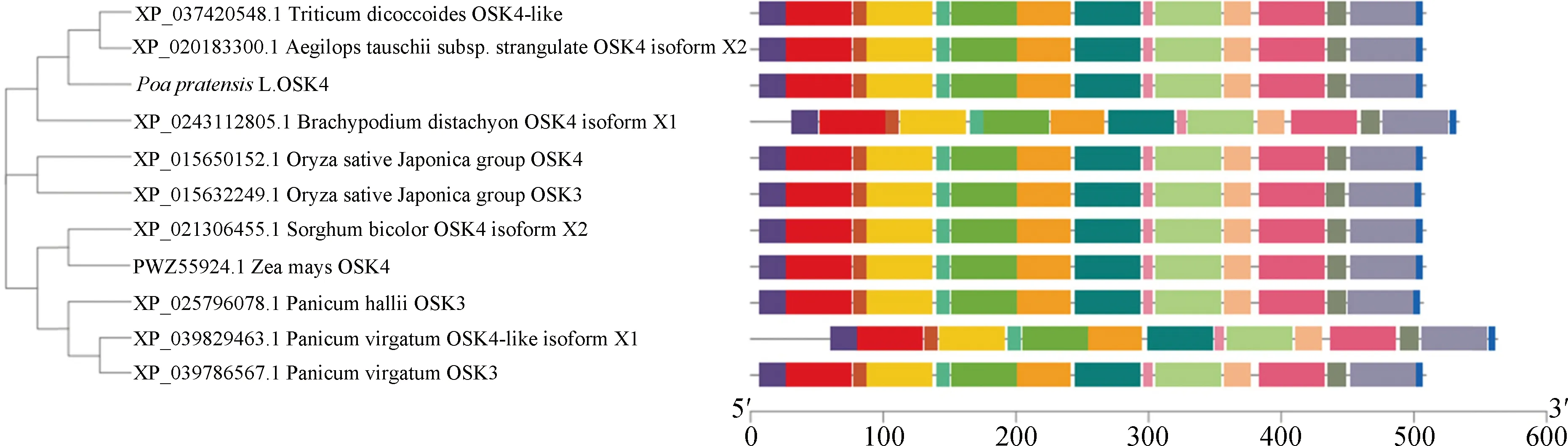

图4 草地早熟禾OSK4氨基酸序列的进化树及MEME分析Fig.4 Phylogenetic tree and MEME analysis of amino acid sequence of OSK4 in kentucky bluegrass
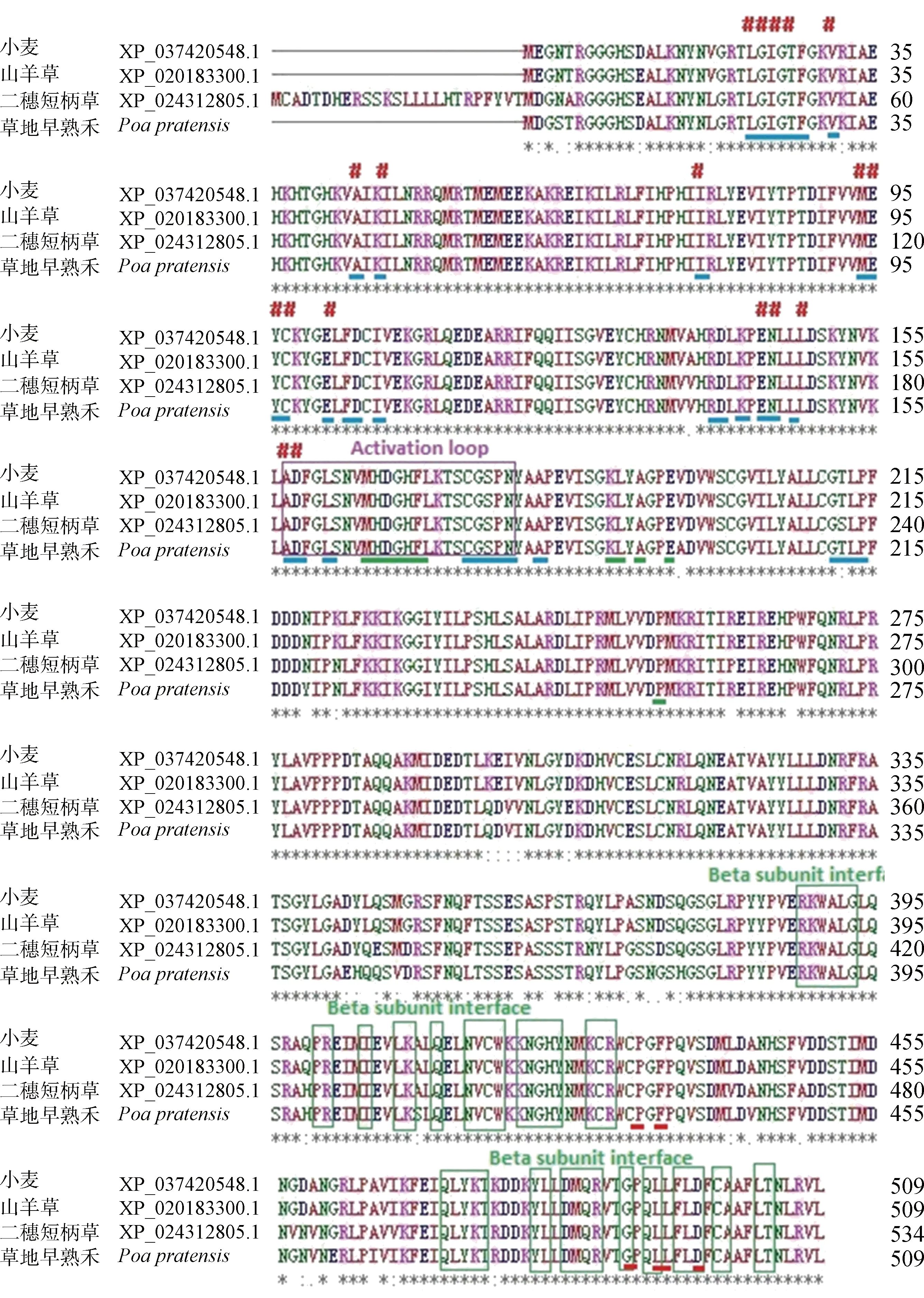
图5 不同植物的OSK4氨基酸多序列比对图Fig.5 Comparison of OSK4 amino acid sequences of different plants注:#代表ATP binding site [化学结合位点],蓝色线代表active site [活性位点],绿色线代表heterotrimer interface [多肽结合位点],紫色框代表activation loop (A-loop),绿色框代表beta subunit interface [多肽结合位点],红色线代表gamma subunit interface [多肽结合位点]Note:# represents ATP binding site [chemical binding site],blue line represents active site [active site],The green line represents the heterotrimer interface [Polypeptide binding site],the purple box represents the activation loop (A-loop),The green box represents beta Subunit Interface [Polypeptide Binding site] and the red line represents Gamma Subunit Interface [Polypeptide binding site]
利用Netphos对磷酸化位点进行预测分析,发现草地早熟禾OSK4氨基酸磷酸化位点含有18个Serine(丝氨酸)、9个Threonine(苏氨酸)和6个Tyrosine(酪氨酸)。利用Wolf-Psort亚细胞定位预测,分析结果显示草地早熟禾OSK4定位于细胞质中。利用SOPMA程序对OSK4蛋白质二级结构预测分析(见图6),结果表明,该蛋白质的二级结构由α-螺旋(35.17%)、不规则卷曲(40.47%)、β-转角(6.09%)和延伸链(18.27%)共同构成。利用SWISS-MODEL在线软件对OSK4蛋白三级结构进行建模(见图7),发现草地早熟禾OSK4蛋白的三级结构中主要由α-螺旋、延伸链、β-转角、无规则卷曲4个部分组成,与蛋白二级结构预测的结果一致。

图6 草地早熟禾OSK4蛋白的二级结构Fig.6 Secondary structure of OSK4 protein in kentucky bluegrass注:图中蓝色表示α-螺旋、红色表示延伸链、绿色表示β-转角、紫色表示不规则卷曲Note:In the figure,blue represents α-helix,red represents extended chain,green represents β-corner,and purple represents irregular curl
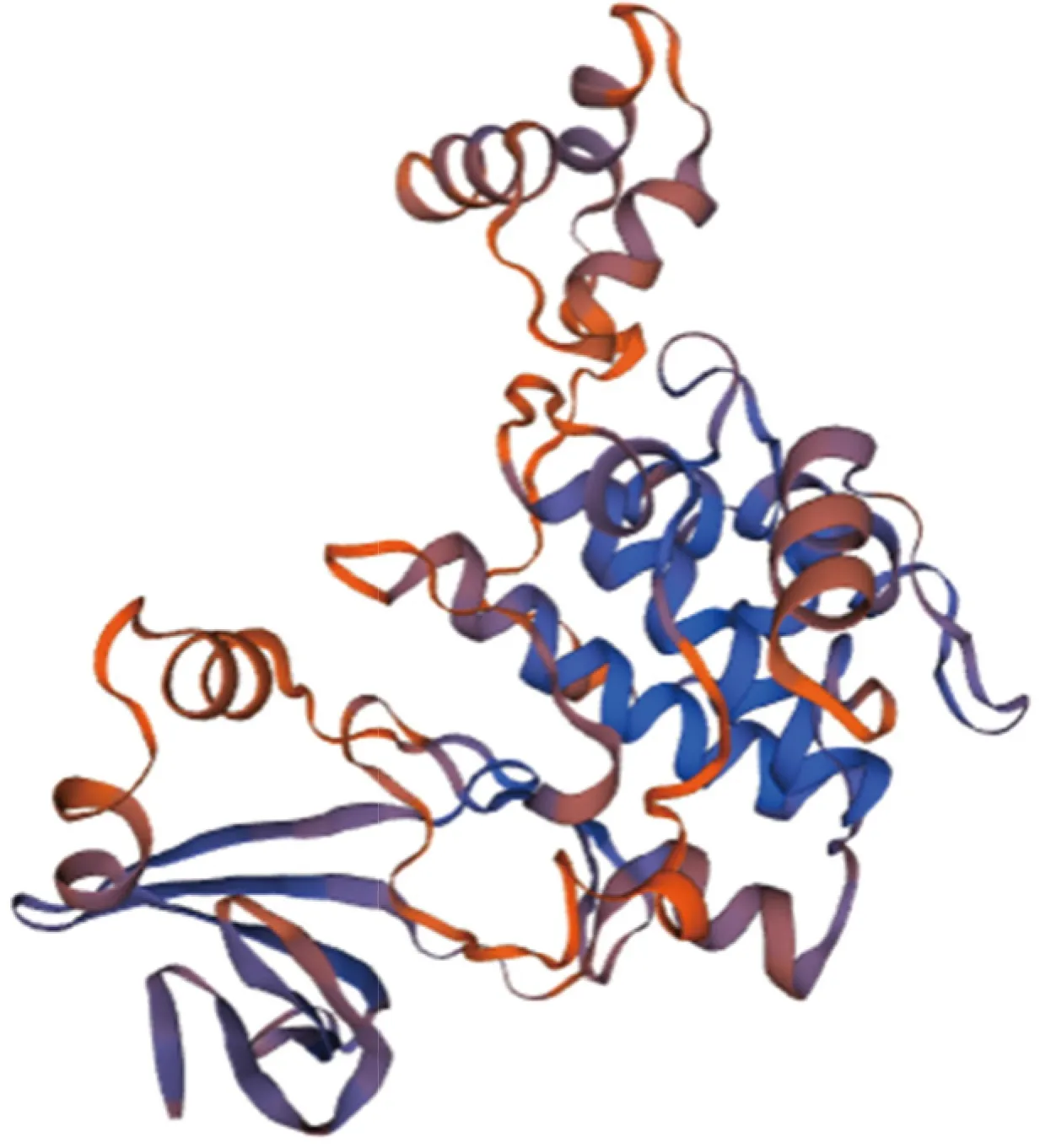
图7 草地早熟禾OSK4蛋白的三级结构Fig.7 Tertiary structure of OSK4 protein in kentucky bluegrass
2.2 草地早熟禾OSK4基因表达水平的分析
2.2.1草地早熟禾OSK4基因组织特异性 由图8a可知,草地早熟禾OSK4基因在不同组织部位中表达量差异显著(P<0.05),OSK4基因相对表达量高低顺序为:根部>茎部>穗>叶部,其中根部表达量最高,叶部表达量最低,根部是叶部的3.87倍。
2.2.2干旱胁迫对草地早熟禾OSK4基因表达水平的影响 本研究利用10%PEG6000模拟干旱胁迫处理草地早熟禾植株,检测干旱胁迫对草地早熟禾根部、叶部OSK4基因表达水平的影响,由图8b可知,干旱胁迫显著降低草地早熟禾根部、叶部OSK4基因的相对表达量。10% PEG6000模拟干旱期间,草地早熟禾根中OSK4基因相对表达量呈现先极速下降后上升的趋势,OSK4基因相对表达量为:0 h>16 h>2 h,2 h的OSK4表达量仅为0 h的24.59%,16 h的OSK4相对表达量是0 h的34.65%。10% PEG6000模拟干旱期间,草地早熟禾叶中OSK4基因呈现持续下降的趋势,OSK4基因相对表达量大小顺序为:0 h>2 h>16 h,16 h的OSK4表达量为0 h的43.44%。
2.2.3氮素胁迫对草地早熟禾OSK4基因表达水平的影响 本研究利用NaNO3水培液处理草地早熟禾植株,观测不同氮素浓度诱导对草地早熟禾植株中OSK4基因表达水平的影响。由图8-c可知,不同梯度氮素浓度处理显著影响草地早熟禾OSK4基因的相对表达量(P<0.05),OSK4基因相对表达量大小顺序为:15 mM NaNO3>1.5 mM NaNO3>0 mM NaNO3,草地早熟禾OSK4基因表达水平随着NaNO3浓度的增加而呈现上调趋势。
2.2.4磷素胁迫对草地早熟禾OSK4基因表达水平的影响 本研究利用KH2PO4水培液处理草地早熟禾植株,观测不同磷浓度诱导对草地早熟禾植株中OSK4基因表达水平的影响。由图8-d可知,不同KH2PO4浓度处理液对草地早熟禾OSK4基因相对表达量影响显著(P<0.05),OSK4基因相对表达量大小顺序为:0.01 mM KH2PO4>0 mM KH2PO4>1 mM NaNO3。草地早熟禾OSK4基因的表达水平随着KH2PO4浓度的增加呈现先升高后降低趋势,0.01 mM KH2PO4处理组中OSK4基因的相对表达量分别是0 mM KH2PO4和1 mM KH2PO4的1.82倍和1.56倍。可见,低磷环境(0.01 mM KH2PO4)可促进草地早熟禾OSK4基因的表达。
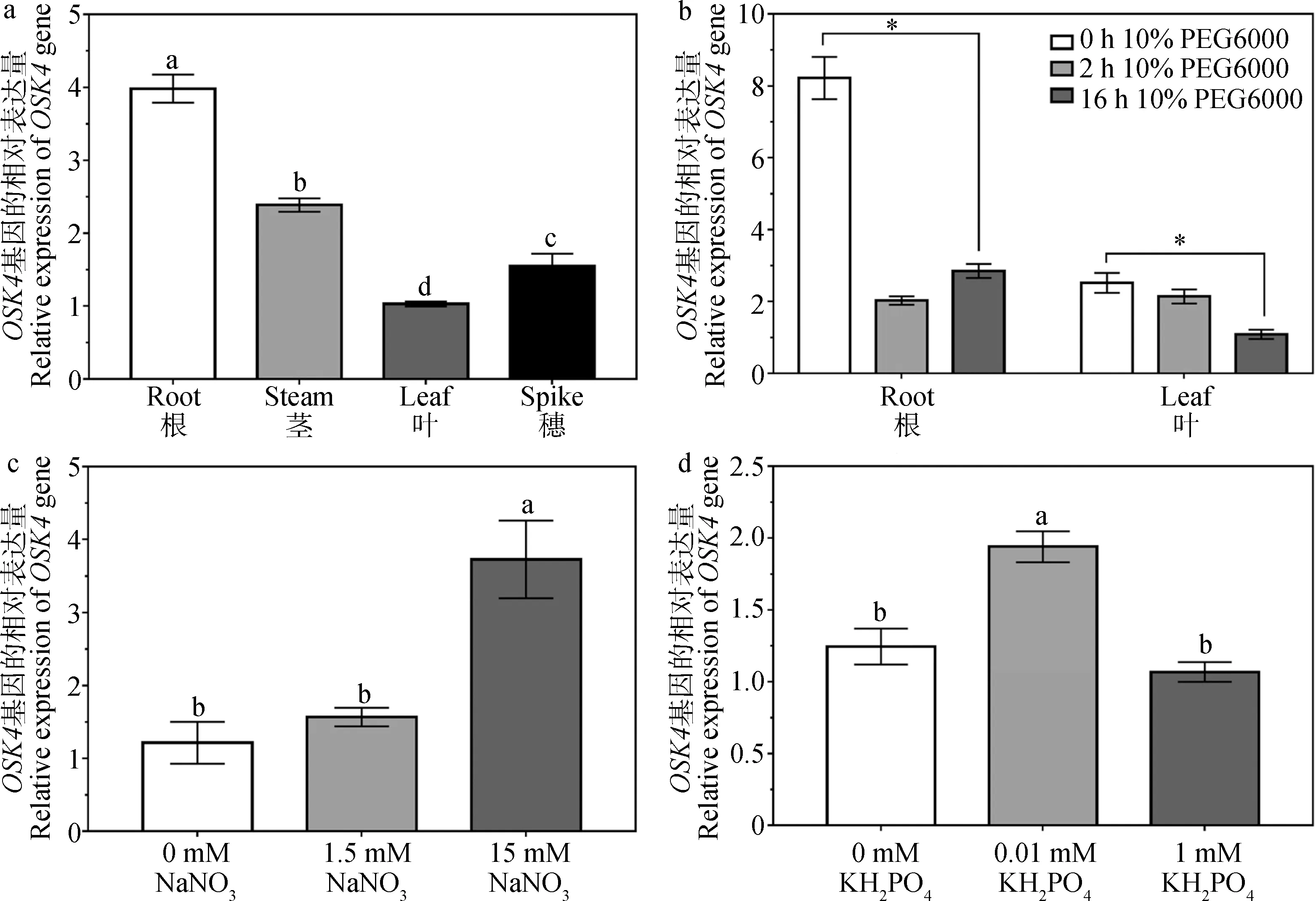
图8 草地早熟禾OSK4基因在组织部位及非生物胁迫下的表达水平分析图Fig.8 Expression level analysis of OSK4 gene in Kentucky Bluegrass under tissue and abiotic stress注:不同小写字母表示不同组织部位、氮素溶液、磷素溶液中该基因的表达量在0.05水平上差异显著,*表示不同干旱胁迫时间中该基因的表达量在0.05水平上差异显著Note:Different lowercase letters indicate significant difference at the 0.05 level,* indicates significant difference at the 0.05 level
3 讨论
3.1 SnRK1家族基因结构特点
拟南芥(Arabidopsisthaliana)、水稻和大麦基因组数据表明,在水稻中鉴定了50个SnRK基因(4个OsSnRK1,11个OsSnRK2和35个OsSnRK3),在拟南芥中鉴定了39个(3个AtSnRK1,10个AtSnRK2和26个AtSnRK3),在大麦中发现了50个(6个HvSnRK1亚家族,10个属于HvSnRK2,34个属于HvSnRK3)[23-24]。研究者将大麦SnRK1亚家族基因划分为3个子类,包括SnRK1a(SnRK1.1和1.2)、SnRK1b(SnRK1.3和1.4)和SnRK1b*(SnRK1.5和1.6)[23]。通常SnRK1的基因实际上编码了一个异三体蛋白的催化亚单位,AMPK也包含三个亚基,一个催化亚基α亚单位和附件β以及γ亚单位[6]。因此,SnRK1基因有时被称为AMPKα,催化剂α亚基包含一个高度保守的N-末端催化结构域和一个可变的C-末端调节结构域,这对于形成复杂的相互作用(与β以及γ亚单位)并调节激酶活性,与SnRK1不同,其他两个亚科(SnRK2s和SnRK3s)在植物中是独特的[25]。本研究发现草地早熟禾SnRK1b亚家族OSK4编码氨基酸具有STKc_AMPK_alpha,UBA_ SnRK1_plant和AMPKA_C典型的结构域,同时,草地早熟禾OSK4编码氨基酸包含典型的多肽位结合位点,与图5中小麦、山羊草、二穗短柄草OSK4编码氨基酸特征一致,可见禾本科物种之间OSK4典型结构域高度相似度,SnRK1氨基酸结构较为保守。
3.2 草地早熟禾OSK4基因组织特异性分析
有研究表明水稻OSK4基因在未成熟的水稻种子中相对表达量最高[26],其次是在其圆锥花序、叶鞘、根部,在萌发的种子及叶部中相对表达量最少[13]。本研究中草地早熟禾根部OSK4相对表达量显著高于叶部,这与水稻相应研究结果相似[13]。水稻OSK4基因在不同部位的表达模式,可能与发育期颖果和发生库源转换的叶鞘中的蔗糖和淀粉代谢密切相关[13]。陈润娟研究发现根茎是草地早熟禾储存碳水化合物的主要场所,不同部位碳水化合物含量为根茎>茎基>叶片,这与本研究的OSK4在不同组织部位中的相对表达量趋势相似[27]。草地早熟禾OSK4基因在不同部位的表达水平可能与碳水化合物代谢过程有关,如蔗糖和淀粉代谢,但仍需进一步研究。
3.3 逆境胁迫对OSK4基因表达模式的影响
Samarina研究发现茶树SnRK1b基因随着干旱程度的增加呈现先增加后降低的趋势,相对表达量的变化为:12 h>6 h>24 h>0 h>3 h>48 h[28]。本研究中草地早熟禾叶部OSK4表达水平与干旱胁迫程度呈负相关关系,干旱胁迫显著降低草地早熟禾根部、叶部OSK4基因的相对表达量,可见,根部受干旱胁迫抑制程度更高。针对大麦SnRK家族基因的启动子的研究发现,HvSnRK1.6基因启动子中包含与干旱相关的调控元件[23]。蛇舌草(Hedychiumcoronarium)HcSnRK1.3,HcSnRK1.4基因启动子中也包含与干旱相关的调控元件[29]。植物SnRK1家族基因积极响应干旱胁迫,可能与其启动子包含干旱胁迫调控元件有关。
研究发现,SnRK1家族基因与植物氮吸收、氮代谢过程有一定的关联性。Wang等研究发现过表达番茄SnRK1基因有利于其碳同化、氮吸收和果实发育过程[30]。Sanagi等研究发现小麦胚中氮代谢相关的天冬酰胺合成酶基因表达水平随着硫水平的下降而上调,SnRK1参与这个调控过程[31]。在低氮环境下拟南芥SnRK1激酶活性下调,可直接磷酸化成花基因BHLH4,而SnRK1在高氮条件下负调控下游成花基因FT的转录水平[32]。可见,SnRK1与植物的氮素调控存在一定的关联。本研究发现,NaNO3浓度的增加可以提高OSK4基因的相对表达量,叶部OSK4表达水平与氮素浓度程度呈正相关关系。当番茄处于磷酸盐(Pi)饥饿环境中,番茄SlSnRK3.10a,SlSnRK3.15a和SlSnRK3.26可被激活[33],磷胁迫显著影响SnRK家族基因的表达。有关于SnRK家族基因响应磷胁迫的研究报道较少,本研究发现草地早熟禾OSK4基因适应磷胁迫的表达模式与氮胁迫的表达不同,草地早熟禾OSK4基因表达水平随着KH2PO4浓度的升高呈现先增加后降低的趋势,低磷环境(0.01 mM KH2PO4)可促进草地早熟禾OSK4基因的表达。
4 结论
本研究利用RT-PCR方法克隆获得草地早熟禾SnRK1亚家族OSK4基因,经生物信息学分析发现禾本科植物的OSK4蛋白具有较高的保守性。草地早熟禾中OSK4基因的相对表达量存在组织特异性,抵御干旱胁迫时OSK4基因在根部、叶部存在不同的响应机制,SnRK家族基因在协调植物生长发育过程的作用仍需进一步研究。草地早熟禾OSK4基因对氮、磷胁迫环境中响应较为敏感,氮素浓度与其表达量呈现正相关关系,低磷处理(0.01 mM KH2PO4)促进OSK4基因上调表达。后续研究可进一步挖掘草坪草SnRK家族基因在植物能量代谢、稳定碳氮平衡中的作用,为草地早熟禾抗逆机理的进一步研究奠定一定理论依据。
