鲴鲢鳙混养系统中微生物对氮素迁移转化的影响
2022-02-25高月香张毅敏陈玲玲郭艳敏朱月明于江华
张 哲,高月香,张毅敏,*,陈玲玲,郭艳敏,朱月明,于江华
鲴鲢鳙混养系统中微生物对氮素迁移转化的影响
张 哲1,高月香1,张毅敏1,2*,陈玲玲1,郭艳敏1,朱月明1,于江华2
(1.生态环境部南京环境科学研究所,江苏 南京 210000;2.南京信息工程大学环境科学与工程学院,江苏 南京 211800)
通过实验室模拟富营养化水体进行鲢()鳙()鲴()混养,结合同位素标记与微生物检测,研究其中沉积物微生物群落的变化及其对氮素迁移转化的影响,揭示鲢鳙鲴混养作用机制.结果表明,鲢鳙鲴混养增加了沉积物中氨化细菌(芽孢杆菌属与假单胞菌属)的数量,实验结束时,与其它组相比,鲢鳙鲴组的芽孢杆菌属()与假单胞菌属()总占比最高,为31.2%,这增强了氨化作用,减少了沉积物中有机氮的含量,使得鲢鳙鲴组的15N (底质沉积物)在实验结束时降至726.8‰.鲢鳙鲴混养提高了沉积物中反硝化菌属(动胶菌属与芽胞杆菌属)的数量,实验结束,鲢鳙鲴组动胶菌属()与芽胞杆菌属()占比显著高于鲢鳙组(<0.05),达到8.17%、33.7%,使得系统中反硝化反应加强,使水体中更多的硝酸盐氮转化为气态氮并移出水体,实验结束时,与其它组相比,鲢鳙鲴组15N(硝酸盐氮)降低至96.3‰,15N总含量也降低至6.94µmol.
鲢鳙鲴;混养;稳定同位素技术;氮素;微生物群落
1996年,Xie[1]提出了“非经典生物操纵理论”,即通过滤食性鱼类鲢()、鳙鱼()摄食控制蓝藻数量[1],之后被广泛应用.但鲢鱼的大量排泄会增加水体氮素水平,影响水质[2],所以鲢鳙鱼控藻引起氮素升高也成为了其应用的限制.微生物群落通过氮矿化、硝化与反硝化作用等方式影响水中氮素的迁移转化[3],并且一般是在沉积物-水界面进行的[4].陈玲玲等[5]采用了鲢鳙鱼与一种底层杂食性鱼类细鳞斜颌鲴()[6-7]混养,利用鲴鱼习惯摄食沉到水体底部的有机碎屑和腐殖质及鱼类粪便[8]的特性,对其在食物链起到的作用进行研究,却忽略了鲴鱼摄食时对沉积物的生物扰动,能够对沉积物-水界面中微生物群落产生影响,并影响系统氮素循环[9].因此,有必要研究鲢鳙鲴混养的沉积物微生物变化及其对氮素迁移转化的影响.
稳定同位素技术因其能够稳定示踪,通常被用作污染物的追踪手段[10].王银平等[11]通过对鱼类喂食15N标记的微囊藻干粉颗粒,进行系统中氮素迁移转化的研究.不仅能够显示出系统氮素迁移转化路径,还能够直观地得到氮素在系统中水相、生物相、沉积物相含量变化.
本文采用室外模拟实验,在模拟水体中混养不同组合的鲢鳙鲴鱼,通过喂食15N标记的微囊藻进行同位素标记并定期检测系统各相同位素比值和沉积物微生物样本,研究鲢鳙鲴混养系统中微生物群落的变化和各相中15N的变化,旨在了解鲢鳙鲴混养系统中微生物群落的变化以及其对氮素迁移转化的影响,为进一步利用鲢鳙鲴混养生物操纵技术控制富营养化水体提供理论参考.
1 材料与方法
1.1 材料
实验鲢鱼()体重: (65.34±2.25)g;体长:(16.54±0.78)cm,鳙鱼()体重:(45.48±3.75)g;体长:(12.24± 0.69)cm,由芜湖红鑫生态农业有限公司提供.实验鲴鱼为细鳞斜颌鲴(),体重:(14.45±4.43) g;体长:(13.25±0.63)m (以下简称鲴鱼),从湖南醴陵市国家鲴鱼良种场引种.实验用水取自富营养化池塘水,并利用200目浮游生物采集网去除浮游生物影响.
15N标记微囊藻颗粒的制备:用1g/L的15NH4Cl (98atom%15N)的溶液培养铜绿微囊藻2周后,冷冻干燥并制成颗粒,平均投放入各桶中,各组平均投放15N含量为13.84μmol.
1.2 实验设计
实验系统由1m3实验用桶、实验用水和模拟底质组成,布置在室外空地.将清洗后的河沙平铺在桶底作为模拟底质,并将实验用水注入850L后静置2周.实验设置3组,每组3个重复,对照组(1#~3#桶)不投放鱼;鲢鳙组(4#~6#桶)投放10尾鲢鱼、6尾鳙鱼;鲢鳙鲴组(7#~9#桶)投放10尾鲢鱼、6尾鳙鱼与15尾鲴鱼.实验进行30d,并24h曝气处理.实验开始前,挑选健康活泼,体型相似的鱼类,进行24h饥饿处理.
实验开始前,将15N标记颗粒用纱布包裹投入桶中,实验期间鱼类生命体征良好.并在试验开始前1d和第1, 5, 10, 15, 20, 25, 30d采集浮游藻类(过滤水样收集)、底部沉积物(利用沉积碎屑捕获器收集)、水样,同步进行稳定同位素比值测定;于实验开始前1d和第1, 5, 10, 15, 20, 30d分别取鲢鳙组鲢、鳙和鲢鳙鲴组鲢、鳙、鲴进行检测,同位素比值单位为‰;并在第15,30d分别取样进行水体沉积物微生物检测,对照组为D1(D1.1,D1.2,D1.3)与D2(D2.1,D2.2, D2.3);鲢鳙组为L1(L1.1,L1.2,L1.3)与L2(L2.1,L2.2, L2.3);鲴鲢鳙组为G1(G1.1,G1.2,G1.3)与G2(G2.1, G2.2,G2.3),共18个样品.
1.3 指标测试与分析方法
微生物检测采用Hiseq2500平台进行16SrRNA 基因高通量测序.
15N同位素比值测定:测量氨氮同位素的样品采用Lehmann等[12]方法进行预处理,测量氨氮同位素的样品采用改进的Holmes法进行预处理[13],浮游藻类、鱼体及底质沉积物采集后冷冻干燥,之后全部送入元素分析仪-同位素比质谱仪联机(FLASH 2000- Thermo Fisher DELTA V advantage,测定精度15N£±0.1‰)中测定同位素,并通过公式[14]计算.
引入来表示样品中同位素比值的变化,单位单位为‰.计算公式为

式中:样品为样品中15N与14N丰度之比;标准为标准物的15N与14N丰度之比,本文采用大气氮标准.

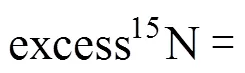

1.4 实验数据处理
数据采用SPSS.2020和Origin2018软件处理并采用单因素方差分析,运用CANOCO 5及QIIME软件进行微生物数据相关处理.
2 结果与分析
2.1 微生物群落分析
第15, 30d的水体沉积物18个样品共产生1449个OUT,为了其数据处理的合理性,对每个样品进行了随机抽平处理.
2.1.1 物种多样性分析 由图1可知,对照组、鲢鱅组和鲴鲢鱅组各组的样品覆盖度均达到0.99,接近1.实验第15, 30d,Chao指数均显示为对照组<鲴鲢鳙组<鲢鳙组,且有鱼组第15d指数大于第30d.实验期间,对照组的Shannon指数并无太大变化,而有鱼组第30d较第15d检测的样品有了明显降低,并表现为鲴鲢鳙组<鲢鳙组.

从下到上5条线分别代表最小值,第1个四分位数,中位数,第3个四分位和最大值
D1:对照组第15d; D2:对照组第30d; L1:鲢鳙组第15d; L2:鲢鳙组第30d; G1:鲢鳙鲴组第15d; G2:鲢鳙鲴组第30d,下同
2.1.2 物种差异性分析 应用QIIME软件并通过迭代算法计算,在不考虑物种丰度的情况下进行计算,得到结果并绘制样品差异性矩阵热图与PCoA分析图.
由图2可知,与有鱼组相比,对照组实验期间物种差异性变化较小,有鱼组差异性较大,且鲢鳙组高于鲴鲢鳙组,实验第15d时,对照组平行样品间的差异性明显大于有鱼组,有鱼组中鲴鲢鳙组差异性最小;实验第30d时,鲢鳙组平行样品间差异性较大,对照组和鲴鲢鳙组相对较小,由图2中的样品差异性矩阵热图可知,实验期间不同系统间表现为有鱼组彼此间差异性小于与对照组间的差异性.
2.1.3 群落组成组成及相对丰度分析 选出代表序列,与已知16S数据库进行比对后进行物种分类,绘制profiling柱状图.
由图3a可知,在门水平上,实验前期,对照组、鲢鳙组和鲴鲢鳙组变形菌门(Proteobacteria)占比最大,除此之外,浮霉菌门(Planctomycetes)和厚壁菌门(Firmicutes)相对占比也较高.随着实验的进行,3组微生物物种在样品中相对百分比有所变化,主要表现为有鱼组中厚壁菌门占比升高,浮霉菌门占比下降,对照组物种在门水平上变化不大,实验结束时,鲢鳙组和鲴鲢鳙组厚壁菌门占比由7.90%、9.48%升高至24.30%、36.53%,而浮霉菌门由15.33%、13.44%降低至 6.13%、1.76%,而且鲴鲢鳙组变化幅度高于鲢鳙组.变形菌门变化不大,仍为优势菌门.
由图3(b)可知,在纲水平上分析,实验期间对照组变化并不明显,而有鱼组β-变形菌占比有小幅上升,同时α-变形菌纲占比小幅下降.有鱼组中芽孢杆菌纲占比升高,浮霉菌纲占比下降,且鲴鲢鳙组变化幅度大于鲢鳙组,实验第30d时,两组芽孢杆菌纲占比分别为24.18%和34.41%,上升幅度分别是17.46和25.52个百分点,而浮霉菌纲占比分别是4.59%和 1.64%,下降了7.24 和 8.45个百分点,同时鲴鲢鳙组中芽孢杆菌纲成为优势菌纲.
由图3(c)可知,在属水平上,实验前期,对照组、鲢鳙组和鲴鲢鳙组均以假单胞菌属()占比最高,有鱼组中浮霉状菌属()占比仅次于假单胞菌.实验后期,鲢鳙组硝化螺菌属()有所上升至8.36%,假单胞菌属占比下降.鲢鳙鲴组中假单胞菌属基本保持不变,但动胶菌属()占比明显上升至8.17%.除此之外,鲢鳙组和鲴鲢鳙组中芽胞杆菌属()占比明显上升,分别达到22.6%和33.7%,并且鲢鳙鲴组的芽孢杆菌属(Bacillus)与假单胞菌属(Pseudomonas)总占比各组最高,为31.2%.

图2 样品差异性矩阵热图与PCoA分析

2.2 氮素在各相中的变化
2.2.1 水相变化(15N(氨氮),15N(硝酸盐氮)) 由图4可知,实验开始后有鱼组15N(氨氮),15N(硝酸盐氮)开始上升,15N(氨氮)在10d达到最大值,鲢鳙组与鲢鳙鲴组分别为688.3‰、586.7‰,之后开始下降,最终鲢鳙组>鲢鳙鲴组;而对于15N(硝酸盐氮)鲢鳙鲴组在20d达到最大值后下降至96.3%,鲢鳙组则一直保持上升,实验结束时,鲢鳙组>鲢鳙鲴组.对照组15N(氨氮),15N(硝酸盐氮)一直小幅上升并低于有鱼组.
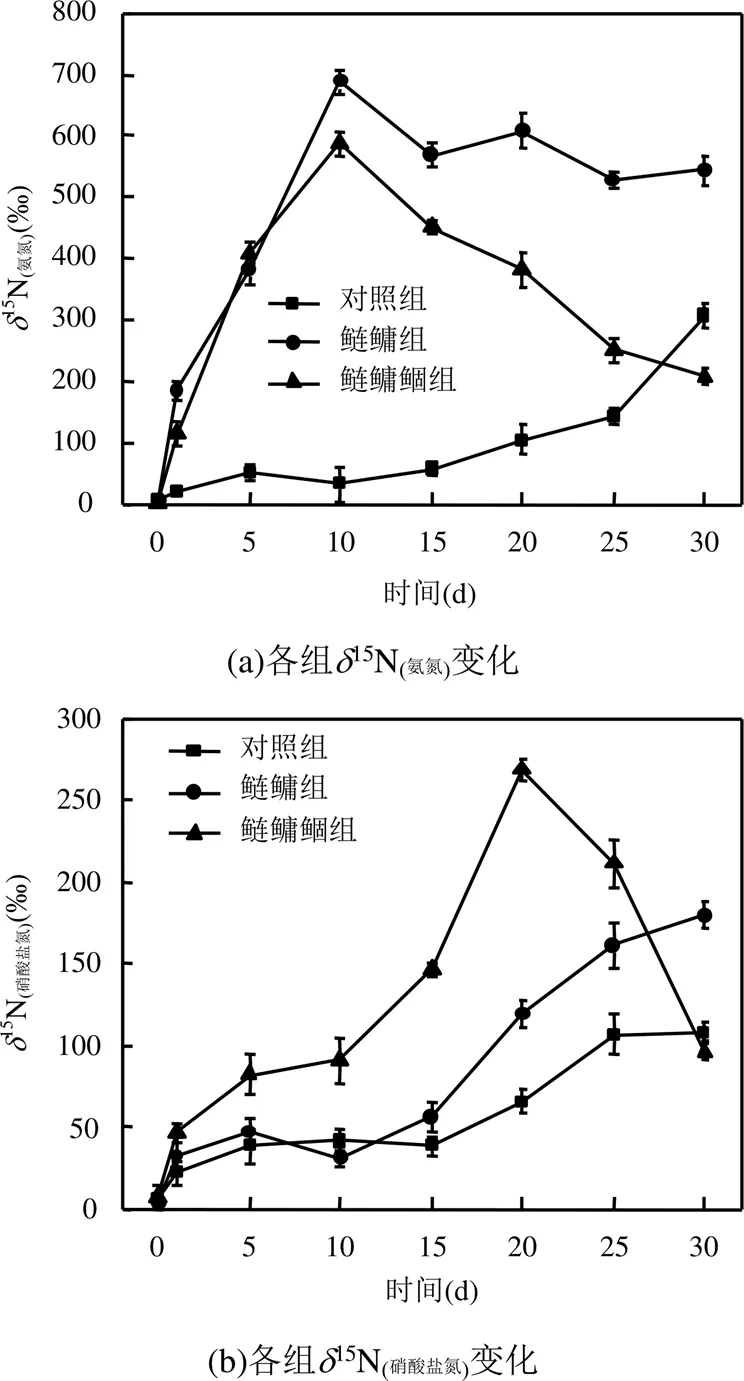
图4 各组水相中δ15N的变化
2.2.2 生物相变化(15N(浮游藻类),15N(鱼体)) 由图5(a)可知,实验开始后有鱼组15N(浮游藻类)开始上升,在第5d来到最大值后开始下降,鲢鳙组在第10d开始停止下降并趋于稳定,而鲢鳙鲴组持续缓慢下降,最终鲢鳙组>鲢鳙鲴组(<0.01);而对照组在前期大幅增长至15d达到最大值933.3‰,之后缓慢下降至实验结束,但仍显著高于实验组(<0.01).
由图5(b)可知,鳙鱼的15N(鱼类)变化基本一致,在前期短暂升高后,在后期略有下降并趋于平缓,而鲴鱼在实验期间不断上升,最终远高于两组鲢鳙鱼体内15N含量(<0.01).

图5 各组生物相中δ15N的变化
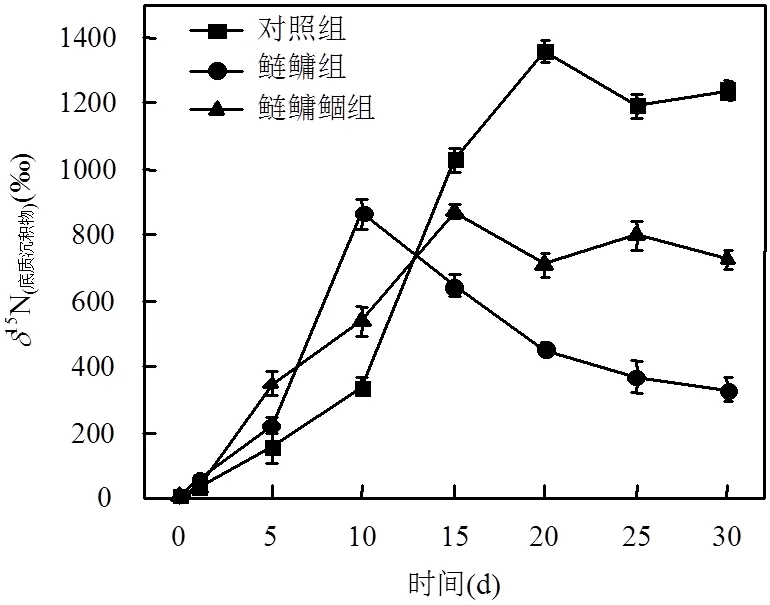
图6 各组沉积物相中δ15N的变化
2.2.3 沉积物相变化(15N(底质沉积物)) 由图6可知,实验初期,鲢鳙组和鲢鳙鲴组的15N(底质沉积物)持续增长,10d时,鲢鳙组最大值865.4‰,随后鲢鳙鲴组在15d也达到了最大值868.8‰,之后,虽然两组值都有所下降,鲢鳙鲴组15N(底质沉积物)在30d时,降至726.8‰,而鲢鳙组下降更加明显并在30d时,鲢鳙组15N(底质沉积物)极显著小于鲢鳙鲴组(<0.01).对照组在前期同样快速增长并在第20d达到峰值1356.8‰,并在实验后期仍维持在较高水平,并极显著高于有鱼组数值(<0.01).
2.3 系统中15N的储存量变化
选取第1,15,30d的数据进行15N含量变化的分析.由表1可知,实验前期各组的沉积物腐殖质中15N储存量均为最高,对照组不断增多,有鱼组在15d达到最大值后,在30d有所下降,与15N-NH4+含量变化一致,而有鱼组15N-NH4+含量不断增加,可能是微生物活动造成的.除此之外,各组15N总量占比在15d达到最大值后,在30d有所下降,对照组达到投放量的70.76%,而鲢鳙组,鲢鳙鲴组仅能达到54.93%与50.21%,这说明系统中15N的含量与投放的15N含量收支间存在差距,相当部分15N已离开实验系统,微生物的活动是影响氮素移出水体的重要因素,进一步说明了微生物与系统氮素迁移转化之间联系密切.

表1 各实验组不同相中15N含量与占比
注:不同字母表示差异显著(<0.05).
2.4 不同细菌群落与水中的15N含量关系
为探究微生物对水体中氮素变化的影响,应用CANOCO 5软件,对微生物中相对丰度较高的菌数进行DCA判断,选择RDA模型对其与水体中的15N(氨氮),15N(硝酸盐氮)进行冗余分析(RDA)[15].

如图7所示,在分析结果中,两因子射线之间夹角小于90°时,为正相关,距离越近,相关性越高,之间夹角大于90°时,则成负相关.对照组与鲢鳙组后期突然增长的偶氮氢菌属()与15N(氨氮),15N(硝酸盐氮)密切相关;鲢鳙组中的硝化螺菌属()与15N(硝酸盐氮)密切相关,但在鲢鳙鲴组中动胶菌属()与芽胞杆菌属()与15N(硝酸盐氮)、15N(氨氮)呈负相关关系.
3 讨论
3.1 微生物群落分析
物种多样性分析与差异性分析有鱼组的沉积物微生物群落与对照组相比具有很大差异.而研究[16]表明养殖团头鲂会通过排放粪便提供大量有机质,从而导致水体沉积物中微生物多样性增加,养殖鲢鳙鲴鱼也可以产生同样影响.而鲢鳙鲴组微生物多样性却低于鲢鳙组,可能是鲴鱼这种刮食性鱼类造成的,Dörte等[17]研究表明,刮食性动物会对沉积物中的细菌进行摄食.但这也表明了,虽然混养鱼类会提高沉积物中微生物的多样性,但由于鲴鱼的摄食,鲢鳙混养要比鲢鳙鲴混养时沉积物中微生物多样性更好.但在之后鲢鳙鲴组微生物群落中,动胶菌属()、假单胞菌属()与芽胞杆菌属()占比显著高于鲢鳙组对应菌属,这是因为鲴鱼等刮食类动物虽然可以摄食微生物,但据研究[18-19]表明这只是造成了生长繁殖缓慢的硝化类细菌的减少,而具有高生长率并且在鱼类肠道内高存活率的反硝化细菌并未受到太大影响.由此可见,混养鲴鱼会降低沉积物微生物群落的多样性但会提高反硝化菌属等的占比.
3.2 系统中氮素的迁移转化
通过对系统中各相15N含量的测量,可以清晰地看到氮素在系统中的迁移转化是一种以食物链为主体的物质循环过程,被标记的微囊藻颗粒进入水体后,一部分被鲢鳙鱼摄食后,以粪便的形式沉入水底,一部分以藻类碎屑的形式直接沉入水底,导致了15N(底质沉积物)前期的增长;随后通过微生物作用释放产生15N-NH4+和15N-NO3-,进入水体后引起了15N(氨氮)和15N(硝酸盐氮)前期大幅增长;水中的浮游藻类吸收了进入的氮素,导致15N(浮游藻类)的增大,这与flynn等[20]的研究结果一致.浮游藻类不论是自身的衰亡还是被鲢鳙鱼摄食,都会重新沉入底部,同时鱼类摄食同化也使15N进入鱼体.这就是系统中氮素的迁移转化过程.对于鲢鳙鲴组,鲴鱼会在底部摄食鲢鳙鱼粪便等碎屑[21]并排放粪便;而对照组由于缺少鱼类形成的食物链,只能通过自然沉降使被标记的微囊藻碎屑进入沉积物中,再进行迁移转化,而各相氮素变化也与陈少莲等[22]的研究结果相符合.但是氮素总量在迁移转化过程中是有所变化的,在15N的收支平衡分析中,系统中的氮素在不断减少,这是因为沉积物中存在微生物,这些微生物会通过硝化-反硝化反应将水体中的氮素转化为气态并移出水体,所以分析微生物在氮素迁移转化过程中的作用成为研究重点.
3.3 沉积物中微生物对氮素迁移转化的影响
3.3.1 微生物对沉积物相中15N的影响 从图6可以看出,对比对照组的15N(底质沉积物),有鱼组的15N(底质沉积物)在后期有了下降趋势并且水平较低,而图3c微生物群落分析中,鲢鳙组与鲢鳙鲴组的沉积物中芽孢杆菌属与假单胞菌属占比总和在后期却有所升高,达到28.6%与41.62%,较第15d相应提高了约2.8倍与4倍.
有研究表明[23],在标记物碎屑沉积到水体底部后,会立即释放大量的溶解性有机氮,这些有机氮75%以上为蛋白质等高分子物质,而更容易被微生物吸收利用的氨基酸、核苷酸等小分子物质只有不到5%[24].从高分子物质到小分子物质,需要许多酶的共同分工作用,氨化细菌分泌的蛋白质水解酶等可能是其中的关键酶[25],它能将蛋白质等高分子物质分解为氨基酸等小分子物质.Bach等[26]发现假单胞菌属中的荧光假单胞菌、噬细胞菌属都可以分泌金属蛋白酶,除此之外,Watanabe等[27]也提出芽孢杆菌属尤其是蜡状芽孢杆菌分泌的肽酶是多肽降解的主要酶种.而当15N标记的高分子含氮物质降解为小分子时,它会被微生物吸收并通过转氨基-解聚-脱氨基作用转化成铵离子(15N-NH4+)排出体外[28].鲢鳙鲴组沉积物中的假单胞菌属与芽孢杆菌属就是通过这种方式减少了底部沉积物中15N的含量,不仅如此,考虑到鲴鱼的摄食活动[24]会导致部分含氮沉积物进入沉积物相深处,造成内源氮素的累积,鲢鳙鲴组沉积物中的氮素仍能有所下降,这也说明了鲢鳙鲴组沉积物中增多的氨化细菌避免了混养鲴鱼造成内源氮素的累积,同时促使沉积物向水中加快释放氮素,促进了氮素的迁移转化.
3.3.2 微生物对水相中15N的影响 水中参与氮素迁移转化过程的15N主要是由15N-NH4+和15N- NO3-形式组成.而沉积物中微生物致使系统氮素无法平衡,正是沉积物中的微生物群落对水中15N- NH4+和15N-NO3-产生影响,使部分15N以气态的形式移出水体.通过占比较大的硝化与反硝化菌属与15N(氨氮)、15N(硝酸盐氮)变化的冗余分析来具体探究导致这种情况的原因.
微生物群落对15N(氨氮)、15N(硝酸盐氮)的影响主要通过硝化、反硝化反应进行.硝化反应主要是通过沉积物中的硝化细菌将沉积物中产生的15N-NH4+氧化为15N-NO3-,硝化反应一般发生在沉积物的上表层[29],是一种普遍的微生物反应,这也导致了各实验组前期15N(硝酸盐氮)的不断上升.图7b显示的鲢鳙组后期占比增加的硝化螺菌属(),与NO3-具有明显的相关性,鲢鳙组后期15N(硝酸盐氮)不断增大,是因为它可以通过硝化反应生成硝酸盐[30-31],使底部释放的15N-NH4+转化为15N-NO3-,从而促进了氮素在系统中的循环.
反硝化作用一般发生在富含硝酸盐氮的区域,也即沉积物表层2~5cm[32].与硝化反应不同,反硝化反应是将硝酸盐直接转化为N2等含氮气体,直接作用于水体中硝酸盐氮[33].通过对鲢鳙鲴组的冗余分析(图7),发现后期占比增加的动胶菌属()与15N(硝酸盐氮)明显负相关,高春娣等[34]研究发现,动胶菌属形成的菌胶团不仅减少水中悬浮物、COD,而且动胶菌属好氧与化能异养的特性,能够使其在厌氧条件下,在电子传递反应中,分泌硝酸还原酶将硝酸盐还原为亚硝酸盐.除此之外,在实验后期占比较大的假单胞菌属()其中一些菌种,也被发现具有反硝化特性,并且还被发现可以在好氧条件下,分泌各种还原酶,使硝酸盐转化为N2O的好氧反硝化反应[35].由此可知,鲢鳙鲴组实验后期15N(硝酸盐氮)呈现的下降趋势,是沉积物中的动胶菌属与假单胞菌属等反硝化细菌增多,导致好氧、厌氧反硝化反应加强的结果.除了直接影响15N(硝酸盐氮)的微生物之外,也有对15N(氨氮)产生影响的,在图7冗余分析中,芽孢杆菌属()与15N(氨氮)呈明显负相关,而芽孢杆菌属是厚壁菌门中具有良好净化能力的菌属,好氧兼性厌氧的特性使其能够利用有机碳源生长,并将水中含氮化合物转化为硝酸盐、亚硝酸盐等,并同时进行好氧反硝化,直接将其异养硝化产物转化为含氮气体[36],蒙海林等[37]在养殖池塘中发现了能够去除氨氮的芽孢杆菌属菌株,这也再次证明,芽孢杆菌属能有效降低鲢鳙鲴组水中的15N-NH4+,造成实验后期鲢鳙鲴组15N(氨氮)持续下降.由此可知,实验后期,鲢鳙鲴组的微生物群落通过增强反硝化反应,将水中15N-NO3-转化为含氮气体移出水体,导致迁移和转化过程中氮素的不平衡.
一直以来,能够对沉积物造成扰动的生物被广泛研究,它们被认为能够加速碎屑中的氮素在水-泥两相中循环,加快氮的迁移转化[38-39].Stief等[40]研究发现大型底栖水生生物通过扰动使底部好氧-厌氧界面增多,使水中的有机质与营养盐进入底部界面中,促进微生物耦合的硝化反硝化过程,使氮素转移出水体[41].鲴鱼的生物扰动也具有相同的效果,除此之外,在对沉积物微生物群落进行研究后,发现鲴鱼还可以通过摄食、排泄等行为提高反硝化菌属的占比.
由上述分析可知,鲴鱼通过其摄食与排泄行为,增加了氨化细菌(假单胞菌属、芽孢杆菌属)的数量,增强了氨化作用,加快底质沉积物中氮素的释放;使生长繁殖缓慢的硝化类细菌减少,而具有高生长率并且在鱼类肠道内高存活率的反硝化细菌并未受到太大影响,造成了反硝化细菌(动胶菌属、芽孢杆菌属等)占比增加,增强了硝化-反硝化反应,使更多的氮转移出水体.
4 结论
4.1 鲢鳙鲴混养增加了沉积物中氨化细菌(芽孢杆菌属与假单胞菌属)的数量,实验结束时,与其它组相比,鲢鳙鲴组的芽孢杆菌属()与假单胞菌属()总占比最高,为31.2%,这增强了氨化作用,减少了沉积物中有机氮的含量,使得鲢鳙鲴组的15N(底质沉积物)在实验结束时降至726.8‰,从而加快了系统中氮素的迁移转化.
4.2 鲢鳙鲴混养提高了沉积物中反硝化菌属(动胶菌属与芽胞杆菌属)的数量,实验结束,鲢鳙鲴组动胶菌属()与芽胞杆菌属()占比显著高于鲢鳙组(<0.05),达到8.17%、33.7%,使得系统中反硝化反应加强,使水体中更多的硝酸盐氮转化为气态氮并转移出水体,实验结束时,与其它组相比,鲢鳙鲴组15N(硝酸盐氮)降低至96.3‰,15N总含量也降低至6.94µmol,加快了系统中氮素的迁移转化,并减少水中的氮素含量.
[1] Xie P. Experimental studies on the role of planktivorous fishes in the elimination of Microcystis bloom from Donghu Lake using enclosure method [J]. Chinese Journal of Oceanology and Limnology, 1996, 14(3).
[2] Starling FLRM. Control of eutrophication by silvercarp (Hypophthalmichthysmolitrix) in the tropical Paranoa Reservoir (Brazil): A mesocosm experiment [J]. Hydrobiologia, 1993,257:143- 152.
[3] Fuhrman J A. Microbial community structure and its functional implications [J]. Nature, 2009,459(7244):193-199.
[4] 韩菲尔.太湖水体浮游植物氮素吸收过程及其影响因素[D]. 苏州:苏州科技大学, 2019.
Han F E. Phytoplankton nitrogen absorption process and its influencing factors in Taihu Lake [D]. University of Science and Technology Suzhou, 2019.
[5] 陈玲玲,高月香,张毅敏,等.鲢鱅鲴混养对水环境及氮素迁移转化的影响[J]. 中国环境科学, 2019,39(3):1181-1188.
Chen L L, Gao Y X, Zhang Y M, et al. The effect of silver carp polyculture on water environment and nitrogen migration and transformation [J].China Environmental Science, 2019,39(3):1181- 1188.
[6] 陈 军,王煜恒,王会聪,等.投放不同密度的细鳞斜颌鲴对河蟹养殖水质和经济效益的影响[J]. 江苏农业科学, 2017,45(23):174-177.
Chen J, Wang Y H, Wang H C, et al. Effects of different densities on the water quality and economic benefits of[J]. Jiangsu Agricultural Sciences, 2017,45(23):174-177.
[7] 刘家辉.鲴鱼养殖效益分析[J]. 农村新技术, 2014(6):46-47.
LIU J H. Benefit analysis of carp breeding [J]. New Rural Technology, 2014(6):46-47.
[8] 贡 毅.池塘泥鳅、细鳞斜颌鲴、鳙鱼高效生态混养试验[J]. 渔业致富指南, 2016,(7):53-55.
Gong Y. High-efficiency ecological polyculture experiment of pond loach, scaly carp, and bighead carp [J]. Fisheries Getting Rich Guide, 2016,(7):53-55.
[9] Papaspyrou S, Gregersen T, Cox R P, et al. Sediment properties and bacterial community in burrows of the ghost shrimp(Decapoda: Thalassinidea) [J]. Aquatic Microbial Ecology, 2005,38:181-190.
[10] Kang C K, Choy E J, et al. Food web structure of a restored macroalgal bed in the eastern Korean peninsula determined by C and N stable isotope analyses [J]. Marine Biology: International Journal on Life in Oceans and Coastal Waters, 2008,153(6):1181-1198.
[11] 王银平,谷孝鸿,曾庆飞,等.食微囊藻干粉鱼类对水环境的影响及氮素迁移转化规律[J]. 湖泊科学, 2015,27(3):475-485.
Wang Y P, Gu X H, Zeng Q F, et al. Effects of microcystis-eating dry meal fish on the water environment and the law of nitrogen migration and transformation [J]. Journal of Lake Sciences, 2015,27(3):475-485.
[12] Lehmann M F, Bernasconi S M, Mckenzie J A. A method for the extraction of ammonium from freshwaters for nitrogen isotope analysis [J]. Analytical Chemistry, 2001,73(19):4717-4721.
[13] 吴俊森,王丽丽,贾瑞宝,等.同位素分析法测定硝酸盐氮预处理方法研究 [J]. 山东建筑大学学报, 2012,27(1):8-10.
Wu J S, Wang L L, Jia R B, et al. Study on pretreatment for measurement nitrate nitrogen in isotopic analysis method [J]. Journal of Shandong Jianzhu University, 2012,27(1):8-10.
[14] 李 柯.蓝藻分解所释放营养盐在沉水植被区的归趋[D]. 武汉:华中农业大学, 2011.
Li K. The fate of nutrients released by the decomposition of cyanobacteria in submerged vegetation areas [D]. Wuhan: Huazhong Agricultural University, 2011.
[15] 赖江山.生态学多元数据排序分析软件Canoco5介绍[J]. 生物多样性, 2013,21(6):765-768.
LAI J S. Introduction of Canoco5, a software for sorting and analyzing ecological multivariate data [J]. Biodiversity Science, 2013,21(6): 765-768.
[16] 李 晓,李 冰,董玉峰,等.精养团头鲂池塘沉积物微生物群落的结构特征及组成多样性分析[J]. 水产学报, 2014,38(2):218-227.
Li X, Li B, Dong Y F, et al.Analysis of sediment microbial communities in Megalobrama amblycephala intensive rearing pond [J].Journal of Fisheries of China, 2014,38(2):218-227.
[17] Dörte A, Stief P, Amann R, et al. In situ distribution and activity of nitrifying bacteria in freshwater sediment [J]. Environmental Microbiology, 2010,5(9):798-803.
[18] Yang C, Zhang W, Liu R, et al. Phylogenetic diversity and metabolic potential of activated sludge microbial communities in full-scale wastewater treatment plants [J]. Environmental & Technology, 2011,45(17):7408-15.
[19] 杨柳燕,王楚楚,等.淡水湖泊微生物硝化反硝化过程与影响因素研究[J]. 水资源保护, 2016,32(1):12-22,50.
Yang L Y, Wang C C, et al. Study on the process and influencing factors of microbial nitrification and denitrification in freshwater lakes [J]. Water Resources Protection, 2016,32(1):12-22,50.
[20] Flynn K J, Hipkin C R, Fasham M J R. Modelling the interactions between ammonium and nitrate uptake in marine phytoplankton [J]. Philosophical Transactions of the Royal Society of London, Series B. Biological Sciences, 1997,352(1361):1625-1645.
[21] 杨四秀.鲴鱼的生物学特性及养殖技术[J]. 内陆水产, 2004,29(5): 28-28.
Yang S X. Biological characteristics and breeding technology of scorpionfish [J]. Current Fisheries, 2004,29(5):28-28.
[22] 陈少莲,刘肖芳.论鲢,鳙对微囊藻的消化利用[J]. 水生生物学报, 1990,14(1):49-59.
Chen S L, Liu XF. On the digestion and utilization ofby silver carp and bighead carp [J]. Acta Hydrobiologica Sinica, 1990, 14(1):49-59.
[23] 罗专溪,魏群山,王振红,等.淡水水体溶解有机氮对有毒藻种的生物有效性[J].生态环境学报, 2010,19(1):45-45.
Luo Z X, Wei Q S, Wang Z H, et al. Bioavailability of dissolved organic nitrogen in freshwater to poisonous algae species [J].Ecology and Environmental Sciences, 2010,19(1):45-45.
[24] Jones D L, Shannon D, Murphy D V, et al. Role of dissolved organic nitrogen (DON) in soil N cycling in grassland soils [J]. Soil Biology & Biochemistry, 2004,36(5):749-756.
[25] Jones D L, Kielland K. Amino acid, peptide and protein mineralization dynamics in a taiga forest soil [J]. Soil Biology & Biochemistry, 2012,55:60-69.
[26] Bach H J, Munch J C. Identification of bacterial sources of soil peptidases [J]. Biology and Fertility of Soils, 2000,31(3/4):219-224.
[27] Watanabe K, Hayano K. Source of soil protease based on the splitting sites of a polypeptide [J]. Soil Science and Plant Nutrition, 1994,40(4): 697-701.
[28] 赵 彤,蒋跃利,闫 浩,等.土壤氨化过程中微生物作用研究进展[J]. 应用与环境生物学报, 2014,20(2):315-21.
Zhao T, Jiang Y L, Yan H, et al. Research progress of microbial action in the process of soil ammonification [J].Chinese Journal of Applied and Environmental Biology, 2014,20(2):315-21.
[29] Li Y, Miao Y, Zhang W, et al. Sertraline inhibits top-down forces (predation) in microbial food web and promotes nitrification in sediment [J]. Environmental Pollution, 2020,267:115580.
[30] Kessel M V, Speth D R, Albertsen M, et al. Complete nitrification by a single microorganism [J]. Nature, 2015,528(7583).
[31] Ma, Yuexin Y, Zichao D, et al. Characterization of Bacterial Community, Ammonia-Oxidizing Bacteria, and Nitrospira During the Operation of a Commercial-Scale Recirculating Aquaculture System for Culturing Pufferfish[J]. Journal of Ocean University of China, 2020,19(6):183-192.
[32] Sybil P. Seitzinger. Denitrification in freshwater and coastal marine ecosystems: Ecological and geochemical significance [J]. Limnology and Oceanography, 1988,33(4part2):702-724.
[33] Yao L, Jiang X L, Chen C R, et al. Within-lake variability and environmental controls of sediment denitrification and associated N2O production in a shallow eutrophic lake [J]. Ecological engineering: The Journal of Ecotechnology, 2016,97251-97257.
[34] 高春娣,张 娜,韩 徽,等.低温下丝状菌膨胀污泥的微生物多样性[J]. 环境科学, 2020,41(7):3373-3383.
Gao C D, Zhang N, Han H, et al. Microbial diversity of filamentous bacteria expanded sludge at low temperature [J]. Environmental Science, 2020,41(7):3373-3383.
[35] 李公伟.准好氧生物反应器填埋场微生物群落多样性及N2O产生途径研究[D]. 青岛:青岛理工大学, 2018.
Li G W. Research on microbial community diversity and N2O generation pathway of semi-aerobic bioreactor landfill [D]. Qingdao: Qingdao Technological University, 2018.
[36] Yang T, Xin Y, Zhang L, et al. Characterization on the aerobic denitrification process of Bacillus strains [J]. Biomass and Bioenergy, 2020,140:105677.
[37] 蒙海林,刘复荣,何敬愉,等.异养硝化细菌的分离鉴定及组合菌群硝化性能分析[J]. 华南理工大学学报(自然科学版), 2019,47(9):113- 120.
Meng H L, Liu F R, He J Y.Isolation and identification of heterotrophic nitrifying bacteria and analysis of nitrification performance of combined bacteria [J].Journal of South China University of Technology (Natural Science Edition), 2019,47(9):113- 120.
[38] Stief P, Polerecky L, Poulsen M, et al. Control of nitrous oxide emission fromlarvae by nitrate and temperature [J]. Limnology & Oceanography, 2010,55(2):872-884.
[39] Gilbertson W W, Martin S, Prosser J I. Differential effects of microorganism–invertebrate interactions on benthic nitrogen cycling [J]. Fems Microbiology Ecology, 2012,(1):11-22.
[40] Stief, P. Stimulation of microbial nitrogen cycling in aquatic ecosystems by benthic macrofauna: mechanisms and environmental implications [J]. Biogeosciences, 2013,10(12):7829-7846.
[41] Marinelli R L, Lovell C R, Wakeham S G. Experimental investigation of the control of bacterial community composition in macrofaunal burrows [J]. Marine Ecology Progress, 2002,235:1-13.
The effects of microorganisms on the migration and transformation of nitrogen in the polyculture system of,and.
ZHANG Zhe1, GAO Yue-xiang1, ZHANG Yi-min1,2*, CHENG Ling-ling1, GUO Yan-min1, ZHU Yue-ming1, YU Jiang-hua2
(1.Nanjing Institute of Environmental Sciences,Ministry of Ecology and Environment,Nanjing 210000, China;2.School of Environmental Science and Engineering, Nanjing University of Information Engineering, Nanjing 211800, China)., 2022,42(2):897~906
The polyculture system of(Xenocypris microlepis),(Silver carp) and(bighead carp) was set up in simulated eutrophic water. The dynamics of microbial communities and their effects on the migration and transformation of nitrogen in the sediments were investigated by applying isotopic labeling and microbial analysis to reveal the underlying mechanism of the polyculture system. The results showed that:The polyculture of Xenocypris microlepis, silver carp and bighead carp couldincrease the proportion ofandinvolved in the ammonification process in the sediments. Comparing with the silver carp and bighead carp group, the total proportion ofandin the polyculture group was the highest (31.2%) at the end of experiment, showing that it enhanced ammonification process and thus, reduced the content of organic nitrogen in the sediments.Consequently, the15N(substrate sediment)of the polyculture group decreased to 726.8‰. The polyculture of Xenocypris microlepis, silver carp and bighead carp couldincrease the proportion of denitrifying bacteria (and) in the sediments. The proportion ofandin the polyculture group was significantly higher than that in the silver carp and bighead carp group (<0.05), reaching 8.17% and 33.7%, respectively. Therefore, the denitrification process was enhanced in the system, converting more nitrate into nitrogen gas in the water and finally releasing this gas from this water. At the end of the experiment, in the polyculture group, the15N(nitrate nitrogen)decreased to 96.3‰ and the total content of15N decreased to 6.94µmol.
,and;polyculture;stable isotope technique;nitrogen;microbial community
X524
A
1000-6923(2022)02-0897-11
张 哲(1996-),男,山东德州人,南京信息工程大学硕士研究生,主要从事水环境治理和流域生态保护研究.发表论文2篇.
2021-05-12
国家水体污染控制与治理重大专项(2017ZX07202006);中央级公益性科研院所基本科研业务专项(GYZX200204)
* 责任作者, 研究员, zym7127@163.com
