斑马鱼Hepcidin-2基因结构的生物信息学分析与抗菌活性优化设计
2022-02-12胡媛媛刘子琦乔子祺陈欣尹华雯姚波
胡媛媛,刘子琦,乔子祺,陈欣,尹华雯,姚波,2
1.重庆科技学院化学化工学院,重庆401331;
2.工业发酵微生物重庆市重点实验室,重庆401331
由于传统抗生素的过度使用和滥用,细菌耐药性在世界范围内引起严重的健康问题,寻找新的有效抗生素变得越来越困难。在这种情况下,抗菌肽(antimicrobial peptide,AMP)提供了一种很有前景的对抗多种病原微生物的策略,既可直接作为抗菌剂,又可作为天然免疫系统的重要调节因子[1-2]。AMP又称宿主防御肽,是几乎所有生物体先天免疫的一部分,具有阳离子性(带正电)和两亲性(亲水和疏水)[3]。阳离子AMP能与带负电荷的细菌细胞膜结合,导致细菌细胞膜上电化学电位变化,引起细胞膜损伤并使蛋白质等内容物泄漏,出现空泡化;通过破坏细胞膜迅速杀死入侵的病原体,使病原体很难产生耐药性[4-6]。
Hepcidin是一种AMP和铁调节激素,在宿主的先天免疫和铁代谢中起双重作用,主要由肝脏产生并释放至血清中[7]。Hepcidin在控制体内铁稳态方面发挥重要作用,其通过与转铁蛋白相互作用,诱导其内化和降解,从而调控铁的稳态。Hepcidin作用广泛,对革兰阴性和阳性细菌均有活性,同时还具有抗病毒、抗寄生虫、抑制真菌和酵母生长等作用。但天然AMP稳定性差,半衰期短,毒副作用强,尤其可能具有严重的溶血活性[8-9]。
对AMP及其生物功能进行预测能有效帮助了解AMP的作用机理,为设计稳定有效的AMP提供理论依据。目前主要使用的AMP预测方法,包括基于经验分析的预测方法和基于机器设计的计算方法[10],前者是根据已知AMP序列的生物信息学分析的数据[11],通过优化新型AMP理化性质、选择性、溶血活性以及抗菌活性等参数进行分子设计;而后者能充分挖掘抗菌活性与生化特性之间的内在线性和非线性关系,适用于复杂模型的大规模AMP预测[12]。但目前对AMP进行序列优化和设计的方法还存在诸多不足,尚有待进一步改进。为了优化AMP的结构,扩大其应用,本研究对天然AMP Hepcidin进行生物信息学分析,揭示其潜在的分子机制,同时采用序列模板法设计优化AMP,克服其天然肽的缺点和潜在的问题,为AMP的应用研究提供针对性和合理选择的思路和方案。
1 材料与方法
1.1H epcidin氨基酸序列收集整理 利用Uniprot数据库(https://www.uniprot.org/)及NCBI的GenBank网站(https://blast.ncbi.nlm.nih.gov)查找鱼类AMP Hepcidin的氨基酸序列,选取斑马鱼中Hepcidin的1个亚类Hepcidin-2(GenBank登录号:Q7T273)作为目的基因,并通过Tblastn(https://blast.ncbi.nlm.nih.gov/)进行多序列同源性比对得到Hepcidin基因家族的核苷酸序列和氨基酸序列。
1.2H epcidin氨基酸序列的生物信息学分析 利用ProtParam蛋白质在线分析网站(http://web.expasy.org/protparam/),计算预测氨基酸等电点、疏水性等,同时利用网站http://web.expasy.org/prootscale.htmL/画出蛋白质的疏水性图谱。运用InterPro在线分析工具(http://www.ebi.ac.uk/interpro/),分析得到蛋白质信号肽N-末端、C-末端、H区组成部分位点。运用SignalP-5.0 Server在线预测软件(http://www.cbs.dtu.dk/services/SignalP)预测其可能存在的切割位点。运用TMHMM 2.0在线跨膜结构分析工具(http://www.cbs.dtu.dk/services/TMHMM/)预测分析蛋白质的跨膜区:基于HMM方法的蛋白质跨膜区,结合TMpred分析跨膜核心位点。运用PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)和Phyre在线分析软件(http://www.sbg.bio.ic.ac.uk/phyre2/ht/page)预测二级结构,运用Phyre在线分析软件预测三级结构。
1.3AM P H epcidin序列模板设计 收集人、家鼠、尼罗罗非鱼、斑马鱼、褐家鼠、欧洲舌齿鲈、大西洋白姑鱼、大菱鲆、尖齿胡鲶及大黄鱼等物种产生的Hepcidin AMP序列,筛选出152条氨基酸长度为80~99 aa的序列。比对后计算每种氨基酸在任意位置出现次数以及比对空格出现的次数,并由此计算氨基酸的频率。提取最大频率的氨基酸,得到序列模板。
1.4AM P H epcidin活性预测
1.4.1AntiBP2预测 AntiBP是采用支持向量机首先分别对N-末端和C-末端15个残基的二进制模式进行AMP预测;其次对N&C-末端残基进行预测;最后利用全肽的氨基酸组成建立基于支持向量机的模型。使用五重交叉验证技术开发所有模型,并在独立数据集上测试模型性能,开发出AntiBP2服务器,有助于发现有效的AMP[13]。
1.4.2DBAASP预测 DBAASP是研究结构与活性关系所需信息的数据库。该数据库提供信息和资源分析,以促进设计具有高治疗指数的抗菌药物。DBAASP搜索页面允许用户根据其结构特征、复杂类型(单体、二聚体和双肽)、来源、合成类型(核糖体、非核糖体和合成)和目标物种来搜索多肽[14],可预测提交的蛋白序列对大肠埃希菌、肺炎克雷伯菌、枯草芽孢杆菌等7种细菌和人红细胞的活性。
1.4.3Antifp预测 Antifp是利用多种肽特征(如氨基酸组成、二元模型和末端残基等)开发的用于预测抗真菌肽(antifungal peptide,AFP)的计算机模型。通过残基位置偏好分析揭示了N-末端特定残基类型(如R、V、K)和C-末端特定残基类型(如C、H)的突出。在成分相似的AFP和非AFP的数据集上对开发的模型和现有方法进行基准测试,在测试数据集和验证数据集上有较高的准确率,所建立的二元模型有更好的预测性能[15]。
2 结果
2.1鱼类AM PHepcidin基因家族的进化Hepcidin-2的开放阅读框编码91个氨基酸,序列N-末端由M(Met)氨基酸开始。该序列包含了Hepcidin-2 AMP全长开放阅读框序列,推测基因组DNA包含3个外显子和2个内含子。Hepcidin-2氨基酸序列中1~24位为信号肽、25~64位为前肽、67~91位为活性肽,二硫键的位置分别为
对鱼类Hepcidin序列构建的系统发育树见图1,分为3大支,大西洋鲱鱼单独形成1支,斑马鱼与黄鳍结鱼、金线鲃、鲫鱼、草鱼、鲈鲤、多鳞白甲鱼、理氏裂腹魚、鳙鱼、鲢鱼、泥鳅、武昌鱼及南亚野鲮为1大支,另1大支由电鳗、巴丁鱼、梭子鱼、海鳟、细鳞鲑、哲罗鲑、红鲑鱼、银大麻哈鱼、虹鳟、北极嘉鱼、乌塘鳢、大鳍弹涂鱼、舌齿鲈、兰副双边鱼、华美达鱼、大黄鱼、淞江鲈、红腹罗非鱼、斑马拟丽鱼、白边锯鳞鱼及长印鱼等物种组成。收集的鱼类AMPHepcidin基因家族进化分析发现,斑马鱼与南亚野鲮Hepcidin序列相似性最高,为72.22%,与大西洋鲱鱼的进化关系最远,相似性为46.15%。见表1。
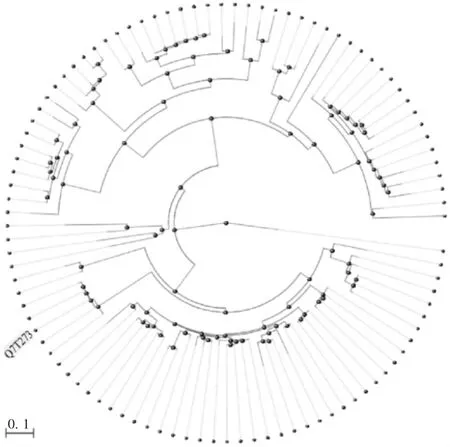
图1 鱼类AMP Hepcidin基因家族进化分析Fig.1 Evolutionary analysis of fish AMP Hepcidin gene family
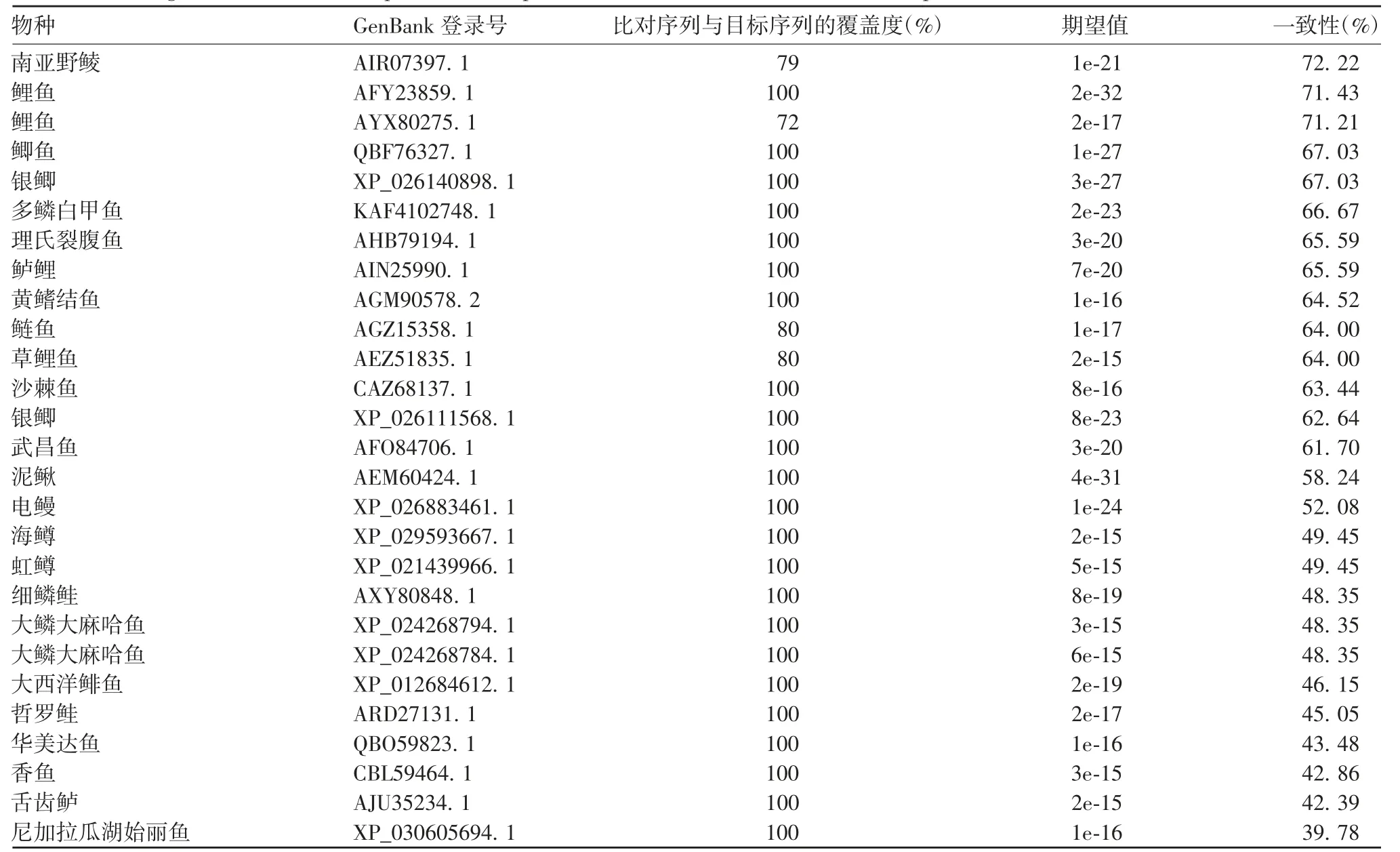
表1 不同物种与斑马鱼Hepcidin AMP氨基酸序列相似性Tab.1 Homologies of amino acid sequences of Hepcidin AMPs from zebrafish and other species
2.2Hepcidin氨基酸序列的生物信息学分析结果
2.2.1氨基酸序列理化性质分析结果 AMP Hepcidin-2序列由91个氨基酸组成,ProtParam在线分析结果显示,其分子式为C459H731N131O130S11,相对分子质量为10 517.31,理论等电点为7.55,总的负电荷残基数(Asp+Glu)=9,总的正电荷残基数(Arg+Lys)=10。该分子的脂肪族指数为88.79,亲水性总平均值为0.041,不稳定性指数为42.82,高于阈值40,为疏水性不稳定蛋白。AMP疏水性图谱见图2,在5~24位氨基酸之间,Score>1.5,呈现明显的疏水性,预测与信号肽有关。
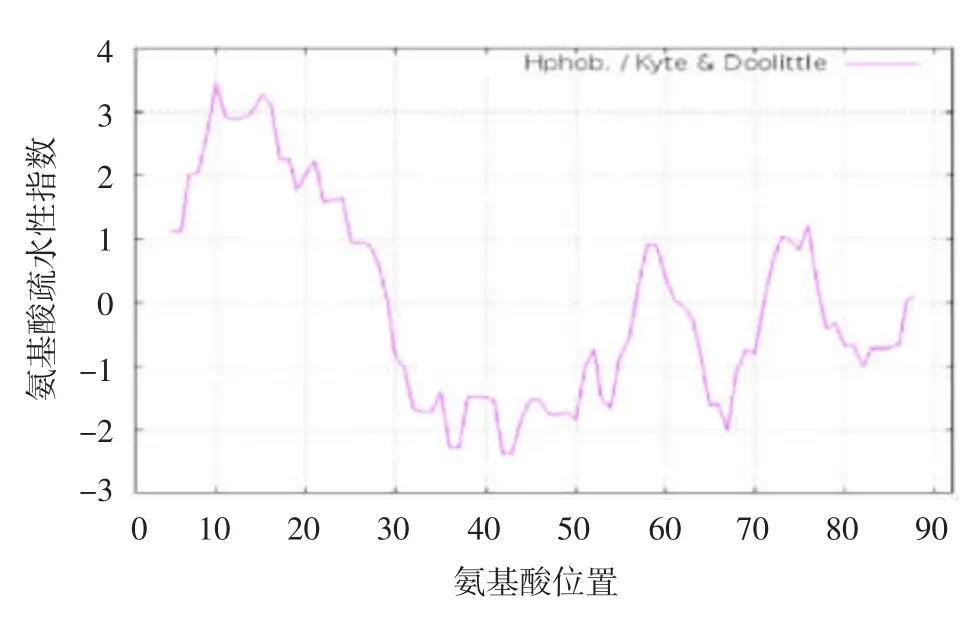
图2 Hepcidin-2 AMP疏水性分析Fig.2 Hydrophobicity of Hepcidin-2 AMP
2.2.2信号肽分析结果 真核生物中,信号肽通常在指导蛋白质插入内质网的膜后被信号肽酶切割。蛋白质中的信号肽可大致分为3类:Sec信号肽(Sec/SPⅠ)、脂蛋白信号肽(Sec/SPⅡ)及Tat信号肽(Tat/SPⅠ)。InterPro在线分析工具分析显示,Hepcidin-2信号肽中1~5位氨基酸为N-末端信号肽,6~19位氨基酸为功能区,20~24位氨基酸为C-末端信号肽,见图3。SignalP-5.0 Server在线预测分析结果显示,其切割位点在Ala24和Val25之间,为Sec信号肽,被SPaseⅠ裂解,见图4。
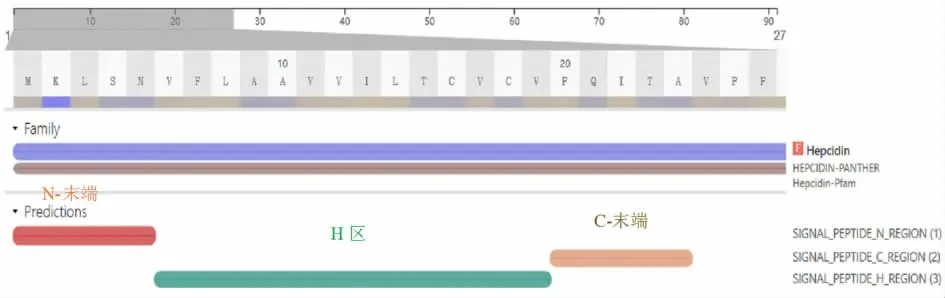
图3 Hepcidin-2信号肽功能区分析Fig.3 Analysis of functional domain of Hepcidin-2 signal peptide
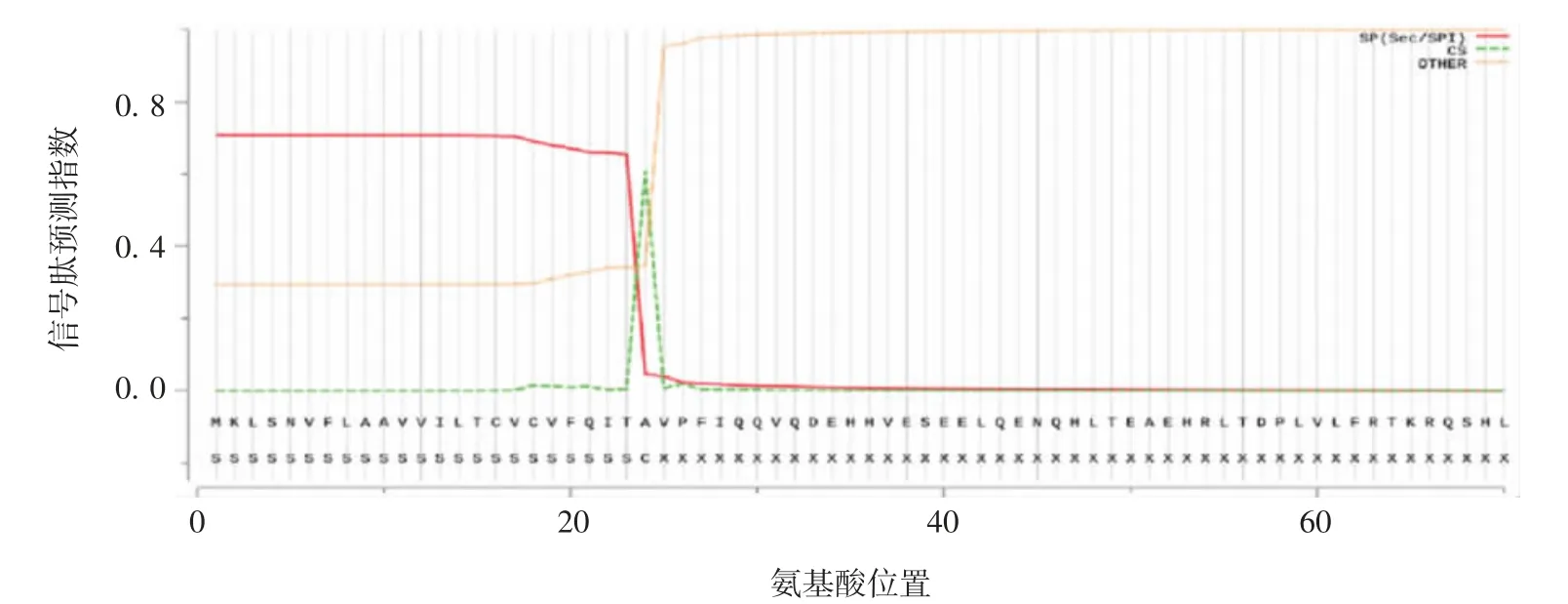
图4 Hepcidin-2 AMP信号肽分析结果Fig.4 Analysis of Hepcidin-2 AMPsignal peptide
2.2.3跨膜结构分析结果 通过2.2.2项对氨基酸序列的分析可知,1~24位为信号肽序列。信号肽引导多肽分子穿过细胞质膜,其位置与TMHMM 2.0分析显示的跨膜螺旋区域(Tm-helix)重合,分析结果中7~29位氨基酸形成跨膜螺旋区。同时,通过TMpred法,基于对Tmbase数据库的统计分析来预测跨膜方向,Score>500被认为有生物学意义,结果显示,预测膜外到膜内的跨膜螺旋1个,由3~22位氨基酸形成,中心位点为13,预测分数为1 990,无偏好性;预测膜内到膜外的跨膜螺旋1个,由6~28位氨基酸形成,中心位点为18,预测分数为2 287,具有偏好性,由此可知,跨膜螺旋方向偏向由膜内向膜外。见图5。
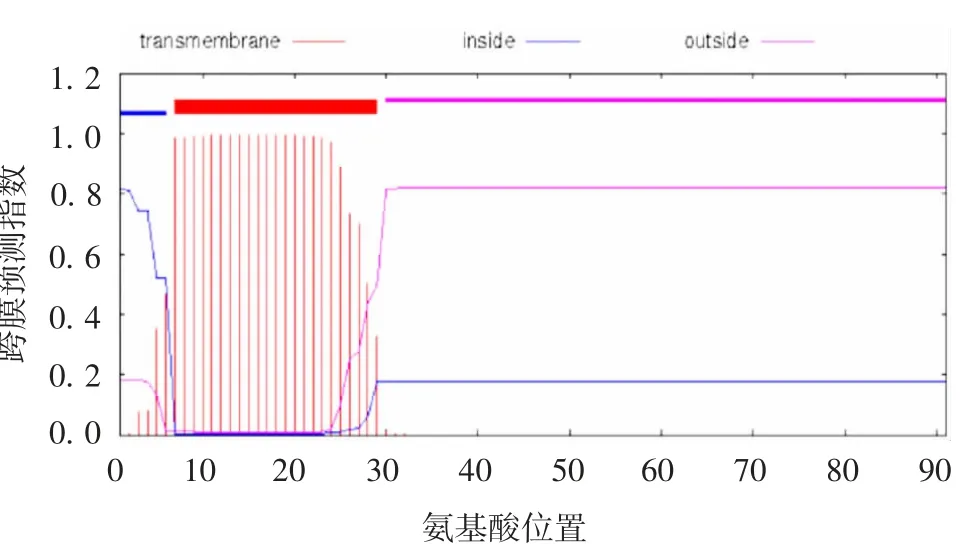
图5 Hepcidin-2 AMP跨膜区分析Fig.5 Analysisof transmembrane region of Hepcidin-2 AMP
2.2.4二级结构预测结果 Phyre分析工具预测结果见图6,Hepcidin-2蛋白序列存在3处α-螺旋富集区,主要集中位于信号肽部分,有较高的置信度;1处β-折叠区域,集中位于成熟肽部分,序列中有35%的区域在数据库无适合的匹配,因此为无规则状态,在图中以“?”表示。
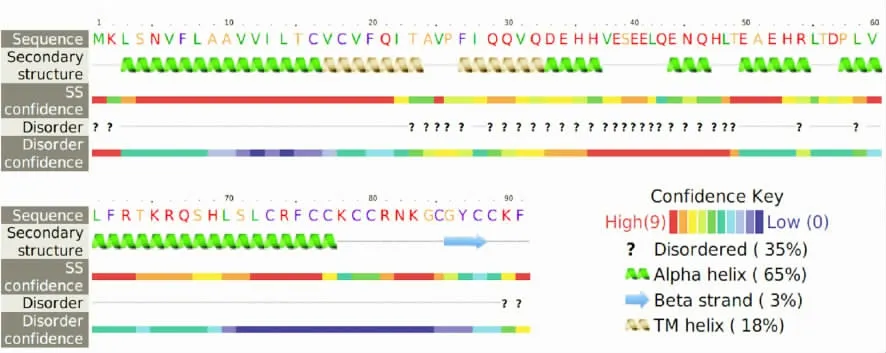
图6 Hepcidin-2 AMP二级结构分析Fig.6 Secondary structure of Hepcidine-2 AMP
2.2.5三级结构预测结果 根据提交的Hepcidin-2氨基酸序列,Phyre在数据库中匹配到最高评分,以该最高评分为模板建模,置信度为98.6%(FoldLibraryid:c1s6wA),保守度为22%(包含20个氨基酸),一致性为70%。成熟肽区域预测分析显示73~76、78、88~90位氨基酸保守性最高,为稳定的α-螺旋和β-折叠。线性置信度在73~80、85~87、88~91位最高。在AMP活性中心位置,空间结构高度保守,见图7。
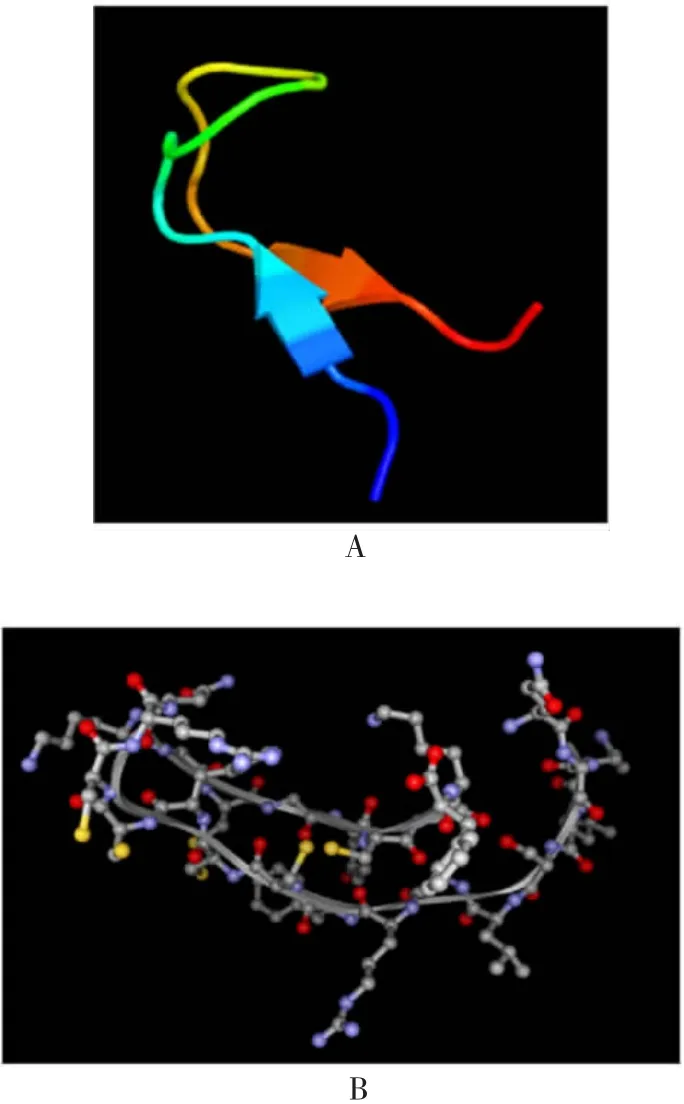
图7 Hepcidin-2 AMP三级结构预测模型Fig.7 Model for prediction of tertiary structure of Hepcidin-2 AMP
2.3序列模板设计结果
2.3.1序列比对结果 经序列比对后确定结构域序列,序列C-末端具有较高的相似性,表明Hepcidin AMP保守性较高,不同物种Hepcidin AMPC-末端大多存在6~8个Cys,形成3~4对二硫键,具有高度保守性。鉴于肽段的序列较长,因此仅分析活性肽功能区序列。为确保功能区结构域的完整性,选取序列比对格式中序列C-末端36位氨基酸,见表2。

表2 序列比对后选取的Hepcidin功能区序列(部分数据)Tab.2 Sequences of functional domains of Hepcidin screened after sequence alignment(partial data)
2.3.2功能区氨基酸分布频率 根据比对后的序列,统计每种氨基酸在相应位置出现的次数,以及序列比对中空格(由“-”表示)出现的次数。根据统计的次数,计算各氨基酸在特定位置出现的频率,其中超过0.05的数据用阴影表示。结果显示,在14、17、18、20、21、30等位置半胱氨酸出现频率均≥78%,表明这些位置具有高度保守性,同时在26、27、29、31位半胱氨酸占比最大;在8~12、34~36位空格的频率≥72%。因此,在最终序列模板里,这些位置将与斑马鱼Hepcidin-2进行比较分析后选择性去除。见表3。
2.3.3序列模板 提取表格中最大频率的氨基酸,得到序列模板为:KRRIH--------CRFCCGCCRNGKCCGCCCF----。按照同样方法,不计算空格的序列模板为:KRRIHHPSSSPKLCRFCCGCCRNGKCCGCCCFFFGF。将两种序列模板与斑马鱼的Hepcidin-2(GenBank登录号:Q7T273)比对,调整序列模板,取序列比对最高分数的Hepcidin模板(Hepcidin Template,HepTP):KRRIHHSLCRFCCGCCRNGKCCGCCCF。
将模板序列与斑马鱼Hepcidin-2进行对比,Hepcidin-2中的半胱氨酸均能被匹配,同时模板序列多出2个半胱氨酸,使得两段序列的不稳定性指数由52.16降至43.35,其他性质差异还有待进一步验证。
根据序列比对最高分数的模板整理各位置频率前3的氨基酸,见图8。
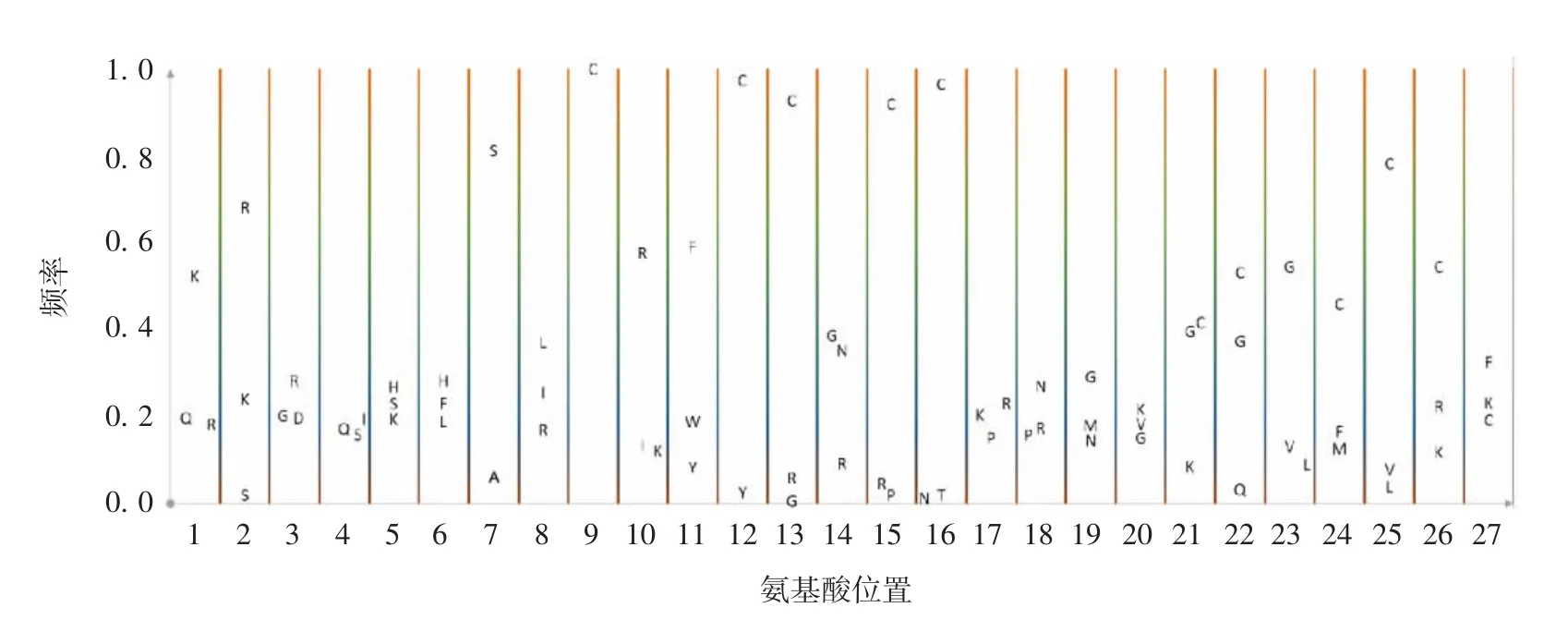
图8 氨基酸频率分布图Fig.8 Distribution of amino acid frequencies
2.4抗菌活性预测结果与分析
2.4.1AntiBP2预测结果 Hepcidin-2模板序列从第1个位置起具有抗菌活性,属于Hepcidin类AMP,预测分数为0.813,预测分数是通过加法矩阵,将每个残基在肽序列特定位置的分数相加来计算。
2.4.2DBAASP预测结果 阳性预测值(positivepredictive value,PPV)表示预测为活性肽,阴性预测值(negative predictive value,NPV)表示为非活性肽。对人红细胞活性的评价,非活性肽被认为是阳性,因此非人类红细胞活性肽表示PPV,反之为NPV。Hepcidin-2模板的预测结果为对肺炎克雷伯菌有抗菌活性和人红细胞有溶血活性。见表4。
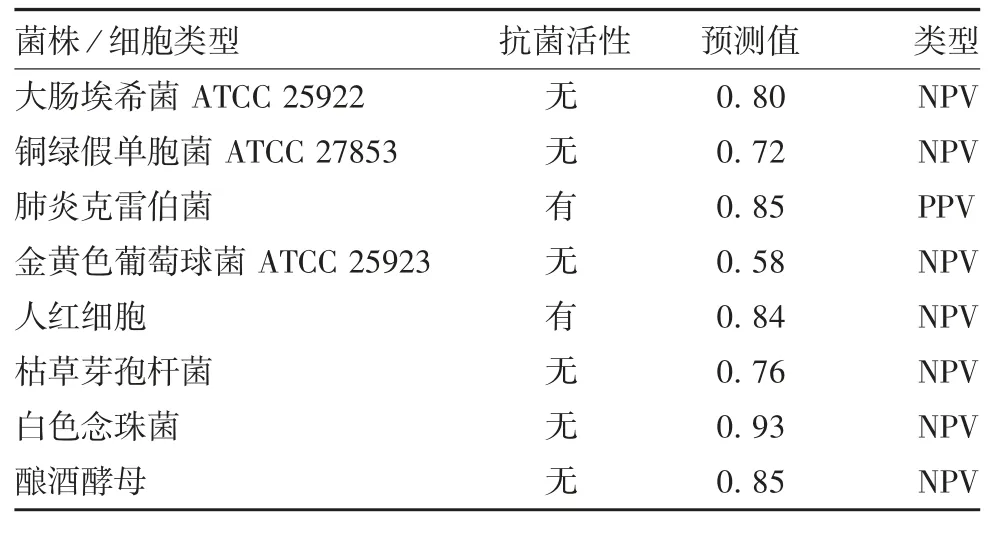
表4 Hepcidin-2模板序列在Dbaasp预测的结果Tab.4 Prediction of Hepcidin-2 template sequence by Dbaasp software
2.4.3Antifp预测结果 Hepcidin-2模板具有抗真菌的活性,机器学习算法根据预测模型给出的分数为1.281,净电荷为7。
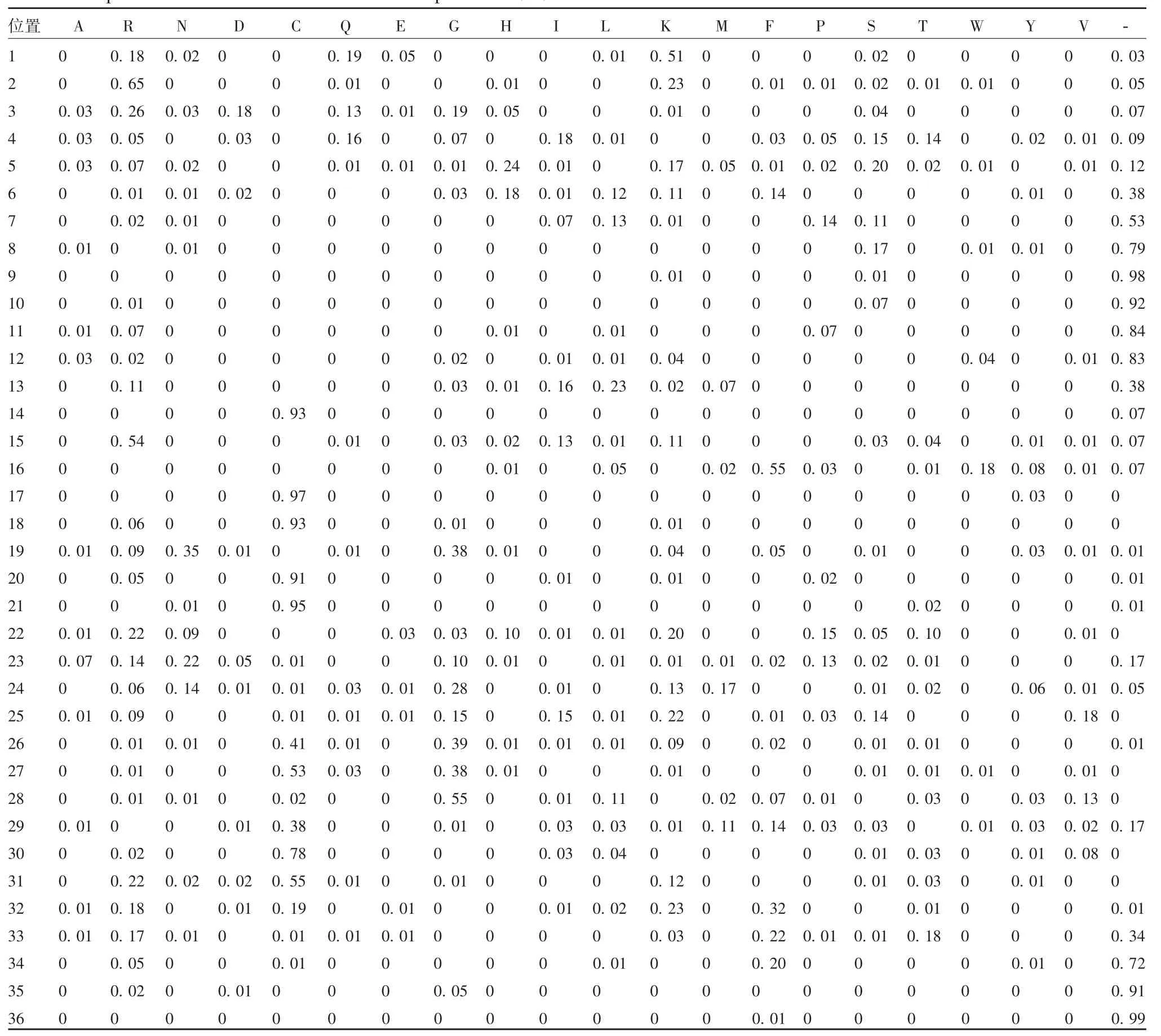
表3每种氨基酸在任一位置的频率(%)Tab.3 Frequencies of each amino acid in various positions(%)
3 讨论
本研究选取斑马鱼Hepcidin-2基因为研究对象,分析了其进化关系、理化性质、信号肽、跨膜结构、二级结构、三级结构等。
Hepcidin AMP由信号肽、前肽和成熟肽3部分组成。信号肽使AMP从细胞内分泌出来,前肽保护自身免受酶解并保持生物活性;其抗菌作用最终体现在成熟肽中[16]。信号肽通常为16~30个氨基酸残基,一般由一段高度疏水的氨基酸序列组成,位于分泌蛋白和膜蛋白的N-末端,包括3个区域:N-末端,带正电的碱性氨基末端;H区,以中性氨基酸为主的一段α-螺旋结构,是信号肽主要功能区;C-末端带负电,具有信号肽酶切割位点[17]。斑马鱼Hepcidin-2蛋白序列是由24个氨基酸组成的信号肽,位于Hepcidin AMPN-末端并具有疏水性;在跨膜区域中,信号肽的N-末端插入膜内,其中间功能区和C-末端呈螺旋结构跨膜区域;整体序列带正电,属于碱性阳离子疏水性AMP。AMP的作用机理取决于许多理化性质,包括氨基酸序列、电荷、双亲性、结构尤其是二级结构等。尽管AMP具有适当的理化特性,研究人员仍认为其与细菌膜结合的能力在研究和应用中起着关键作用[18]。SHEN等[19]计算Hepcidin成熟肽区域中,多肽空间结构含32%的β-折叠结构和8个半胱氨酸残基组成的4个二硫键,形成稳定的发夹结构。另有个例仅存在4个半胱氨酸,发现于南极的物种中,有研究认为极端的天气,如寒冷可能导致Hepcidin进化,减少半胱氨酸数量以适应外界环境[20]。半胱氨酸的侧链上有活泼的巯基,一般情况下不会被其他任何氨基酸取代,因此具有保守性。半胱氨酸通过空间约束改变二级结构,形成的二硫键桥可稳定三级结构,其拓扑结构是蛋白质一级结构同源性分析的基础[21]。
斑马鱼Hepcidin-2蛋白成熟肽三级结构预测结果显示,其置信度为98.6%,保守度为22%(包含20个氨基酸),一致性为70%。在AMP活性中心位置,空间结构高度保守。对编码蛋白质结构的分析中,在N-末端主要为α-螺旋,C-末端集中为β-折叠。经计算得到的序列模板的抗菌活性预测结果显示,其具有抗肺炎克雷伯菌和抗真菌活性及其对人类红细胞毒性。
在任一蛋白序列中,每种氨基酸在任意位置的随机分布频率约5%,显著高于或低于该频率值表示该位置对给定氨基酸的偏好或不相容,这些氨基酸可能是通过进化选择的,因此与肽的功能有关[22]。本研究采用的序列模板法,提取各位置频率最大的氨基酸组成模板正符合这一规律。
序列模板法除了可用于指导SAR(structureactivity relationship)研究中AMP设计之外,还可用于在数据库中搜索具有潜在抗菌活性的新序列。ZELEZETSKY等[22]使用ScanProsite工具用模板扫描Swiss-Prot数据库,在Pilosulin 1中鉴定出1个含有56个残基的片段,该片段是来自跳投蚁(Myrmeciapilosula)毒液的细胞毒性肽,与模板密切相关。该片段N-末端20个残基显示出有效的广谱抗菌活性,其抗菌谱包括耐多药的和普通的革兰阳性、阴性菌以及白色念珠菌,证实了该搜索方法的有效性。
在序列模板法的基础上,通过单个氨基酸残基取代,研究调节取代位氨基酸的性质进行选择性的优化,而同时又不影响对多种耐药细菌和酵母指示菌株的抗菌活性,从最小抑菌浓度、膜透性动力学和对红细胞裂解等方面讨论结构特征(侧链的大小/疏水性以及构象和灵活性)和生物活性之间的关系[23]。使用蛋白氨基酸各种参数的计算来进行肽的设计,这一原理可开发有效且降低细胞毒性的AMP。该方法通用性强,允许使用非蛋白残基的取代,明显扩展了物理化学和结构变化的范围,以调整生物特性。
研究表明,改变关键位置的氨基酸残基可调节侧链尺寸、疏水性等特性,以及主链在此位置的构象和柔韧性。ZELEZETSKY等[23]设计的AMP类似物中,将第7位氨基酸由蛋白源性Gly、Pro、非蛋白源性β-Ala、D-Nle氨基酸、STA或PST模块(modules)取代,以此调节蛋白特性及主链的构象。在50%TFE或模拟SDS胶束的膜存在下,均显示出良好的结构水平;在抑菌活性试验中所有肽对测试微生物均具有广谱活性,最小抑菌浓度为8μmol/L甚至更低。非蛋白源性肽对大肠埃希菌的内外膜均显示出快速的通透性,所有蛋白源肽均显示出对大肠埃希菌外膜的通透性,而仅非蛋白源性肽Ala7和蛋白源肽Gly7取代对大肠埃希菌和金黄色葡萄球菌的细胞质膜有明显通透性。由此,提出了调节AMP使细菌裂解的方法,并通过影响最小的氨基酸替换使其对微生物细胞更具特异性。
本研究建立了运用序列模板法优化AMP的技术路线,可为预测其抗菌活性和细胞毒性提供参考,有助于改造天然AMP,根据特定情况有针对性地设计肽,调节不同肽的活性,为扩大其在临床和生产中的应用提供理论支持和技术支撑。
