基于mtDNA D-Loop区和Cytb基因的清水江斑鳜群体遗传多样性分析
2022-02-11麻智芳潘秋芝
麻智芳,潘秋芝,安 苗,李 珊,黄 胜,余 科
(1.贵州大学动物科学学院,贵阳 550025;2.贵州农业职业学院,贵阳 551499)
斑鳜(Sinipercascherzeri)隶属于鲈形目(Perciformes),鮨科(Sinipercidae),鳜属,俗称花鲈、黑鳜等,是东亚特有的淡水经济鱼类,也是鳜类分布最广的群体之一,南起越南红河,北至辽宁南部、朝鲜半岛均有分布[1]。斑鳜分布广、个体较大、数量也较为丰富,是我国鳜属主要的经济种类之一[2]。20世纪90年代学者研究发现,野生斑鳜资源由于不合理捕捞和生境遭到破坏而呈现下降趋势[3-4]。近年来,由于野生资源量骤减和人工定向繁育等原因,鳜类的种质资源呈现退化现象[5-6]。因此,开展野生斑鳜的遗传多样性调查对斑鳜的种质资源保护与合理开发利用具有重要的意义。
清水江位于苗岭山脉东段北面的云贵高原向湘桂丘陵过渡的斜坡地带上,是长江流域洞庭湖水系沅江的上游,在贵州省境内长459 km,流域面积17 157 km2,是贵州省境内第二大河流,其干流在贵州境内由上至下以黄平县重安江汇口和锦屏六洞河汇口为界划分为上、中和下游。20世纪末,伍律[7]、王大忠等[8]、辜永河和黎道洪[9]等先后对清水江鱼类资源状况展开了调查,进入21世纪,代应贵和陈毅峰[10-11]采取实地调查与文献查阅相结合的方法调查表明清水江共有鱼类86种(亚种),其中有鳜(S.chuatsi)、大眼鳜(S.kneri)、斑鳜、波纹鳜(S.undulata)、暗鳜(S.obscura)、长体鳜(S.roulei)、中国少鳞鳜(C.whiteheadi)和漓江少鳞鳜(C.loona)等鳜类8种,斑鳜是清水江的优势种,具有一定的渔业利用价值。因此,掌握清水江野生斑鳜的遗传结构及多样性水平,能够为该流域野生斑鳜资源的可持续利用提供科学数据。
由于鱼类线粒体具有DNA分子小、结构简单、进化速度快、不同区域进化速度不同等特点,使其作为分子标记在鱼类进化遗传学、群体遗传结构、分子生态学和保护生物学等研究领域中取得了很多有意义的成果[12]。其中细胞色素b(cytochromeb,Cytb)和线粒体控制区(control region displacement loop, D-Loop)变异速率快且存在差异,是检测水生生物的遗传多样性和系统发育的常用分子手段[13-16]。因此,本研究基于mtDNA D-Loop区和Cytb基因测序技术,来探究清水江野生斑鳜群体的遗传结构、遗传多样性和群体历史动态,以期为斑鳜种质资源的保护和开发利用提供理论依据。
1 材料与方法
1.1 样品采集
2015年3月至2017年11月在清水江干流设置了下司、凯里、旁海、柳川、锦屏和远口6个站点,支流设置了台盘、南哨、小江和大同4个站点。10个站点共采集野生斑鳜287尾(图1,表1)。每尾鱼测定其基础生物数据后,取背部肌肉3~5 g用无水乙醇固定于离心管中,置于-80 ℃冰箱中保存待用。

表1 清水江野生斑鳜样本信息
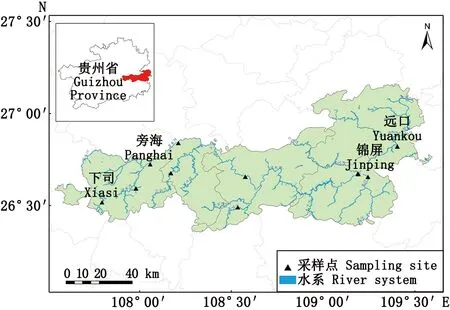
图1 清水江野生斑鳜采样点
1.2 DNA提取
采用(肌肉、血液、组织)基因组DNA提取试剂盒(北京天根)提取野生斑鳜基因组DNA。然后用微量紫外分光光度计检测其浓度及吸光值,1.0%琼脂糖凝胶电泳检验其完整性,最后将成功提取的DNA置于-20 ℃保存备用。
1.3 PCR扩增及测序
野生斑鳜线粒体D-Loop控制区和Cytb基因的扩增引物参考LEE等[17]和CAO等[18]。引物均由生工生物工程(上海)股份有限公司合成。PCR体系为25 μL:模板DNA 2 μL;1×TaqPCR Master Mix 15 μL;正反引物各1 μL;其余用超纯水补足。PCR反应条件为:94 ℃预变性3 min;35 个循环,每个循环包括94 ℃变性40 s,58.6 ℃(D-Loop)/49.6 ℃(基因Cytb)退火1min,72 ℃延伸50 s;最后72 ℃延伸7 min。两者产物都在1%琼脂糖凝胶电泳中检测,以每个PCR产物2 μL为模板,用marker标记对扩增片段长度进行验证。
把目标条带清晰的产物送至生工生物(上海)股份有限公司进行纯化和双向测序,测序引物与扩增引物一致。
1.4 数据处理
利用DNAStar(DNAS-tar,Inc)软件包中SeqMan、EditSeq程序对测序结果进行拼接,Cytb和D-Loop基因序列分别参考Genebank中的斑鳜序列(DQ648645和AP014527)手动校正,保留有效片段。采用Mega6.0软件分别统计两个基因区的碱基组成,基于Kimura 双参数法(Kimura 2-parameter, K2p)模型计算群体内(genetic distance within populations)和群体间的遗传距离(genetic distance among populations),利用邻接法(neighbor-joining,NJ),Bootstrap 1 000次自举检验构建单倍型间的系统发育树。使用DnaSP5.0进行遗传多样性相关分析。使用Network10.2软件以中介邻接网络算法(median-joining networks, MJ)分别按照两个基因的单倍型比例绘制网络图。运用Arlequin3.5进行群体分子遗传变异分析(analysis of molecular variance,AMOVA)和群体间遗传分化指数(genetic differentiation,Fst)计算,并对种群进行中性检验及核苷酸不配对分析,判断种群变动情况。利用式(1)[19]估算种群扩张时间,式(2)为基因流计算公式。
T=t×(代时数);τ= 2ut
(1)
式(1)中:T为种群扩张时间,t为每个世代种群的扩张时间;其中,τ(Tau)为种群扩张参数,由Arlequin3.5计算得出;斑鳜的代时数取3;u=μk;k表示序列长度;μ为鱼类 mtDNA 基因的变异速率, 按每百万年2%[20]计。
Nm≈(1-Fst)/(4Fst)
(2)
式(2)中,Nm为基因流,Fst为遗传分化系数。
2 结果与分析
2.1 清水江斑鳜群体mtDNA两个基因的序列变异
287尾斑鳜样品的D-Loop区序列有840 bp和841 bp两种长度类型,序列中A、T、G和C 4种碱基的平均含量分别为34.0%、30.1%、15.8%和20.1%(表2)。A+T的含量(64.1%)明显高于G+C(35.9%)的含量,呈碱基偏倚性。287条D-Loop区同源序列共定义了43种单倍型(表3),有44个多态位点(表4),其中单一多态位点5个,简约信息位点39个,有3个碱基缺失,无碱基插入,碱基的转换/颠换比值为10.19,上游河段群体(upper reaches of Qingshui River, UQ)(37)、中游河段群体(middle reaches of Qingshui River, MQ)(37)和下游河段群体(lower reaches of Qingshui River, LQ)(38)3个群体的变异位点数相差也基本持平(表4)。

表2 斑鳜群体线粒体D-Loop区和Cytb基因序列的碱基组成
Cytb基因序列长度为1 141 bp,序列中A、T、G和C 4种碱基的平均含量分别为24.3%、27.3%、15.5%和32.4%。A+T的含量(51.6%)略高于G+C(48.4%)的含量。287条Cytb同源序列共定义了25种单倍型(表3),有23个多态位点(表4),其中单一多态位点7个,简约信息位点16个,无碱基的插入或缺失,碱基的转换/颠换比值为4.60,UQ(15)、MQ(17)和LQ(15)3个群体的变异位点数相差不大(表4)。
3个群体单倍型数量及其分布情况见表3。D-Loop区的43个单倍型中,有一半以上(22个)为UQ、MQ和LQ 3个群体独有,10个为其共享,11个为两两共享,hap1(18.81%)和hap2(17.07%)为3个群体共享的频率最高单倍型,MQ的单倍型最多(28种),LQ(23)和UQ(22)次之。Cytb基因的25个单倍型中,有一半以上(14个)为3个群体独有,8个为其共享,3个为两两共享,共享的hap1频率最高(36.93%),其次是hap4(13.94%)和hap3(11.15%),UQ、MQ和LQ 3个群体单倍型数相差不大。上、中和下游3个群体单倍型无地理分布格局。

表3 斑鳜3个群体线粒体基因的单倍型分布
2个基因的单倍型多样性指数(Hd)和核苷酸多样性指数(π)等遗传多样性参数统计结果见表4。斑鳜群体mtDNA D-Loop区的Hd、π和K分别为0.919、0.008 3和6.968;Cytb基因为0.821、0.002 0和2.311。上游、中游和下游3个群体的D-Loop区遗传多样性呈UQ > MQ> LQ递减趋势,而Cytb基因以MQ遗传多样性最高,大小顺序为MQ>LQ >UQ。
2.2 清水江斑鳜群体线粒体DNA的遗传结构
群体内和群体间的遗传距离见表5。群体内,D-Loop区上游、中游和下游3个群体内遗传距离从0.009 2±0.001 7递减到0.007 4±0.001 5,其遗传变异程度也逐渐变低;而Cytb基因以MQ的变异最高,UQ和LQ的变异程度相同。群体间,D-Loop区的UQ与MQ的遗传差异最大,MQ与LQ的遗传差异最小;而Cytb基因3个群体间和群体间的差异程度相近。
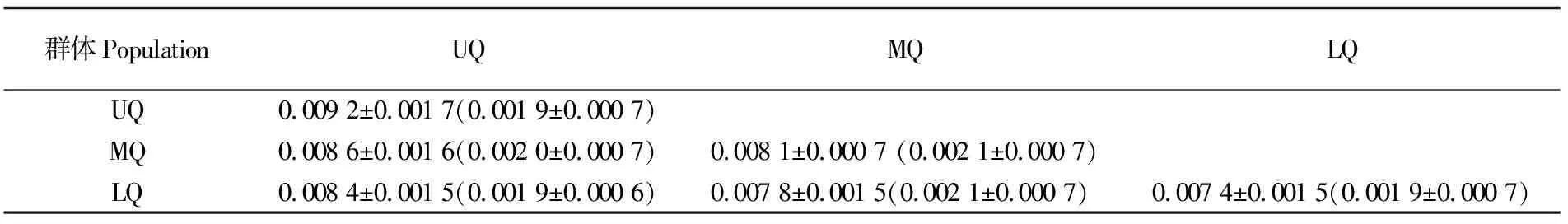
表5 斑鳜群体的遗传距离
群体间的遗传分化指数(Fst)和基因流(Nm)见表6,UQ和LQ之间的Fst明显高于UQ-MQ和MQ-LQ,而Nm明显低于后者。总的来说,基于Fst和Nm2个参数,清水江上游群体与下游的分化程度要高于其他2个河段群体间,这与地理距离远近一致。

表6 斑鳜群体间的遗传分化指数和基因流
基于AMOVA分析(表7),结果显示,斑鳜群体内的遗传变异占97.32%(Cytb为97.54%),而10个站点群体间的变异占2.95%(Cytb为2.47%)。这说明清水江野生斑鳜的遗传变异主要来源于群体内个体间。
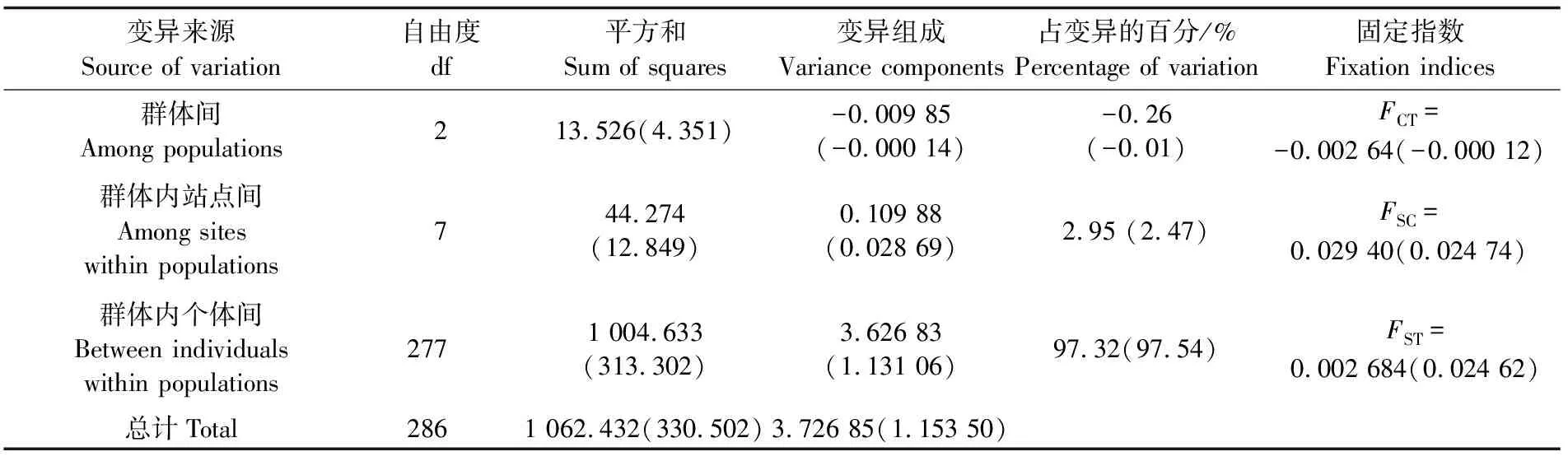
表7 斑鳜群体的分子方差
2.3 清水江斑鳜群体线粒体DNA单倍型的神经网络图和系统树
基于MJ法,Cytb基因单倍型构建的神经网络图显示(图2-A),hap1和hap4两个中心单倍型间因3个节点(或单倍型缺失)而分为2个区域,它们分别进化出17个和6个单倍型,进化的单倍型呈星状散射分布;D-Loop区的43个单倍型构建的神经网络图(图2-B)未呈现出单一星状散射分布,但以Hap1为中心进化出分支节点复杂、突变步骤长的42个单倍型。
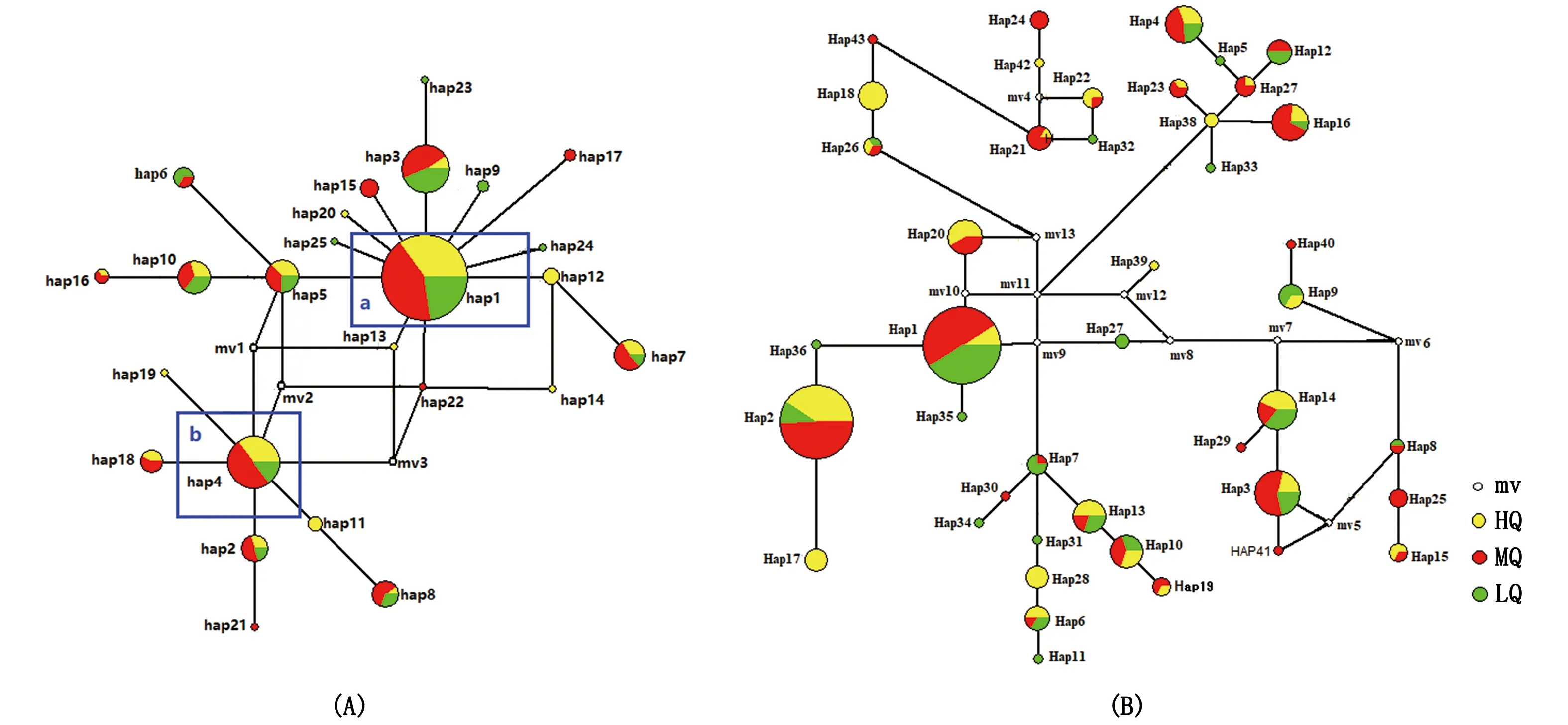
图2 斑鳜群体单倍型的神经网络图(A: Cytb;B: D-Loop)
总体来看,清水江斑鳜群体2个基因的单倍型缺乏明显的地理分布格局。
以中国少鳞鳜为外群(Cytb: JN315581; D-Loop: KJ149811),基于K2p模型构建的2个基因单倍型NJ进化树见图3和图4,显示Cytb和D-loop基因均聚为2支,树枝上的单倍型没有形成明显的地理划分,Cytb树上显示清水江斑鳜与长江流域的洞庭湖水系斑鳜具有较高亲缘性。
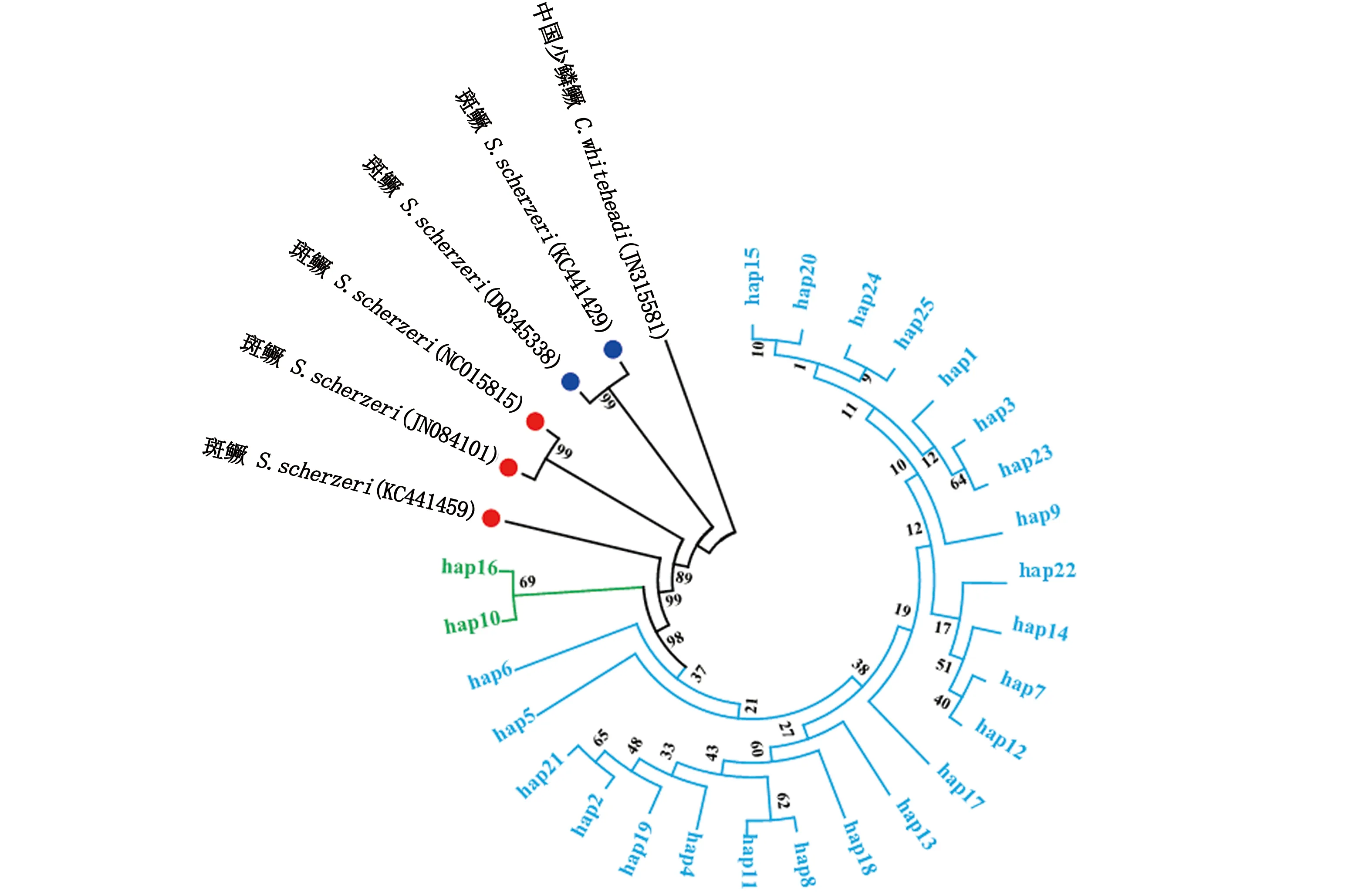
图3 斑鳜群体mtDNA Cytb基因单倍型的NJ进化树
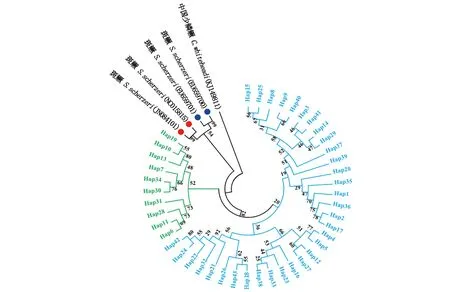
图4 斑鳜群体mtDNA D-Loop区单倍型的NJ进化树
2.4 清水江斑鳜的种群动态
鉴于上文,清水江野生斑鳜群体并没有发生明显的遗传分化现象,所以从整体角度分析清水江斑鳜的群体动态。mtDNACytb和D-Loop区的Fu’sFs值见表4,其中Cytb呈显著负值,检测出了种群扩张,其核苷酸不配对分析图呈现单峰(图5),进一步说明清水江斑鳜群体在近期发生过群体扩张。据公式推算出清水江斑鳜于0.176Ma年前发生了扩张,随后处于相对稳定状态。D-Loop区符合中性进化的假设,未检测到种群扩张。

表4 斑鳜群体的遗传多样性和中性检验
3 讨论
3.1 遗传多样性
遗传多样性是指群体之间以及群体内部的遗传差异[21],是物种进化和环境适应的基础,遗传多样性水平较低可能会使该物种的资源衰退甚至濒临灭绝,而较高的遗传多样性能够为该物种进化提供充足的潜力,以确保物种的延续[22]。清水江野生斑鳜群体mtDNA D-Loop区和Cytb基因的Hd和π分别为0.919/0.82 1和0.008 3/0.002 0。根据WAS和 BOWEN[23]制定的单倍型多样性0.5和核苷酸多样性0.005为界的分类标准,D-Loop区属高Hd和高π,Cytb基因为高Hd和低π,依据mtDNA D-Loop区的高Hd高π遗传多样性特点,预示清水江斑鳜群体是一个大而稳定的种群,经过长时间演化积累了丰富的遗传多样性。
从整体来看,D-Loop区多样性水平要明显高于Cytb基因。2个基因在遗传多样性分析上的差异,与D-loop 区为非编码序列、其进化速率比Cytb基因更快有关,因而在遗传多样分析上,其灵敏度高于Cytb基因[12]。但也存在控制区D-Loop变异速率低于Cytb的现象,比如在陈康等[22]对大獭蛤(Lutrariamaxima)的5个不同地理群体的遗传多样性研究中Cytb单倍型多样性指数(0.834 5±0.030 3)大于D-Loop区(0.445 6±0.049 8)。从河段来看,3个群体的Cytb基因序列遗传多样性水平呈MQ> LQ>UQ的现象,而D-Loop区则呈UQ>MQ> LQ的趋势,由于mtDNA 不同区域的进化速率不同,所以采用 mtDNA 不同区域分析得到的序列遗传变异特征会存在差异[24]。
3.2 遗传分化
清水江野生斑鳜群体内的遗传距离为(0.007 4±0.001 5)~(0.009 2±0.001 7)(D-Loop)和(0.001 9±0.000 7)~(0.002 1±0.000 7)(Cytb),群体间的遗传距离为(0.007 4±0.001 5)~(0.008 6±0.001 6)(D-Loop)和(0.001 9±0.000 6)~(0.002 1±0.000 7)(Cytb),群体内和群体间的遗传距离十分接近,且远低于2%的种间遗传分化界限[25],没有发生明显遗传分化。AMOVA分析结果也得到同样的结论,其遗传变异绝大部分来源于群体内个体间,只有少量来源于群体间和河段间。其河段间的遗传分化指数分别为Fst=0.001 15~0.009 07(Cytb)和Fst= 0.007 21~0.025 53(D-Loop),处于0~0.05的无分化范围内[26],整条清水江的斑鳜存在较频繁的基因交流。通常来说,鱼类的遗传分化和地理隔离有关[27],人类修建的水利工程会使河流中的原物种的有效群体缩小和孤立,起到地理隔离的作用,从而影响其遗传多样性[28-29]。清水江3个群体的遗传多样性水平十分接近,可能是清水江干流中的梯级电站建成的时间没有足够长,对斑鳜种质资源的影响尚未显现。但是相对而言,上游群体(UQ)和下游群体(LQ)之间的基因交流更少,出现这种现象的原因可能有以下两点:一是上下游之间的地理距离遥远;二是沅江的三座大型梯级电站三板溪、白市和挂治均位于下游,斑鳜无法自由向上游或者下游迁移,从而影响其遗传分化[30]。此外,清水江上游主要是产优质杉木的林业区,而下游则主要为农业区[31],下游人类活动更为频繁,人为因素的影响使得中上游的斑鳜群体遗传多样性高于下游群体。
清水江是长江流域中洞庭湖水系的支流沅江的上游,从进化树上看,Cytb基因显示和长江上游的洞庭湖水系具有较高的亲缘性,但其又单独于洞庭湖中的野生斑鳜独立成支,遗传分化表现明显。王伟伟等[32-33]利用线粒体Cytb基因和D-Loop区分析结果表明,我国不同地理斑鳜群体可以根据地理屏障划分为南北两个群体,CHU 等[34]则将我国斑鳜分为长江群和珠江群,而LIANG等[35]认为前者的研究样本量太少,南北群体划分和长江群珠江群的划分都过于简化,他的研究结果表明我国斑鳜存在两大谱系,但不能简单地按照地理屏障划分,根据他的划分,清水江斑鳜群体属于长江谱系,但在长江谱系内部存在一定的分化。从Cytb基因和D-Loop区定义的单倍型的分布来看,清水江没有形成明显的地理划分,这一点,在NJ系统进化树和神经网络图上也有体现,但是其向不同谱系进化的趋势较为明显,其结果与李珊[36]的研究结论相同。
3.3 群体历史动态
单倍型神经网络图可以用来简单判断某群体是否发生过种群扩张,如单倍型神经网络图呈单一星状散射分布,说明该群体曾经在越到瓶颈后迅速扩增。更为准确的种群动态检测方法通常有两种:一种是基于无限位点模式(infinite-site model)的碱基不配对分析,当碱基不配对分布曲线呈明显的单峰形时被认为群体历史有扩张现象;第二种是Tajima’sD和Fu’sFs中性检验,二者呈负值且差异显著的话则表明该群体有群体扩张史[37],其中Tajima’sD检验是检测早期的种群事件,而Fu’sFs检验则是对最近的种群事件更为敏感[38]。本研究中,斑鳜群体的mtDNACytb的单倍型神经网络图呈现以hap1和hap4为中心的两个星状辐射,显示清水江斑鳜群体在历史上曾发生过局部扩张;而且其Tajima’sD和Fu’sFs值均呈负值,但Tajima’sD不显著,Cytb的Fu’sFs值呈显著负值和核苷酸不配对分析图呈单峰均表明了清水江斑鳜近期发生过种群扩张。D-Loop区则符合中性进化的假设,未检测到种群扩张。据平均τ值估算,清水江斑鳜约在0.176百万年前发生了扩张,这个时期属于更新世中晚期,而更新世冰期与间冰期的更迭产生的气候波动对许多动植物的遗传多样性和分布格局产生了影响[39]。
总的来说,清水江斑鳜群体遗传多样性高,虽向着不同谱系进化,但未形成明显的地理分布格局,群体分化不明显。综合比较长江水系其他斑鳜群体,清水江斑鳜群体拥有较高的遗传多样性,可作为一个管理保护单位。近年来,沅江梯级电站的开发势必对清水江生态环境造成巨大的影响,从而对斑鳜的种质资源形成潜在的威胁。因此,有必要对该地区的野生斑鳜资源进行保护,以期得到合理的开发利用。
