鳗弧菌(Vibrio anguillarum)核酸适配体的筛选及其结合蛋白的分离鉴定*
2022-02-10刘慧敏黄力行林筱钧彭雪云江兴龙周建传汤学敏
郑 江 刘慧敏 黄力行 林筱钧 彭雪云 江兴龙 周建传 汤学敏
(1)集美大学水产学院,鳗鲡现代产业技术教育部工程研究中心,福建省水产生物育种与健康养殖工程研究中心,厦门 361021;2)福建省特种水产配合饲料重点实验室,福清350308)
核酸适配体是采用SELEX 筛选技术获得的对靶目标有较高亲和特异性的寡核苷酸序列[1]。该技术以人工合成的随机寡核苷酸文库为初始文库,利用寡核苷酸可在空间形成多种多样的三维结构,将寡核苷酸文库与靶目标经过多轮的结合、分离、扩增等筛选进化过程,最终筛选出能与靶目标特异性结合的寡核苷酸分子,即核酸适配体[1]。与抗体蛋白相比,核酸适配体具有亲和特异性好、稳定性高、靶分子范围广等多种优点,在细菌、病毒、蛋白质等的识别鉴定和药物开发方面都展现出较好的应用潜力[2‑7]。
鳗弧菌(Vibrio anguillarum,V.anguillarum)属于弧菌科、弧菌属,是致病性较高的一种条件致病菌,可以感染多种海洋或淡水环境中的鱼类、贝类等,给水产业造成巨大的经济损失[8‑9]。对鳗弧菌进行快速的识别鉴定是其病害防治的前提和基础。但由于弧菌种类多、遗传变异快、基因特征又相似,早期的感染症状也非常相似,因此传统的微生物培养法和16S rDNA 等方法对其进行检测鉴定的效果并不理想[10‑12]。核酸适配体作为一种分子识别工具,早期主要用于肿瘤标志物的识别鉴定和肿瘤细胞的发现,近年来发现其在微生物的检测方面也有较好的应用效果[12‑14],并可作为一种分子标记工具应用于微生物相关靶标位点的分析及其侵染机制的研究[15‑17]。例如,Yu等[17]利用核酸适配体标记石斑鱼虹彩病毒的衣壳蛋白,研究了该病毒的侵染致病机制。因此,筛选鳗弧菌的核酸适配体,利用核酸适配体对鳗弧菌相关位点进行分析鉴定,不仅能为鳗弧菌的检测鉴定提供一个新的手段,对于了解鳗弧菌的这些位点和研究这些位点在鳗弧菌病害防治中的作用也具有重要意义。
本研究对鳗弧菌核酸适配体的1~5轮筛选产物进行高通量测序,从其中的高频序列中挖掘出候选的核酸适配体序列,然后通过亲和特异性研究、亲和常数、饱和亲和力等指标对相应的核酸适配体进行验证和表征,最后通过磁分离、聚丙烯酰胺凝胶电泳(PAGE)和质谱对核酸适配体的结合蛋白进行分离和鉴定,相关研究对于鳗弧菌的检测鉴定及其病害防治都具有重要意义。
1 材料与方法
1.1 实验材料
鳗弧菌(V.anguillarum)、迟缓爱德华氏菌(Edwardsiella tarda)、溶藻弧菌(Vibrio alginolyticus)、哈维氏弧菌(Vibrio harveyi)、大肠杆菌(Escherichia coli)、嗜水气单胞菌(Aeromonas hydrophila),均由集美大学病原生物实验室提供。随机ssDNA 文库为:5′‑TCAGTC‑GCTTCGCCGTCTCCTTC(N35)GCACAAGAG‑GGAGACCCCAGAGGG‑3′,长度为82 nt,N35为含35个随机碱基的核苷酸序列。PCR用的引物P1:5′‑TCAGTCGCTTCGCCGTCTCCTTC‑3′;P2:5′‑CCCTCTGGGGTCTCCCTCTTGTGC‑3′。文中实验用到的核酸适配体序列为:5′‑TCAGTCGCTT‑CGCCGTCTCCTTC(N)GCACAAGAGGGAGAC‑CCCAGAGGG‑3′,两端是固定序列,不同核酸适配体的中间序列N 不同(表1)。随机文库、引物和核酸适配体序列均由生工生物工程(上海)股份有限公司(简称上海生工)合成和标记。含有DNA聚合酶、dNTP、Mg2+以及PCR稳定剂和增强剂组成的PCR 缓冲预混体系2×Super Pfx MasterMix,购于福建泉州华诺生物科技有限公司。链霉亲和素磁珠购于中科雷鸣(北京)科技有限公司。20×结合缓冲液(pH 7.4,100 ml)的配制:NaCl 5.844 g、KCl 3.725 g、Tris‑HCl 6.06 g、MgCl2·6H2O 2.033 g,以上物质全部溶于超纯水中,调节pH至7.4,并用超纯水定容至100 ml,使用时用超纯水稀释为2×和1×结合缓冲液。

Table 1 Middle sequences of aptamers
1.2 核酸适配体的SELEX筛选
SELEX 筛选流程和方法参照文献进行[18]。初始随机文库及每轮的筛选文库在筛选前都先在95℃热变性5 min,然后冰浴复性10 min,以消除原有结构的影响,保证筛选效果。筛选共进行5轮,前3轮以鳗弧菌为靶目标,每轮筛选中筛选文库与鳗弧菌的结合时间为2 h,洗涤1 次,每轮筛选获得的菌沉淀经重悬、加热、离心后,取上清,上清即为该轮的筛选产物和下一轮的筛选文库;第4 轮是以哈维氏弧菌和溶藻弧菌为靶目标的反筛,筛选文库分别与哈维氏弧菌结合1 h,洗涤2 次,弃菌沉淀,上清再与溶藻弧菌结合1 h,洗涤2次,弃菌沉淀,该上清即为第4轮的筛选产物,并作为筛选文库再进行第5轮筛选;第5轮筛选是以鳗弧菌为靶目标,筛选文库与鳗弧菌的结合时间为1 h,洗涤2 次,菌沉淀经重悬、加热、离心后,取上清,即为该轮的筛选产物。最后将这5轮的筛选产物送上海生工进行高通量测序,选择测序结果中的高频序列进行分析研究。
筛选过程中采用不对称PCR 进行扩增,反应体系 为:模板4 μl,10 μmol/L 的P1 引物4 μl,0.4 μmol/L 的P2 引物4 μl,含 有DNA 聚合酶、dNTP 的PCR 缓冲预混体系2×Super Pfx MasterMix 25 μl,加双蒸水至50 μl。不对称PCR 的热力学循环参数为:98℃预变性3 min,98℃变性10 s,60℃退火30 s,72℃延伸15 s,30 个循环,最后72℃延伸5 min。
1.3 核酸适配体的亲和力测定
核酸适配体在使用前先在95℃热变性5 min,再冰浴复性10 min,然后按照文献的方法[19],通过测定目标菌上结合的ssDNA 浓度来反映其亲和力。
1.4 核酸适配体的亲和常数和饱和(最大)亲和力的测定
取10 μmol/L的核酸适配体,用2×结合缓冲液将其稀释成不同的浓度梯度,然后按照1.3的亲和力测定方法,测定每个浓度梯度下核酸适配体的亲和力,再以核酸适配体的浓度为横坐标,对应的亲和力为纵坐标,采用Origin 8.0 软件、选择反比例函数(Hyperbola 函数)进行非线性拟合,从而获得相应核酸适配体的饱和结合曲线及其拟合方程,从拟合方程中可以得到相应核酸适配体的亲和常数(Kd)值和饱和亲和力(Am)值。
1.5 核酸适配体结合蛋白的分离和鉴定
根据1.3的测定结果,选取亲和力较高的核酸适配体H5,通过构建核酸适配体‑磁珠复合物,采用磁分离法对其结合蛋白进行分离,再用质谱对分离纯化出的蛋白质进行鉴定,具体如下:
鳗弧菌全菌蛋白液的制备:取5ml、浓度为5×1011个/L 的鳗弧菌,用超声波细胞粉碎机(JY92‑IIDN,宁波新艺)进行细胞破碎,相应的工作参数为:功率40%,工作时间2 s,间隙时间3 s,持续时间40 min,从而制备出鳗弧菌的细胞破碎液或全菌蛋白液。
磁珠‑适配体复合液的制备:取5µmol/L、5′端标记生物素的核酸适配体40µl,95℃加热5 min后再冰浴10 min,然后与200µl、2 g/L 的磁珠混合,于28℃、200 r/min 的摇床中孵育结合2 h,得到240µl的磁珠‑适配体复合液。
核酸适配体结合蛋白的分离、洗涤及电泳:取鳗弧菌全菌蛋白液100µl,与240µl的磁珠‑适配体复合液混合后,于28℃摇床200 r/min 孵育结合2 h,磁分离后弃上清,沉淀部分即为磁珠‑适配体‑结合蛋白复合物,然后向该沉淀中加入50µl 2×结合缓冲液重悬沉淀,即得到未洗涤的结合蛋白样品。未洗涤的结合蛋白样品经磁分离后,弃上清,沉淀再加入50~100µl 2×结合缓冲液洗涤重悬,即得到洗涤1次的样品,同样方法可以得到洗涤2次以上的结合蛋白样品。取上述样品液40µl,加入5×加样缓冲液8 µl,在100 V 电压下进行PAGE 1 h,电泳结束进行染色、脱色、割胶,割下来的含有蛋白质样品的胶条送上海生工生物工程公司进行质谱分析和鉴定,可得到相应的候选蛋白及其氨基酸的排列顺序。
1.6 核酸适配体及其结合蛋白的结构分析和亚细胞定位
登录 Structurewang 网站(http://rna.urmc.rochester.edu/RNAstructureWeb/),输入核酸适配体H5的核苷酸序列,选择类型为DNA,即可获得该核酸适配体最可能出现的二级结构。
将质谱分析得到的具有最大可能性的结合蛋白序列命名后输入Prabi 网站(https://npsa‑prabi.ibcp.fr/cgi‑bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html),选择参数如output、width 等为默认值,可对其二级结构进行预测分析。
在Phyre2 网站(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)中输入结合蛋白的完整氨基酸序列,选择建模模式为normal,可得到结合蛋白的三级结构预测图。再通过PyMOL 软件打开该三级结构图的PDB 文件,对其中的二级结构分布位置进行颜色标记,从而可得到更为细化、精确的三级结构图。
参考丁秀敏等[20]的方法,使用Psortb 3.0线上网站(http://www.psort.org/psortb/)对核酸适配体结合蛋白在鳗弧菌中的定位进行预测。具体方法如下:进入网站后,选择细菌里的革兰氏阴性菌,按照fasta 格式插入结合蛋白的氨基酸序列即可进行预测。
1.7 统计数据分析
实验数据采用t检验来进行组间的差异分析,P<0.05为显著差异,P<0.01为极显著差异。
2 结果
2.1 核酸适配体富集库的亲和力变化
核酸适配体富集库是筛选产物经不对称PCR扩增得到的PCR 产物。随着筛选轮数的增加,富集库对鳗弧菌的亲和力呈现逐渐上升的趋势,从第1 轮的1.60 mg/L 上升至第5 轮的12.03 mg/L,上升了6.52倍(图1),说明筛选取得了较好效果。

Fig.1 Affinity changes of aptamer enrich library in the selection
2.2 候选核酸适配体序列的选择
高通量测序后会得到数万条序列,其中有些序列是出现两次以上的,称之为高频序列。表2统计了前5 轮出现的高频序列,从中可看出,前5 轮中,H5 在每一轮都有出现,H1 则出现了4 轮,只在第3 轮没有出现,H21 出现了3 轮(第1、4、5轮),而H6、H12、H25、H26、H28、H33、H38、H42则都出现了两轮(第4、5轮),说明这些高频序列对鳗弧菌有较高的亲和特异性或较好的扩增性,从而能在各轮的筛选中保留下来成为优势序列,因此这些高频序列大概率是具有较高亲和特异性的核酸适配体序列,后续则选择H1、H5、H6、H12、H25、H26、H28、H33、H38 和H42 等10 个序列进行亲和特异性研究。
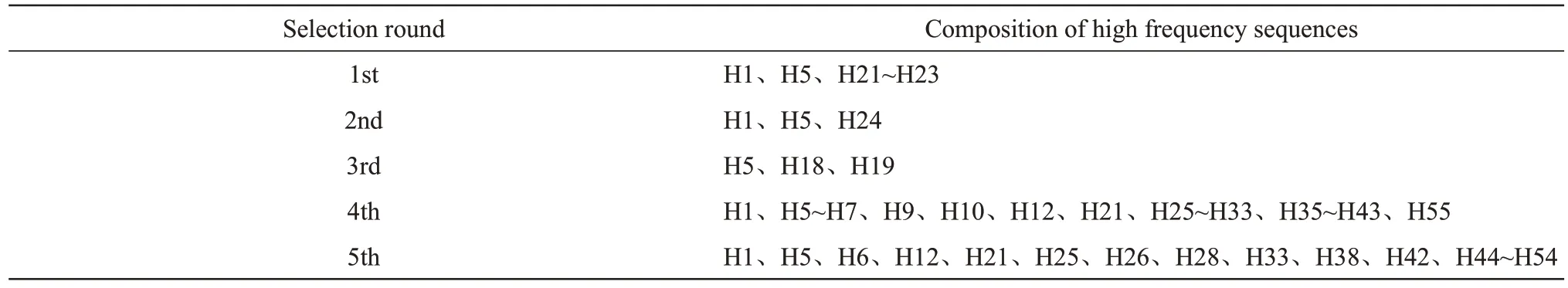
Table 2 High frequency sequences in the first 5 rounds of selections
2.3 核酸适配体的亲和特异性研究
图2 显示了10 个高频序列(H1、H5、H6、H12、H25、H26、H28、H33、H38、H42)的亲和特异性,从中可看出,10 个高频序列对目标菌鳗弧菌的亲和力均显著高于其他5 种非目标菌(P<0.01),表明这10个高频序列对鳗弧菌都具有较好的亲和特异性,都是鳗弧菌的核酸适配体。10个核酸适配体对鳗弧菌的亲和力从高到低依次为H5、H25、H26、H33、H38、H42、H6、H12、H28、H1,相应的亲和力大小依次为13.75、11.38、10.10、9.72、9.30、8.61、6.50、5.73、5.31、3.83 mg/L。比较而言,前5 个核酸适配体(H5、H25、H26、H33、H38)对鳗弧菌的亲和力是对其他菌的5倍以上,亲和特异性更好。而核酸适配体H1 的亲和力虽然是最低的,但它在前5 轮中出现了4 轮,仅次于H5,排第二位,说明它在筛选进化的竞争中也具有一定的优势,因此后续研究亲和常数时,我们选择H1、H5、H25、H26、H33、H38这6个核酸适配体进行研究。
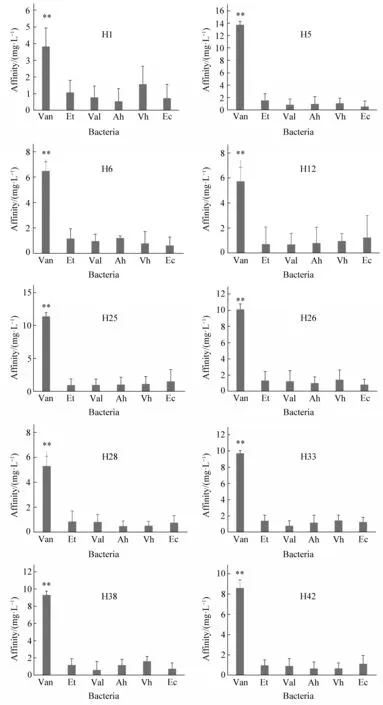
Fig.2 Affinities of 10 aptamers(H1,H5,H6,H12,H25,H26,H28,H33,H38 and H42)towards the bacteria V.anguillarum(Van),E.tarda(Et),V.alginolyticus(Val),A.hydrophila(Ah),V.harveyi(Vh)and E.coli(Ec)
2.4 核酸适配体的亲和常数(Kd)和饱和亲和力(Am)
对出现频率较高的核酸适配体H1 以及亲和特异性较高的5个核酸适配体H5、H25、H26、H33、H38进行了亲和常数(Kd)和饱和亲和力(Am)的测定,相应的饱和曲线如图3,亲和常数和最大亲和力则汇总在表3 中。从图3 可以看出,饱和曲线的拟合系数都在0.95 以上,显示出较好的拟合效果。6 个核酸适配体的亲和常数Kd在50~350 nmol/L 之间,饱和亲和力Am在200~1 000 nmol/L 之间。其中Kd值最大的是H26,最小的是H1,Am值最大的是H5,最小的是H1。
通常,Kd值越低,对靶目标的结合能力就越强;Am越高,说明靶目标上的结合位点就越多,能结合的核酸适配体数量就越多。因此,Kd越小,Am越大,核酸适配体最终表现出的亲和力就越大。不过,对于同一个核酸适配体,这两个参数表现的并不完全一致。如核酸适配体H1,其Kd值较低,说明对鳗弧菌的结合能力较强,但其Am却较低,说明鳗弧菌上H1的结合位点较少,能结合H1的数量并不多,因此最终H1 对鳗弧菌的表观亲和力并不高(图2,H1);核酸适配体H5,其Kd值较高,说明对鳗弧菌的结合能力并不强,但其Am较高,说明鳗弧菌上H5 的结合位点较多,菌上能结合的H5较多,因此最终体现出H5对靶目标鳗弧菌的表观亲和力却是最高的(图2,H5)。因此,核酸适配体最终的表观亲和力应该是Kd和Am共同作用的结果,而不能仅仅只看其中一个参数。

Fig.3 Saturation curves of the affinity constant of aptamers against V.anguillarum
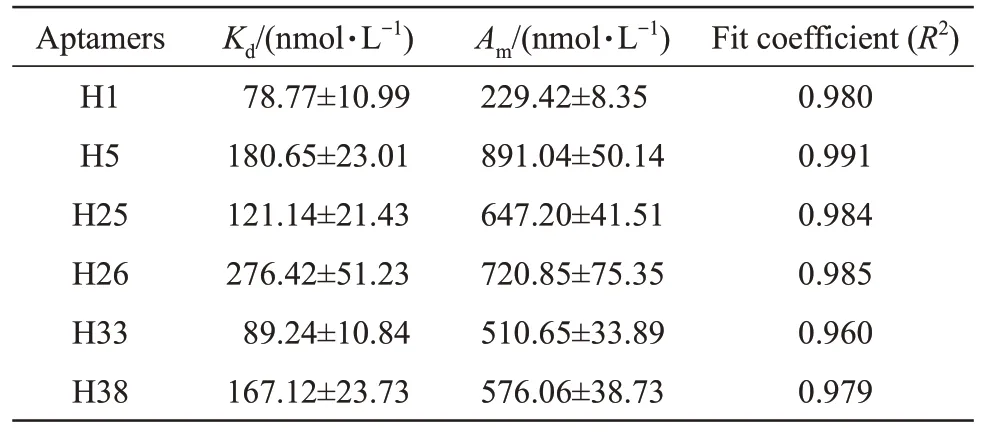
Table 3 Affinity constant(Kd)and maximum affinity(Am)of aptamers against V.anguillarum
2.5 核酸适配体H5结合蛋白的分离与鉴定
对磁分离后的结合蛋白样品进行电泳,未洗涤的结合蛋白样品有多个蛋白质条带(图4a 的N 泳道),说明杂蛋白较多;洗涤后的结合蛋白样品只在100 ku 和15 ku 附近各有一个较浅的条带,其他分子质量的蛋白质条带则几乎都已消失(图4a 的Y泳道),说明这两个蛋白质可能与核酸适配体H5或者磁珠的结合较为紧密,并没有因为洗涤而消失;进一步研究发现,磁珠对照的电泳中也出现了15 ku的条带,但没有出现100 ku的条带(图4a的MB 泳道),说明100 ku 的蛋白质不是磁珠自带的蛋白质,而15 ku的蛋白质应该是链霉亲和素磁珠自带的亲和素蛋白,后续质谱鉴定也证实了该15 ku 的蛋白质是磁珠的亲和素蛋白。由此基本可以确定100 ku 的蛋白质很可能是核酸适配体H5 的结合蛋白。为进一步确认,我们降低了对结合蛋白样品的洗涤力度,得到图4b 的电泳图,从中可看出,结合蛋白样品经过1 次和2 次洗涤后,在100 ku附近仍然有一条较为明显条带,而其他分子质量的蛋白质条带几乎完全消失(图4b中的1和2泳道),说明该100 ku的蛋白质与核酸适配体H5的亲和力较高、结合较为紧密,并没有因为洗涤而消失,应该是核酸适配体H5的结合蛋白。
对100 ku 的核酸适配体H5 的结合蛋白进行质谱鉴定,其可能的候选蛋白如表4所示。由表中可看出,得分最高、匹配度最高的是丙酮酸脱氢酶E1组分,其分数为810分,远高于其他蛋白质的得分,说明H5 的结合蛋白最有可能就是鳗弧菌中的丙酮酸脱氢酶E1组分。

Fig.4 PAGE of bacterial protein binding with aptamer H5

Table 4 Possible binding proteins of aptamer H5 analyzed by mass spectrum
2.6 核酸适配体H5与其结合蛋白的结构分析和亚细胞定位
核酸适配体H5 的主要功能区是3 个环,在5′端的环由9 个核苷酸构成,在3′端的环由6 个核苷酸构成,中间的环较大,由17 个核苷酸组成。如果按照两个核苷酸的间距为0.34 nm 进行粗略估算,则5′端环的直径约0.97 nm,中间环的直径约1.73 nm,3′端环的直径约0.65 nm。由于3′端环较小,而5′端是与磁珠连接的,因此与结合蛋白作用的部位很可能是H5 上较大的中间环(许净等.海洋与湖沼,2021,52(5):1315‑1322)。
蛋白质的一级结构是其氨基酸的组成和排列顺序。质谱分析表明,核酸适配体H5 的结合蛋白应该是丙酮酸脱氢酶E1 组分。该蛋白的编号为F7YPI5,通过Uniprot 数据库(https://www.uniprot.org)查询其一级结构,发现该蛋白是由891 个氨基酸组成的。
蛋白质二级结构主要有α 螺旋、β 折叠、β 转角以及无规卷曲等4种形式。分析表明,该结合蛋白的891个氨基酸中,有403个氨基酸形成α螺旋,124个氨基酸形成β折叠,97个氨基酸形成β转角,其余267 个氨基酸形成无规卷曲,4 种二级结构在总氨基酸序列中的占比分别为45.2%、13.9%、10.9%和30.0%。
图5是该结合蛋白的三级结构预测图。图5a的三级结构是按照数据库中评分最高的模板以100%的可信度建立的,用于建模的氨基酸数目为860个,占总氨基酸数目的96.5%,图中颜色由红→蓝表示的是结合蛋白的N 端→C 端。图5b 是在图5a基础上采用PyMOL 软件对其中的二级结构进行更为清晰的标记和展示,从中可看出红色的α螺旋占比较高,黄色的β折叠主要处于蛋白质的内部,绿色的以环状形式出现的无规卷曲结构则大多分布在该结合蛋白的外部。
由于蛋白质中α螺旋的结构较为刚性,核酸适配体的结构主要是茎环等结构,因此核酸适配体与蛋白间的结合很可能是通过核酸适配体上环结构套在蛋白质中的α螺旋上来实现的。已知一个α螺旋的直径是1.2 nm,核酸适配体H5 的三个环中只有中间的大环可以套在α螺旋上,从蛋白的三维结构上看,图5 箭头所指的3 处α 螺旋的结构很可能是与H5 中间环嵌套结合或互相作用的区域。其中,中间和右侧箭头所指的α‑螺旋,由于外围还有较灵活的无规卷曲等的二级结构包裹,其与核酸适配体H5中间环的结合可能要更紧密些。

Fig.5 Tertiary structure of the protein binding with aptamer H5 and the binding regions(pointed by arrow)
通过在线网站对该核酸适配体结合蛋白进行亚细胞定位,结果显示该结合蛋白,即丙酮酸脱氢酶E1 组分有99.7%的可能性是位于鳗弧菌的细胞质中,有0.1%的概率是位于鳗弧菌的质膜上。这表明核酸适配体H5能以某种方式进入鳗弧菌的内部,并与菌内的丙酮酸脱氢酶E1组分结合。
3 讨论
SELEX 筛选通常都要经过少则5 轮、多则10多轮的筛选,使富集文库中的核酸适配体得到足够多的富集后再进行测序[21‑26]。多轮筛选不仅增加了工作量,而且由于筛选过程中涉及结合、分离、PCR 等多个步骤,只要有一个步骤出现问题都会导致后续的筛选失败,因此大大增加了筛选的难度和风险。本文采用“每轮测序”的方法,从第1轮开始就对每轮的筛选产物都进行高通量测序,并选择测序结果中的高频序列进行研究,结果表明,有较高亲和力的核酸适配体H1和H5在第一轮的高频序列中就已呈现出来,因此理论上只需一轮的筛选和测序就可获得相应的核酸适配体序列,这极大地提高了筛选的效率;而且该方法对筛选富集的要求大大降低了,只要有少许富集,就能在测序结果中的高频序列上体现出来,即使富集文库整体的亲和力并没有上升,也能获得预期的核酸适配体序列。比如本文的第1轮筛选,筛选产物的亲和力是非常低的,但高通量测序却发现其中已经有了5个高频序列,而亲和力较高的核酸适配体H1和H5在第一轮测序中就已经被挖掘出来了。因此,通过这种“每轮测序”并聚焦高频序列的方法,可以快速发现核酸适配体的候选序列,大大提高了筛选效率。当然,虽然理论上只要进行一轮的筛选和测序就有可能获得期望的核酸适配体序列,但为保险起见,进行3~5轮的筛选和高通量测序,选择多轮次出现的高频序列作为核酸适配体的候选序列进行验证分析,应该是一种较为稳妥可靠的策略。
有关高频序列的亲和力问题,不同学者的研究结果似乎并不一致。Jing 等[25]筛选硼酸钠的核酸适配体时发现,出现频率最高的两个核酸适配体,它们的亲和力也是最高的;Lu 等[13]的SELEX 筛选也是优先选择测序中出现频率较高的序列进行研究的;不过,Zhang 等[14]分析肠炎沙门氏菌的核酸适配体时发现,频率最高的序列,其亲和力却比较低;Stuart 等[27]筛选一种蛋白质的核酸适配体时也发现,频率最高的S1 序列,其亲和力却是最差的;还有学者认为,一些亲和力相对较低、但容易扩增的序列,经过多次PCR后会成为高频序列,而一些亲和力高但不易扩增的序列则可能随着筛选的进行而不断丢失,最后可能被淘汰或成为低频序列[28]。我们认为,少数高频序列的亲和力不高是正常的现象,因为核酸适配体筛选进化中存在两种策略,一种是以高亲和力为主的策略,一种是以高扩增性为主的策略。如果某些高频序列是通过后一种进化策略得到的,则其亲和力不高是完全可能的,但还有相当多的高频序列是通过高亲和力为主的策略得到的,有些可能是两种策略兼有的,这些高频序列则大概率都是有较高亲和力的核酸适配体。我们的前期研究也表明,高频序列大多都是亲和特异性较好的序列[18]。本文的研究也表明10个高频序列中有9个对目标菌的亲和力是较高的。因此,从高频序列中找到高亲和力核酸适配体的概率是相当高的。更关键的是,高频序列在总序列中的占比通常都是很低的,因此,从高频序列中挑选候选序列进行验证能极大提高筛选验证的效率,大大节约筛选验证的成本。当然,选择高频序列,不可避免的会失去那些只出现一次、但却有较高亲和力的序列。不过这种舍弃应该是值得的,因为舍弃的只是出现频率较低、扩增性较差的高亲和力序列,大量高频、扩增性较好的高亲和力序列仍然在候选序列库中,而且这个舍弃能换来筛选验证效率的大幅提升和成本的大幅下降,完全是值得的。
在核酸适配体的研究方面,通常对其亲和力的主要评价参数是亲和常数Kd,而对饱和亲和力参数Am则较少涉及和关注[29‑31]。本文则发现最终的表观亲和力A不仅与Kd有关,还与Am密切相关。根据公式,表观亲和力,其中[Apt]是适配体的浓度,核酸适配体最终的表观亲和力应该是Kd和Am这两个参数共同作用的结果。因此,单纯根据Kd值来判断适配体的表观亲和力是不全面的。从前面亲和特异性的研究中也可以看出,Kd值较低、单位结合能力较高的核酸适配体H1,由于Am较低的原因,其最终的表观亲和力并不高;而Kd值较高、单位结合力较低的核酸适配体H5,因为其Am较高,最终的表观亲和力却是最大的,说明Am在核酸适配体表观亲和力中的影响不仅不能忽略,有时Am影响甚至超过了Kd,起到了主要作用。
有关核酸适配体的结合蛋白,从已有的报道来看,以细胞表面的膜蛋白居多[15,32‑34]。不过,Zhang 等[35]研究发现,核酸适配体是能够进入小鼠胚胎成纤维细胞,并与解旋酶、GTP 酶、被膜蛋白复合物等蛋白质结合的,并推测核酸适配体有可能是先结合到细胞表面的膜蛋白,再通过胞吞作用进入到细胞内部,与上述胞内蛋白结合的;Yu等[15‑17]的研究也认为,核酸适配体应该是先与膜蛋白结合,再通过网格蛋白介导的胞吞作用进入细胞内部的。因此,本研究中的核酸适配体H5 很可能也是先与鳗弧菌表面的某种蛋白质结合,再通过胞吞作用进入细胞质,与细胞质中的丙酮酸脱氢酶E1 组分结合。另外,亚细胞定位显示,鳗弧菌的质膜上也有可能分布有少量的丙酮酸脱氢酶E1 组分,因此鳗弧菌表面首先与核酸适配体H5 结合的蛋白质也有可能就是丙酮酸脱氢酶E1 组分及其类似物。当然,这些推理猜测还需要进一步的实验验证。但可以明确的是,核酸适配体能够以胞吞等形式进入细菌内部与其靶标分子结合,这个结合有可能会对细菌的生长、代谢产生影响。这也为新型核酸适配体药物的开发提供了一个新的思路。
4 结论
本文采用每轮测序的筛选策略,选择其中出现轮数较多、频率较高的高频序列作为核酸适配体的候选序列,建立了一种快速筛选核酸适配体的方法,获得了一系列对靶目标鳗弧菌有较好亲和特异性的核酸适配体(H1、H5、H6、H12、H25、H26、H28、H33、H38 和H42),并重点研究和测定了其中6 个核酸适配体(H1、H5、H25、H26、H33、H38)的亲和常数Kd和最大亲和力Am,结果发现核酸适配体最终的表观亲和力是Kd和Am共同作用的结果。通过磁分离技术和PAGE分离得到了核酸适配体H5 的结合蛋白,质谱鉴定表明该结合蛋白为鳗弧菌细胞质中的丙酮酸脱氢酶E1 组分,该蛋白质中的α螺旋在总氨基酸序列中的占比高达45.2%,β 折叠主要位于该蛋白质内部,二者共同构成了该结合蛋白的主要骨架,而环状的无规卷曲结构则主要分布在该蛋白质外部。核酸适配体能够以胞吞等形式进入细菌内部与其靶标分子结合,这也为鳗弧菌病害防治及新型核酸适配体药物的开发提供了一个新的思路。
猜你喜欢
杂志排行
生物化学与生物物理进展的其它文章
- Protein Aggregation and Phase Separation in TDP-43 Associated Neurodegenerative Diseases*
- 细菌的信号转导系统及其在耐药中的作用*
- CRBGP Inhibited The Activity of Glioma U251 Cells Through Suppressing FAK-AKT Pathway and The Secretion of Interleukin-6*
- Bradykinin Upregulated The Expression of Cyclooxygenase-2 in The Submucosal Plexus of Enteric Nervous System of Guinea Pig*
- 阿尔茨海默病体外诊断纳米技术*
- α-Synuclein as a Diagnostic Marker and Therapeutic Target for Parkinson Disease*
