基于宏基因组学的分子生态网络揭示活性污泥微生物相互作用*
2022-01-27邹奎,张鹏
邹 奎, 张 鹏
(湘潭大学 环境与资源学院,湖南 湘潭 411105)
0 引言
传统活性污泥法(AS)、移动床生物膜反应器(MBBR)和膜生物反应器(MBR)等一系列以活性污泥微生物群落为基础的生物处理技术已被广泛应用于城市污水处理[1].在众多生物处理技术中,活性污泥因其形态不同可分为絮体污泥、颗粒污泥和附着生物膜等.活性污泥中微生物群落物种繁多且组成极其复杂,不同种类的微生物通过物质、能量和信息交换而相互作用,例如竞争、共生、寄生等[2].同样地,污染物的去除不仅仅依靠单一微生物而是需要多种微生物之间的协同作用.这种复杂的生态关系可以表示为微生物分子生态网络,以物种为节点,物种相互作用关系为纽带[3].近年来,针对活性污泥微生物生态学研究逐步开展:不同污染物对序批式反应器中活性污泥微生物群落的影响;不同消毒方式对自来水管道中污垢微生物群落组成影响;大空间尺度下探究不同气候条件对污水厂活性污泥微生物群落变化影响等[4-6].而在小空间尺度开展针对不同工艺段活性污泥微生物群落分子生态网络的差异分析却鲜有报道.因此深入剖析不同生物处理单元微生物分子生态网络组成机制对定向调控活性污泥微生物群落和提高生活污水处理效率有着重要意义.
近年来随着高通量测序技术飞速发展,使得宏基因组学在分析土壤、水体、空气等环境中的微生物组成方面占有显著优势[7].环境微生物宏基因组学是以环境中所有微生物的基因组文库数据为基础开展具有针对性的生物信息学分析,从而解决了传统方法中因部分环境微生物不可纯培养而导致的分析不完善问题[8-9].因此通过宏基因组技术探究不同聚集形态下活性污泥微生物分子生态网络组成机制变得可行.
本研究通过宏基因组学对湘潭市河东污水处理厂生物处理单元中的两种不同聚集形态活性污泥样本进行了高通量测序,基于物种分类学注释结果构建活性污泥微生物分子生态网络,分析不同形态下分子生态网络拓扑属性、稳定性的差异,为更好地促进活性污泥法的应用与发展提供了理论指导.
1 材料与方法
1.1 样本采集与处理
本研究分别采集了湘潭市河东污水处理厂好氧池中絮体污泥和附着生物膜样本.湘潭市河东污水处理厂主体工艺为A2O+MBBR工艺,好氧池中以蜂窝环作为生物膜载体.采样成功后分装于10 mL离心管并编号为附着生物膜1~附着生物膜3(AB1~AB3),絮体污泥1~絮体污泥3(FS1~FS3),经初步处理后置于-80℃低温保存.
1.2 样本DNA提取
本研究采用磁珠法提取絮体污泥和附着生物膜样本的基因组DNA.样本微生物细胞在裂解液和蛋白酶的共同作用下被分解和消化,而后在结合液和磁珠的共同作用下吸附纯净DNA,最后通过洗涤、洗脱过程进行回收,收集的DNA用于宏基因组文库的构建.DNA完整性通过凝胶电泳(1%琼脂糖,电压:200 V,时间:30 min)检测,通过Qubit2.0定量浓度.
1.3 高通量测序
基因组DNA片段化后,按照以下步骤构建文库:DNA片段末端修复(末端补充和3'末端加A尾)、连接接头、磁珠分选和连接产物纯化、文库扩增和文库纯化.通过测量文库大小、浓度和长度分布来进行文库质量控制.文库测序通过Illumina Hiseq平台进行[10].原始序列数据已存放在国家基因组科学数据中心(NGDS,登录号:CRA003823,https://ngdc.cncb.ac.cn/).原始测序数据通过FastQC(V0.11.2)软件评估,Trimmomatic(V0.36)软件过滤,得到相对准确有效的数据.使用 IDBA_UD(V1.1.2)软件将每个样本的纯净序列(Clean reads)拼接并组装成长重叠群.根据读数之间的重叠,获得重叠群(Contigs).然后,对多个长为K的序列(K-mer)组装结果进行了综合评定,选择最佳的序列组装结果.最后进行物种与功能注释.
1.4 数据分析方法
香农指数和辛普森指数用于表征样品的物种多样性和基因多样性.T检验用于检验统计显着性,当P< 0.05 时,则认为显着.通过“Protest”函数校正P值,通过Vegan包进行非度量多维标度分析(Non-metric multidimensional scaling,NMDS).上述过程在R3.6.3环境中运行.
利用Pearson相关系数(ρ)探索微生物群落之间的网络共生关系,并使用Matlab2019软件中的 'Corr' 函数计算微生物群落物种之间的ρ和P值.最后,在 Gephi 0.9.2软件中完成数据可视化.
剩余物种的丰度加权平均作用强度(Abundance Strength,AS)计算公式:
式中:ASi为物种i丰度加权平均作用强度;aj为与物种相关的所有物种;βij为物种i与物种j的相互作用强度,即Pearson相关系数.ASi= 0表示与物种相关的所有物种均被移除,物种i将不能继续生存;ASi< 0表示在其他物种的竞争影响下,物种i将自然灭绝.
2 结果与讨论
2.1 活性污泥微生物物种组成
根据NR数据库(https://www.biostars.org/p/342521/)比对结果,本研究分析了活性污泥微生物在纲分类水平上的物种组成特征.考虑到活性污泥生态系统微生物种类繁多,本研究仅选择了相对丰度前15的物种分别为:Betaproteobacteria,Alphaproteobacteria,Gammaproteobacteria,Nitrospira,Deltaproteobacteria,Planctomycetia,Chitinophagia,Anaerolineae,Chloroflexi_noname,Acidobacteria_noname,Bacteria_noname,Actinobacteria_class,Cytophagia和Flavobacteriia.其他纲水平物种被归类为Other,如图1所示.

图1 活性污泥微生物物种组成Fig.1 Composition of microbial species in activated sludge
由图1可知,对物种数据进行归一化处理之后,活性污泥微生物表现出了明显的分类簇,以Alphaproteobacteria,Chloroflexi_noname和Actinobacteria_class等为代表的絮体污泥(FS)微生物群落,以Deltaproteobacteria,Alphaproteobacteria和Planctomycetia等为代表的附着生物膜(AB)微生物群落,这与Li等[11]学者所报道的结果具有相似性.除此之外,Deltaproteobacteria,Alphaproteobacteria,Planctomycetia和Nitrospirae作为有机物去除和硝化过程中的关键微生物,在附着微生物中的丰度远高于絮体污泥,这表明附着生物膜相较絮体污泥有着更强的污染物处理能力.
考虑到样本微生物丰度注释结果中存在部分零值,为了避免“马蹄形效应”,本研究采用非度量多维标度(NMDS)分析了附着生物膜和絮体污泥样本微生物群落之间的关系,如图2所示.

图2 活性污泥微生物非度量多维标度分析Fig.2 Non-metric multidimensional scaling (NMDS) analysis of activated sludge microbes
由图2可知,不同絮体污泥微生物群落样本之间存在高度相似性,而附着生物膜则在NMDS2尺度上表现出了一定的差异性,这可能是由于附着生物膜生态系统中更为旺盛的新陈代谢过程所导致多样性.而造成两种不同形态的活性污泥微生物生态系统的差异主要由于Bacteroidetes,Acidobacteria,Planctomycetes和Gemmatimonadetes等特征微生物的分布不同,这与图1所得到结果一致.
2.2 活性污泥微生物分子生态网络
为了进一步剖析不同形态下活性污泥微生物生态系统,本研究基于物种丰度之间的Pearson相关系数(β)构建了微生物分子生态网络[12],当|β| > 0.6且具有显著性差异时(P< 0.05)认为可构建网络,如图3和图4所示.
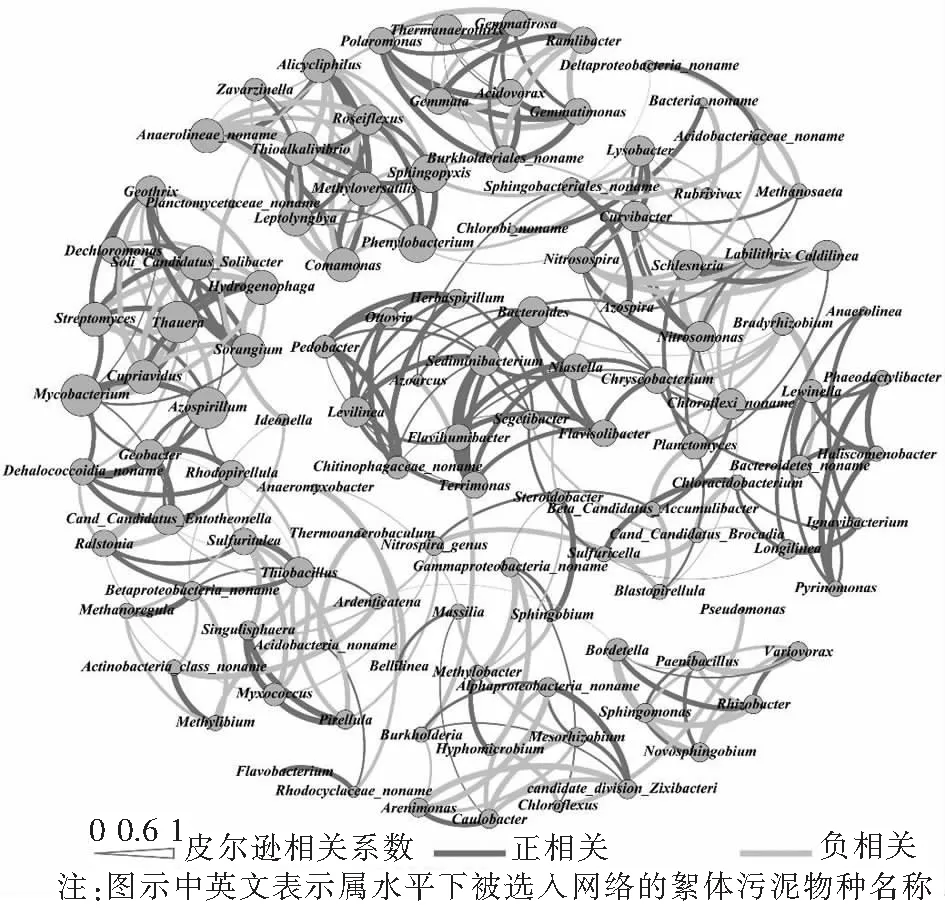
图3 附着生物膜微生物分子生态网络Fig.3 Molecular ecological network of attached biofilm microbes

图4 絮体污泥微生物分子生态网络Fig.4 Molecular ecological network of floc sludge microbes
共有117种属级微生物被选入到网络中,生物膜、絮体污泥微生物分子生态网络中分别包含341和347边,且均表现出了明显的模块化特征(模块化指数分别为0.826和0.828).其中生物膜微生物分子生态网络中以Thauera,Alicycliphilus,Chloroflexi_noname和Anaerolineae_noname为主要的关键物种,絮体污泥微生物分子生态网络则是以Gemmatimonas,Methylibium,Steroidobacter和Sulfuritalea为主要的关键物种,分别与10,9,8,8和11,10,9,9种微生物有着强的相关性.从微生物分子生态网络的组成上不难看出附着生物膜样本中微生物相较于絮体污泥存在较为稀疏的连接,这在一定程度上说明了生物膜网络存在相对较小的相互作用.
2.3 活性污泥微生物分子生态网络差异分析
本研究通过研究网络拓扑属性剖析附着生物膜、絮体污泥微生物分子生态网络之间的差异性,如表1所示.在形成分子生态网络后,拓扑属性可直接通过Gephi软件计算得到[13].在总节点数、正相关边数和负相关边数等属性上表现了一致性,但是在连接部件、平均聚类系数(avgCC)和平均路径强度(HD)却表现出了较大的差异性.

表1 分子生态网络拓扑属性Tab.1 Topological properties of molecular ecological networks
本研究所构建的活性污泥微生物分子生态网络均表现出了“小世界特性”(HD > 3.3~3.8),这表明两种不同形式的活性污泥均具有较强的抗干扰能力,可以迅速将干扰分布到整个网络中,从而提高整个网络的效率.除此之外,有研究表明HD值越高,avgCC的值越小,表明网络成员之间存在稀疏的连接[14].因此本研究推论絮体污泥中微生物之间的相互作用比附着生物膜更为强烈.
2.4 活性污泥微生物分子生态网络稳定性分析
本研究所构建的活性污泥微生物分子生态网络均表现出了高模块化特性,这对微生物所介导的“污水厂生态功能”将有着深远影响.本研究通过随机移除网络中的现在物种并计算网络鲁棒性(抵抗节点丢失能力),来探究活性污泥微生物不同形态下分子生态网络的稳定性.结合前人的研究,本研究将网络稳健性(Robustness)定义为随机移除网络中物种后剩余物种的比例[2].此外,为了描述移除物种后对剩余物种的影响,本研究计算了剩余物种的丰度加权平均作用强度(Abundance Strength,AS),详细计算过程见1.4节.结果如表2和图5所示.

表2 活性污泥微生物分子生态网络鲁棒性检验Tab.2 Robustness test of molecular ecological network of activated sludge microbes
从表2可以看出,在随机移除活性污泥分子生态网络中的50个节点之后,将分别导致生物膜和絮体污泥分子生态网络中的35和44个物种的灭绝,最后网络中仅仅剩余27.4%和19.7%的物种.结合前人研究结果与图5结果,本研究推测附着生物膜上的微生物群落比絮体污泥更为“健壮”[15].

图5 活性污泥微生物分子生态网络鲁棒性Fig.5 Robustness of molecular ecological network in activated sludge microbes


图6 随机移除物种后活性污泥微生物分子生态网络Fig.6 Molecular ecological network of activated sludge microbes after random removal of species
3 结论
在本研究中,基于宏基因组物种分类结果构建了A2O+MBBR工艺中不同形态的活性污泥微生物分子生态网络,结果表明附着生物膜与絮体污泥在微生物组成、分子生态网络拓扑属性以及分子生态网络稳定性方面有着显著差异.絮体污泥微生物群落相较附着生物膜有更为强烈的相互作用,而附着生物膜相较絮体污泥则有着更为丰富的污染物去除物种以及更为健壮的生态网络.综上所述,本研究进一步揭示了活性污泥微生物群落作用机制,为进一步发展活性污泥法在城市污水处理中的应用提供了新的见解.
