环境DNA在湖泊生物多样性研究中的应用
2022-01-20邢迎春高婉茹赵亚辉
邢迎春 高婉茹 白 洁 赵亚辉
(1. 上海海洋大学水产与生命学院, 上海 201306; 2. 中国水产科学研究院资源与环境研究中心, 北京 100141; 3. 中国科学院动物研究所动物进化与系统学重点实验室, 北京 100101)
中国湖泊数量众多, 是陆地水生态系统的重要组成部分, 具有重要的文化、经济和生态价值[1]。中国拥有1 km2以上的自然湖泊2693个, 总面积超过81414.6 km2, 占我国陆地总面积的0.9%[2]。最近30年, 随着我国工农业发展和人类活动的频繁影响,湖泊退化现象严重, 我国东部湖区面积下降了8.2%[3]。湖泊生物多样性水平也随之下降, 物种组成变动较大[4—7]。通常对于湖泊生物多样性的研究多采用传统分类学调查方法, 对采集获得的生物样品进行形态学鉴定, 确定物种分类地位。这样的调查方法虽然已很成熟, 但还是会出现由于采样工具和地理环境限制, 一些珍稀濒危或隐秘物种很难被采集到的问题; 而且利用形态学进行物种鉴定更依赖于科研人员的分类学经验和水平。
环境DNA(Environmental DNA, eDNA)是指从土壤、水、冰川或沉积物等环境样品中提取的DNA, 可揭示过去和现今生物多样性信息[8]。这一方法被用于生物多样性监测, 与传统调查方法相比,具有更高的检出率, 特别是对传统调查中很难发现的隐秘物种; 在调查区域范围较大的情况下, 获得eDNA更加简便和经济; 利用eDNA监测湖泊生物多样性, 可避免对珍稀濒危物种的伤害和对自然环境的干扰; 随着第二代测序技术的发展, 开展eDNA研究的成本也大大降低[9—14]。湖泊环境相对于河流,流速更缓, DNA富集量更高, 因此eDNA在湖泊生物多样性研究方面具有更广阔的应用前景。
1 eDNA应用于湖泊生物多样性的研究回顾
与其他类型水体相比, eDNA技术在湖泊上的应用较早, 于20世纪90年代末就被应用到湖泊生物多样性的研究, 是近20多年来该领域发展起来的新方法、新技术, 科研人员也在应用过程中对实验方案进行不断改进, 对研究结果的准确性和方法的有效性进行不断探讨, 逐步摸索和形成更加成熟的技术方案。
eDNA技术的研究对象广泛, 从细菌、真核微生物、低等动植物到高等动植物, 最初在湖泊中应用是开展细菌类群的研究, 如Brinkhoff等[15]通过设计Thiomicrospira属细菌的16S rRNA特异性引物,采集希腊西奈半岛太阳湖的沉积物和水样品及德国达克斯坦淡水池塘、德国阿尔滕盐泉和智利康塞普西奥陆架环境的沉积物, 提取eDNA分析了该属物种间的系统发生关系。通过开发适用于高等生物类群检测的特异性和通用引物, eDNA逐步应用到研究植物和动物等高等生物方面。Thomsen等[16]从欧洲湖泊、池塘和溪流中检测到两栖动物和鱼类的DNA, 表明eDNA可作为珍稀和濒危物种的监测手段; Parducci等[17]比较了花粉方法和eDNA元条形码技术, 分析了采自位于斯堪的纳维亚地区中部挪威和瑞典边界的斯坎德山脉两侧, 伦特约纳湖和克洛克湖中全新世植物组成, eDNA结果为传统的利用花粉开展分析提供了补充; Bista等[18]通过eDNA分析获得170个属于动物界的分类单元, 其中91个为节肢动物(42个属于昆虫纲); Zhang等[19]利用eDNA分析获得北京未名湖、昆明湖和官厅水库中30、35和41种鱼类。
从样品类型角度分析, 研究湖泊生物多样性的eDNA样品多为沉积物和水。早期研究多集中在对湖泊沉积物中提取的古DNA, 利用其研究湖泊历史和生物多样性的演化。Matisoo-Smith等[20]从新西兰霍克湾圆湖(Round Lake, Hawke’s Bay)未被扰动的沉积物内核中, 在陶波火山层(AD 217—247)上的沉积物中检测到了细菌DNA, 其与人类肠道和粪便细菌高度相似; 同时也检测到了只在新西兰分布的大鮈塘鳢(Gobiomorphus cotidianus)的DNA, 这些研究结果充分展示了eDNA作为一项革命性技术对于第四纪研究的重要性; Anderson-Carpenter等[21]从采自全新世北美五大湖西部地区的7个小型湖泊沉积物中提取的126个古DNA样品中成功获得64个样品的测序数据, 建立了相关实验方法和DNA文库的构建方法; Parducci等[17]从格陵兰南部湖泊沉积物中提取eDNA, 结合基于12个放射碳建立的年代模式分析, 该湖泊生物记录可追溯到距今10650年;Giguet-Covex等[22]从法国阿尔卑斯山区安特恩湖沉积物中提取eDNA, 提供了新石器时代以来该地区植被和畜牧业历史, 提出了人为因素对景观尺度变化影响的新前景。
通过湖泊水体提取的eDNA对于了解湖泊现今水生生物多样性则更为重要。随着高通量测序技术(High-throughput sequencing, HTS)的广泛应用,利用eDNA作为传统调查的补充或替代方法开展生物多样性研究成为更经济和有效的方式, 且这一趋势在2015年后变得更加明显。比较具代表性的研究包括: Thomsen等[16]在欧洲范围内的池塘、湖泊和溪流采集了98个样点的水样品, 开展了一系列eDNA实验, 结果表明eDNA获得结果与实际的物种记录情况基本一致, 所有两栖动物和鱼类的DNA都可被检测到, 该方法可作为珍稀和濒危物种的监测手段。Bista等[18]从英国Llyn Padarn湖采集葡萄球菌和水样品, 选择COI基因中的2个长度不同的片段开展eDNA研究, 分析获得170个属于动物界的分类单元, 其中91个为节肢动物(42个属于昆虫纲); 结果还揭示了eDNA的时间动态变化, 验证了eDNA宏条形码在追踪生态系统季节性多样性方面的实用性。Goutte等[23]利用eDNA对法国巴黎马恩河和塞恩河鱼类多样性和丰富度进行了分析, 结果表明与每年度电鱼监测相比, eDNA宏条形码技术是更好的监测手段; eDNA对于物种的监测比3—6年内开展的电鱼监测更有效, 但与14年长期电鱼监测结果相似或略有差距。近年来中国学者开始应用eDNA技术, 对湖泊鱼类多样性开展研究。Qu等[24]采集江西鄱阳湖和湖北天鹅洲自然保护区的水样品, 使用16S rRNA鱼类通用引物进行PCR扩增, 通过高通量测序和数据分析, 共鉴定出75种鱼类, 创建了一种调查长江流域鱼类多样性的新方法。舒璐等[25]首次应用eDNA宏条形码技术评估洱海鱼类多样性及空间分布, 通过使用鱼类通用引物MiFish-U进行扩增, 共检测出17种鱼类, 其中包括5个土著种和12个外来物种。Zhang等[19]选择北京未名湖、昆明湖和官厅水库作为小型、中型和大型湖泊的代表, 采集了水样品提取eDNA, 利用12S rRNA片段确定鱼类物种组成分别为30、35和41种, 并分析了鱼类空间分布与其生活的典型栖息地具有相关性。
eDNA技术可应用于对湖泊中特定物种或多个生物类群的检测。对特定物种进行检测的典型案例就是在北美五大湖流域和小型湖泊关于入侵物种亚洲鲤的研究, Mahon等[26]利用eDNA检测五大湖地区入侵物种鲢(Hypophthalmichthys molitrix)和鳙(Hypophthalmichthys nobilis), 分别于2000年在伊利湖桑达斯基湾和2010年在距离卡卢梅特湖4和6 km的位置检测到鳙。Eichmiller等[27]监测位于密西西比河上游流域小型湖泊Lake Staring中的入侵鲤(Cyprinus carpio), 检测概率为75%, 且在物种活动区域几十米以外也能通过eDNA的方法进行监测。近年来eDNA更多地被应用在对湖泊中多个生物类群的研究, 对生物多样性进行分析。
2 eDNA研究湖泊生物多样性的实验设计
eDNA研究湖泊生物多样性的主要步骤包括样品的采集和保存、eDNA提取、PCR扩增、测序、数据处理及分析等, 关键实验步骤如图 1所示。

图 1 利用eDNA研究湖泊生物多样性的关键实验步骤Fig. 1 Key experimental steps for detecting lake biodiversity based on eDNA
2.1 eDNA在湖泊生物多样性研究中的样品采集和保存技术
在利用eDNA研究湖泊生物多样性时, 如何选择合适的样品采集量、采集方法和保存方式, 是保证样品质量的3个重要方面。采集湖泊水样品提取eDNA时, 每个样品采集量不等, 且通常需要至少采集3个重复。Thomsen等[16]通过在丹麦的湖泊、池塘和溪流中采集水样品, 检测到两栖类、鱼类、哺乳类、昆虫和甲壳类中的濒危物种DNA, 研究采用的采样方法是每个水样品采集15 mL, 并进行3次重复采样。Agersnap等[28]利用eDNA对丹麦、挪威和芬兰3个湖泊中的螯虾属(Astacus)四个分类单元(Astacus astacus,Pacifastacus leniusculu,Astacus leptodactylusclade I and III)进行了监测, 实验过程中采集的每个水样品量在500—1000 mL。Fujii等[29]从日本北海道31个回水湖泊记录有鱼类分布的水面各采集1000 mL水样品, 进行eDNA实验, 并将测序结果与传统采样方法获得的结果进行了比较,eDNA检出率超过50%。Muha等[30]比较了过滤15、100、250、1000和2000 mL水样品获得的eDNA产量, 利用12S rRNA短片段进行聚合酶链式反应(Polymerase Chain Reaction, PCR)和实时荧光定量PCR(Quantitative Real-time PCR, qPCR)扩增,结果显示2000 mL水样品获得的eDNA产量最多, 且相较于100和250 mL样品的qPCR扩增效率最高, 因此研究建议根据环境中颗粒物的尺寸来确定过滤膜孔径大小和可行的过滤水量, 尽可能多的采集水样品进行eDNA实验。为减少采样时的污染, 采样一定要佩戴口罩和手套, 并在采样前用10%漂白剂清洁所有用品和实验场地, 采样时用纯净水做阴性对照, 以监测是否存在污染[16,29]。
目前采集eDNA主要有3种方法: 过滤法、沉淀法和离心法。过滤法通过将样品进行抽滤, 使eDNA截留在滤膜上, 从而对样品中的eDNA进行浓缩和收集[31—34]; 沉淀法通过在样品中加入一定比例乙酸钠(CH3COONa)和无水乙醇(C2H5OH), 使eDNA发生聚集沉淀[16,35]; 离心法通过使用离心机将一定体积的样品进行离心处理, 使eDNA发生沉降, 从而达到浓缩和收集eDNA的目的[36]。Deiner等[37]利用过滤法和沉淀法从河流和湖泊中采集水样品, 比较后表明利用过滤法可获得更多的eDNA。Eichmiller等[27]比较了3种采样方法获得的鱼类DNA产量, 结果表明利用过滤法可获得更高的鱼类DNA产量, 其次是离心法, 沉淀法获得的鱼类DNA产量最低。虽然相关研究表明, 过滤的水量越多, 能获得的eDNA产量越高, 可检测出的物种多样性越高[30]。但如果水中的悬浮物或杂质过多, 很容易阻塞小孔径的滤膜, 科研人员尝试通过预过滤或增加滤膜数量的方法来解决这一问题。Ma等[38]在利用eDNA开展长江江豚种群监测实验过程中, 为防止水中悬浮物堵塞过滤孔, 先用灭菌过的医用纱布对水样品进行预过滤, 以保证足够的过滤水量。Hunter等[39]比较了用一张滤膜过滤200 mL水量获得的目标片段拷贝数, 和用4张滤膜过滤800 mL水量后合并提取DNA获得的目标片段拷贝数, 过滤过程中最大限度的避免了沉积物阻塞过滤孔, 结果表明用4张滤膜所得目标片段的拷贝数是用单一滤膜所得拷贝数的4.4倍, 表明在避免了过滤孔阻塞的情况下, 可获得更多的目标片段, 且分多张滤膜对同一体量的水样品进行过滤, 获得的过滤效果更好。
最近的eDNA研究更倾向于通过采集水样品,对湖泊中的生物多样性进行监测, 往往采集大量水样品进行过滤, 分析得到的物种种类更为精确, 采集量最多的已达到45 L水量, 以获得更多eDNA[40]。滤膜的孔径和材质会对过滤时间和eDNA浓度有一定影响, 目前的研究多采用玻璃纤维素滤膜(GF)、混合纤维素滤膜(MCE)、硝酸纤维素滤膜(NC)、尼龙滤膜(NL)、聚碳酸酯滤膜(CF)或聚醚砜滤膜(PES), 孔径在0.2—8.0 μm。Eichmiller等[27]比较了用1.5 μm GF和0.2、0.6、1.0及5.0 μm CF在过滤水样品后获得的鱼类DNA产量, 结果表明使用1.5 μm CF能获得最多的DNA产量。Li等[41]比较了利用不同孔径的固体封闭式GF研究池塘中鱼类种群结构的有效性和准确性, 结果表明0.45 μm GF表现最佳,可获得更多的eDNA产量; 但与0.8 μm GF相比, 需要更长的过滤时间; 而0.8 μm GF更适用于过滤浑浊、富营养化和鱼类密度高的水体。
选择适当的环境样品保存方式有助于减慢eDNA的降解速度, 提高eDNA提取效率, 获得更理想的实验结果。Renshaw等[42]研究了在eDNA采样和运输过程中不具备冷藏或冷冻条件, 可以使用哪些试剂进行样品保存, 结果表明用十六烷基三甲基溴化铵(CTAB)和Longmire’s两种裂解缓冲液在室温(20℃)可以保存eDNA两周以上。Hinlo等[43]比较研究了过滤后将滤膜放入100%酒精保存在室温环境和未加酒精保存在-20℃的eDNA保存效果, 提出与其他保存方式相比, 优先使用95%酒精保存的结论,这可以解决在野外用冰块很难将样品进行快速冷冻, 及使用CTAB或Longmire’s试剂不能成功保存DNA的问题, 且酒精的成本较低。Majaneva等[44]从湖泊和河流生态系统采集eDNA样品, 比较了低温冷冻保存(-20℃)、使用酒精或裂解缓冲液(Lysis buffer)和硅胶干燥等不同样品保存技术所获得的生物多样性, 结果显示干燥或使用lysis缓冲液获得一致的类群组成结果; 此外与PES相比, 利用MCE采样能获得更一致的结果。
2.2 湖泊环境样品中eDNA的提取
eDNA提取的过程是为了将环境样品中的DNA从完整的细胞和细胞器中释放出来, 并且能够有效去除PCR反应抑制物(如: 腐殖酸和腐殖质)[27]。由于环境样品中的生物复杂性高, 如何有效地提取DNA是科研人员面临的最大挑战[8]。eDNA提取过程中最需要关注2个方面, 一是获得最高的eDNA产物; 二是控制抑制成分, 这些成分可能是细胞复合物(如: 蛋白酶)或环境样品中的其他物质(如: 腐殖质)[45]。eDNA提取可以采取配制方法或商业试剂盒, 需针对不同研究对象采取合适的提取方法。早期从环境中提取古DNA, 多针对研究对象的不同,采用酚氯仿法(Phenol-chloroform method, PCI法)提取eDNA, 如Ochsenreiter等[46]利用eDNA研究高盐湖泊中古细菌多样性时, 在对样品进行过滤、经裂解液重悬和加入蛋白酶K后, 利用PCI法提取DNA。一直以来, 还是有很多eDNA的研究是采用PCI法提取DNA, 且也有研究表明利用PCI法从GF或NC提取的eDNA量比利用Qiagen DNeasy Blood and Tissue DNA extraction kit(简称DNeasy试剂盒)获得的更多, 且成本更低[27,42]。但因为PCI法会应用到苯酚和氯仿这一类有害物质, 因此在实验操作时要特别注意对试剂进行处理和实验废物的处置, 相比之下, 还是更推荐使用商业试剂盒进行eDNA提取[47]。
我们对湖泊eDNA多种提取方法进行了整理和比较(表 1)。尽管由于研究对象的不同, 不同试剂盒提取DNA的效率不尽相同, 但在湖泊eDNA研究方面还是有很多成功案例[16,48]。目前eDNA提取多采用DNeasy试剂盒、MoBio PowerWater DNA Extraction Kit(简称PowerWater试剂盒)、MoBio PowerSoil DNA Extraction Kit(简称PowerSoil试剂盒)、MP Biomedicals FastDNA SPIN Kit(简称Biomedicals试剂盒)、MP Biomedicals FastDNA SPIN Kit for soil(简称Biomedicals for soil试剂盒)、PowerMax Soil(简称PowerMax试剂盒)、Qiagen QIAamp DNA Stool Mini Kit(简称QIAamp试剂盒)和DNeasy mericon Food Kit(简称DNeasy mericon试剂盒)等。针对不同目标物种, 可选择不同的试剂盒, 达到更好的DNA提取效率。近些年, 多项研究都是针对哪种试剂盒提取什么样的eDNA样品可获得更高提取效率进行的。Deiner等[37]利用从河流和湖泊采集的水样品, 选择6套用于检测两栖动物和大型哺乳动物的eDNA采样和提取方案检测生物多样性, 结果表明对于静水环境, 利用过滤法获得eDNA样品并配合PCI法提取DNA, 可获得更高的eDNA产量; 沉淀法获得eDNA配合PowerWater试剂盒提取的eDNA产量最低; 从物种检出率看, 利用过滤法获得eDNA配合DNeasy试剂盒可以检测到更多的目标物种。Eichmiller等[27]在利用eDNA对鲤(Cyprinus carpio)进行监测和定量分析时, 比较了6种DNA提取试剂盒的效果, 对于井水和湖泊环境,利用Biomedicals试剂盒可以获得最多的鲤DNA, 但产量没有明显高于Biomedicals for Soil和DNeasy两个试剂盒; PowerSoil试剂盒对于井水和湖泊采集样品的提取效率是一致的, 适用于更广泛的生境类型;在综合分析了不同eDNA采集和提取方法后, 推荐使用1.5 μm GF并用Biomedicals试剂盒提取DNA;应用eDNA对鲤或其他鲤科鱼类进行定量分析时,建议使用0.2—0.6 μm CF, 使用PowerSoil试剂盒是最适宜的。Hinlo等[43]比较了通过不同材质滤膜过滤水样品, 经-20℃或酒精室温保存, 利用DNeasy和PowerWater两种试剂盒提取eDNA并进行qPCR分析, 结果表明无论哪种保存方式, 均是用DNeasy试剂盒提取DNA得到的结果更好。
环境样品中的抑制成分通常包括酚类化合物、腐殖酸和重金属, 更广泛的抑制成分还包括细菌细胞、非目标DNA和污染物等[45]。这些抑制成分会干扰DNA提取过程中的细胞裂解, 或与DNA一起被提取出来, 影响其提取纯度[8,45]。因此,是否产生抑制作用也是评价eDNA提取方法优劣的指标。Eichmiller等[27]对研究所用的6种试剂盒提取eDNA产生的抑制作用进行了分析, 结果表明PowerWater和Biomedicals试剂盒可观察到轻微的抑制作用, 并可通过将eDNA提取产物稀释两倍来缓解; DNeasy试剂盒产生最多的抑制作用, 只有将eDNA提取产物稀释五倍才能得到缓解。

表 1 湖泊样品中eDNA提取的常用方法Tab. 1 Common methods for extracting eDNA from lake samples
2.3 湖泊样品中的eDNA检测
回顾基于eDNA开展的湖泊生物多样性研究,可以将eDNA检测分为两大类: 一类是利用eDNA对特定物种或类群进行检测; 另一类是利用eDNA宏条形码技术(eDNA metabarcoding)对多种生物类群进行检测。
特定物种或类群研究eDNA技术一般被应用于湖泊中珍稀、濒危和外来物种的检测, 对于珍稀和濒危物种的研究包括对中国长江中下游的江豚(Neophocaena asiaeorientalis asiaeorientalis)[38],及欧洲丹麦、瑞典、德国、爱沙尼亚和波兰等地的池塘、水库和溪流中分布的棕色锄足蟾(Pelobates fuscus)、北冠毛蝾螈(Triturus cristatus)、水獭(Lutra lutra)、欧洲纵带泥鳅(Misgurnus fossilis)、胸大隐骨虫(Leucorrhinia pectoralis)和蓝带鱼(Lepidurus apus)等物种及其分布范围进行监测[16];外来物种的检测涉及法国西南部佩里戈尔-利穆赞自然公园池塘中的北美牛蛙(Lithobates catesbeianus)[57]、日本池塘中的蓝鳃翻车鱼(Lepomis macrochirus)[58,59]和美国五大湖地区的梅花鲈(Gymnocephalus cernua)[53]等。
对于特定物种或类群的eDNA检测主要通过设计特异性引物进行PCR, 利用目标片段进行物种鉴定[11]。一般用线粒体DNA片段开展湖泊生物检测,因为其含有较高的拷贝数且区分物种的保守序列较多; 由于核基因的敏感性不高, 因此很少使用核基因进行eDNA检测[60,61]。经常用到的线粒体基因包括Cytb、COⅠ、12S rDNA、16S rDNA、Dloop等, 片段大小在80—200 bp[16,37,62,63]。特异性引物可根据目标物种或类群的特定线粒体DNA序列来设计, 目的片段长度要适合高通量测序平台的要求, 需具有一定特异性, 能有效区分近缘种。常用的引物设计软件包括Primer Express 3.0.1、Primer premier 6.0和PrimerHunter等[13,28]。对湖泊中特定物种或类群开展检测时, 目标片段的扩增多采用PCR、qPCR和微滴式数字PCR(Droplet digital PCR,ddPCR)方法[51,64—66]。PCR比较传统和普遍地应用于eDNA研究, 可以对目标物种进行定性分析, 虽然可通过电泳条带的亮度推测eDNA浓度, 但并不精确且存在污染的可能性[47]。qPCR是具有更高特异性和敏感性的定量方法, 近些年已作为eDNA研究的主流方法, 应用于定量要求高的研究, 如评估生物多样性和分析种群密度等[16,58,67,68]。ddPCR是一种核酸分子绝对定量技术, 可直接推算eDNA目的片段的拷贝数; ddPCR的优点是DNA浓度和PCR抑制物不会影响DNA定量分析, 因此该方法可被用作qPCR的替代方法, 可用于eDNA定量分析[51]。
eDNA宏条形码随着HTS技术的发展, 宏条形码应运而生, 在生物多样性较为丰富的环境中,更适合开展eDNA宏条形码研究。DNA宏条形码可以更快速、准确和自动化地进行物种鉴定, 这一技术可同时分析一定环境中的生物多样性, 且更经济和简便, 相关技术方法也在不断改进和发展[16,69,70]。在进行eDNA宏条形码研究时, 通常利用生物类群的通用引物进行扩增, 这些通用引物通常被设计在线粒体或核基因DNA某一基因区段的保守区域内[11]。根据湖泊内不同的生物类群, 通用引物的选择有所不同: 针对微生物种群和群落的研究, 多选择16S rRNA基因设计引物; 对于底栖动物和浮游动物,COⅠ基因是更好的分类鉴定基因; 浮游植物则优先选择18S rDNA基因, 该基因具有更高的物种覆盖率及较完善的条形码数据库, 已被广泛应用于真核藻类的检测; 鱼类引物设计通常选用的基因片段为Cytb、12S rRNA或16S rRNA等线粒体基因, 其中12S rRNA基因被证实表现更为出色[47,71—75]。Zhang等[75]基于计算模拟PCR方法(Silico PCR)比较了22对硬骨鱼类的eDNA宏条形码通用引物, 并在北京水体中进行了应用, 结果表明不同引物在对不同生物类群的扩增范围和比例、检测鱼类物种丰富度和群落结构等方面均存在明显差异, 12S rRNA区段引物对鱼类多样性的扩增表现优于其他区段引物。但由于物种的个体差异、引物存在偏好性和测序深度等问题, eDNA宏条形码技术在开展物种精确定量方面还存在不足[73,74]。
eDNA样品扩增得到的PCR产物经等量混合和纯化后, 构建文库。目前, 湖泊eDNA研究中的文库构建均采用商业试剂盒进行, 常选用的建库试剂盒包括: NEBnext DNA Sample Prep Master Mix Set2(New England Biolabs, Ipswich, MA)[16]、Sequal-Prep Normalization Plate Kit (Invitrogen, Life Technologies)[76]和Agilent SureSelect Target Enrichment Kit[54]等。我们利用eDNA研究鄱阳湖鱼类物种多样性时, 选用的建库试剂盒是适用于Illumina测序平台的Illumina TruSeq DNA PCR-free LT Sample Prep kit。现在市场上可选择的建库试剂盒种类繁多, 如何选择是与测序平台的类型和所需数据深度密切相关的。已有研究中用到的高通量测序平台主要包括: Roche GS FLX 454、Illumina Miseq和Hiseq等[16,54,72,76]。在我们开展湖泊eDNA研究时,选用过Illumina HiSeq X Ten测序平台, 利用12S rRNA序列分析鱼类物种多样性。根据数据量的大小, 高通量测序平台一般被分为超高通量和中等通量测序, 如Roche GS FLX 454和Illumina Hiseq 2000、2500和HiSeq X Ten属于超高通量测序平台,适合大规模基因组学研究; Illumina Miseq是中等通量测序平台, 适合靶向和小型基因组测序。Illumina测序平台是目前最常被使用到的, 近期该公司又推出了介于Hiseq和Miseq之间的NextSeq 500测序仪, 可在高产量和中等产量2个模式下进行测序。
eDNA宏条形码技术依赖于成熟的DNA数据库, 可以将用于物种鉴定的靶序列与数据库中的序列进行比对来确定物种信息。如果DNA数据库中涵盖的信息较少或缺少某些物种的DNA信息, 靶序列通常聚类为分子可操作分类单元(Molecular Operational Taxonomic Units, MOTUs), MOTUs往往更容易鉴定到科和属级水平[11,47]。Deiner等[77]对利用eDNA宏条形码开展动植物群落调查进行了详细综述, 其中利用湖泊沉积物的古DNA和宏条形码检测植物多样性, 重建生物群落; 微生物宏条形码可用于检测真菌和细菌多样性。eDNA宏条形码或将成为监测动植物多样性的标准方法, 可结合传统调查方法快速开展生物多样性监测。
利用eDNA宏条形码进行物种鉴定时需要对测序序列进行生物信息学分析, 主要分析流程如图 2所示, 目前常用的分析软件包括PhyloPythia[78]、Mothur[79]、Cutadapt[80]、Usearch[81]、QIIME[82]和OBITools[75]等。通常进行eDNA宏条形码分析, 需要科研人员具有一定的序列分析和计算机编程能力。但最近新开发的一些HTS数据分析平台可以提供更友好、便捷的序列分析方案, 科研人员只需要在线提交数据就可以完成分析, 如MiFish Pipeline (http://mitofish.aori.u-tokyo.ac.jp/)鱼类多样性在线数据分析平台[47]。
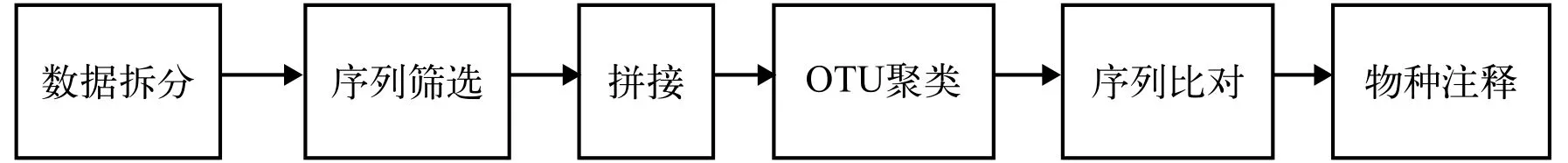
图 2 eDNA高通量测序序列主要分析步骤Fig. 2 Major analysis steps of eDNA high-throughput sequences
影响eDNA检测的因素基于eDNA的原理,eDNA是否能被检测到是与其释放、扩散和降解有着密切关系[34,83,84]。生物和环境条件, 即水温、pH、微生物活性、生物量和生物活动等, 是影响eDNA释放和降解的重要因素。Thomse等[16]在水族馆和实验池塘条件下, 以鲤(Cyprinus carpio)为研究对象, 分析了水温、生物量和鲤的活动与eDNA释放量和浓度之间的关系, 结果表明eDNA释放量会随鲤活动的增加而增加; 在水族馆条件下, 水温的变化不会影响eDNA浓度; 在水族馆和实验池塘条件下, 鲤的生物量与eDNA浓度呈正相关。Klymus等[36]在实验条件下, 以鲢(Hypophthalmichthys molitrix)和鳙(Hypophthalmichthys nobilis)为研究对象,研究了水温、生物量和摄食如何影响eDNA释放率, 结果表明生物量和摄食与eDNA释放率存在正相关, 但水温对其没有明显影响。Lacoursiere-Roussel等[85]比较了在7和14℃水温条件下鱼类eDNA释放量, 结果表明温水中的释放量高于冷水。Lacoursiere-Roussel等[86]对采自加拿大魁北克12个自然湖泊的水样品进行了eDNA检测, 分析了水温、溶氧、pH、污浊度、生物量与eDNA浓度之间的关系, 结果表明eDNA浓度与几个环境指标没有明显相关性, 但与生物量有正相关性。Tsuji等[87]研究了水中pH高低对eDNA过滤量的影响, 结果表明: pH在5—9, eDNA浓度与pH呈负相关, 在酸性条件下eDNA量相对较高。Jo等[88]利用实验鱼缸研究了水温和鱼类生物量对eDNA释放、降解率和片段大小的影响, 结果表明水温越高、生物量越大,eDNA释放率越高, 但降解率也随之升高; 将鱼从鱼缸中取走, 大于10 μm的eDNA片段会减少, 但更小的片段会增加。
eDNA在水中的降解率也与目的片段大小存在密切关系。Bylemans等[89]利用实验鱼缸研究了水中鱼类不同长度线粒体eDNA和核eDNA短片段的降解情况, 结果表明在有鱼的情况下, 线粒体eDNA短片段的丰富度高于长片段和核eDNA短片段; 在鱼被取走的情况下, 各种片段的降解表现出相似的情况。Jo等[90]比较了eDNA长片段(719 bp)和短片段(127 bp)拷贝数的时间变化, 结果显示长片段相较于短片段更容易降解, 但长片段的浓度可以更好地被用于生物量的估算。
3 eDNA应用于湖泊生物多样性研究面临的挑战及前景展望
eDNA作为一个新兴领域, 虽然相较传统生物多样性调查方法具有很多优势, 但也因为其样品的复杂性和对实验操作的严格性和数据分析的困难性, 使得这一研究仍存在很多技术性问题需要我们在未来去解决。
3.1 最佳实验方案的选择
通过回顾自20世纪90年代以来eDNA在湖泊生物多样性方面的研究, 选择效果最佳、最为经济、简便的样品采集和eDNA提取方案一直是科研人员的关注焦点, 且这一趋势在近年表现得尤其突出。随着eDNA技术的发展和更多不同国家科研人员的加入, 可选择的采样和实验方案越来越多, 且正在不断更新和发展。因此针对湖泊这一生态环境和不同目标类群, 如何选择和开发最佳采样和实验方案将是未来该领域发展的一个重要趋势。
3.2 污染
污染是eDNA研究面临的最大挑战, 其可能发生在采样、DNA提取、PCR扩增等各个环节, 造成假阳性结果的出现。避免采样环境和实验室污染发生, 可以利用10%漂白剂和75%酒精进行清洁, 并注意操作人员佩戴口罩和手套。但是在实验过程中, 由于试剂药品和操作过程中造成的交叉污染几乎是不能避免的, 或多或少会对实验结果造成一定误差, 特别是通过HTS测序, 很容易得到少量由于交叉污染产生的片段。针对这一问题, 在研究的各个环节都需要设置阴性对照(空白对照), 监控污染情况。另外, 开发一些新的采样技术也是降低甚至避免污染的有效途径。目前已有研究尝试利用无人机技术开展eDNA采样[91], 使得交叉污染基本被避免。
3.3 抑制反应
环境样品中包含的腐殖酸、腐殖质和有机复合物等抑制因子在eDNA提取过程中被一同提取出来, 将对后续PCR产生一定抑制作用。且像湖泊这样相对静止的环境, eDNA中更容易产生这些抑制因子, 影响实验的顺利开展。eDNA研究快速发展的这二十年中, 科研人员也发现了抑制因子的存在,针对这一问题摸索出一些降低抑制反应的方法, 如利用稀释或纯化等方法去除抑制因子[49,92], 或在PCR过程中添加BSA、RSA和Tween 200等反应加强剂[93,94]。但随着eDNA研究中环境样品量不断增加的趋势, 如何降低抑制反应的发生仍然是该领域未来重要的研究方向。
3.4 误差和错误
误差和错误的产生除了由于实验过程中发生交叉污染, eDNA样品的长期保存、PCR和测序过程中都有可能产生误差或错误。PCR导致的错误主要是由于嵌合子、异源双链分子的形成和TaqDNA聚合酶错误[95]。测序过程中也有更多机会产生错误。针对这一问题, 通过对测序所得原始序列进行过滤和去除假阳性等方法可以在一定程度上降低误差和错误的发生。一方面, 开发高效的扩增引物并发展高通量测序序列分析方法将是未来eDNA研究中减少错误和误差发生的重要趋势; 另一方面, eDNA技术可结合传统调查方法, 用于监测湖泊生物多样性, 有利于更为高效准确地开展生态调查。
3.5 DNA分类数据库的完善
eDNA研究特别是eDNA宏条形码研究离不开DNA分类数据库, 一个好的参考数据库, 可以保证分类鉴定结果的准确性, 是物种鉴定重要的参考系。NCBI数据库(http://www.ncbi.nlm.nih.gov/genbank)正在快速发展和完善, 一些针对特定类群建立的DNA数据库也在不断出现(如: 世界鱼类world’s fish, www.fishbol.org; 哺乳动物DNA数据库www.mammaliabol.org和鸟类条形码数据库www.barcodingbirds.org)[11]。尽管如此, 这些国际数据库中的分类学数据仍存在很大欠缺, 特别是中国生物类群的相关数据。作者团队在利用eDNA研究中国洞穴鱼类多样性过程中就发现, NCBI数据库中记录的洞穴鱼类DNA条形码数据仅占到所有洞穴鱼类不到一半。这就需要科研人员在这方面投入更多研究, 对数据库进行不断完善的同时, 也要增强数据库的准确性, 能够使每条序列与对应标本匹配。建议涉及对特定湖泊生物多样性开展长期性监测, 可考虑建立地区性数据库, 有针对性地整理DNA分类数据; 通过不同数据库跨库间的比对筛选, 可识别出存在鉴定问题的物种, 再对数据库进行针对性改善; 加强各实验室数据库共享。
