基于MAGIC群体的水稻镉含量全基因组关联分析
2022-01-18董铮王雅美黎用朝熊海波薛灿辉潘孝武刘文强魏秀彩李小湘
董铮 王雅美 黎用朝 熊海波 薛灿辉 潘孝武 刘文强 魏秀彩 李小湘, *
基于MAGIC群体的水稻镉含量全基因组关联分析
董铮1, 2, #王雅美3, #黎用朝1, 2熊海波1, 2薛灿辉1潘孝武1, 2刘文强1, 2魏秀彩1, 2李小湘1, 2, *
(1湖南省农业科学院 水稻研究所, 长沙 410125;2农业部长江中下游籼稻遗传育种重点实验室, 长沙 410125;3中国农业科学院 农业基因组研究所, 深圳 518120;#共同第一作者;*通信联系人, E-mail: xiaoxiang66196@126.com)
【】基于水稻MAGIC-Hei(Multi-parent advanced generation inter-cross)群体,在多环境下挖掘镉含量相关新位点/基因,并筛选含有低镉等位基因的优良株系,为选育低镉积累品种提供新的基因和种质资源。将由8个亲本衍生的MAGIC群体分别于2017、2018、2019和2020年度种植于湖南长沙并开展稻米镉含量表型测试分析。利用GBS(genotyping by sequencing)简化基因组测序获得基因型数据,对稻米镉含量开展全基因组关联分析(genome-wide association analysis,GWAS),发掘QTL位点,解析其遗传机制。检测到了14个镉积累相关的QTL位点,除了第8染色体之外,其他11条染色体上均有分布。其中6个位点与已报道基因一致,8个为新发现位点。另外,这8个位点分布在第2、4、7、9和12染色体上,均可以在两个及以上环境中检测到,效应较为稳定,可用于下一步精细定位及功能研究。结合基因注释和基因表达分析结果,推测、、和为镉含量相关位点候选基因,这些基因与重金属转运和积累等功能相关。另外,我们筛选到10个携带有利等位基因的优良株系,可用于低镉积累水稻材料的创制。发掘了8个水稻镉积累相关性状的QTL位点和低镉优异材料,对于镉积累相关遗传研究和利用分子标记辅助选育低镉积累品种具有一定意义。
MAGIC;QTL;镉含量;分子标记辅助选择育种;候选基因
镉(Cd)元素可通过食物在人体内富集,严重危害人类身体健康。镉含量超标会引起剧烈的关节疼痛以及严重的脊柱变形[1-2]。水稻是我国最为重要的粮食作物之一,镉被水稻根系吸收后富集在籽粒中,严重影响粮食安全生产[3]。我国西南和中南地区是水稻的主要种植区域,也是我国重要的矿产基地,采矿排放的大量污水导致当地水稻籽粒中镉含量较高[4]。镉在水稻中的积累是个极其复杂的生物学过程,受到一系列相关基因的共同调控,不同的水稻品种对镉的吸收和积累能力也有很大差异[5-6]。当前,针对镉积累相关性状已开展一系列遗传研究,并利用连锁和关联分析的方式检测到一批遗传位点[6-7]。Kashiwagi等[8]利用Kasalath和日本晴构建的回交群体检测到了3个与水稻地上部镉积累相关的QTL。Huang等[9]通过协青早B和东乡野生稻构建回交重组自交系群体筛选出了一批镉相关QTL,其中13个仅在糙米中被发现,多数位点的效应较低且表现不稳定。
选育镉低积累水稻品种是减轻镉危害最为经济、绿色的途径。目前对低镉水稻的遗传机制与设计育种的研究无法满足当前低镉育种的迫切需要。因此,发掘新的镉含量相关基因,对于选育低镉积累品种具有重要意义。镉含量是一个典型的数量遗传性状,受多个微效基因控制[5-7]。基于双亲群体的传统连锁分析是水稻基因挖掘的经典手段,该方法具有遗传背景稳定、作图精准等优点,但同时也面临群体构建耗时长、工作量大、遗传背景单一等弊端,且不能充分地反映自然界的遗传多样性[10-11]。近年来,基于连锁不平衡(Linkage disequilibrium, LD)的全基因组关联分析(Genome-wide association analysis,GWAS)已广泛应用于水稻数量遗传性状基因挖掘研究中,与连锁分析相比,GWAS具有耗时短、成本低和遗传信息来源广泛等优点,但同时存在关联位点假阳性高和等位变异检出率低等缺点[10-11]。而多亲本杂交和多代互交的MAGIC群体则有效聚合了常规连锁分析和GWAS分析的优势,遗传背景丰富,在水稻复杂农艺性状基因挖掘中已成熟应用,在农艺性状、产量性状和耐逆相关性状遗传研究中起着重要作用[12-14]。
目前,部分镉积累相关基因已被克隆,如、、和等[6-7]。(Heavy metal ATPase 3)编码水稻重金属ATP酶,定位于根部液泡膜上,是Cd2+的重要转运蛋白,可通过将Cd2+转运至液泡来减少Cd2+向其他组织的运输[15]。(Cd accumulation in leaf 1)编码植物类防御素蛋白,主要在根外皮层和木质部薄壁细胞表达,可通过对Cd2+的螯合作用来调节水稻叶片中的镉含量[16]。属于MFS(Major facilitator superfamily)类蛋白,主要在根部表达,可通过调节对Cd2+的转运影响镉在水稻籽粒中的积累,的V449D突变是造成一些籼稻和粳稻品种之间籽粒镉含量不同的主要原因[17]。耐镉基因(Cd tolerance 1)定位在细胞核中,是硫酸盐/硒酸盐吸收和同化的负调控因子,可通过调控根和地上部半胱氨酸和谷胱甘肽等化合物的含量来影响水稻的耐镉性[18]。
有关重金属镉吸收、转运以及积累的分子机制的探索为筛选水稻中的低镉品种奠定了理论基础。然而,水稻中有关镉积累的基因报道还很少,限制了人们对低镉积累水稻品种的选育以及创制。因此,亟需发掘水稻镉含量相关新的基因和位点。本研究以MAGIC-Hei群体为材料[13],分别在2017、2018、2019和2020年种植在湖南省长沙市,利用GWAS发掘水稻镉含量关联位点并挖掘其候选基因,筛选低镉含量优质株系,为水稻镉积累遗传机制研究及低镉水稻育种提供新的基因位点和种质资源。
1 材料与方法
1.1 实验材料和种植方法
MAGIC-Hei群体由Fedearroz 50, Shan-Huang Zhan-2(SHZ-2), IR64633-87-2-2-3-3(PSBRc82), IR4630-22-2-5-1-3,IR45427-2B-2-2B-1-1,IR 84196- 12-32(SAMBHA MAHSURI+SUB1),IR77298-14- 1-2-10和IR77186-122-2-2-3(PSBRc158)共8个亲本经多年多次杂交组合而成,含有395个株系。将该群体及亲本分别于2017、2018、2019和2020年度在湖南省水稻研究所马坡岭试验田种植。试验田四周和区组间开深沟,田面平整,保障田面水的深浅和土壤含水量基本一致。种子催芽开始25 d后移栽至田中,每份材料种植30株,均匀分成3行,株距为20.0 cm×26.5 cm,并设置两次生物学重复。田间肥料管理和晒田前的水分管理按照当地一季晚稻常规种植管理技术进行,并及时开展病虫鸟害防治;分蘖末期晒田后灌浅水,自然干裂后再灌浅水,至收种前田间保持干干湿湿。
1.2 镉含量测定方法
待种子黄熟时收种,收获稻谷自然晒干清选后加工成精米,用高速粉碎机粉碎成粉末。粉碎样品经纯硝酸和高氯酸混合溶液(4∶1)消化后,用原子吸收光谱仪(Thermo Fisher iCE3500)测定样品的镉浓度,单位为mg/kg。
1.3 基因型分析
使用CTAB法提取新鲜叶片中的DNA,利用限制性内切酶K I(NEB)对基因组DNA进行酶切,采用IluminaHiseq测序平台进行双末端测序,以日本晴基因组为参考基因组,通过GBS方式获得MAGIC-Hei群体基因型。MAGIC-Hei群体基因型数据之前已有报道[12-14]。
1.4 数据分析
用SAS 9.0对镉含量开展基本统计分析和差异显著性分析(<0.05)。基于IciMapping 4.3开展4个年度的BLUP(Best linear unbiased predictors)值计算,并作为后续分析表型值。
1.5 关联分析
采用基于R语言的GAPIT内置的MLM(Mixed linear model)模型对MAGIC-Hei群体镉含量进行GWAS分析[19],协变量为主成分分析(PCA)和亲缘关系(K)。阈值选择<1.0×10−3筛选与镉含量显著关联的标记。关联分析的可视化结果由基于R语言的CMplot(R 3.2.2)软件包绘制曼哈顿图(Manhattan Plot)和Q-Q图(Quantile-quantile plot)。2代表该位点所解释的表型变异率。
1.6 候选基因挖掘和表达分析
参考日本晴数据库基因注释的结果,本研究在新发现的重复性较好的8个位点附近进行基因筛选,挖掘镉积累相关的未克隆基因作为候选。采用TRIzol法提取总RNA,根据浓度值确定RNA模板加样量。参照Thermo Scientific公司提供的反转录试剂盒(RevertAid First Strand cDNA Synthesis Kit)反转合成20 μL cDNA,并将cDNA模板用无菌双蒸水稀释5~10倍,配制qPCR体系,使用2法进行基因表达水平数据分析。每个样品设置3次重复,以、和管家基因为内参基因对基因表达数据进行归一化分析,计算候选基因的相对表达量。
2 结果与分析
2.1 MAGIC-Hei群体镉含量分析
对2017−2020连续4年的MAGIC-Hei群体株系镉含量进行分析(表1、图1)。2017年8个亲本镉积含量变幅为0.514~1.412 mg/kg,群体均值为0.651 mg/kg,群体变幅为0.148~1.336 mg/kg;在2018年环境下,亲本镉含量变幅为0.579~1.667 mg/kg,群体镉含量平均值为0.672 mg/kg,群体变幅范围为0.068~1.516 mg/kg;在2019年环境下,亲本镉含量变幅为0.291~0.823 mg/kg,群体镉含量平均值为0.327 mg/kg,群体变幅为0.070~1.188 mg/kg;在2020年环境下,亲本镉含量变幅为0.286~0.903 mg/kg,群体镉含量平均值为0.534 mg/kg,群体变幅为0.192~1.125 mg/kg。以上结果表明,镉含量为典型的数量遗传性状,在MAGIC- Hei亲本及株系间差异广泛。

表1 MAGIC-Hei群体在不同环境下镉含量表型分析
数值后不同小写字母表示差异显著(﹤0.01)。
Different lowercase letters indicate significant differences(﹤0.01).
A, Fedearroz 50; B, Shan-Huang Zhan-2 (SHZ-2); C, R64633-87-2-2-3-3 (PSBRc82); D, IR4630-22-2-5-1-3; E, IR45427-2B-2-2B-1-1; F, IR 84196-12-32 (SAMBHA MAHSURI+SUB1); G, IR77298-14-1-2-10; H, IR77186-122-2-2-3 (PSBRc 158).
2.2 镉含量关联分析及优良株系的筛选
基于MLM模型,对MAGIC-Hei群体开展镉含量GWAS分析,在2017、2018、2019和2020环境下共计检测到14个镉含量相关位点,分别位于除第8染色体外的其他11条染色体上,可解释6.62%~10.57%的表型变异(表2、图2~3)。其中,、、、、、、和在两个及以上环境中均可检测到。基于镉含量表型数据,结合基因型数据,筛选到10个携带多个有利等位基因的低镉积累优良株系(镉含量<0.40 mg/kg且优异等位基因数目>10),这些株系可用于下一步的育种工作。
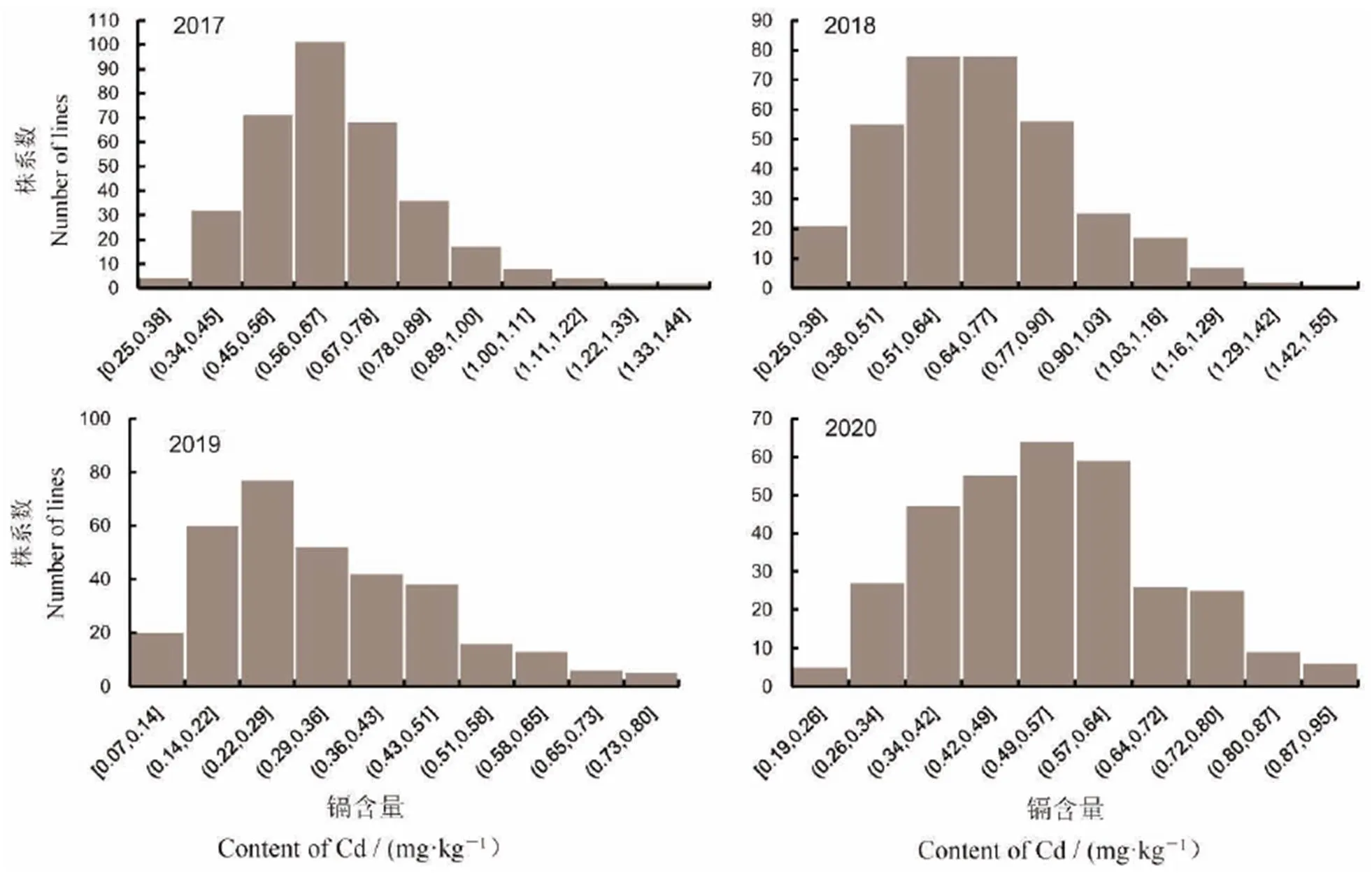
图1 MAGIC-Hei群体镉含量的分布
Fig. 1. Cadmium content distribution in MAGIC-Hei population.
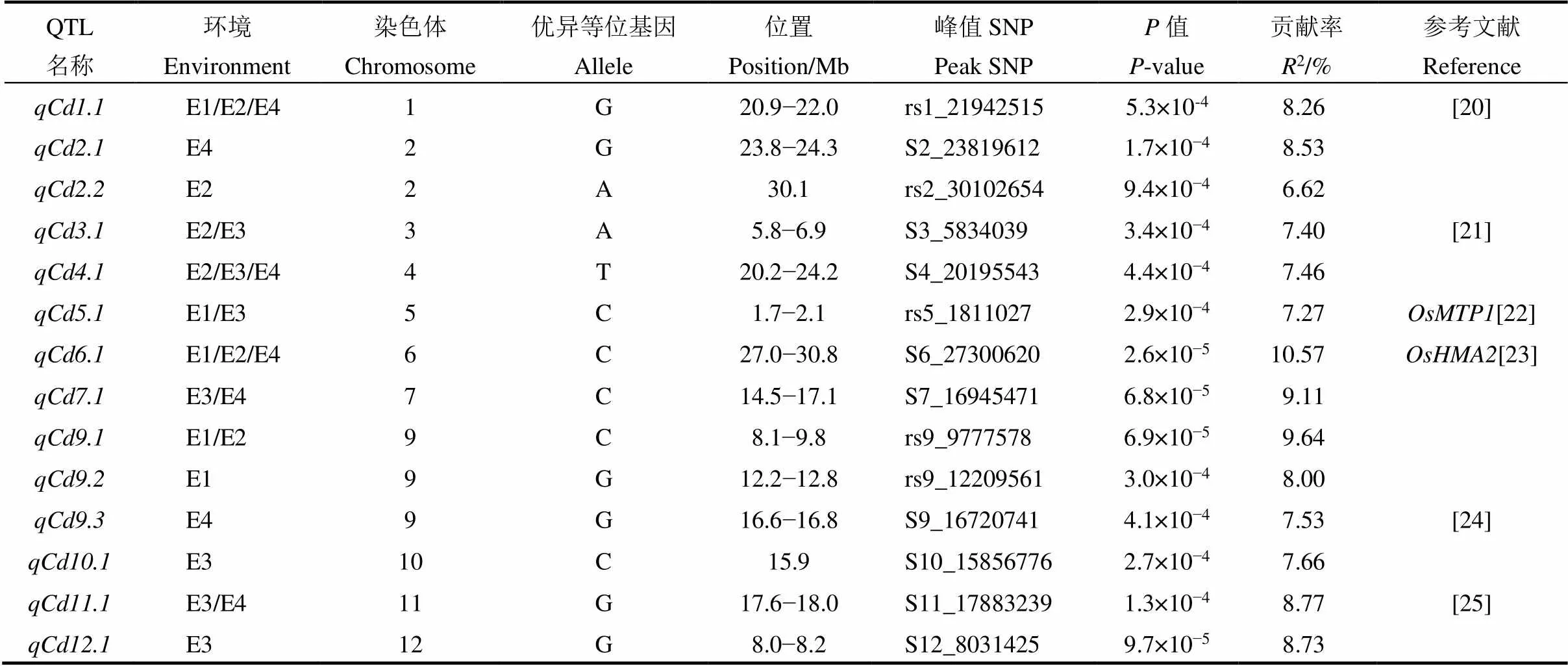
表2 MAGIC-Hei群体中通过全基因组关联分析检测到与镉积累显著关联的位点
E1−E4代表不同环境,分别代表2017, 2018, 2019和2020年。
E1−E4 indicated the various environments of 2017, 2018, 2019 and 2020, respectively.
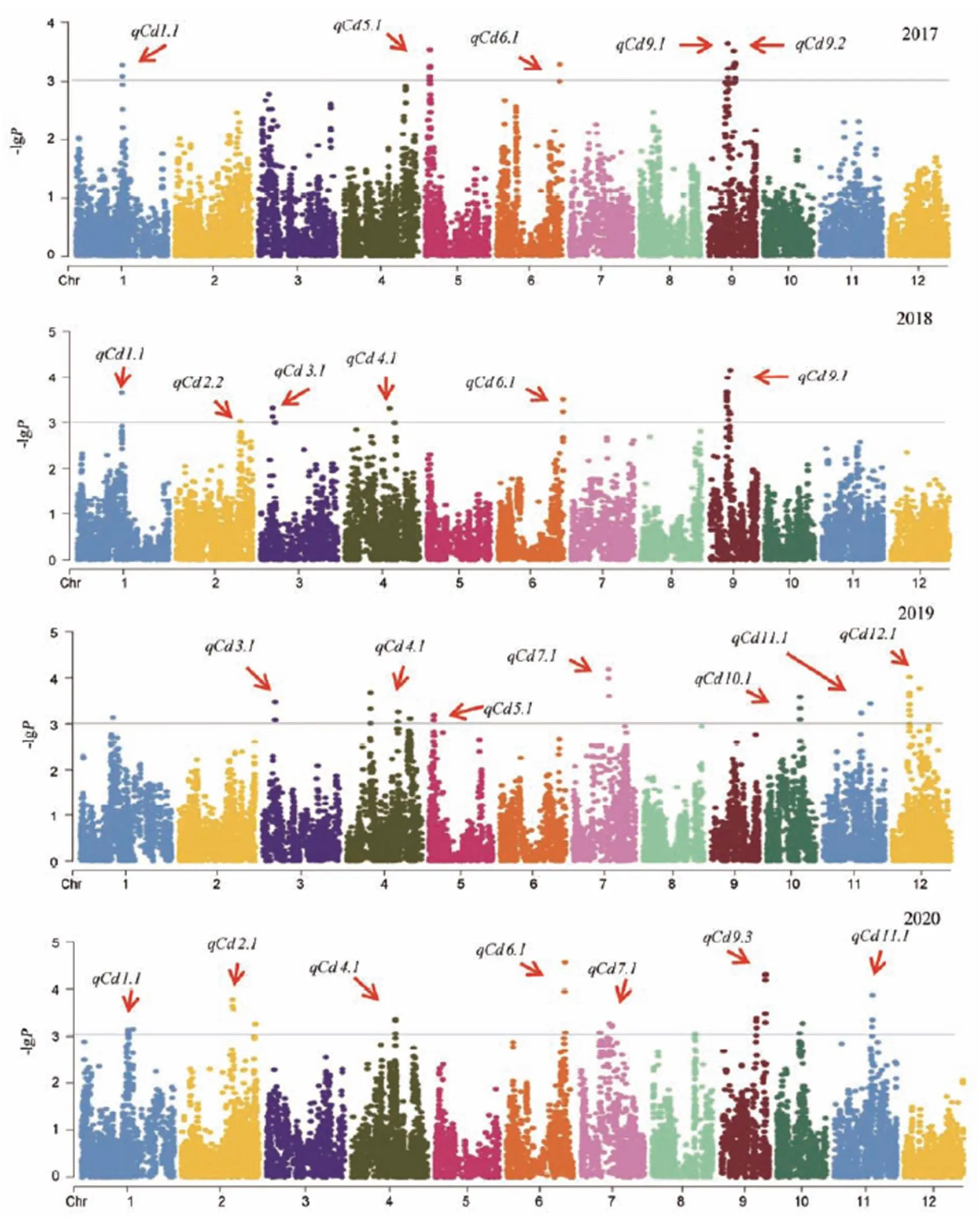
图2 MAGIC-Hei群体镉含量的曼哈顿散点图
Fig. 2. Manhattan plot of Cadmium content in MAGIC-Hei.
2.3 候选基因注释及表达量分析
结合日本晴基因组注释信息筛选在重要位点筛选到14个候选基因。采用qRT-PCR对上述基因进行表达模式验证。结果表明,有4个基因在高镉亲本和低镉亲本中的表达量呈现显著差异(表3、图4)。其中和在高镉亲本中的表达水平显著高于在低镉亲本的表达情况;这个结果说明和的转录水平随着水稻体内的镉含量的增加而升高(图4-A、B)。和在高镉亲本中的表达水平则显著低于在低镉亲本中,表明这两个基因可能参与了水稻中镉含量低积累的调控过程(图4-C、D)。
图3 MAGIC-Hei群体镉含量的Q-Q散点图
Fig. 3. Q-Q plot of cadmium content in MAGIC-Hei.

A, Fedearroz 50; B, IR 84196-12-32 (SAMBHA MAHSURI+SUB1); C, R64633-87-2-2-3-3 (PSBRc82); D, IR4630-22-2-5-1-3; E, IR45427-2B-2-2B-1-1; F, Shan-Huang Zhan-2 (SHZ-2); G, IR77298-14-1-2-10; H, IR77186-122-2-2-3(PSBRc 158).
A~D为高镉亲本,E~F为低镉亲本。内参基因为-Actin。*表示在<0.05水平上显著相关(≥3)。
A-D are high Cd content parents, E-F are low Cd content parents. The internal reference gene is. *Significant correlation at<0.05 (≥3).
图4 候选基因在MAGIC-Hei不同亲本间的表达量差异
Fig. 4. Expression difference of candidate genes among different parents in MAGIC-Hei.

表3 水稻镉含量相关候选基因
3 讨论
目前,我国糙米中镉含量安全标准为0.2 mg/kg,在生产过程中,田间轻微的镉污染就会造成稻米镉超标。而低镉品种的大田生产效应在一定程度上不如水分管理和pH值调控见效快,在很长一段时间内得不到有效重视和推广[26]。但随着水稻安全生产越来越被重视及水稻镉积累鉴定的田间种植管理越来越规范,人们发现水稻品种资源之间吸收积累镉量差异很大;低镉品种的种植不会增加生产成本,不需田间一直保持有水层而浪费水资源,不用调整稻米生产工艺,也不造成田间二次污染而更利于可持续发展。同时水稻重测序和GWAS分析等生物学技术的快速发展,更是加快了镉含量等数量性状遗传解析的进程[10]。这些都有助于低镉基因的挖掘,为低镉品种的选育和推广奠定基础。
本研究基于MAGIC-Hei群体和GBS测序数据,在多环境条件下,尝试通过GWAS对水稻籽粒镉含量进行遗传解析,发现了14个与镉含量显著关联的位点,8个新发现的位点都能在两个及以上环境中检测到,另外6个位点与已报道过的基因或位点一致。其中,和与已克隆基因一致:位点内存在一个已克隆的镉转运相关基因(Metal tolerance protein 1),OsMTP1定位在液泡上,为CDF(Cation diffusion facilitato)家族重要成员,影响水稻对Zn2+和Cd2+的耐受性[22, 27];位点内存在(Heavy metal ATPase 2)基因,该基因与同源,OsHMA2在水稻植株Zn2+和Cd2+由地下部分向地上部分转运过程中发挥重要作用,过表达可有效降低水稻籽粒镉含量[23]。与Xu等[20]利用两批导入系材料在水稻第1染色体上检测到的一个与Zn2+、Cd2+和Fe2+转运的相关位点一致;与Zhang等[21]利用连锁分析在水稻第3染色体上发现的位点接近;与Yan等[24]在水稻第9染色体上检测到的一个与Ca2+和Cd2+转运相关的位点一致;与陈志德[25]在第11染色体上检测到的一个Cd2+转运相关位点接近。这些位点经研究和验证,存在镉含量相关基因的可能性较大,但尚未克隆,其遗传调控机制还有待研究。另外,前人针对水稻籽粒镉含量也开展了大量研究,在水稻第1、2、3、4、6、7、9和11染色体上发现多个镉含量相关遗传位点,由于采用的标记为传统的RFLP、SSR或者DArT标记,无法与现有SNP或InDel标记进行可靠的对比,暂时无法确定是否与本研究发现位点一致。
综合注释信息和表达模式验证结果,我们在关联位点区间内鉴定到4个候选基因,其中,和基因的表达量在正常生长的高镉积累水稻中要高于在低镉材料中的情况,和的转录水平则在低镉积累水稻中显著高于在高镉积累水稻中的情况。编码重金属转运蛋白,编码bZIP转录因子,编码重金属相关结构域蛋白,编码重金属转运因子Nramp6。镉元素在水稻籽粒中的积累主要是通过根系吸收,随后通过木质部的装载和运输转移到地上部,再经过韧皮部的运输和转移积累到籽粒中。已有研究表明,NRAMP蛋白、重金属ATP酶和锌铁转运蛋白(Zinc transporter,ZIP)家族参与了镉吸收和积累过程[6-7, 28]。NRAMP家族中的OsNramp5已被证明参与了Mn2+、Fe2+和Cd2+在凯氏带和木质部维管束之间的转运,缺失OsNramp5在降低镉积累的同时,也显著降低了水稻生长发育水平和籽粒产量[6][7]。重金属转运蛋白可将由根吸收到的Cd2+封存在根部细胞液泡中,减少了Cd2+从木质部向地上部的运输,降低了籽粒中的镉含量[15]。
本研究发现了数个在多环境下存在的镉含量关联位点,其中、、、、、、和相对更为可靠,可进一步转化为KASP或STARP标记,用于分子标记辅助育种和QTL精细定位。10个株系在多环境重复检测过程中具有较低的镉含量(<0.40 mg/kg),且含有较多优异等位基因(>10个),可作为亲本以导入或聚合优异等位基因,降低育种材料的镉含量,为低镉水稻MAS育种做出了贡献。
谢辞:感谢国际水稻研究所叶国友博士提供MAGIC-Hei群体资源及其基因型。
[1] Canli M, Furness R W. Toxicity of heavy metals dissolved in sea water and influences of sex and size on metal accumulation and tissue distribution in the norway lobster[J]., 1993, 36(4): 217-236.
[2] Grant C A, Clarke J M, Duguid S, Chaney R L. Selection and breeding of plant cultivars to minimize cadmium accumulation[J]., 2008, 390(2/3): 301-310.
[3] Yao H, Xu J, Huang C. Substrate utilization pattern, biomass and activity of microbial communities in a sequence of heavy metal-polluted paddy soils[J]., 2003, 115(1): 139-148.
[4] Li Z, Ma Z, der Kuijp T J, Yuan Z, Huang L. A review of soil heavy metal pollution from mines in China: Pollution and health risk assessment[J]., 2014, 468-469: 843-853.
[5] Clemens S, Aarts M G, Thomine S, Verbruggen N. Plantscience: The key to preventing slow cadmium poisoning[J]., 2013, 18(2): 92-99.
[6] 丁仕林, 刘朝雷, 钱前, 高振宇. 水稻重金属镉吸收和转运的分子遗传机制研究进展[J]. 中国水稻科学, 2019, 33(5): 383-390.
Ding S L, Liu C L, Qian Q, Gao Z Y. Research advances on molecular genetic mechanism for Cadmium absorption and transportation in rice[J]., 2019, 33(5): 383-390. (in Chinese with English abstract)
[7] UraguchiS, Fujiwara T. Cadmium transport and tolerance in rice: Perspectives for reducing grain cadmium accumulation[J]., 2012, 5(1): 1-8.
[8] Kashiwagi T, Shindoh K, Hirotsu N, Ishimaru K. Evidence for separate translocation pathways in determining cadmium accumulation in grain and aerial plant parts in rice[J]., 2009, 9(8): 1-10.
[9] Huang D R, Fan Y Y, Hu B L, Xiao Q Y, Chen D Z, Zhuang J Y. Assessment and genetic analysis of heavy metal content in rice grain using an×backcross inbred line population[J]., 2018, 98(4): 1339-1345.
[10] Zhao K, Tung C, Eizenga G C, Wright M H, Ali M L, Price A H, Norton G J, Islam M R, Reynolds A, Mezey J, McClung A M, Bustamante C D, McCouch S R. Genome-wide association mapping reveals a rich genetic architecture of complex traits inL[J]., 2011, 2(1): 467.
[11] Liu J D, He Z H, Rasheed A, Wen W E, Yan J, Zhang P Z, Wan Y X, Xie C J, Xia X C. Genome-wide association mapping of black point reaction in common wheat (L.)[J]., 2017, 17(1): 220-232.
[12] Elshire R J, Glaubitz J C, Sun Q, Poland J A, Kawamoto K, Buckler E S, Mitchell S E. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species[J]., 2011, 6(5): e19379.
[13] Bandillo N, Raghavan C, Muyco P A, Sevilla M A L, Lobina I T, Dilla-Ermita C J, Tung C W, McCouch S, Thomson M, Mauleon R, Singh R K, Gregorio G, Redoña E, Leung H. Multi-parent advanced generation inter-cross (MAGIC) populations in rice: Progress and potential for genetics research and breeding[J]., 2013, 6(1): 1-15.
[14] Raghavan C, Mauleon R, Lacorte V, Jubay M, Zaw H, Bonifacio J, Singh R K, Huang B E, Leung H. Approaches in characterizing genetic structure and mapping in a rice multiparental population[J]., 2017, 7(6): 1721-1730.
[15] MiyadateH, Adachi S, Hiraizumi A, Tezuka K, Nakazawa N, Kawamoto T, Katou K, Kodama I, Sakurai K, Takahashi H., a P1B-type of ATPase affects root-to-shoot cadmium translocation in rice by mediating efflux into vacuoles[J]., 2011, 189(1): 190-199.
[16] Luo J, Huang J, Zeng D, Peng J S, Zhang G B, Ma H L, Guan Y, Yi H Y, Fu Y L, Lin H X, Qian Q, Gong J M. A defensin-like protein drives cadmium efflux and allocation in rice[J]., 2018, 9(1): 645.
[17] Yan H, Xu W, Xie J, Gao Y W, Wu L L, Sun L, Feng L, Chen X, Zhang T, Dai C H, Li T, Lin X N, Zhang Z Y, Wang X Q, Li F M, Zhu X Y, Li J J, Li Z C, Chen C Y, Ma M, Zhang H L, He Z Y. Variation of a major facilitator superfamily gene contributes to differential cadmium accumulation between rice subspecies[J]., 2019, 10(1):1-12.
[18] Chen J, Huang X, Salt D E, Zhao F J. Mutation inenhances cadmium tolerance and enriches selenium in rice grain[J]., 2020, 226(3): 838-850.
[19] Tang Y, Liu X, Wang J, Li M, Wang Q, Tian F, Su Z, Pan Y, Liu D, Lipka A E, Buckler E S, Zhang Z. GAPIT Version 2: An enhanced integrated tool for genomic association and prediction[J]., 2016 9(2): 1-9.
[20] Xu Q, Zheng T Q, Hu X, Cheng L R, Xu J L, Shi Y M, Li Z K. Examining two sets of introgression lines in rice (L.) reveals favorable alleles that improve grain Zn and Fe concentrations[J]., 2015, 10(7): e0131846
[21] Zhang M, Pinson S R M, Tarpley L, Huang X Y, Lahner B, Yakubova E, Baxter I, Guerinot M L, Salt D E. Mapping and validation of quantitative trait loci associated with concentrations of 16 elements in unmilled rice grain[J]., 2014, 127(1): 137-165.
[22] Yuan L, Yang S, Liu B, Zhang M, Wu K. Molecular characterization of a rice metal tolerance protein, OsMTP1[J]., 2012, 31(1): 67-79
[23] Takahashi R, Ishimaru Y, Shimo H, Ogo Y, Senoura T, Nishizawa N K, Nakanishi H. Thetransporter is involved in root-to-shoot translocation of Zn and Cd in rice[J]., 2012, 35(11): 1948-1957.
[24] Yan Y F, Lesta I P, Lee K J, Kim M Y, Lee S H, Lee B W. Identification of quantitative trait loci for cadmium accumulation and distribution in rice () [J]., 2013, 56(4): 227-232.
[25] 陈志德. 水稻不同品种耐镉性鉴定及耐镉胁迫相关性状的QTL定位[D]. 南京: 南京农业大学, 2010
Chen Z D. Screening of rice varieties with cadmium tolerance and mapping of QTLs related to cadmium stress in rice[D]. Nanjing: Nanjing Agricultural University, 2010
[26] 陈艳彩, 唐文帮. 筛选和培育镉低积累水稻品种的进展和问题探讨[J]. 农业现代化研究, 2018, 39(6): 1044-1051
Chen C Y, Tang W B. A perspective on the selection and breeding of low-Cd rice[J]., 2018, 39(6): 1044-1051.
[27] Lan H, Wang Z, Wang Q H, Wang M M, Bao Y M, Huang J, Zhang H S. Characterization of a vacuolar zinc transporterin rice (L.)[J]., 2013, 40(2): 1201-1210.
[28] 鄂志国, 张玉屏, 王磊. 水稻镉胁迫应答分子机制研究进展[J]. 中国水稻科学, 2013, 27(5): 539-544.
E Z G, Zhang Y P, Wang L. Molecular mechanism of rice responses to cadmium stress[J]., 2013, 27(5): 539-544. (in Chinese with English abstract).
Genome-wide Association Analysis of Cadmium Content in Rice Based on MAGIC Population
DONG Zheng1, 2, #, WANG Yamei3, #, LI Yongchao1, 2, XIONG Haibo1, 2, XUE Canhui1, 2, PAN Xiaowu1, 2, LIU Wenqiang1, 2, WEI Xiucai1, 2, LI Xiaoxiang1, 2, *
(1Hunan Rice Research Institute, Hunan Academy of Agricultural Science, Changsha 410125, China;2Key Laboratory of indica Rice Genetics and Breeding in the Middle and Lower Reaches of Yangtze River Valley, Ministry of Agriculture, Changsha 410125, China;3Agricultural Genomics Institute, Chinese Academy of Agricultural Sciences, Shenzhen 518120, China;#These authors contributed equally to the work;*Corresponding author, E-mail: xiaoxiang66196@126.com)
【】Based on the MAGIC-Hei (Multi parent advanced generation inter cross) population of rice, we explored new loci/genes related to cadmium (Cd) content in multiple environments, and screened excellent lines with low Cd alleles to provide new genes and germplasm resources for breeding low Cd varieties.【】MAGIC-Hei, which is composed of eight parents, was planted in Changsha, Hunan Province in 2017−2020, and the Cd content was measured. Based on genotypic data by using genotyping-by-sequencing, we carried out genome wide association analysis (GWAS) on Cd content and explored QTLs to analyze their genetic mechanism. 【】Fourteen QTLs controlling Cd accumulation were detected. Except for chromosome 8, the 14 QTLs distribute on the other 11 chromosomes. Among them, six loci were consistent with reported genes, and eight loci were newly found. In addition, the eight loci on chromosomes 2, 4, 7, 9 and 12, can be detected in two or more environments and the effects are relatively stable, which can be used for further fine mapping and functional research. According to the results of gene annotation and gene expression analysis,,,andare candidate genes for Cd content related sites, which are related to heavy metal transport. In addition, we screened 10 excellent lines carrying favorable alleles, which can be used to create low Cd rice materials. 【】Eight new QTLs for Cd accumulation related traits and excellent materials with low Cd content in rice were found in this study, which is of great significance for the genetic study of Cd accumulation and the breeding of low Cd varieties by marker assisted selection (MAS).
MAGIC; QTL; cadmium content; MAS breeding; candidate gene
10.16819/j.1001-7216.2022. 210504
2021-05-24;
2021-08-16。
国家重点研发计划资助项目(2016YFD0100101-12);国家现代农业产业技术体系建设专项(CARS-01-14);湖南省农业科技创新资金项目(2019LS05-2);湖南省农业科技创新资金项目(2020CX17)。
