基于通路活性的抗癌药物敏感性预测
2022-01-07高冲秦玉芳陈明
高冲,秦玉芳,陈明
1.上海海洋大学信息学院,上海201306;2.农业农村部渔业信息重点实验室,上海201306
前言
恶性肿瘤(癌症)是严重影响人类健康的疾病之一[1]。虽然传统治疗癌症的方法(放疗、化疗)有明显的治疗效果,但是大量研究表明肿瘤具有异质性[2],患有相同癌症的病人使用相同的治疗方法却有不同的疗效。基于此,个性化医疗应运而生,它关注每一位患者的特异性特征,其中测量患者对药物的反应是一个关键问题[3-4]。
随着高通量基因组学技术的发展,药物基因组学成为测量患者对药物反应的一个重要方法[5]。研究者通常通过基因或蛋白质表达谱等分子图谱来测量细胞对药物的反应,进而建立相应的计算模型预测药物反应[6]。Gillet 等[7]发现在细胞系模型和临床具有相关性的前提下,这些计算模型能识别决定药物反应的分子因素,并对患者群体进行相应的个性化药物治疗。许多研究机构开发了诸如癌症细胞系百科全书(Cancer Cell Line Encyclopedia, CCLE)和肿瘤药物敏感性基因组学(Genomics of Drug Sensitivity in Cancer,GDSC)等包含基因表达数据和拷贝数变异等基因组学数据以及药物反应值在内的大型数据库,这些大型数据集为识别新的药物靶点和药物反应标记物提供了更多的可能性[8];同时,这也为开发药物反应计算模型提供了依据,如Papillon-Cavanagh 等[9]利用CCLE 和癌症基因组计划(Cancer Genome Project,CGP)数据集建立预测药物反应的线性模型,发现基因组预测因子能够验证对特定药物的反应;Masica等[10]利用CCLE数据集构建多变量组合改变组织(MOCA)模型来识别药物反应的组合生物标志物;Menden 等[11]利用GDSC 数据集和机器学习算法建立基于细胞系的基因组特征和药物化学特性的药物敏感性预测模型,并通过对实验结果和已有事实的对比验证该模型的有效性。
近年来,许多研究者根据基因水平特征建立抗癌药物敏感性预测模型[6]。如Costello等[12]把基因表达谱或拷贝数变异等基因组学数据用于预测抗癌药物反应,发现基于基因表达数据建立的抗癌药物敏感性预测模型具有很好的预测性能;Geeleher等[13]采用岭回归算法建立抗癌药物反应预测模型,同时使用独立数据集验证了该模型的有效性。这些方法大多基于基因表达数据等基因水平特征,在独立研究中的重复性有限,这对生物学解释提出了挑战[14]。有研究表明考虑基因间相互作用行为比仅仅关注单个基因行为在预测药物反应上具有更好的预测效果[15]。通路数据库是系统分析基因功能,联系基因组信息和功能信息的知识库。通路作为基因功能集合能够提高预测模型的预测能力和解释能力[16]。Wang 等[17]把通路数据和基因表达谱应用到药物敏感性预测,研究表明在CCLE 数据集的24 种药物中,基于通路的模型较基于基因的模型具有更好的预测性能,并且基于通路的模型能识别更多药物相关的基因或通路,具有更好的生物学解释;然而该方法仅仅把通路作为基因集合,没有考虑通路中基因互相作用关系。
针对以上问题,本研究提出一种整合通路网络中高连接度基因和基因表达数据推断通路活性,建立抗癌药物敏感性预测模型,简记为PHG(Pathway Hub Gene)。首先利用通路数据和STRING数据库得到每个通路的基因相互作用网络表,从该网络表中选择高连接度基因;然后分别计算每一个通路的活性向量;最后合并所有通路的活性向量,得到通路活性特征矩阵,以此作为抗癌药物敏感性预测模型的输入。10折交叉验证的实验结果表明,在17-AAG等大多数抗癌药物上,并不是通路中所有基因都对药物敏感性预测有帮助,考虑通路中的关键基因较通路全部基因构建预测模型具有更好的预测效果,同时验证了基于通路的模型较基于基因的模型能给出更好的生物学解释。
1 数据集及预处理
本研究的基因表达和药物IC50 值数据来自于CCLE 数据库,下载地址为 https://portals.broadinstitute.org/ccle/data;同时为了独立检验,也下载了GDSC 数据库中的基因表达和药物IC50 数据,下载地址为https://www.cancerrxgene.org/。为消除实验技术和实验平台所导致的基因表达量误差,采用以基因为中心的RMA 标准化算法对基因表达谱进行标准化处理。经过标准化后,CCLE 基因表达谱共有18 900 个基因和1 036 个细胞系样本,GDSC 基因表达谱中共有9 920个基因和697个细胞系样本。
本研究使用IC50 值衡量药物敏感性,类似于Wang 等[17]的做法,对药物反应IC50 值做log 变换。由于基因表达数据中的一些细胞系样本在药物反应数据里不存在,所以本研究选取在基因表达数据和药物反应数据中同时存在的细胞系进行分析。例如,对于药物AEW541 来说,NCIH2196_LUNG 细胞系存在基因表达谱中,但在AEW541 药物反应数据中没有该细胞系,所以在做AEW541 药物的敏感性预测时需去除该细胞系。
本研究使用的通路数据来自京都基因和基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)中的通路数据库,在该数据库中下载每个通路的基因集,最终的通路数据集包括389 个通路,共有14 097 个基因。通路中基因间相互作用关系表可从STRING 数据库中获得,下载地址为https://www.string-db.org/cgi/download,STRING数据库包含5 090个物种、24 584 628 种蛋白和3 123 056 667 个相互作用关系[18],本研究下载的数据来自数据库最新版本(Version 11.0)。
2 模型
2.1 基于通路高连接基因的通路活性推断
为推断通路活性,本研究不仅考虑通路中每个基因的表达水平,还关注了通路中基因间相互作用关系,基因相互作用关系在预测药物敏感性具有更好的鲁棒性[15]。首先从STRING 数据库中得到每个通路(基因集)中基因间互相作用网络表,表中的(Gi,Gj)表示基因Gi和基因Gj在通路中是相互连接的;接着根据通路互相作用网络表计算通路活性向量。
计算每个基因在通路互相作用网络表中的度,由于通路网络中高连接度的Hub 基因对整个通路的功能起着更关键的作用[19],所以从该网络表中选择高连接度Hub 基因来进行分析。将基因的度降序排序,选择排名在前10%的基因作为Hub 基因,图1中的Gh1,Gh2,…,Ghk为通路p1的Hub 基因;计算Hub基因表达值的平均值作为活性值,然后合并每个细胞系样本中的活性值得到该通路的活性向量。活性值计算公式如下:

其中,hk表示通路P1中Hub基因的数量;gij表示基因i在细胞系样本j中表达值;p1j表示细胞系样本j在通路p1中的活性值。
按照上面的方法,计算所有通路的活性向量,合并得到通路活性矩阵(列为细胞系样本,行为通路)。假定有l 个通路,分别记为P1,P2,…,Pl,按照上述方法计算所有通路的活性向量后得到通路活性矩阵(pij),其中i为通路,j为细胞系样本。
总的来说,可将基因表达谱和通路中关键基因信息分析整合得到通路活性矩阵,以此来预测癌症药物敏感性。基于通路中高连接度基因模型的流程如图1所示。
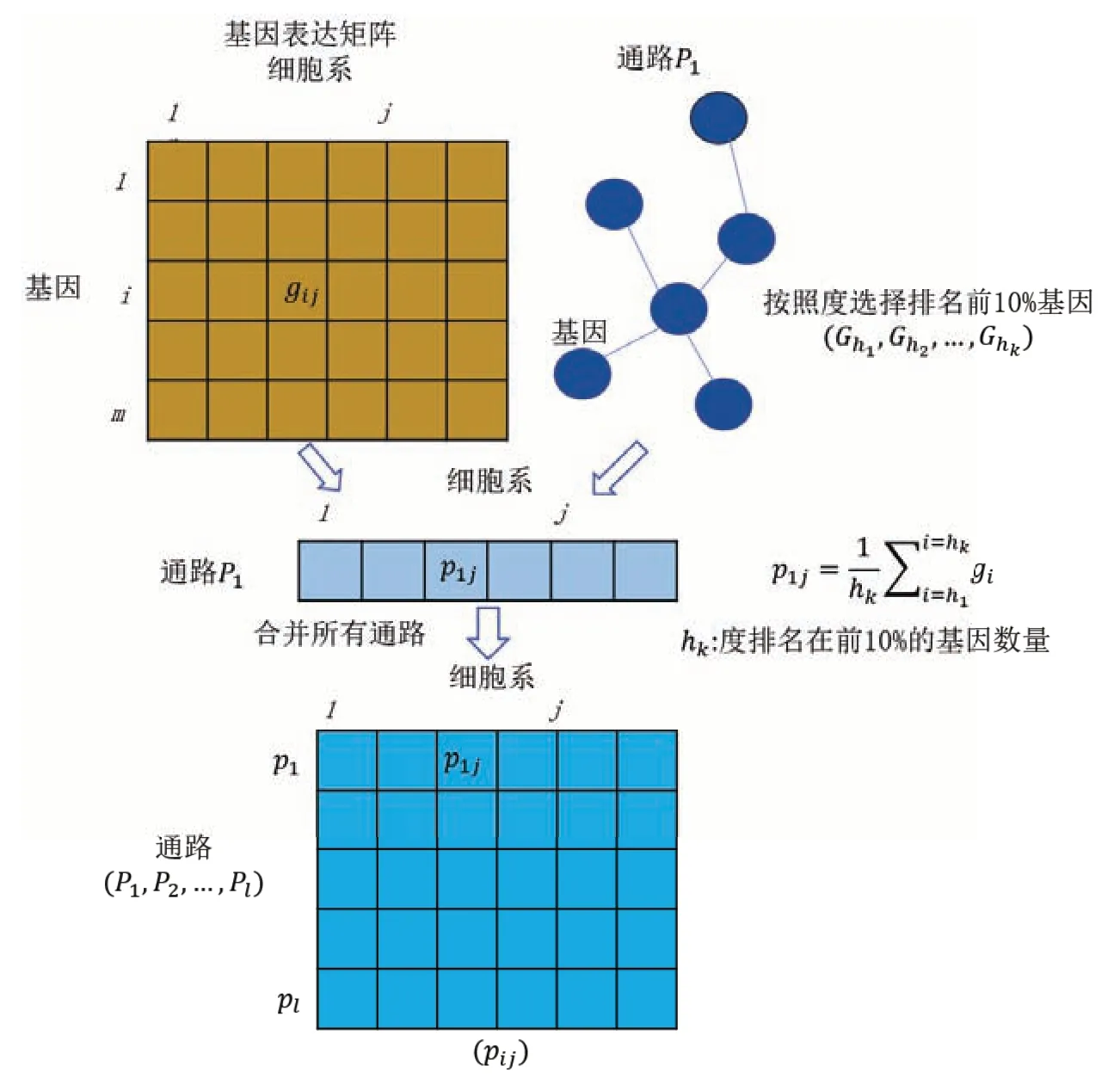
图1 利用通路中高连接度基因表达推断通路活性Fig.1 Using the expression of genes with high connectivity in the pathway to infer pathway activity
2.2 建立药物反应预测模型
将得到的通路活性特征矩阵作为预测模型的输入,药物敏感性水平作为模型输出,根据均方误差(Mean Square Error, MSE)来调试优化模型的参数,并进行训练与预测。本研究采用机器学习中的弹性网作为预测算法。
弹性网是一种使用L1 和L2 范数作为先验正则项训练的线性回归模型[20]。这种组合可以学习到类似于Lasso 的一个稀疏模型,同时还保留岭回归的正则化属性,既能实现重要特征变量的选择,又能处理强相关性特征数据,具有较好的群组效应,结合了岭回归和Lasso 回归的优点。因此,弹性网尤其适用于有多个特征彼此相关的场合。在基于通路/基因的预测模型中,作为特征的通路/基因相互之间实际上都是有联系的。因此,本研究选用弹性网回归算法来构建预测模型,并使用R语言中glmnet包实现弹性网算法。调整和优化模型主要通过网格搜索,在1 000个参数中寻找最优参数,其中α:[0.1,1 ]设置10 个参数,λ:[exp-5,exp5]设置100 个参数,使用10 折交叉验证选取最优参数。
3 结果与讨论
3.1 通路之间的重叠性
本研究使用Jaccard 指数来评价两个通路之间的重叠性。通过对通路间重叠性的研究,分析通路是否具有特异性,是否对实验产生较大的误差。Jaccard指数计算公式如下:
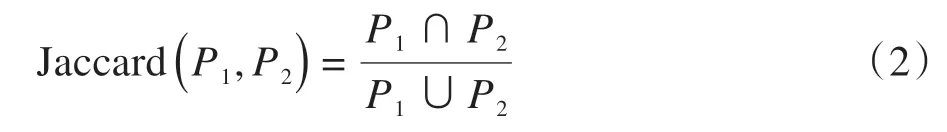
其中,P1∩P2表示同时存在于通路P1和通路P2的基因;P1∪P2表示存在于通路P1或P2的基因。由式(1)可以发现,当两个通路完全不同时,即两个通路没有相同的基因,则Jaccard 指数为0,当两个通路的基因集完全相同时,则Jaccard 指数为1。因此,所有通路对的Jaccard 指数在0 到1 变化不等。计算所有通路对的Jaccard 指数,结果显示约30%通路对的重叠性小于0.6,大多数通路的Jaccard 指数小于0.2,这说明通路之间的重叠性较低,降低了因通路之间的重叠过高而引起的模型误差。
3.2 基于通路活性的药物敏感性预测性能
比较分析文献[17]中的方法(DiffRank),本研究提出基于通路中所有基因推断通路活性的方法,即PAG(All Gene of Pathway)。为了把基于通路模型和基于基因模型进行对比,还提出基于基因模型的方法AG(All Gene)。
PAG 方法和PHG 方法的不同在于PHG 方法在推断通路活性时使用的是通路中高连接度的关键基因,而PAG 方法使用通路中所有基因来计算活性值,进而得到通路活性矩阵,以此作为预测模型的输入。此外,基于基因模型的AG 方法是直接使用基因表达矩阵作为药物敏感性预测模型的输入,而不考虑通路信息,基因模型中的细胞系为样本,基因表达值为特征。
本研究使用弹性网算法训练通路活性矩阵,10折交叉验证选择最优参数,并使用最优参数下的MSE 作为预测模型性能的评价标准。图2 给出了基于CCLE 数据集中24 种药物在4 种模型下进行药物敏感性预测的结果。PHG 方法在17-AAG 等12 种药物上具有最好的预测效果,在AZD6244 等6 种药物上的预测效果是次好的;PAG 方法在Irinotecan 等4种药物上具有最好的预测效果,在17-AAG等11种药物上具有次好的效果。通过PHG 和PAG 对比分析,发现并不是通路中所有基因都会对药物敏感性预测有帮助,只选取通路中连接紧密的基因进行预测可能更具有鲁棒性。AG 方法在AZD6244 等7 种药物上具有最好的效果,在Erlotinib 等6 种药物上的预测效果是次好的。对比基于通路模型和基于基因模型可以发现基于通路模型有较好的预测性能。总的来说,对于一些药物,使用基于通路中高连接度基因的计算分析方法取得了最好的预测效果,更有利于药物敏感性预测。

图2 不同模型对CCLE中24种药物的预测性能Fig.2 Predictive performance of different models for 24 kinds of drugs in CCLE
3.3 模型再现性
本研究中的模型再现性是指在一个数据集上训练数据,在另一个数据集上测试数据,然后再交换数据集重新训练和测试。与CCLE 数据集相比,GDSC数据集中基因表达矩阵和通路数据推断通路活性矩阵的特征数量较少。为了实验的有效性,本研究从基于CCLE 基因表达谱推断通路活性矩阵中随机抽取和GDSC相同数量的特征,以便训练和预测。
对于给定的抗癌药物,随机选择50 次相等数量的特征数据输入到药物敏感性模型,然后计算MSE,把50 次MSE 的平均值作为验证模型再现性预测性能。共计算了24 种药物在CCLE 数据集和GDSC 数据集上的预测性能,表1 中列出了Paclitaxel 等4 种药物在CCLE 数据集和GDSC 数据集上的预测性能。在基于药物Paclitaxel 敏感性预测的模型再现性中,当以GDSC 数据作为训练集,CCLE 数据作为测试集时,MSE 为5.91;当以CCLE 数据为训练集,GDSC 数据为测试集时,MSE 为5.52,这表明PHG 方法在药物Paclitaxel 上的药物敏感性预测具有较好的模型再现性。
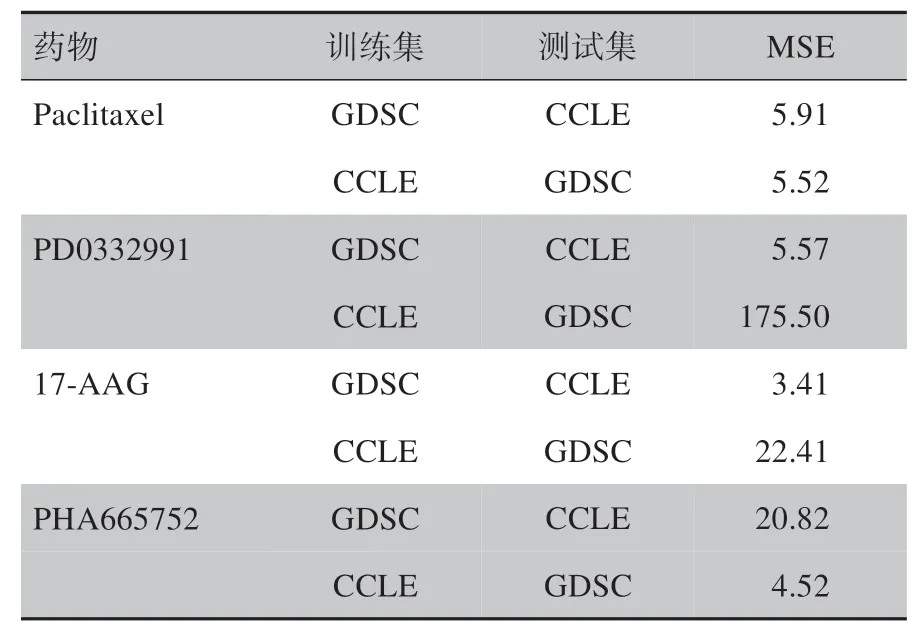
表1 PHG方法在4种药物的模型再现性Tab.1 Model reproducibility of PHG method in 4 kinds of drugs
另外,对于药物17-AAG和PD0332991,以GDSC数据作为训练集训练模型,同时用此模型测试CCLE数据,发现具有较低的MSE,即较好的预测性能,然而当以CCLE 数据训练模型,再以GDSC 数据测试模型,则有较高的误差,这表明PHG 方法在这两种药物上使用基于GDSC 基因表达谱作为训练集时会得到较好的模型,具有较好的预测性能。相反,对于药物PHA665752,以CCLE 基因表达数据作为训练集构建药物敏感性预测模型则会得到较好的预测性能。
3.4 识别药物相关联基因的通路
本研究把通路数据和基因表达谱整合得到通路活性评分,并以此构建预测模型,进一步识别癌症标记物,从而给出生物学解释。当利用通路中高连接度基因数据和弹性网算法建立预测模型时,弹性网中非零系数对应的特征是预测细胞对药物反应的重要数据[1]。因此,本研究采用了弹性网算法中非零系数统计与抗癌药物相关联基因的通路数量。在24种药物中,19 种药物包含靶向基因的通路都能识别出来(表2)。

表2 药物相关基因的通路数量Tab.2 Number of pathways for drug-related genes
例如,对于药物Lapatinib,使用PHG 方法能识别弹性网中非零系数对应的MicroRNAs in cancer、Breast cancer 和 EGFR tyrosine kinase inhibitor resistance 等9 个特征通路,其中MicroRNAs in cancer通路包含ABCB1、EGFR 和ERBB2 等靶向基因,Breast cancer 通路包含EGFR 和ERBB2 等靶向基因。总的来说,基于通路高连接度基因的药物敏感性预测模型能够识别药物相关联基因的通路,具有更好的生物学解释能力。
4 结论
本研究提出一种基于通路中高连接度基因的抗癌药物敏感性预测方法(PHG);对基因表达谱、通路数据和药物敏感性IC50 值进行综合分析,综合考虑不同因素的作用,提取高连接度基因集合,然后计算通路活性矩阵,进而通过机器学习技术进行抗癌药物敏感性预测分析,并把识别的标记与已有研究进行对比分析,验证基因/通路与药物之间的联系。实验表明,基于通路中高连接度基因模型相比其他通路或基因模型有更好的预测性能。通路中并不是所有的基因都对药物敏感性预测起到促进作用,而是一些关键基因更为重要。本研究提出的计算方法为通路活性预测模型的发展提供了参考。