适于检测非洲猪瘟病毒的点亮Spinach-p54 RNA适配体的设计及应用
2022-01-06韩程程夏凯龚茹莹吴栩涵张蕾梁新乐
韩程程,夏凯,龚茹莹,吴栩涵,张蕾,梁新乐
适于检测非洲猪瘟病毒的点亮Spinach-p54 RNA适配体的设计及应用
韩程程1,2,夏凯1,3,龚茹莹1,吴栩涵1,张蕾1,2,梁新乐1,2
1. 浙江工商大学食品与生物工程学院,杭州 310018 2. 浙江工商大学食品生物工程研究所,杭州 310018 3. 浙江科技学院生物与化学工程学院,杭州 310023
非洲猪瘟病毒(African swine fever virus, ASFV)是近年来流行的一种主要致病病毒,对我国的国民经济生活造成了严重影响。本研究以非洲猪瘟病毒的编码保守蛋白基因序列片段为检测目标,结合点亮 Spinach RNA适配体的开关功能,设计了Spinach-p54的嵌合式RNA适配体。其特异地结合目标序列并产生荧光,实现了在RNA水平上对非洲猪瘟病毒的快速检测。研究结果表明,核酸适配体浓度200 nmol/L,反应温度为37 ℃,该方法检出限为200 nmol/L,线性范围为200~400 nmol/L。该方法特异性强,操作简单,灵,可用于市场和养殖场的现场快速检测。
核酸适配体;RNA;turn-on;非洲猪瘟
非洲猪瘟是由非洲猪瘟病毒(African swine fever virus, ASFV)引起的一种在家猪和野猪身上表现为急性出血热症状的高传染性的致命疾病[1~4]。世界动物卫生组织(Office International Des Epizooties, OIE)将猪瘟列为必须报告的动物传染病之一,我国将其列为一类动物疫病[5]。我国从2018年辽宁省沈阳市首例非洲猪瘟疫情的确诊至2020年共有32个省份报告了165起疫情,约119.3万头生猪被扑杀,极大地影响了生猪供应,并给养殖业带来了巨大的打击[6]。ASFV病毒基因组为170~190 kb,基因易变异,病毒蛋白种类多、存活能力强、可通过多种方式和途径进行有效传播,这是导致ASFV在染疫国家和地区常呈地方流行的重要原因之一[7]。因此对于ASFV的早期诊断以及现场快速检测对非洲猪瘟的防控显得尤为重要。
目前,对于非洲猪瘟的检测主要采取病原学检测和血清学抗体检测[8]。病原学检测主要利用病毒分离培养、荧光抗体检测和红细胞吸附实验等传统鉴定方法。这些传统鉴定方法尽管简单易行,但所需时间较长,并受人为因素干扰,不适宜现场检测。新发展的病毒核酸检测主要有PCR、qRT-PCR、环介导等温扩增技术(loop-mediated isothermal amplification, LAMP)、等温重组酶聚合酶扩增结合侧向流条法(isothermal recombinase polymerase amplification with lateral flow dipstick, RPA-LFD)等[5~9, 11],此类方法检测时间短、灵敏度高,但对操作环境要求高。血清学抗体检测技术主要有酶联免疫吸附法、胶体金免疫层析技术等[12~14],这种基于抗原-抗体特异性结合的检测技术具有特异性强,易于观察,灵敏度高等优点,但制备抗体时间较久、成本较高,且不稳定。近年来,由于核酸适配体与抗体相比具有成本低,稳定性好等优点,它被作为抗体分子的替代分子,受到诸多领域的关注。其中 Spinach是一种类似增强绿色荧光蛋白(enhanced green fluorescent protein, EGFP)的核酸适配体[15]。该核酸适配体能够与荧光染料3,5-二氟-4-羟基亚苄基咪唑啉酮(DFHBI)特异性结合产生1000倍的荧光强度,该染料无毒且具有良好的细胞通透性。Spinach已在RNA成像和小分子代谢物的传感器等方面得到应用[16-17]。除此之外,Spinach已被开发为报告核酶的自切割活性,并应用于研究 RNA-RNA的体外组装[18,19]。
本文以截短的 Spinach序列为基础[20],在其5′端和3′端分别联接上基因的短序列,形成具有特异性识别基因序列RNA适配体— Spinach-p54,该适配体与基因互补配对后形成的二级结构能结合荧光团DFHBI,产生强烈的荧光信号,从而完成快速定量检测ASFV保守基因的特异性快速检测(图1)。
1 材料与方法
1.1 仪器与试剂
96孔板购自上海生工生物工程股份有限公司;酶标仪购自美国BioteK公司;NanoPro超微量紫外可见分光光度计购自天津鼎昊源生物科技有限公司;超纯水装置系统Milli-Q购自美国Millipore公司。

图1 点亮Spinach-p54 RNA适配体检测非洲猪瘟示意图
pUC-Spinach-p54-1/2/3质粒,pUC-p54质粒由苏州金唯智生物科技有限公司合成;HiScribe T7快速高效RNA合成试剂盒购自美国NEB公司;柱式RNA快速浓缩纯化试剂盒,SanPrep柱式PCR产物纯化试剂盒,SanPrep柱式质粒DNA小量抽提试剂盒,硫酸卡那霉素,DEPC-treated Water购自上海生工生物工程股份有限公司;Ⅰ和Ⅰ限制性内切酶购自北京宝日医生物技术有限公司;DFHBI购自美国Sigma公司;KCl,MgCl2,NaH2PO4购自上海阿拉丁生化科技股份有限公司。
1.2 Spinach-p54 RNA适配体设计
Spinach-p54 RNA适配体与ASFV的基因碱基互补配对的序列应具有高度特异性,以防止不必要的分子内或分子间杂交[21]。利用NUPACK (http://www.nupack.org/)对Spinach-p54 RNA适配体与RNA在等浓度条件下进行建模,两者结合后形成一个复合物的概率应为 100%。基于上述原则对Spinach-p54 RNA适配体进行筛选,共设计了在最小自由能条件下结合后形成稳定的二级结构的3个Spinach-p54 RNA适配体(表1)。
1.3 Spinach-p54 RNA适配体及p54 RNA制备
将大肠杆菌() DH5α(分别含有pUC-Spinach-p54-1/2/3和 pUC-p54质粒)接种于LB培养基(含硫酸卡那霉素,终浓度为50 μg/mL)于37 ℃,180 r/min的条件下培养12~16 h,利用SanPrep柱式质粒DNA小量抽提试剂盒提取质粒。将提取的pUC-Spinach-p54-1/2/3质粒和pUC-p54质粒分别利用Ⅰ和Ⅰ限制性内切酶于37 ℃,酶切4 h。利用SanPrep柱式PCR产物纯化试剂盒对酶切产物进行纯化并利用NanoPro测定酶切产物浓度。利用T7快速高效RNA合成试剂盒进行体外转录,体外转录体系为:酶切产物1 μg,NTP buffer Mix 10 μL,T7 RNA polymerase Mix 2 μL,用 DEPC-treated Water补齐至30 μL;转录条件为37 ℃ 16 h。利用柱式RNA快速浓缩纯化试剂盒对转录产物进行纯化并利用NanoPro测定RNA浓度。RNA于–80 ℃保存,备用。
1.4 Spinach-p54 RNA适配体检测反应体系
将Spinach-p54 RNA适配体取出后进行复性,条件为95 ℃变性5 min,静置于冰上10 min[22]。在96孔板内加入100 μL反应体系,并于37 ℃避光反应30 min,测量在37 ℃,激发波长460 nm,发射波长502 nm下的荧光强度。反应体系包括:30 μL缓冲液(140 mmol/L KCl,1 mmol/L MgCl2,10 mmol/L NaH2PO4),200 nmol/L RNA适配体(Spinach-p54- 1/2/3),200 nmol/LRNA,1 μL 20 μmol/L DFHBI,最后用 DEPC- treated Water补齐至100 μL。其中设置阴性对照,阴性对照组中不存在RNA。
1.5 Spinach-p54-1 RNA适配体反应温度实验
按照1.4方法将反应体系加入96孔板内,分别于25 ℃、30 ℃、37 ℃避光反应30 min,并分别检测荧光强度。
1.6 Spinach-p54-1 RNA适配体检测灵敏度实验
按照1.4方法中的条件添加缓冲液、Spinach- p54-1 RNA适配体和DFHBI,而RNA的浓度按20、100、200、300、400 nmol/L进行添加,其中相应浓度的拷贝数分别为11、55、110、165、220拷贝/μL,并用DEPC- treated Water补齐至100 μL。在37 ℃避光反应30 min,37 ℃条件下检测荧光强度。

表1 适配体名称和序列
1.7 Spinach-p54-1 RNA适配体检测p54 RNA 的特异性实验
以大肠杆菌和李斯特菌()的总RNA模拟干扰信号,并加入到上述检测体系中,考察Spinach-p54-1 RNA适配体检测的特异性。
1.8 Spinach-p54-1 RNA适配体转录后直接检测ASFV p54序列
将纯化后的pUC-Spinach-p54-1酶切产物1 μg,NTP buffer Mix 10 μL,T7 RNA polymerase Mix 2 μL,用DEPC- treated Water补齐至30 μL,于37 ℃条件下转录16 h。转录结束后的Spinach-p54-1产物分别进行下列操作(所有产物均未测定浓度):(1)将转录体系于70 ℃加热10 min对T7转录酶进行失活,将失活后的样品直接加入反应体系中;(2)将转录体系直接加入到反应体系中。Spinach-p54-1 RNA适配体按上述方法操作结束后加入96孔内,再将方法1.4中的反应体系加入96孔板中。在37 ℃避光反应30 min,并于37 ℃条件下每隔1 min检测荧光强度,共检测45 min。
2 结果与分析
2.1 Spinach-p54 RNA适配体建模及筛选
采用NUPACK设计并选择了3种Spinach-p54 RNA适配体单个序列进行建模。3种Spinach-p54 RNA适配体在37 ℃条件下所形成的二级结构如图2所示,它们最小自由能分别为–24.20 kcal/mol、–27.40 kcal/mol和–24.50 kcal/mol (表2)。当Spinach-p54-1/2/3 RNA适配体与RNA在37 ℃结合后,最小自由能都显著下降,分别为–52.88 kcal/mol、–51.38 kcal/mol和–46.28 kcal/mol。这表明单个适配体是不稳定的,与RNA序列结合后增强了RNA适配体的稳定性。Spinach-p54 RNA适配体与RNA浓度均为200 nmol/L时的结合实验结果表明,在加入DFHBI后均显著地产生荧光,与对照组明显区分(图3)。这表明形成了可产生荧光的Spinach-p54 RNA适配体-RNA-DFHBI复合物,即Spinach-p54 RNA适配体可与RNA特异地结合。但是,由于本反应体系中其他物质的存在,实验结果会产生较大的背景噪声,需要在后续研究中进一步优化。
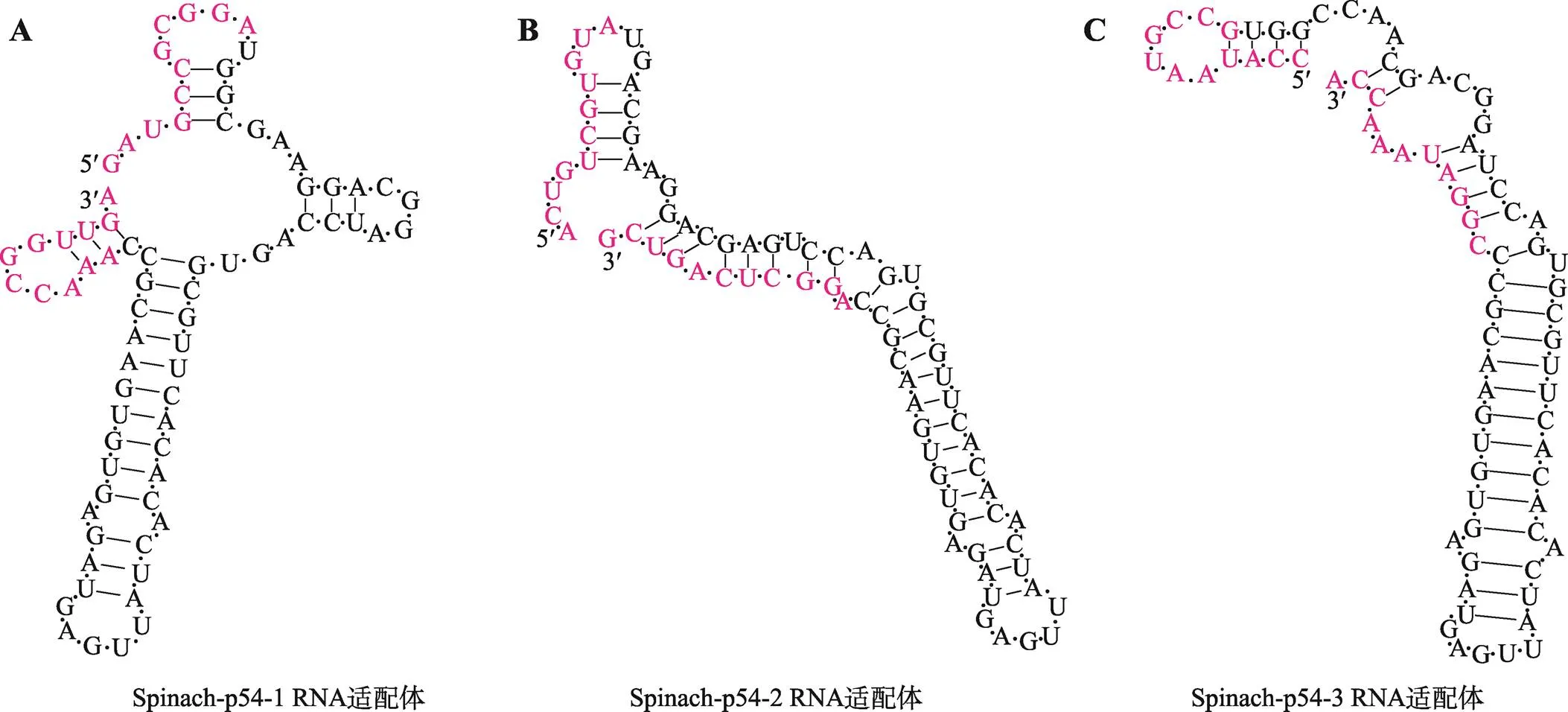
图2 点亮Spinach-p54 RNA适配体二级结构

表2 Spinach-p54 RNA适配体二级结构的最小自由能
2.2 温度显著影响Spinach-p54 RNA适配体特异结合p54 RNA
温度是影响化学反应速度及 RNA二级结构形成与稳定的关键因素之一。在此首先考察了3种不同温度对Spinach-p54 RNA适配体特异结合RNA的影响。在没有RNA存在的情况下,单个RNA适配体的背景荧光定义为阴性对照。结果显示,在探究的3种温度下 Spinach-p54-1/2/3 RNA适配体与RNA浓度均为200 nmol/L的情况下均能产生明显区分于阴性对照的荧光(图3)。尽管在25 ℃条件下3个RNA适配体产生的荧光值与阴性对照未产生显著性差异,但在30 ℃和37 ℃条件下3个RNA适配体产生的荧光值与阴性对照相比具有显著性差异(<0.05)其中在37 ℃条件下产生的荧光值最强,表明Spinach-p54 RNA适配体-RNA-DFHBI复合物形成的最适温度为37 ℃。尽管37 ℃为最适温度,但因与RNA结合序列的不同,在该温度下不同的RNA适配体产生的荧光强度存在差异,其中Spinach-p54-1 RNA适配体产生的荧光值最大。表明Spinach-p54-1 RNA适配体为3个适配体中较为适合用于检测ASFV的RNA适配体。因此后续实验以Spinach-p54-1 RNA适配体作为实验对象,除特殊说明外。

图3 Spinach-p54 RNA适配体反应温度优化
在上述实验基础上,对3种RNA适配体与RNA在不同温度条件下形成的二级结构进行了拟合计算。3种RNA适配体在3个不同温度下共形成两种不同的二级结构,故以Spinach-p54-1 RNA适配体为例绘制二级结构图,其中25 ℃和30 ℃形成的二级结构相同。Spinach RNA适配体中与DFHBI
结合部分的结构为G-quadruplex,G-quadruplex序列在Spinach-p54 RNA适配体的位置为G20、G23、G24、G25、G50、G53、G55、G57。其中G25(对应于原始Spinach适配体G28位置)碱基堆积在四链体G24(对应于原始Spinach适配体G27位置)上,并与DFHBI形成氢键[23,24]。在25 ℃和30 ℃条件下形成的二级结构(图4A)其特征为存在5个茎环结构,其中 G25位于最大的茎环结构中,该结构由 12 个碱基构成。在37 ℃条件下形成了包含4个茎环的二级结构(图4B),其中G25位于由18个碱基构成的最大茎环中。二级结构图结合实验数据表明在37 ℃条件下形成的二级结构能更好的达到检测ASFV的目的。
2.3 Spinach-p54-1 RNA适配体检测范围实验
灵敏度是检验检测方法可行性的一个主要参数,因此进一步探究了Spinach-p54-1 RNA适配体对RNA最低检测浓度。当Spinach-p54-1 RNA适配体的浓度为200 nmol/L (110拷贝/μL)时,反应强度在20~400 nmol/L (11~220拷贝/μL)RNA范围内随着RNA浓度升高而升高,但浓度在20~100 nmol/L (11~55拷贝/μL)时产生的荧光值与阴性对照无法进行明显区分,当浓度高于200 nmol/L (110拷贝/μL)时产生的荧光与阴性对照明显区分(图5A)。当RNA浓度高于Spinach-p54-1 RNA适配体浓度时,产生的荧光值仅略微高于两者浓度相等,表明当Spinach-p54-1 RNA适配体与RNA浓度为1∶1时为产生荧光的最佳条件。当Spinach-p54-1 RNA适配体浓度为400 nmol/L,RNA浓度范围在200~400 nmol/L (110~220拷贝/μL)时荧光强度与RNA的浓度存在良好线性关系(图5B),校正曲线方程为=0.2059–3.4804,2=0.9863。

图4 Spinach-p54 RNA适配体-p54 RNA复合物不同温度下二级结构图
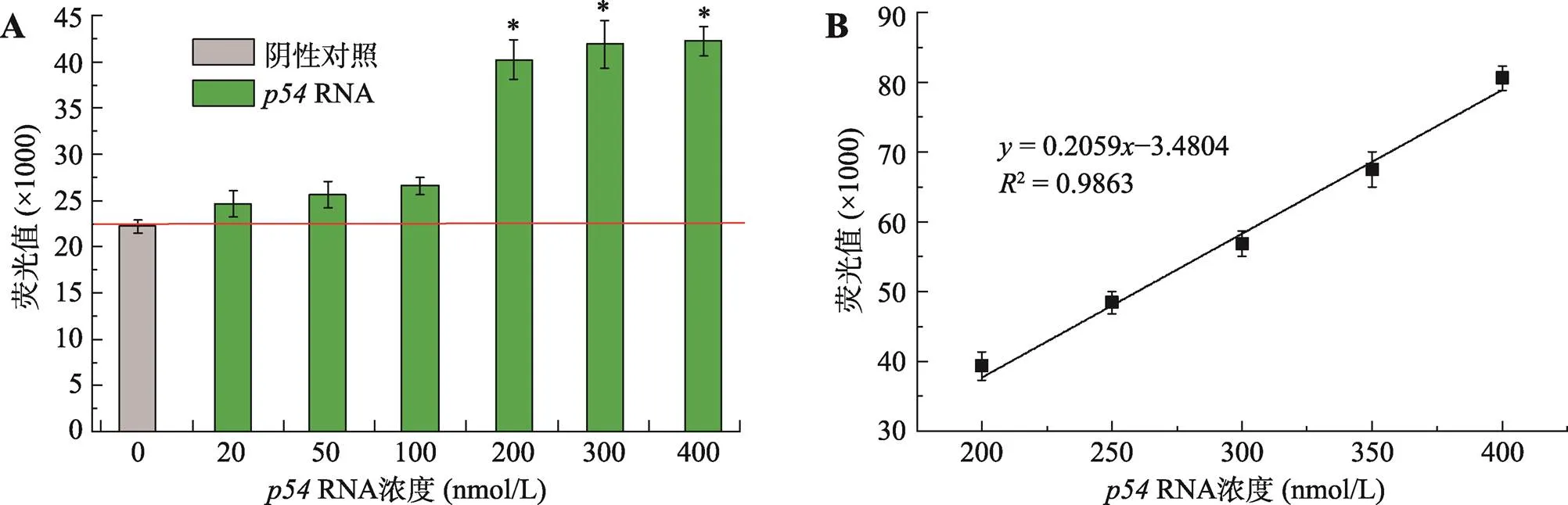
图5 Spinach-p54-1 RNA适配体检测ASFV的灵敏度
2.4 Spinach-p54-1 RNA适配体识别特异性
为了评估筛选出的Spinach-p54-1 RNA适配体的特异性,利用大肠杆菌和李斯特菌的总RNA作为干扰信号。结果显示,仅添加了大肠杆菌和李斯特菌总 RNA后产生的荧光值与阴性对照无法进行有效区分,而在存在RNA的体系中再添加大肠杆菌和李斯特菌总RNA后产生的荧光值能明显区分于阴性对照,且与仅有RNA存在的条件下产生的荧光值相似(图6)。这表明Spinach-p54-1 RNA核酸适配体即使存在细菌RNA干扰仍对RNA具有较好的专一性。
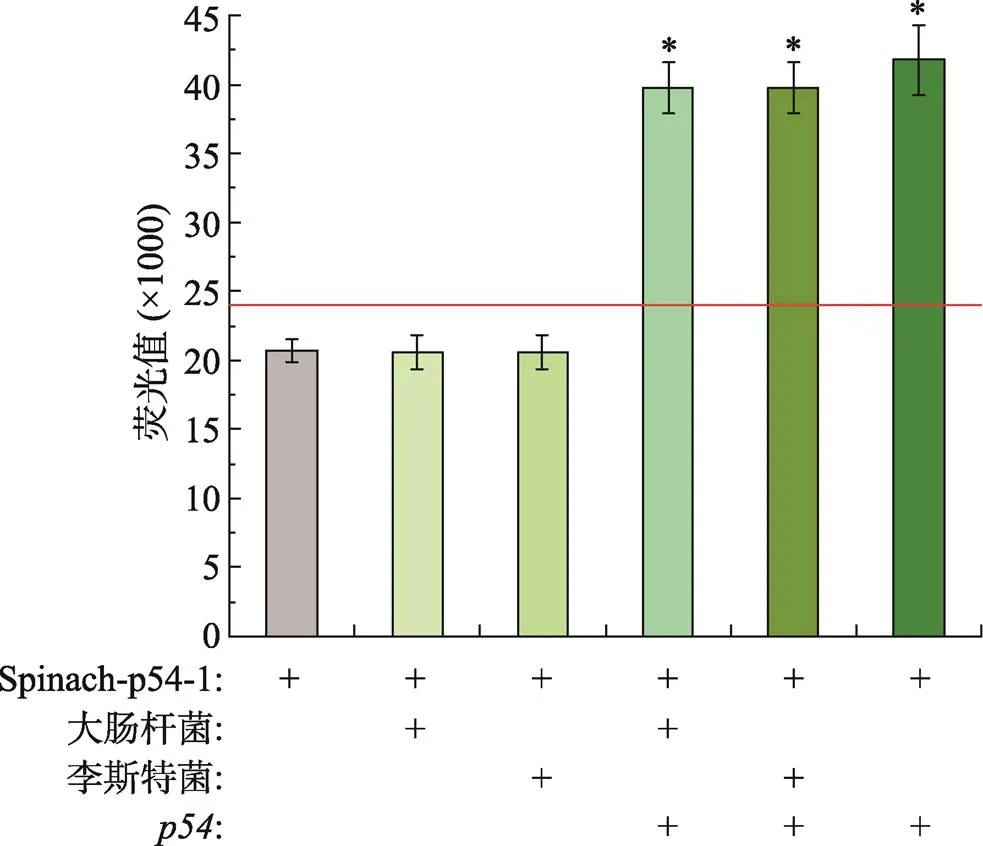
图6 Spinach-p54-1 RNA适配体检测ASFV特异性
2.5 Spinach-p54-1 RNA适配体制备过程的简化
为提高 Spinach-p54-1 RNA适配体应用性和经济性,本研究进一步对Spinach-p54-1 RNA适配体制备过程进行了简化,即在制备RNA适配体时删除了试剂盒纯化RNA的步骤,直接采用转录混合物。结果表明,对Spinach-p54-1 RNA适配体体外转录过程中的T7转录酶进行失活处理后,发现其与RNA结合后产生的荧光值略高于阴性对照,但未产生明显的区分(图7)。对转录体系不做任何处理,直接应用后发现,其产生的荧光能与阴性对照产生明显区分,不过其产生的荧光随着时间的延长逐渐减弱,在30 min后其荧光值无法与阴性对照进行区分。但其前30 min荧光能与阴性对照进行明显区分,表明该Spinach-p54-1 RNA适配体能够在转录结束后直接应用,可略过纯化步骤,减少了实验流程,缩短了时间。
3 讨论
RNA适配体是一种可形成发夹、茎环和 G-quadruplex等结构并与靶分子特异性结合的RNA寡核苷酸序列,最初被应用于检测 RNA水平上的基因表达和调控[22]。由于 RNA核酸适配体具有灵活性和方便性使其能够作为新型报告基因应用于体内和体外成像、以及细菌、病毒的快速检测领域[25~27]。本研究探索了RNA适配体用于非洲猪瘟病毒保守序列的快速检测方法。

图7 Spinach-p54-1 RNA适配体制备过程简化后对ASFV的诊断性能
本研究设计了3种特定 Spinach-p54 RNA适配体,每个适配体与RNA结合后产生的荧光能与阴性对照进行明显区分,进而达到检测 ASFV的目的。已有的研究表明温度能够影响适配体的折叠和荧光强度[28],因此,通过探究不同温度下Spinach- p54 RNA适配体的性能,发现3个适配体在37 ℃条件下形成的Spinach-p54 RNA适配体-RNA- DFHBI复合物所产生的荧光值均高于25 ℃和30 ℃,该结果与形成Spinach2-DFHBI-1T复合物的最佳温度相同[29]。为了检测Spinach-p54-1 RNA适配体的荧光分析效率,当RNA浓度为200 nmol/L时即可检测到明显区分于阴性对照的荧光,整个检测过程需要40 min。其中RNA为人工全合成,利用T7启动子体外转录获得。而在真实临床样本中RNA浓度较低的情况下,可在前期通过磁珠富集[30~32]后进行检测,该部分需要今后的进一步研究。
Zsak检测是ASFV的高敏感、特异和快速的检测技术。Wang等[33]开发了一种新的Zsak检测技术,该检测方法能够覆盖98%已分离的菌株,且检测限为6拷贝,具有很高的特异性。该方法在提取 DNA/RNA后还需要其他操作才能达到检测 ASFV的目的,而利用 Spinach-p54-1 RNA适配体可在提取RNA后直接进行检测。RPA-Cas12a方法中最低检测DNA浓度为0.05 nmol/L,该检测过程仅需 30~40 min[34];除此以外Cas12a结合免疫层析条技术的最低检测限为20个拷贝,该方法需要1 h才能完成检测[35],利用Cas12a检测 ASFV都需要对Cas12a蛋白进行纯化。本研究对 Spinach-p54-1 RNA适配体制备过程进行简化后,发现其转录后无需纯化,即能达到检测ASFV的目的。以上结果表明,基于点亮Spinach-p54-1 RNA适配体的检测方法具有较好的灵敏度、特异性和高效性。受限于生物安全法规对生物技术研究、开发与应用安全的要求,本研究没有使用临床样本验证该检测方法的检测效果,后续将在不同生物样本中对该方法的检测性能进行进一步评估。
综上所述,本研究设计了一种点亮 Spinach-p54 RNA适配体,可以用于快速检测 ASFV中的保守基因,通过检测RNA产生的荧光强度对ASFV进行定量检测。该方法处理时间短(仅为40 min)、灵敏度高、特异性强,为ASFV分子检测手段提供了一种新的方法,具有广阔的应用前景。
[1] Normile D. Arrival of deadly pig disease could spell disaster for China., 2018, 361(6404): 741.
[2] Ma J, Chen H, Gao X, Xiao JH, Wang HB. African swine fever emerging in China: distribution characteristics and high-risk areas., 2020, 175: 104861.
[3] Galindo I, Alonso C. African swine fever virus: a review., 2017, 9(5): 103.
[4] Giudici SD, Franzoni G, Bonelli P, Bacciu D, Sanna G, Angioi PP, Ledda M, Pilo G, Nicolussi P, Oggiano A. Interaction of historical and modern Sardinian African swine fever viruses with porcine and wild-boar monocytes and monocyte-derived macrophages., 2019, 164(3): 739–745.
[5] Yang F, Cheng LQ, Xu XG, Zhang WM, Zhang Q. Establishment and preliminary application of a real-time fluorescence quantitative PCR for CSFV detection., 2021, 42(1): 1–5. 杨峰, 陈立强, 许信刚, 张为民, 张琪. 猪瘟病毒实时荧光定量PCR检测方法的建立及初步应用. 动物医学进展, 2021, 42(1): 1–5.
[6] Zhu YS, Shao N, Chen JW, Qi WB, Li Y, Liu P, Chen YJ, Bian SY, Zhang Y, Tao SC. Multiplex and visual detection of African swine fever virus (ASFV) based on Hive-Chip and direct loop-mediated isothermal amplification., 2020, 1140: 30–40.
[7] Guinat C, Gogin A, Blome S, Kei G, Pollin R, Pfeiffer DU, Dixon L. Transmission routes of African swine fever virus to domestic pigs: current knowledge and future research directions., 2016, 178(11): 262–267.
[8] Wang ZY, Dan K, Shi YG, Kan SF, Zhong LX, He QH. Research progress on rapid detection methods of African swine fever virus., 2020, 11(10): 3215–3220. 王梓莹, 但琨, 施远国, 阚式绂, 钟雷响, 何庆华. 非洲猪瘟病毒的快速检测方法研究进展. 食品安全质量检测学报, 2020, 11(10): 3215–3220.
[9] Cui BB, Li T, Qiu SY, Mei L, Han XQ, Wu SQ. Establishment of a real-time PCR for detection of African swine fever virus E184L gene., 2020, 41(1): 8–14. 崔贝贝, 李霆, 仇松寅, 梅琳, 韩雪清, 吴绍强, 林祥梅. 基于E184L基因的非洲猪瘟病毒实时荧光定量PCR检测方法的建立. 动物医学进展, 2020, 41(1): 8–14.
[10] Wang SJ, Liu MF, Yan RQ, Ban FG, Zhao XL, Ma ZY, Wang HJ, Wang C, Zhao MJ. Establishment and application of real-time quantitative PCR assay for detection of classical Swine fever virus., 2017, 44(7): 2112–2118. 王淑娟, 刘梅芬, 闫若潜, 班付国, 赵雪丽, 马震原, 王华俊, 王翠, 赵明军. 猪瘟病毒实时荧光定量PCR检测方法的建立与应用. 中国畜牧兽医, 2017, 44(7): 2112–2118.
[11] Yan L, Song S, Zhang JH, Tang XL, Zhang HY, Guo KP. Establishment and application of droplet digital PCR for detection of African swine fever virus., 2020, 29(4): 344–351. 严礼, 宋晟, 张继红, 唐小兰, 张海韵, 郭焜鹏. 非洲猪瘟病毒微滴式数字PCR检测方法的建立与应用. 激光生物学报, 2020, 29(4): 344–351.
[12] Bergeron HC, Glas PS, Schumann KR. Diagnostic specificity of the African swine fever virus antibody detection enzyme-linked immunosorbent assay in feral and domestic pigs in the United States., 2017, 64(6): 1665–1668.
[13] Huang J, Huang JJ, Cheng XF, Li GX, Gao F, Tong GZ. Expression of p30 protein of African swine fever virus in prokaryotic cells and preparation of polyclonal antibodies., 2020, 28(5): 37–41. 黄剑, 徐晶晶, 程雪飞, 李国新, 高飞, 童光志. 非洲猪瘟病毒p30蛋白的原核表达及其多克隆抗体制备. 中国动物传染病学报, 2020, 28(5): 37–41.
[14] Liu XT, Wang ZY, Xin T, Guo XY, Jiang YT, Cui S, Yu HN, Zhu HF, Jia H. Prokaryotic expression and polyclonal antibody preparation and identification of African swine fever virus B438L protein., 2021, 48(3): 991–1000. 刘雪婷, 王召阳, 鑫婷, 郭晓宇, 姜一曈, 崔帅, 于海男, 朱鸿飞, 贾红. 非洲猪瘟病毒B438L蛋白的原核表达及其多克隆抗体的制备与鉴定. 中国畜牧兽医, 2021, 48(3): 991–1000.
[15] Bouhedda F, Autour A, Ryckelynck M. Light-up RNA aptamers and their cognate fluorogens: from their development to their applications., 2017, 19(1): 44.
[16] Rogers TA, Andrews GE, Jaeger L, Grabow WW. Fluorescent monitoring of RNA assembly and processing using the split-spinach aptamer., 2015, 4(2): 162–166.
[17] Chandler M, Lyalina T, Halman J, Rackley L, Lee L, Dang D, Ke WN, Sajja S, Woods S, Acharya S, Baumgarten E, Christopher J, Elshalia E, Hrebien G, Kublank K, Saleh S, Stallings B, Tafere M, Striplin C, Afonin KA. Broccoli fluorets: split aptamers as a user-friendly fluorescent toolkit for dynamic RNA nanotechnology., 2018, 23(12): 3178.
[18] Kikuchi N, Kolpashchikov DM. Split Spinach aptamer for highly selective recognition of DNA and RNA at ambient temperatures., 2016, 17(17): 1589–1592.
[19] Ausländer S, Fuchs D, Hürlemann S, Ausländer D, Fussenegger M. Engineering a ribozyme cleavage-induced split fluorescent aptamer complementation assay., 2016, 44(10): e94.
[20] Ong WQ, Citron YR, Sekine S, Huang B. Live cell imaging of endogenous mRNA using RNA-based fluorescence “Turn-On” probe., 2017, 12(1): 200–205.
[21] Bhadra S, Ellington AD. Design and application of cotranscriptional non-enzymatic RNA circuits and signal transducers., 2014, 42(7): e58.
[22] Gui HL, Jin QR, Zhang YJ, Wang XD, Yang YC, Shao CY, Cheng CY, Wei FF, Yang Y, Yang MH, Song HH. Development of an aptamer/fluorescence dye PicoGreen-based method for detection of fumonisin B1., 2015, 31(9): 1393–1400. 桂海娈, 金庆日, 张亚军, 王晓杜, 杨永春, 邵春艳, 程昌勇, 卫芳芳, 杨杨, 杨梦华, 宋厚辉. 基于荧光染料PicoGreen和核酸适配体的伏马毒素B1检测方法. 生物工程学报, 2015, 31(9): 1393–1400.
[23] Fernandez-Millan P, Autour A, Ennifar E, Westhof E, Ryckelynck M. Crystal structure and fluorescence properties of the iSpinach aptamer in complex with DFHBI., 2017, 23(12): 1788–1795.
[24] Huang H, Suslov NB, Li NS, Shelke SA, Evans ME, Koldobskaya Y, Rice PA, Piccirilli JA. A G-quadruplex- containing RNA activates fluorescence in a GFP-like fluorophore., 2014, 10(8): 686–691.
[25] Truong L, Ferré-D'Amaré AR. From fluorescent proteins to fluorogenic RNAs: tools for imaging cellular macromolecules., 2019, 28(8): 1374–1386.
[26] Matsunaga KI, Kimoto M, Lim VW, Thein TL, Vasoo S, Leo YS, Sun W, Hirao I. Competitive ELISA for a serologic test to detect dengue serotype-specific anti-NS1 IgGs using high-affinity UB-DNA aptamers., 2021, 11(1): 18000.
[27] Chen H, Park SG, Choi N, Kwon HJ, Kang T, Lee MK, Choo J. Sensitive detection of SARS-CoV-2 using a SERS-based aptasensor., 2021, 6(6): 2378–2385.
[28] Jeng SCY, Chan HHY, Booy EP, McKenna SA, Unrau PJ. Fluorophore ligand binding and complex stabilization of the RNA Mango and RNA Spinach aptamers., 2016, 22(12): 1884–1892.
[29] Filonov GS, Moon JD, Svensen N, Jaffrey SR. Broccoli: rapid selection of an RNA mimic of green fluorescent protein by fluorescence-based selection and directed evolution., 2014, 136(46): 16299–16308.
[30] Parra-Guardado AL, Sweeney CL, Hayes EK, Trueman BF, Huang Y, Jamieson RC, Rand JL, Gagnon GA, Stoddart AK. Development of a rapid pre-concentration protocol and a magnetic beads-based RNA extraction method for SARS-CoV-2 detection in raw municipal wastewater., 2021.
[31] Modh H, Scheper T, Walter JG. Aptamer-modified magnetic beads in biosensing., 2018, 18(4): 1041.
[32] Wei Y, Li K, Lu DR, Zhu HX. Optimization of CUT&Tag product recovery and library construction method., 2021, 43(4): 362–374. 韦晔, 李科, 卢大儒, 朱化星. CUT&Tag产物回收和建库方法的优化. 遗传, 2021, 43(4): 362–374.
[33] Wang Y, Xu LZ, Noll L, Stoy C, Porter E, Fu JP, Feng Y, Peddireddi L, Liu XM, Dodd KA, Jia W, Bai JF. Development of a real-time PCR assay for detection of African swine fever virus with an endogenous internal control., 2020, 67(6): 2446–2454.
[34] Fu JY, Zhang YP, Cai G, Meng G, Shi SB. Rapid and sensitive RPA-Cas12a-fluorescence assay for point-of-care detection of African swine fever virus., 2021, 16(7): e0254815.
[35] Wang XJ, Ji PP, Fan HY, Dang L, Wan WW, Liu SY, Li YH, Yu WX, Li XY, Ma XD, Ma X, Zhao Q, Huang XX, Liao M. CRISPR/Cas12a technology combined with immunochromatographic strips for portable detection of African swine fever virus., 2020, 3(1): 62.
Design and application of lightening Spinach-p54 RNA apatmer to detect African swine fever virus
Chengcheng Han1,2, Kai Xia1,3, Ruying Gong1, Xuhan Wu1, Lei Zhang1,2, Xinle Liang1,2
African swine fever virus (ASFV) is a major and prevalent pathogenic virus in livestock. Its outbreak in the recent years has seriously affected the national economic life. In this study, a specific chimeric RNA aptamer Spinach-p54 corresponding to the conserved protein p54 of ASFV, was designed and used in assembling a toehold RNA module with a truncated lightening Spinach switch. They were used to detect thegene. The aptamer bound rapidly and paired specifically with the ASFVtarget sequence and produced fluorescence with the addition of DFHBI substrate. The results showed that the limit of detection was as low as 200 nmol/Lfragment, under the conditions of 200 nmol/L Spinach-p54 aptamer and 37 ℃, suggesting that the current method can provide a simple, specific, sensitive, and rapid on-site detection of ASFV at the RNA level in the markets and farms.
aptamer; RNA; turn-on; African swine fever viru
2021-09-21;
2021-11-30
韩程程,博士研究生,研究方向:食品生物技术。E-mail: 707884766@qq.com
张蕾,博士,讲师,研究方向:分子免疫学。E-mail: zhanglei@zjsu.edu.cn
梁新乐,博士,教授,研究方向:食品生物技术。E-mail: dbiot@mail.zjgsu.edu.cn
10.16288/j.yczz.21-335
2021/12/13 18:40:49
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20211210.1302.002.html
(责任编委: 卢大儒)
