利用全基因组重测序技术鉴定五指山猪GHR突变体转基因插入位点
2022-01-06魏强奥岩杨漫漫陈涛韩虎张兴举王然夏秋菊姜芳芳李勇
魏强,奥岩,杨漫漫,陈涛,韩虎,张兴举,王然,夏秋菊,姜芳芳,李勇
利用全基因组重测序技术鉴定五指山猪突变体转基因插入位点
魏强1,2,奥岩3,杨漫漫1,2,陈涛1,2,韩虎1,2,张兴举1,2,王然1,2,夏秋菊1,姜芳芳1,2,李勇1,2
1. 深圳市华大农业应用研究院,深圳 518120 2. 深圳华大生命科学研究院,深圳市动物基因组辅助育种工程实验室,深圳 518083 3. 华中农业大学,农业动物遗传育种与繁殖教育部重点实验室,武汉 430070
转基因插入位点及其侧翼序列的分子特征对转基因动物新品系培育和生物安全评价至关重要。(growth hormone receptor)显性抑制突变体转基因猪(T274)是本实验室前期制备的一种表型显著的矮小症模型,目前已形成了多世代群体,但该模型中外源突变体的插入位点尚未鉴定。本研究利用BGI-seq500测序平台,对T274转基因猪进行全基因组重测序,获得超过289 Gb的数据(106×)。以转基因载体和猪基因组序列作为参考序列进行比对分析,获得该外源突变体的插入位点,并利用PCR扩增和Sanger测序进行验证。所有的分析数据都支持外源显性抑制突变体插入在五指山猪1号染色体269,513,538~269,514,371之间。序列保守性及功能元件分析表明,该插入位点可能位于转录激活相关的基因间区域。不同世代个体的7种组织中该外源突变体的表达水平和表达一致性都较高,说明该插入位点可以作为一个潜在的转基因“友好位点”。本研究表明全基因组重测序技术可以高效的鉴定转基因插入位点,该插入位点的获得为后期转基因猪新品系的培育奠定了基础,同时也为行业提供了一个可靠的基因组定点整合位点。
转基因插入位点;全基因组重测序;转基因猪;遗传稳定性
转基因是利用分子生物学方法将感兴趣的目标外源基因导入宿主细胞中,且该外源基因能在宿主基因组中稳定表达并遗传给下一代,利用该方法获得的动物称为转基因动物。自20世纪80年代以来,转基因技术得到了快速发展,目前已经获得了包括猪在内的多种转基因动物[1]。转基因技术主要用于基因功能研究,重要蛋白质生产,人类疾病动物模型制备等方面[2]。转基因动物对于家畜品种改良、动物疾病模型创制以及异种器官移植等方面的发展具有重要的推动作用[3]。家猪()在遗传、解剖和生理方面同人类有许多相似之处,被认为是异种器官移植的合适供体,利用转基因技术创制的疾病模型在人类疾病的治疗中具有十分重要的作用[4]。此外,在农业育种实践中,由于传统的家猪育种存在周期长、遗传资源有限等弊端,转基因技术可以快速突破物种限制,创制并培育符合人类健康需求的新品种(系)[5]。
转基因动物虽然为解决这些问题提供了有效方法,但仍存在许多问题,其中外源基因的稳定遗传和表达是最主要的制约因素之一,亟需开发更多转基因友好位点来解决这方面的问题。转基因友好位点是指在基因组中某些没有特定生理功能的DNA序列,其缺失和插入外源基因都不会对周围的基因结构及表达产生影响,且可以保障外源基因的稳定遗传和表达[6]。为了避免干扰内源基因正常表达,理想的转基因整合位点应该位于基因间区,且周围序列处于转录激活状态。在家猪基因组中,常见的友好位点包括等[7~9],然而在转基因动物制备过程中,外源基因的插入大多以随机整合的方式进行,而这类随机整合常常导致表达的不可预测性和表型的不稳定性[10]。因此,在转基因动物研究中,很重要的一项工作就是鉴定外源基因在基因组中的插入位点,然后再根据插入位点在基因组中位置分布来确定位点整合的规律。
目前在转基因插入位点鉴定研究中,外源基因整合位点鉴定的方法包括荧光原位杂交、反向PCR、半随机引物PCR、交错式热不对称PCR以及基因组测序方法等[11~14]。近年来,测序技术已成为解决生物学问题的热门技术,利用全基因测序技术鉴定转基因动植物插入位点的报道越来越多[15~20]。尽管如此,利用全基因组重测序技术鉴定转基因猪插入位点尚未见报道。
本研究对手工克隆(handmade cloning, HMC)产生的转基因五指山猪进行了全基因组重测序,经过序列比对与分析,获得了外源基因的插入位点(五指山猪1号染色体269,513,538~269,514,371之间);并发现在F1、F2代中该位点可以稳定遗传、稳定表达,提示该位点是一个潜在转基因友好位点。对该位点进行鉴定、稳定性表达研究,对转基因动物培育及应用具有一定的推动作用。
1 材料和方法
1.1 样本及基因组测序
显性抑制突变体(dominant-negativemutation,GHR)转基因猪(T274)是本实验室前期制备的1头表型显著的矮小症模型猪,其简要制备方法如下:构建pCAG-GHR-IRES--neo载体,转染到雄性五指山猪胎儿成纤维细胞系,用G418筛选出单克隆并进行传代、鉴定和保存。利用手工克隆技术,将去核的卵母细胞胞质体与阳性细胞进行融合,培养5~6 d后的重构胚经手术移植到代孕母猪体内,怀孕112 d后分娩,获得经PCR鉴定的阳性个体。由于该显性抑制突变体可以与野生竞争结合体内生长激素,抑制生长激素的下游通路,从而使个体出现矮小的症状[21]。后代群体以T274为系祖来构建(图1),具体方法如下:首先利用T274与野生五指山母猪配种获得F1代,F1代经PCR鉴定获得阳性留种个体;随后利用阳性F1半同胞互配获得F2代,对F2代同样经PCR鉴定进行选留。
采集T274个体血液,提取DNA后采用MGIEasyTMDNA文库制备试剂盒(深圳华大智造科技有限公司)进行文库构建,在BGIseq500平台进行PE100测序。F1代和F2代样本进行DNA提取后 –20℃保存,进行后续实验。

图1 T274及其后代系谱分析图

表1 插入位点鉴定引物信息
1.2 插入位置鉴定
将测序原始数据进行质量控制和过滤,去除:(1) N碱基含量超过5%的reads;(2)质量值低于20且比例超过30%的reads。利用SOAP1.5.0软件将过滤后的reads与猪参考基因组(sus11.1)进行比对,提取不完全比对的reads,再比对到pCAG-GHR载体序列上。提取既能比对到参考基因组又能比对到载体上的reads进行BLAST分析,确定插入位点的位置。
1.3 插入位点验证
根据截断reads的比对模式,结合其对应的PE reads,对插入序列进行拼接和比对,并根据拼接序列设计引物用于插入位点的鉴定。引物设计的原则是跨拼接区域:左侧鉴定引物的上游位于参考基因组上,下游位于载体序列上;右侧鉴定引物的上游位于载体序列上,下游位于参考基因组上。引物具体信息见表1。通过琼脂糖凝胶电泳和Sanger测序进行验证,检测T274及其阳性后代的序列插入情况。
1.4 外源基因的表达验证
随机选取T274后代F1、F2各3头,提取肝、心、肾、肺、肌肉、睾丸、附睾组织总RNA,利用TIANScript RT Kit cDNA第一链合成试剂盒(天根生化科技(北京)有限公司)反转录为cDNA,通过RT-PCR检测野生型生长激素受体基因(wild type, W)和转入突变型生长激素受体基因(GHR)表达情况。通过琼脂糖凝胶电泳检测。引物序列和PCR产物大小见表2。

表2 表达稳定性检测引物信息
2 结果与分析
2.1 全基因组测序结果
T274经过全基因组重测序后,共产生3067.8 Mb reads,通过SOAP1.5.0软件对原始数据进行过滤,获得clean data 289.5 Gb,Q20为96.85%,Q30为88.16%,结果显示测序数据质量较高。通过比对猪参考基因组,结果发现基因组比对率和覆盖率分别为98.19%和98.53%,GC含量为42.43%,测序深度约为106×。
2.2 插入位点鉴定
根据与参考基因组和载体的比对结果,再结合BLAST分析,本研究初步确定载体的插入位点位于五指山猪1号染色体269,513,538~269,514,371之间(图2A)。根据截断reads的比对模式,结合其对应的PE reads,对插入后的序列进行拼接和比对(图2:B,C),用于后续分析。
序列分析结果表明该插入区域位于猪(immediate early response 5 like)基因上游57,972 bp处,(chromosome 1homolog)基因下游 294,575 bp处,序列保守性分析发现该区域序列保守性较低(图3)。通过比对UCSC Genome Browser (https://genome.ucsc.edu/),发现插入位点与基因之间存在大量的候选顺式调控元件(candidate-regulatory elements, cCREs),如promoter- like signature、proximal enhancer-like signature、distal enhancer-like signature等,及丰富的表观修饰标记(图3)。这些结果说明该区域是一个转录激活区域,有利于外源基因的高效稳定表达。
2.3 插入位点验证
为了验证T274转基因序列的准确插入位置,本研究在插入位点两侧各设计1对引物:左侧断点扩增产物长度459 bp,右侧1732 bp;野生型预期仅能扩增出基因组序列1298 bp (表1)。电泳结果显示,T274左右两侧断点扩增长度与预期一致,并能够扩增出基因组序列,说明T274是转基因杂合型;野生型个体未能扩增左右两侧断点,仅能扩增出基因组序列,与预期一致(图4A)。随后,本研究对扩增片段进行Sanger测序,进一步将测序数据进行比对分析。结果发现该区域猪基因组发生834 bp的缺失,载体两端序列也发生了部分缺失,其中左侧的基因缺失944 bp,右侧PBR322_ori区域发生153 bp的缺失(图4:B,C)。这些结果证实基因组随机断裂和外源基因插入过程中基因组序列和外源载体序列都发生了部分丢失。
2.4 外源插入片段遗传稳定性分析
为了检测该插入位点的遗传稳定性,选择T274的F1代、F2代个体利用断点检测引物进行PCR扩增,其中左侧断点扩增产物长度为459 bp,右侧为1732 bp;野生型为1298 bp (图5红色箭头所示)。电泳结果显示,9个F1代个体(M103、M116、M131、M150、M151、M167、M169、M171、M172)左、右侧断点以及野生型检测引物都可以扩增出目标片段(图5),说明9个F1个体都获得了单等位基因插入。随后,本研究利用同样的方法对10个F2代个体(M611、M612、M614、M620、M631、M635、M640、M647、M651、M652)进行插入位点检测。电泳结果显示,10个F2代个体左侧、右侧断点以及野生型都能扩增出片段(图5)。所有转基因阳性后代的插入位点均与T274个体一致,说明该位点插入的外源基因可以稳定遗传。

图2 跨越基因组与载体融合位置的测序reads

图3 插入位点周围区域序列保守性及功能分析

图4 转基因插入位点左右断点鉴定及Sanger测序结果
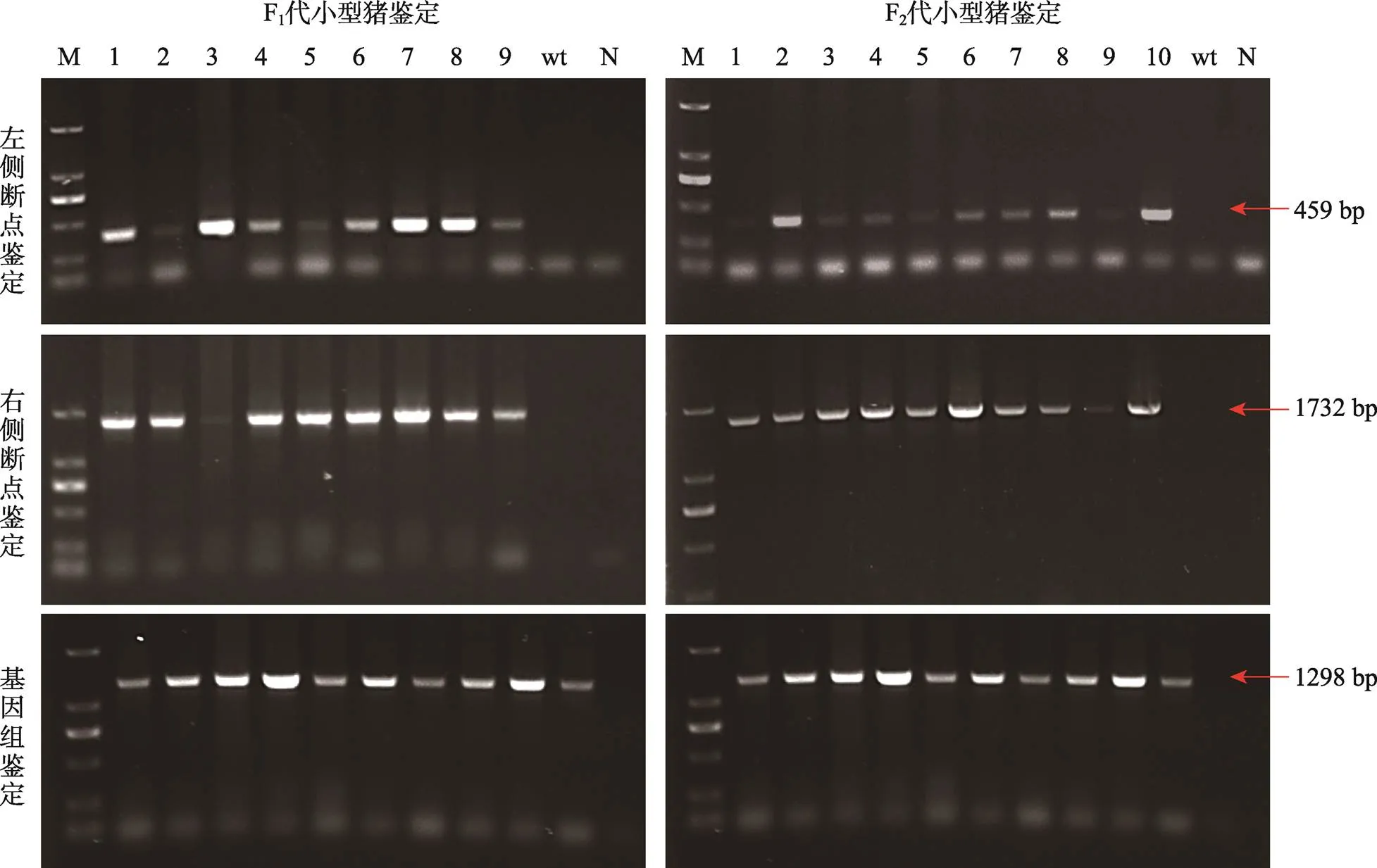
图5 T274后代插入位点遗传稳定性验证
2.5 插入位点表达稳定性分析
对插入位点周围序列分析表明,该位点位于潜在的基因表达激活区域,有利于外源基因的稳定高效表达。为了验证外源基因在不同世代、不同组织的表达稳定性,本研究在F1和F2代中随机选取杂合子,设计RT-PCR引物,检测内源基因(W)和插入基因(GHR)的表达情况,以作为内参,内源W作为对照,扩增片段大小分别为243 bp,201 bp和252 bp (图6,红色箭头所示)。结果显示,GHR在F1和F2后代的不同组织中均可稳定表达,且GHR的表达量要高于内源W(图6),后代个体的表达模式与T274类似。本研究结果与Ji等[19]研究结果一致。
3 讨论
转基因技术在家猪新品种培育以及生物医学模型制备等方面应用越来越广泛,特别是近年来与基因编辑技术的结合,进一步提升了转基因猪的制备效率。然而早期的转基因猪大多是通过显微注射或转基因克隆方法制备的,这些方法最大的劣势是插入位置的随机性,需要通过后代群体大规模的筛选才能获得稳定遗传的种群[22]。因此,插入位点的鉴定是后期转基因新品系培育的关键,同时友好插入位点的获得也有利于制备基因编辑介导的定点整合转基因动物。本研究利用全基因组重测序技术对先前研究获得GHR转基因猪(T274)进行插入位点鉴定,在106×测序深度下获得跨越基因组与载体的reads,说明高深度测序能捕获跨越断点的reads来满足位点鉴定需求。不过从经济性角度看,可以降低测序深度,一般来说10×以上就足够鉴定到插入位点,如转基因植物和转基因小鼠的结果显示8×以上测序深度都可以鉴定到插入位点[18,19]。

图6 GHR突变体在T274个体后代不同组织的表达情况
本研究利用比对reads的序列信息推测断点区域的融合序列,并利用Sanger测序进行验证。序列分析结果显示该突变体的插入位点位于五指山猪1号染色体269,513,538~269,514,371之间,该区域位于猪基因约58 kb上游,属于基因间区域,物种间保守性较低。进一步序列分析发现,插入区域基因组序列发生了834 bp的缺失,而载体两端的序列也发生了部分缺失。有趣的是,线性化载体的左侧原核抗性片段基因完全缺失,但保留了完整的外源基因表达框。这些缺失的形成在转基因小鼠中也很常见[19,23,24]。外源基因的随机插入主要是由于某些基因组区域的双链断裂(double- strand break, DSB)修复而形成,修复的过程会导致基因组区域InDel产生和外源基因片段的丢失[25]。此外,表观遗传沉默仍然是外源基因表达抑制的主要因素[26]。Yin等[27]的研究发现转基因表达的变异受到整合位点的影响,位置效应变异可引起表观遗传修饰的差异,例如DNA甲基化和组蛋白乙酰化。一般来说,原核序列越少插入片段的遗传稳定性越高;此外,插入在富含简单重复序列的基因组区域常常会导致外源基因的表达沉默[28]。本研究发现尽管插入位置的基因组序列保守性较低,但插入区域周围50 kb的基因组富含潜在的顺式调控元件和转录激活相关的表观修饰标记H3K27Ac。这说明该插入点位于转录活跃区域,有利于外源基因的稳定遗传和表达。最后,外源基因的插入也有可能导致基因组调控元件的删除或染色质三维空间构象的改变,进而影响插入位点周围内源基因的表达,如转基因小鼠实验中,即使内源基因距离插入位点超过500 kb,依然会受到外源插入表达框的影响,导致内源基因表达的下调[29]。本研究中,也发生了834 bp基因组片段的丢失,但该序列物种间保守性很低(数据未展示),参与表达调控的可能性较低。尽管如此,该外源插入基因对区域染色质三维空间构象和附近内源基因表达的影响还需要后期实验验证。
本研究通过PCR方法检测了T274后代F1和F2代的左右两侧断点序列,发现外源突变体在不同世代可以稳定遗传。同时,RT-PCR的结果也表明GHR表达框在该区域可以高效、稳定表达,且不同组织的表达一致性较高。转基因猪T274个体是通过CAG强启动子驱动GHR表达,该启动子是普遍型表达[30],这与本研究获得的不同组织表达效率结果一致。与目前已鉴定到的“友好位点”类似,不同组织间外源基因表达的效率相对一致[5]。此外,该区域高效表达GHR可以抑制内源性W的功能,从表达水平看,突变体表达高于野生型,这也是T274及其后代体重在15~30 kg之间的原因[21]。遗传稳定性和高效表达的数据显示该区域与序列分析预测的类似,属于较为理想的转录激活区域,可以作为候选的转基因友好位点。未来,结合基因编辑技术,人们可以将各种感兴趣的目的基因定点插入到该位点,获得稳定遗传和表达的转基因或基因修饰猪,避免随机整合带来的诸多问题。
[1] Maksimenko OG, Deykin AV, Khodarovich YM, Georgiev PG. Use of transgenic animals in biotechnology: prospects and problems., 2013, 5(1): 33–46.
[2] Kong QR, Wu ML, Zhang L, Wang F, Yin Z, Mu YS, Liu ZH. Transgene insertion affects transcription and epigenetic modification of flanking host sequence in transgenic pigs., 2011, 57 Suppl: OL1505– 1512.
[3] Clark J, Whitelaw B. A future for transgenic livestock., 2003, 4(10): 825–833.
[4] Niemann H, Petersen B. The production of multi- transgenic pigs: update and perspectives for xenotransplantation.,2016, 25(3): 361–374.
[5] Ruan JX, Xu J, Chen-Tsai RY, Li K. Genome editing in livestock: are we ready for a revolution in animal breeding industry?,2017, 26(6): 715–726.
[6] Friedrich G, Soriano P. Promoter traps in embryonic stem cells: a genetic screen to identify and mutate developmental genes in mice., 1991, 5(9): 1513–1523.
[7] Li XP, Yang Y, Bu L, Guo XG, Tang CC, Song J, Fan NN, Zhao BT, Ouyang ZM, Liu ZM, Zhao Y, Yi XL, Quan LQ, Liu SC, Yang ZG, Ouyang HS, Chen YE, Wang Z, Lai LX. Rosa26-targeted swine models for stable gene over- expression and Cre-mediated lineage tracing.,2014, 24(4): 501–504.
[8] Ruan JX, Li HG, Xu K, Wu TW, Wei JL, Zhou R, Liu ZG, Mu YL, Yang SL, Ouyang HS, Chen-Tsai RY, Li K. Highly efficient CRISPR/Cas9-mediated transgene knockin at the H11 locus in pigs.,2015, 5: 14253.
[9] Ma LY, Wang YZ, Wang HT, Hu YQ, Chen JY, Tan T, Hu M, Liu XJ, Zhang R, Xing YM, Zhao YQ, Hu XX, Li N. Screen and verification for transgene integration sites in pigs.,2018, 8(1): 7433.
[10] Yang DS, Wang CE, Zhao BT, Li W, Ouyang Z, Liu ZM, Yang HQ, Fan P, O'Neill A, Gu WW, Yi H, Li SH, Lai LX, Li XJ. Expression of Huntington's disease protein results in apoptotic neurons in the brains of cloned transgenic pigs., 2010, 19(20): 3983–3994.
[11] Lu YF, Tian C, Deng JX. Research progress in identification technology of genetically modified animals., 2000, 20(3): 60–61.卢一凡, 田靫, 邓继先. 转基因动物鉴定技术的研究进展. 生物工程进展, 2000, 20(3): 60–61.
[12] Fujimoto S, Matsunaga S, Murata M. Mapping of T-DNA and Ac/Ds by TAIL-PCR to analyze chromosomal rearrangements.,2016, 1469: 207–216.
[13] Groenen MAM, Archibald AL, Uenishi H, Tuggle CK, Takeuchi Y, Rothschild MF, Rogel-Gaillard C, Park C, Milan D, Megens HJ, Li ST, Larkin DM, Kim H, Frantz LAF, Caccamo M, Ahn H, Aken BL, Anselmo A, Anthon C, Auvil L, Badaoui B, Beattie CW, Bendixen C, Berman D, Blecha F, Blomberg J, Bolund L, Bosse M, Botti S, Bujie Z, Bystrom M, Capitanu B, Carvalho-Silva D, Chardon P, Chen C, Cheng R, Choi SH, Chow W, Clark RC, Clee C, Crooijmans RPMA, Dawson HD, Dehais P, De Sapio F, Dibbits B, Drou N, Du ZQ, Eversole K, Fadista J, Fairley S, Faraut T, Faulkner GJ, Fowler KE, Fredholm M, Fritz E, Gilbert JGR, Giuffra E, Gorodkin J, Griffin DK, Harrow JL, Hayward A, Howe K, Hu ZL, Humphray SJ, Hunt T, Hornshøj H, Jeon JT, Jern P, Jones M, Jurka J, Kanamori H, Kapetanovic R, Kim J, Kim JH, Kim KW, Kim TH, Larson G, Lee K, Lee KT, Leggett R, Lewin HA, Li YR, Liu WS, Loveland JE, Lu Y, Lunney JK, Ma J, Madsen O, Mann K, Matthews L, McLaren S, Morozumi T, Murtaugh MP, Narayan J, Nguyen DT, Ni PX, Oh SJ, Onteru S, Panitz F, Park EW, Park HS, Pascal G, Paudel Y, Perez-Enciso M, Ramirez-Gonzalez R, Reecy JM, Rodriguez-Zas S, Rohrer GA, Rund L, Sang YM, Schachtschneider K, Schraiber JG, Schwartz J, Scobie L, Scott C, Searle S, Servin B, Southey BR, Sperber G, Stadler P, Sweedler JV, Tafer H, Thomsen B, Wali R, Wang J, Wang J, White S, Xu X, Yerle M, Zhang GJ, Zhang JG, Zhang J, Zhao SH, Rogers J, Churcher C, Schook LB. Analyses of pig genomes provide insight into porcine demography and evolution.,2012, 491(7424): 393–398.
[14] Meurens F, Summerfield A, Nauwynck H, Saif L, Gerdts V. The pig: a model for human infectious diseases.,2012, 20(1): 50–57.
[15] Park D, Park SH, Ban YW. Kim YS, Park KC, Kim NS, Kim JK, Choi IK. A bioinformatics approach for identifying transgene insertion sites using whole genome sequencing data.,2017, 17(1): 67.
[16] Park D, Kim D, Jang G, Lim J, Shin YJ, Kim J, Seo MS, Park SH, Kim JK, Kwon TH, Choi IY. Efficiency to discovery transgenic loci in GM rice using next generation sequencing whole genome re-sequencing.,2015, 13(3): 81–85.
[17] Niu L, He HL, Zhang YY, Yang J, Zhao QQ, Xing GJ, Zhong XF, Yang XD. Efficient identification of genomic insertions and flanking regions through whole-genome sequencing in three transgenic soybean events.,2021, 30(1): 1–9.
[18] Guo BF, Guo Y, Hong HL, Qiu LJ. Identification of genomic insertion and flanking sequence of G2-EPSPS and GAT transgenes in soybean using whole genome sequencing method., 2016, 7: 1009.
[19] Ji Y, Abrams N, Zhu W, Salinas E, Yu ZY, Palmer DC, Jailwala P, Franco Z, Roychoudhuri R, Stahlberg E, Gattinoni L, Restifo NP. Identification of the genomic insertion site of Pmel-1 TCR α and β transgenes by next- generation sequencing.,2014, 9(5): e96650.
[20] Jacobsen JC, Erdin S, Chiang C, Hanscom C, Handley RR, Barker DD, Stortchevoi A, Blumenthal I, Reid SJ, Snell RG, MacDonald ME, Morton AJ, Ernst C, Gusella JF, Talkowski ME. Potential molecular consequences of transgene integration: the R6/2 mouse example.,2017, 7: 41120.
[21] Li FD, Li Y, Liu H, Zhang XJ, Liu CX, Tian K, Bolund L, Dou HW, Yang WX, Yang HM, Staunstrup NH, Du YT. Transgenic Wuzhishan minipigs designed to express a dominant-negative porcine growth hormone receptor display small stature and a perturbed insulin/IGF-1 pathway.,2015, 24(6): 1029–1042.
[22] Liu W, Lu G. Progress in technology of transgenic animal models., 2001, 23(3): 289–291.刘薇, 卢光. 转基因动物技术的研究进展. 遗传, 2001, 23(3): 289–291.
[23] Chandler KJ, Chandler RL, Broeckelmann EM, Hou Y, Southard-Smith EM, Mortlock DP. Relevance of BAC transgene copy number in mice: transgene copy number variation across multiple transgenic lines and correlations with transgene integrity and expression., 2007, 18(10): 693–708.
[24] Le Saux A, Houdebine LM, Jolivet G. Chromosome integration of BAC (bacterial artificial chromosome): evidence of multiple rearrangements.,2010, 19(5): 923–931.
[25] Hu W, Zhu ZY. Research progress of BAC and genetic modification., 2001, 21(4): 3–7.胡炜, 朱作言. BAC及其转基因研究进展. 生物工程进展, 2001, 21(4): 3–7.
[26] Kong QR, Wu ML, Wang ZK, Zhang XM, Li L, Liu XY, Mu YS, Liu ZH. Effect of trichostatin A and 5-Aza-2'- deoxycytidine on transgene reactivation and epigenetic modification in transgenic pig fibroblast cells.,2011, 355(1–2): 157–165.
[27] Yin Z, Kong QR, Zhao ZP, Wu ML, Mu YS, Hu K, Liu ZH. Position effect variegation and epigenetic modification of a transgene in a pig model., 2012, 11(1): 355–369.
[28] Kong QR, Liu ZH. Inheritance and expression stability of transgene in transgenic animals., 2011, 33(5): 504–511.孔庆然, 刘忠华. 外源基因在转基因动物中遗传和表达的稳定性. 遗传, 2011, 33(5): 504–511.
[29] Laboulaye MA, Duan X, Qiao M, Whitney IE, Sanes JR. Mapping transgene insertion sites reveals complex interactions between mouse transgenes and neighboring endogenous genes., 2018, 11: 385.
[30] Niwa H, Yamamura K, Miyazaki J. Efficient selection for high-expression transfectants with a novel eukaryotic vector.,1991, 108(2): 193–199.
Identification of genomic insertion of dominant-negativemutation transgenes in Wuzhishan pig using whole genome sequencing method
Qiang Wei1,2, Yan Ao3, Manman Yang1,2, Tao Chen1,2, Hu Han1,2, Xingju Zhang1,2, Ran Wang1,2, Qiuju Xia1, Fangfang Jiang1,2, Yong Li1,2
Molecular characterization of sequences flanking the transgenic insertion site is essential for safety assessment and breeding a novel strain of transgenic animal.The growth hormone receptor (GHR) dominant-negative mutant transgenic pig (T274) is a Laron syndrome model, which was previously established in our laboratory and maintained in multi-generational populations. However, the insertion site of the exogenous mutant in the genome of this model has not yet been identified. In this experiment, the BGI-seq500 sequencing platform was used to re-sequence the whole genome of the T274 model. More than 289 Gb of data (106×) was obtained. Then, the transgenic vector and porcine genome sequences were used as references for alignment analysis, and the insertion site of the exogenousmutant was obtained, and verified by PCR amplification and Sanger sequencing. The results showed that the insertion site of the exogenous mutant was located in chromosome 1 (269,513,538-269,514,371) of the Wuzhishan pig. Conservation and functional element analysis indicated that the insertion site could be located in the intergenic region associated with transcription activation. The expression levels of the exogenous mutant in seven tissues of offspring in different generations are high, indicating that the insertion site can be used as a potential safe site for transgene targeting. This study shows that whole-genome resequencing can efficiently identify transgenic insertion sites. The insertion site identified in T274 could be useful in establishing and breeding new transgenic pig lines, as well as a reliable safe site for transgenic pig research.
transgenic insertion site; whole genome sequencing; transgenic pig; genetic stability
2021-06-24;
2021-08-30
广东省重点领域研发计划项目(编号:2018B020203002)和广东省基础与应用基础研究基金项目(编号:2019B1515210028)资助[Supported by the Science and Technology Innovation Strategy Projects of Guangdong Province (No. 2019B020203002) and Guangdong Province Basic and Applied Basic Research Fund (No. 2019B1515210028)]
魏强,硕士,工程师,研究方向:动物基因编辑,动物遗传育种。E-mail: weiqiang@genomics.cn
李勇,博士,副研究员,研究方向:动物基因编辑,动物遗传育种。E-mail: liyong3@genomics.cn
10.16288/j.yczz.21-221
2021/10/19 12:23:31
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20211019.0935.002.html
(责任编委: 李明洲)
