新生荷斯坦犊牛后肠道微生物母源传递特征的研究
2021-12-08杨敏娜高伟星曲永利
杨敏娜 朱 焕 高伟星 曲永利
(黑龙江八一农垦大学动物科技学院,大庆 163000)
对人类与啮齿类动物肠道早期定植菌群结构和来源的分析显示,哺乳动物肠道微生物的早期定植具有很强的母源特征,母代来源的微生物在子代肠道菌群形成过程中发挥了重要作用[1]。肠道微生物在早期发育过程中对天然免疫系统的成熟具有重要影响[2]。此外,胃肠道微生物区系具有重要的代谢和营养作用[3],并且影响动物的健康和生产性能,对动物的整体福利有很大的影响[4-5]。
然而,哺乳动物新生儿肠道微生物的起源仍然存在争议。有研究报道在妊娠期母体不同部位均发现了微生物区系,包括羊水、脐带血、子宫内膜、胎膜[6-9]等部位,对应的婴儿胎粪中也发现微生物的存在[10]。Quercia等[11]的研究结果表明,从胎儿时期起,母马不同部位的微生物群落在马驹肠道中进行了定植。Ferretti等[12]通过菌株分析也表明某些菌株可能从母代垂直传递到子代并进行定植。以上这些研究说明了在妊娠期间,母体各个部位的微生物可能通过某种内源途径传递给子代。目前有关子代微生物母源传递的研究绝大部分是关于人的,奶牛相关研究的报道很少。本试验利用16S rDNA扩增子测序技术,结合SourceTracker方法,对母牛粪便、初乳、胎盘、羊水、脐带和胎粪的微生物区系进行分析,探讨母牛不同部位的微生物来源对犊牛后肠道微生物区系的影响,深入了解母牛微生物对犊牛肠道定植的作用,将有助于设计对早期犊牛肠道微生物群落结构干预的策略,为改善犊牛健康状况,特别是在免疫系统发育方面奠定科学的理论基础。
1 材料与方法
1.1 试验动物
本试验在黑龙江省某荷斯坦奶牛场进行,选取15头健康且预产期相近的经产奶牛(2胎)进行试验。试验期内所有的母牛均在同一牛舍内饲养,并在预产期前1周左右集体转移至产房。所有妊娠期的奶牛均在农场工人的监督下产犊。按照试验采样标准要求,最终选取6对荷斯坦母牛及其所生犊牛的样品进行研究。所有样品均由同一名经验丰富的兽医取样。
1.2 样品采集
母牛分娩后立即对所有样品进行采集。胎盘与脐带取样:采用无菌设备对胎盘(脐带)快速处理,从胎盘(脐带)内部切取样本,尽量减少环境中微生物污染的可能性。羊水取样:当羊水囊清晰可见时,使用无菌手套和20 mL无菌注射器,用70%乙醇拭子擦拭胎膜后,采用穿刺法取出至少10 mL羊水。粪便取样:用无菌手术手套对母牛和尚未被饲喂初乳的新生犊牛进行直肠取样。初乳取样:在分娩后,立即在犊牛第1次哺乳前,乳房用肥皂水消毒后,随后用无菌生理盐水进行擦洗,通过弃去头几滴乳汁来冲洗乳腺导管,收集5 mL的乳汁于无菌管中。所有样品均放于无菌管中,分装编号后迅速投入液氮中保存备用。
1.3 DNA提取
所有工作都在严格控制、分离和无菌的工作场所进行。采用十六烷基三甲基溴化铵(CTAB)方法对样本的基因组DNA进行提取,通过琼脂糖凝胶电泳检测DNA的纯度和浓度,将离心管中适量的样本DNA用无菌水稀释样本至1 ng/μL。
1.4 PCR扩增和产物的混样和纯化
以稀释后的基因组DNA为模板,通过使用带Barcode的特异引物和高效高保真酶对测序区域进行PCR。用2%浓度的琼脂糖凝胶对PCR产物进行电泳检测,并用Qiagen公司提供的胶回收试剂盒回收产物。
1.5 文库构建和高通量测序
1.6 数据分析
数据下机后,从中拆分出各样本数据,通过使用FLASH[13]对每个截去Barcode和引物序列的样本reads进行拼接得到原始Tags数据;利用Qiime 1.9.1软件[14]对Tags进行质控,使用物种注释数据库对Tags序列[15]进行比对检测,并去除其中的嵌合体序列,得到有效数据。利用Uparse 7.0.1001软件[16]将所有序列以97%的一致性聚类成为操作分类单元(OTU),用Mothur方法与SILVA132[17]的SSUrRNA数据库[18]对OTU分析中出现频数最高的代表序列从门到属进行物种注释(设定阈值为0.81)。
为了评估测序深度和生物多样性丰富度,建立了所有样品的稀疏曲线。通过R 3.0.3软件中的VennDiagram包绘制花瓣图。计算样品间Shannon指数和Chao1指数,并用wilcox秩和检验进行差异显著性分析,P>0.05为差异不显著,P<0.05为差异显著,P<0.01为差异极显著。基于Unweighted Unifrac距离来进行主坐标分析(PCoA),评价不同样本类型之间微生物区系的结构差异[19]。本研究通过R 3.4.1软件中的SourceTracker包分析母牛不同部位微生物来源对犊牛胎粪中微生物的相对贡献度。结果以平均值±标准差表示。
2 结果与分析
2.1 特异性OTU
从胎盘、脐带、羊水、母牛粪便、胎粪和初乳等6类36个样品中产生了3 447 210条原始读码。将有效数据以97%的一致性进行OTU聚类。图1为所有样本类型的花瓣图,其中羊水的特异性OTU最多(801个),其次是初乳(641个)、胎盘(630个)、胎粪(343个)、脐带(266个)、母牛粪便(97个),所有组共享916个OTU。
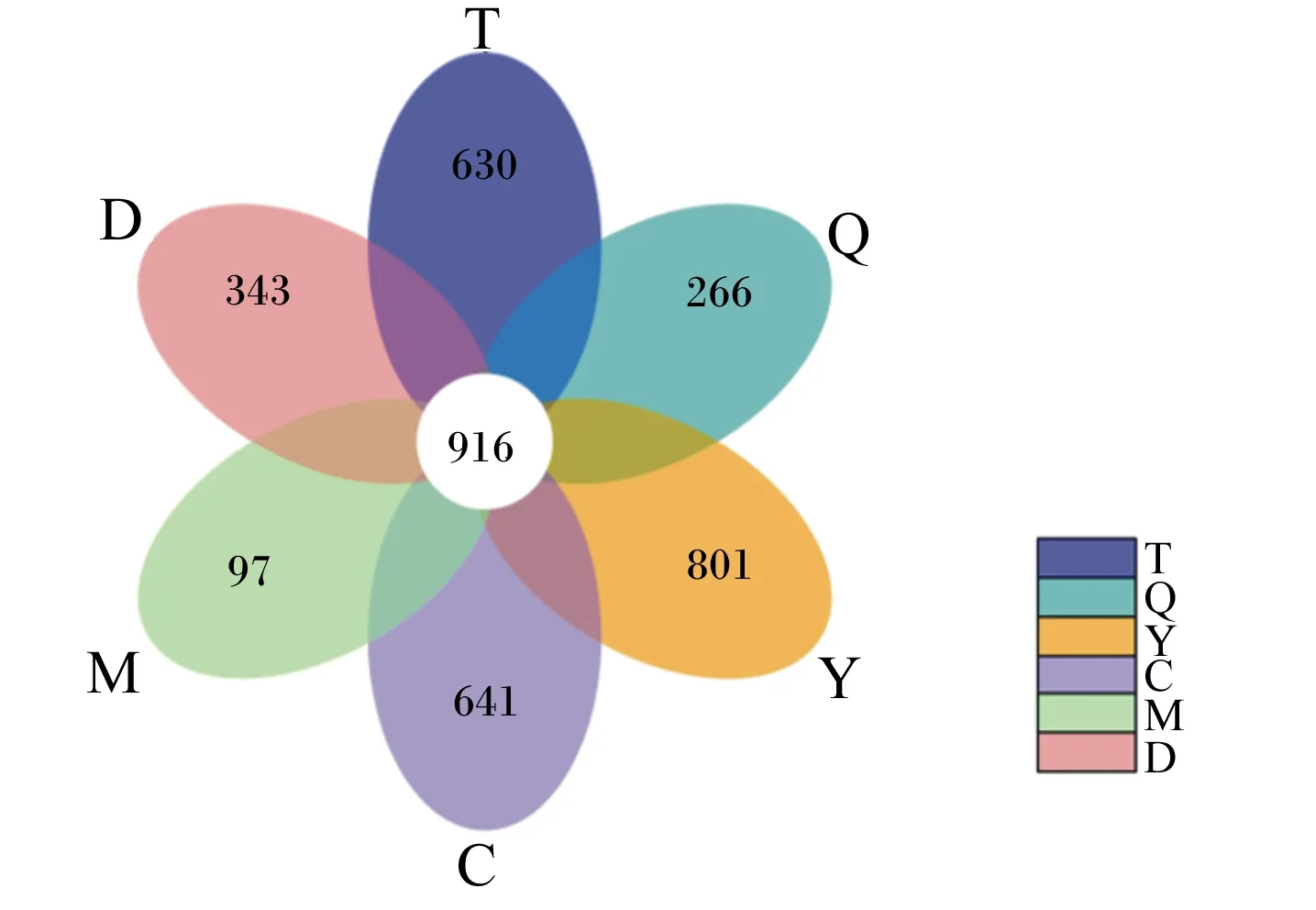
T:胎盘;Q:脐带;Y:羊水;C:初乳;M:母牛粪便;D:胎粪。下图同。T:placenta;Q:umbilical cord;Y:amniotic fluid;C:colostrum;M:cow feces;D:meconium.The same as below.图1 OTU花瓣图Fig.1 OTU petals diagram
2.2 母子不同类型样本微生物区系的α多样性
Shannon多样性指数曲线趋于平稳(图2),表明本试验的测序深度足以捕捉具有代表性的微生物多样性。在取样深度为424 824时,用wilcox秩和检验对Shannon指数和Chao1指数进行差异显著性分析(表1)。Shannon指数差异显著性分析表明,除胎粪和羊水、胎粪和胎盘组间差异显著(P<0.05)外,其余样本类型间差异均不显著(P>0.05)。Chao1指数差异显著性分析表明,胎粪除了和母牛粪便差异不显著(P>0.05)外,和其余母牛样本类型差异均显著或极显著(P<0.05或P<0.01);胎粪和初乳、胎盘、脐带样本类型间差异显著(P<0.05),胎粪和羊水间差异极显著(P<0.01);母牛粪便和其他母体样本类型差异极显著(P<0.01);其余样本类型间差异均不显著(P>0.05)。
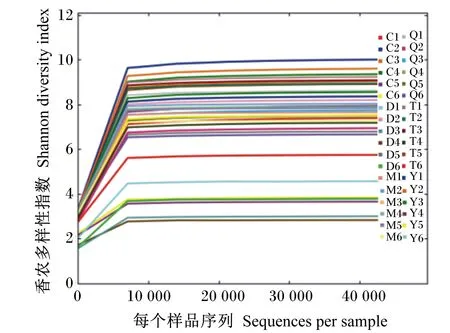
图2 Shannon多样性指数稀释曲线Fig.2 Rarefaction curves of Shannon diversity index
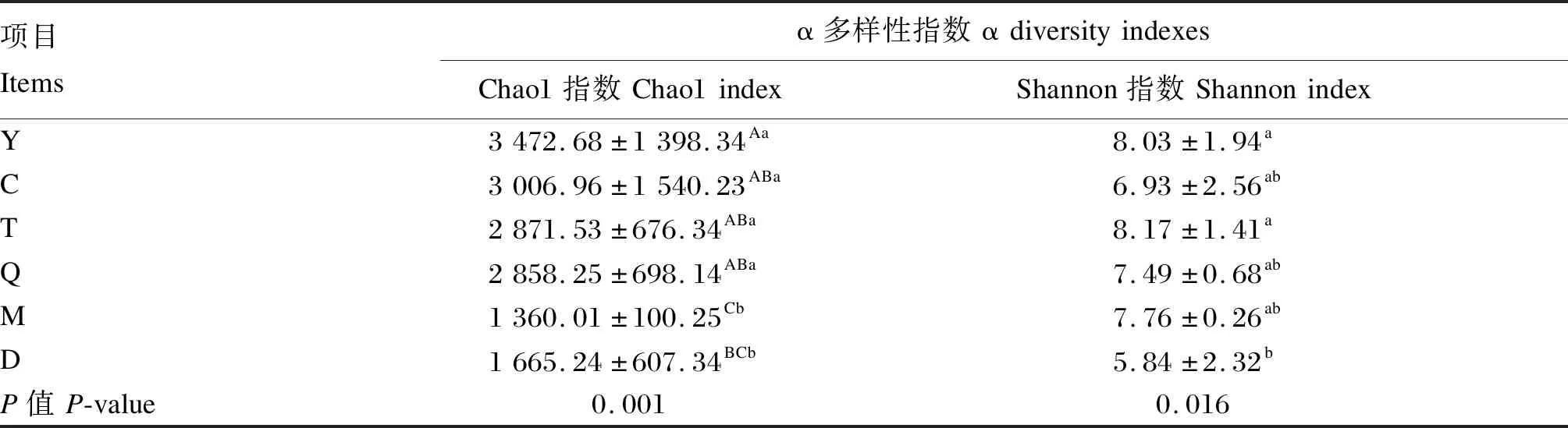
表1 不同样本类型的α多样性分析Table 1 α diversity analysis of different sample types
2.3 母子不同类型样本微生物区系的组成特征
将本试验所得有效序列在门和属水平上进行物种注释和统计(图3和图4),结果表明,在门水平上,羊水、初乳、脐带和和胎粪的主要优势菌门相同,为变形菌门(Proteobacteria),次要优势菌门也相同,为厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)。胎盘的主要优势菌门为Proteobacteria和Firmicutes,Bacteroidetes和Actinobacteria是胎盘的次要优势菌门。母牛粪便主要是由Firmicutes和Bacteroidetes组成。在属水平上,假单胞菌属(Pseudomonas)是羊水、初乳、胎盘和脐带中相对丰度最高的优势菌属,但在母牛粪便和胎粪中是第2优势菌属。Limnobacter和短波单胞菌属(Brevundimonas)是羊水、初乳和胎盘中的次要优势菌属,脐带、母牛粪便和胎粪中也含有这些菌属,但是这些菌属的相对丰度都很低。在母牛粪便中,所有物种的相对丰度都很低,不动杆菌属(Acinetobacter)是相对丰度最高的菌属。嗜盐单胞菌属(Halomonas)是胎粪中相对丰度最高的菌属。
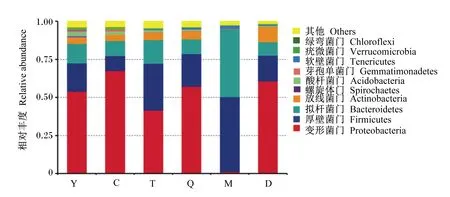
图3 门水平上的物种相对丰度Fig.3 Relative abundances of species at phylum level
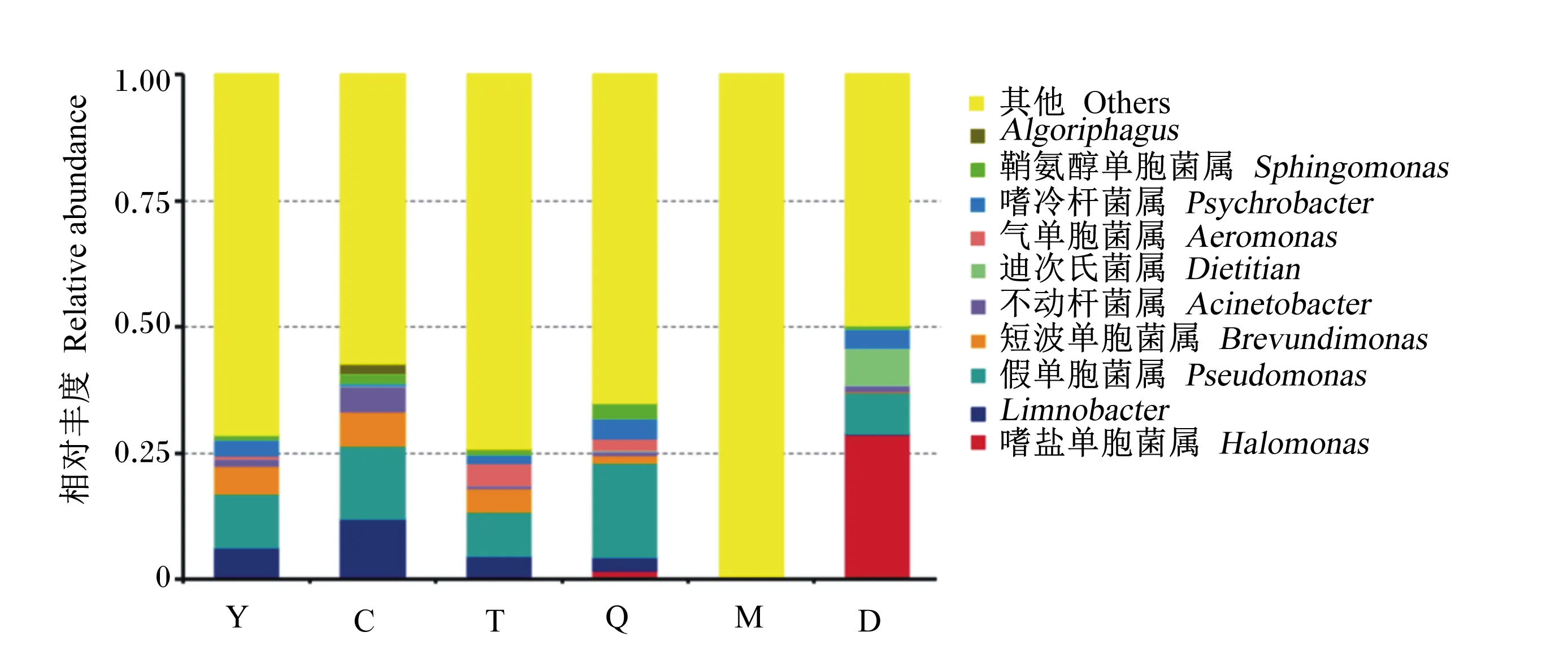
图4 属水平上的物种相对丰度Fig.4 Relative abundances of species at genus level
2.4 母子不同类型样本微生物区系的β多样性特征分析
利用基于Unweighted Unifrac距离来进行PCoA的方法,评价胎粪与母体样本微生物区系结构的特征差异(图5)。母牛粪便的个体聚集程度最高,表明母牛粪便的组内相似度比其他样本类型都高。母牛粪便和胎粪都不与其他母体样本类型位置相似,表明母体粪便和胎粪都有独特的微生物区系。羊水和初乳、胎盘和脐带的位置相似,表明羊水和初乳、胎盘和脐带微生物区系结构相似。
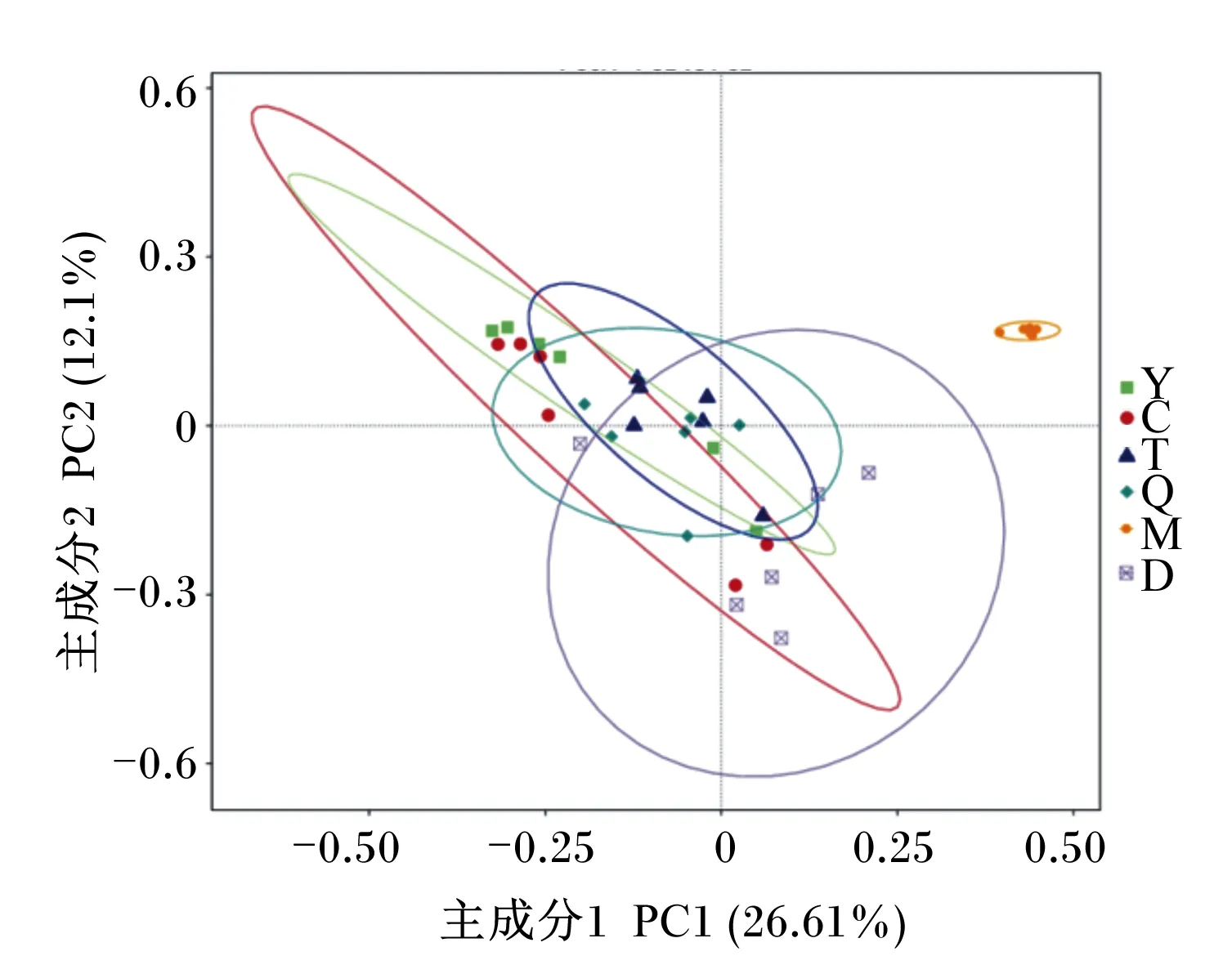
图5 PCoA聚类分析Fig.5 Cluster analysis by PCoA
2.5 用SourceTracker预测胎粪微生物的母体来源
利用基于贝叶斯方法的SourceTracker分析估计母体不同部位潜在的微生物来源对新生犊牛胎粪中微生物贡献的比例。SourceTracker分析结果表明,母牛的胎盘、脐带、羊水、初乳和粪便中的微生物均对新生犊牛胎粪菌群的定植有贡献,按贡献比例由高到低依次为脐带(23.45%)、胎盘(16.35%)、初乳(14.29%)、羊水(11.45%)、母牛粪便(9.93%)。其胎粪群落主要由来自脐带的微生物组成;未知来源微生物贡献占24.53%(图6)。
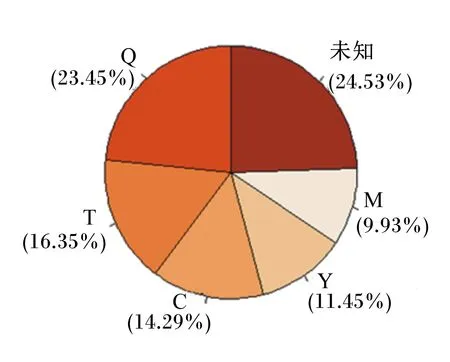
图6 SourceTracker分析不同微生物来源对犊牛胎粪微生物的贡献度Fig.6 SourceTracker analysis of potential microbial sources of meconium microbiota of calves
3 讨 论
为了尽量减少污染微生物样本的机会,在犊牛出生后立即对母牛粪便、初乳、羊水、胎盘、脐带和胎粪样本进行无菌采集。与以往收集新生犊牛出生后几天粪便样本的研究不同,本研究分析的是胎粪样本,其代表犊牛出生时的肠道内容物,不受饮食等环境影响[20]。在此研究中,6种样本类型都检测到了微生物群,α多样性的显著性差异分析表明,犊牛胎粪除了和羊水、胎盘间微生物群落多样性差异显著外,和其余母体样本类型间微生物群落多样性差异均不显著;胎粪除了和母牛粪便间微生物群落丰富度差异不显著外,和其余母牛样本类型微生物丰富度差异均显著或极显著。从β多样性分析可以看出每种样本类型的微生物区系都表现出不同的特征。此外,在所有样本类型中均发现了只出现于某一样本类型的特异性OTU。胎粪中有343个特异性OTU,这些OTU并非来自本研究中的母体部位,很有可能来自于其他未被研究的母体部位。胎盘、脐带和羊水的第3优势菌门均是Bacteroidetes,此菌门也是Moore等[21]研究中青年母牛子宫内优势菌门之一。在初乳中发现了相对丰度较小的Halomonas,其不仅是鹿乳汁微生物的优势微生物之一,也是奶酪表面的优势微生物,其可能与乳汁中其他微生物产生互作,并且受到乳汁环境的影响[22]。本研究发现胎盘和胎粪中均存在乳杆菌属(Lactobacillus)。Satokari等[23]也在胎盘及胎粪中发现乳杆菌属(Lactobacillus),说明在妊娠期间母体的羊水、脐带血、胃肠道等可能通过某种内源途径将自身菌群垂直传递给子代。SourceTracker分析表明母牛的粪便、胎盘、脐带、羊水和初乳中的微生物均是犊牛胎粪菌群的主要来源,其中脐带微生物对犊牛胎粪微生物定植的贡献最大。产生这种结果的原因可能是由于产前母代和子代的主要联系是脐带,所以其不仅是母代和子代之间进行代谢废物和营养物质交换的通道,而且有可能是母代和子代之间进行微生物传递的主要途径。羊水中的微生物可能是通过子代吞咽羊水的过程进入子代肠道并进行定植,最终成为胎粪生态系统的一部分。这一结果和He等[24]发现羊水是胎粪菌群主要来源的结论一致。以上研究表明胎粪微生物区系可能来自于多个母体部位[11-12,24],具有强烈的母源特征,但母体不同部位间微生物的传递途径仍不清楚。本研究发现犊牛胎粪中未知来源微生物(24.53%)贡献占比较高,但由于本研究母体样本类型的局限性,需要进一步研究来确定未知来源微生物的母体来源。
传统观点认为新生犊牛肠道微生物的最初定植来源于出生后2 d内初乳或其他外界环境中的微生物。但本试验数据表明,胎粪菌群是在胎儿生命期间已经定植。初乳中的微生物可能是通过某种途径到达胎儿肠道,而不是在犊牛出生后通过吮吸乳汁的方式直接对胎粪微生物区系产生影响,其更有可能的原因是胎粪和初乳微生物有共同的母体来源。这一观点与上述不同母体部位的菌群通过某种传递途径进入子代肠道进行定植的观点一致,但具体途径也有待进一步研究。
4 结 论
在本研究中,围产期母牛胎盘、脐带、羊水、初乳和粪便中的微生物均是犊牛肠道微生物的主要来源,其中脐带微生物对胎粪微生物的定植贡献最大;犊牛胎粪中还有许多未知来源且相对贡献度(24.53%)较高的微生物,其母体来源有待深入研究。
