山溪鲵属的水样环境DNA分析方法的建立
2021-12-04马红英张红星王启军
马红英,张 晗,邓 捷,赵 虎,孔 飞,姜 维,张红星,王启军
山溪鲵属的水样环境DNA分析方法的建立
马红英,张 晗,邓 捷,赵 虎,孔 飞,姜 维,张红星,王启军*
(陕西省动物研究所,陕西省秦岭珍稀濒危动物保育重点实验室,西安 710032)
通过优化建立一套符合山溪鲵属物种环境DNA(environmental DNA,eDNA)分析的操作方法,为后期山溪鲵属资源调查及保护提供理论依据。以太白山溪鲵为研究对象,在室内养殖条件下,进行水样过滤及目标物种线粒体细胞色素b(Cytb)基因的扩增,根据测序序列构建系统发育树,来判定水样DNA中目标物种的归属。结果表明:(1)通过酒精直接浸泡、锡箔纸包裹、唾液收集管收集这3种方法对过滤养殖水后的滤膜进行贮存,发现酒精直接浸泡法可增加后期eDNA的提取量;(2)在进行目的片段的PCR扩增时发现高保真Mix的扩增成功率大于普通Mix;(3)应用属间特异引物进行不同物种扩增后,可通过系统发育分析来确定种间的分类。通过优化首次建立了山溪鲵属物种的水样eDNA分析方法,可为后期生活在相同水质且种间分化较小的物种进行eDNA分析提供参考。
环境DNA;山溪鲵属;系统发育分析
山溪鲵属()隶属于两栖纲(Amphibia)有尾目(Caudata)小鲵科(Hynobiidae),有重要的药用价值,也因此被大量猎杀[1-2],目前该物种已被列入中国国家林业局2000年8月1日发布的《国家保护的有益的或者有重要经济、科学研究价值的陆生野生动物名录》。我国存在7个山溪鲵种群,分别为山溪鲵()、西藏山溪鲵()、盐源山溪鲵()、龙洞山溪鲵()、无斑山溪鲵()、弱唇褶山溪鲵()和太白山溪鲵(),而在陕西境内存在的山溪鲵属有3种,分别为山溪鲵、西藏山溪鲵和太白山溪鲵[3]。
黑河是陕西省西安市周至县境内渭河的最大支流[4],海拔跨度大(600~3 500 m以上),与山溪鲵属生存的生态吻合[3]。不仅如此,黑河流域大部分地区森林茂密,有多处被划为国家自然保护区,水源充沛,水质清纯,几乎没有任何污染,是西安市民饮用水和附近农民的农田灌溉的主要水源[5]。
了解黑河水域中的山溪鲵属的资源分布情况,可选择环境DNA(environmental DNA,eDNA)这种方便快捷的研究方式[6]。水样环境DNA是指水生和半水生物种通过皮肤粘液、尿液、粪便及相关残留物释放到水环境中的DNA分子印迹[6]。Ficetola是最早将eDNA技术应用到水生物种(青蛙)上,鉴定了其在自然环境中的分布状况[6],随后eDNA技术在水生生物上使用越来越频繁,涉及到的研究方向也越来越多样化,如保护生物学调研[7-9]、物种入侵调研[10-16]、物种资源分布及多样性调研[17-22]及特定环境中的生物量调研[23]。eDNA的技术流程主要是从水样的获得、eDNA的提取及目标物种的鉴定这些方面进行的。水样获得会根据水质和试验条件进行水样直接冷冻[6,7,9,14]和水样现场过滤冷 冻[8,11-12,17,21,23-25]。eDNA提取有多种方式,有用组织样/血样DNA提取试剂盒提取的[6,7,9,11,14,17,22-25],有用水样专用DNA提取试剂盒提取的[10,16,18,20-21],有用传统的苯酚-氯仿-异戊醇方法提取的[19],还有一些研究将多种提取方法进行比较[8,15,26],但就所有的eDNA提取研究中使用率最高且提取效果最优的还属DNeasy blood and tissue kit (Qiagen, Germany)这个试剂盒。目标物种鉴定方面会根据种间特异引物进行常规PCR或qPCR,前者将目的条带进行测序后在NCBI中进行比对确定是否为目标物种的序列[6,8,15,26],后者通过扩增曲线来估测物种的存在或通过拷贝数来估测物种存在多少[9,11,13-14,17,21-22,24-25]。目标物种鉴定方面还有根据属间特异引物进行常规PCR扩增,随后将目标物种的序列与其他同属物种序列进行系统发育分析,根据聚类结果来判定目标物种的归属[16]。然而应用eDNA分析技术对山溪鲵属的资源调查目前尚鲜见报道。
本研究拟建立一套适用于山溪鲵属水样eDNA的分析方法,便于后期对山溪鲵属物种进行资源调查,可为山溪鲵属的保护提供理论依据。
1 材料与方法
1.1 试验材料
2020年9月,于陕西周至县厚畛子镇钓鱼台地区的黑河流域中发现2尾山溪鲵属物种,参照《中国两栖动物彩色图鉴》[27]和赵虎等[28]相关材料进行鉴定,均为太白山溪鲵。将这两尾太白山溪鲵装入已盛有3 L黑河水的养殖箱中,并在当天带回实验室进行人工养殖,养殖箱规格为50 cm×38 cm×22 cm。
在实验室条件下养殖2 d后,所有太白山溪鲵自然死亡。根据姜维等的研究方法用0.45 μm 孔径的硝酸纤维素滤膜(cellulose nitrate,CN)过滤养殖箱中的水样,将滤膜取出后分别用酒精直接浸泡法[29]、锡箔纸包裹浸泡法[30]、唾液收集管收集法[22]进行储存,每种储存方法设计2个重复,每个重复过滤的水样大约500 mL。同时过滤相同体积的灭菌去离子水,设置2个空白对照。将所有收集到的滤膜置于–20 ℃保存备用。

表1 太白山溪鲵Cytb基因部分序列扩增引物
1.2 试验方法
1.2.1 组织样采集及其DNA提取 采集自然死亡的两条太白山溪鲵的肌肉组织各5份,使用血液/细胞/组织基因组DNA提取试剂盒(天根生化科技(北京)有限公司生产)进行组织DNA的提取。取5 μL DNA溶液,利用1 %琼脂凝胶电泳检测。
1.2.2 引物的设计与筛选 参照NCBI数据库中太白山溪鲵基因的部分序列(GenBank 登录号:EU296330.1),应用primer 5.0软件设计引物,鉴于相关研究中提到水样eDNA扩增的片段长度不易大于400 bp[7],因此本研究设计了4对目的片段长度在100~300 bp之间的引物,并委托北京擎科新业生物技术有限公司进行引物合成。参照马红英研究中的内容进行所有引物的梯度PCR扩增[31],其中Tm设置范围为46~59 ℃。所有引物信息见表1。
1.2.3 水样eDNA的提取与检测 鉴于相关研究经验所得,本研究也使用提取效果较优的DNeasy blood and tissue kit (Qiagen, Germany)进行水样DNA的提取[22,25,29,32-33],除最后一步使用50 μL无菌ddH2O进行洗脱外,其余步骤与该试剂盒说明书一致。取10 μL DNA溶液,利用1%琼脂凝胶电泳和核酸分析仪进行检测,剩余溶液–20℃保存。
1.2.4 水样eDNA的PCR反应体系及扩增程序的确定 选取3个提取效果较好的目标水样eDNA作为模板,2个目标物种DNA为阳性对照,2个去离子水样eDNA为阴性对照。
比较高保真Mix与常规Mix之间的扩增效率。反应体系参考说明书,其中常规Mix反应体系为:10 μL 2×Taq MasterMix(汇天东方,北京),7 μL无菌ddH2O,2 μL DNA模板,上下游引物各0.5 μL。高保真Mix反应体系为:12.5 μL 2×T10 FastLong PCR Mix(汇天东方,北京),9 μL无菌ddH2O,2.5 μL DNA模板,上下游引物各0.5 μL。
PCR反应程序也因Mix不同而不同,常规Mix的PCR反应程序为:95 ℃变性8 min,50个循环(95 ℃,30 s;52 ℃,30 s;72 ℃,1 min),72 ℃延伸8 min。高保真Mix的PCR反应程序为:94 ℃变性3 min,50个循环(94 ℃,10 s;52 ℃,10 s;72 ℃,10 s),72 ℃延伸3 min。
扩增结果用2%的琼脂糖凝胶检测,随机挑选阳性扩增,送至北京擎科新业生物技术有限公司进行测序。
1.2.5 测序结果分析 将测序合格的样品序列通过ChromasProV1.4.1软件转化存储为FASTA格式;利用Mega软件将测序结果与NCBI下载的山溪鲵、太白山溪鲵、西藏山溪鲵的Cytb序列进行对比。应用Mega软件和BEAST软件对所有序列进行聚类分析[34-36],进一步确定本研究的目标物种与其水样DNA扩增结果的归属。
2 结果与分析
2.1 组织样DNA检测
提取的2条太白山溪鲵的各5个重复,除个别提取效果欠佳之外,其余都符合后续的试验要求(图1)。
2.2 PCR引物筛选
随机选取提取效果较优的组织DNA作为模板,对合成的4对引物进行筛选,由图2可知,4对引物都能进行良好扩增,52 ℃可作为所有引物的最佳退火温度,而且引物P2的扩增效果最好,在后期水样eDNA扩增中选取P2为目标引物。

泳道1-5为太白山溪鲵个体一的组织DNA;泳道6-10为太白山溪鲵个体二的组织DNA;M为Marker。
Figure 1 Electrophoresis result of genomic DNA sample
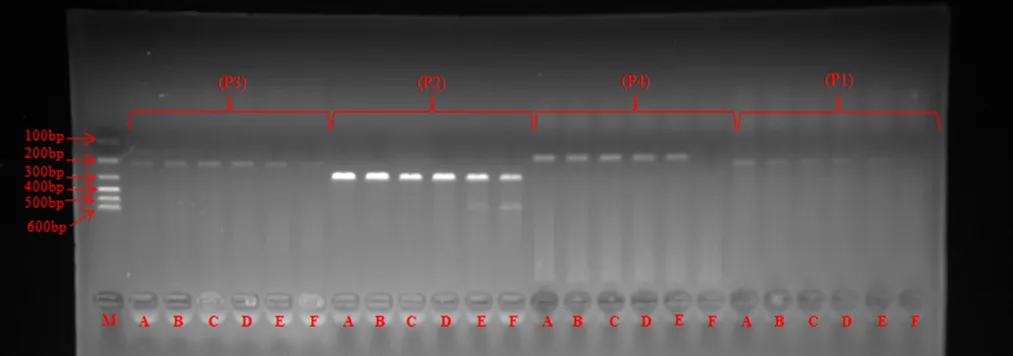
P1-P4为所有引物,具体信息见表1;M为Marker;A-F为设置的不同退火温度,对应值分别为:46 ℃、49 ℃、52 ℃、54 ℃、56 ℃、59 ℃。
Figure 2 Primer screening result
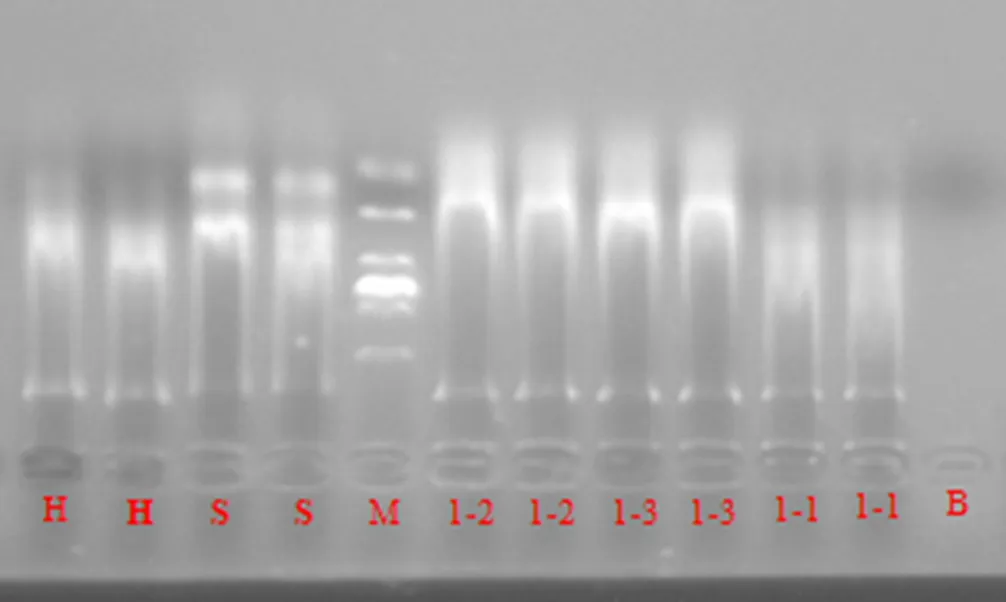
H、S为阳性对照;M为Marker;1-1为唾液收集管收集法储存的滤膜eDNA;1-2为酒精直接侵泡法存储的滤膜eDNA;1-3为锡箔纸包裹存贮的滤膜eDNA;B为阴性对照。
Figure 3 Electrophoresis result of environment DNA sample
2.3 水样eDNA的提取与检测
使用DNeasy blood and tissue kit(Qiagen, Germany)试剂盒对酒精直接浸泡法、锡箔纸包裹浸泡法和唾液收集管收集法进行储存的滤膜进行eDNA提取(为避免交叉污染水样DNA提取和组织样DNA提取在不同时间段和不同操作间进行),结合凝胶电泳图(图3)和核酸分析仪检测结果,不同的滤膜存储方法的DNA提取效果的优先级为:酒精浸泡法(818 ng·μL-1)>锡箔纸包裹法(655 ng·μL-1)>唾液收集管收集法(252 ng·μL-1)。

上半部分为普通Mix扩增结果,下半部分为高保真Mix扩增结果;1、2为阳性对照;3、4、5为目标物种水样eDNA扩增结果;6为阴性对照;M为Marker。
Figure 4 PCR results of different Mixes for water eDNA sample
2.4 水样eDNA的PCR检测
选3个提取效果较好的目标水样eDNA、2个目标物种DNA及1个去离子水样eDNA作为扩增模板,分别应用高保真Mix和普通Mix对本研究中的4对引物(见表1)进行扩增,比对不同Mix的扩增效率,结果如图4所示,使用高保真Mix扩增效果更高,且4对引物中P2特异性更强,扩出了大部分的目标水样的eDNA。
2.5 测序结果的聚类分析
将使用高保真Mix及P2引物对扩出的水样eDNA样本进行大量扩增,同时也将2条通过形态鉴定推断为太白山溪鲵的目标样本进行扩增后,送去北京擎科新业生物技术有限公司进行测序。测序结果在NCBI中进行blast比对后发现无论组织样序列还是水样序列都只比对到山溪鲵属物种上,说明这3种山溪鲵属物种差异较小,且属间特异引物的特异性好,后期通过分子系统学方法对具体种进行分类。本研究将新测得的序列和GenBank中下载的所有山溪鲵、西藏山溪鲵和太白山溪鲵的Cytb基因序列进行比对,分别使用Mega软件和BEAST软件构建系统发育树,通过聚类发现本研究测序得到的水样序列和组织样序列都与GenBank中的太白山溪鲵聚在一起(图5),结合外形判定可进一步断定本研究收集到的是2尾太白山溪鲵,同时水样eDNA中扩增出的序列也是如此。因此在种间差异较小的物种间可结合eDNA和系统发育分析的方法对其分布及归属进行判定。
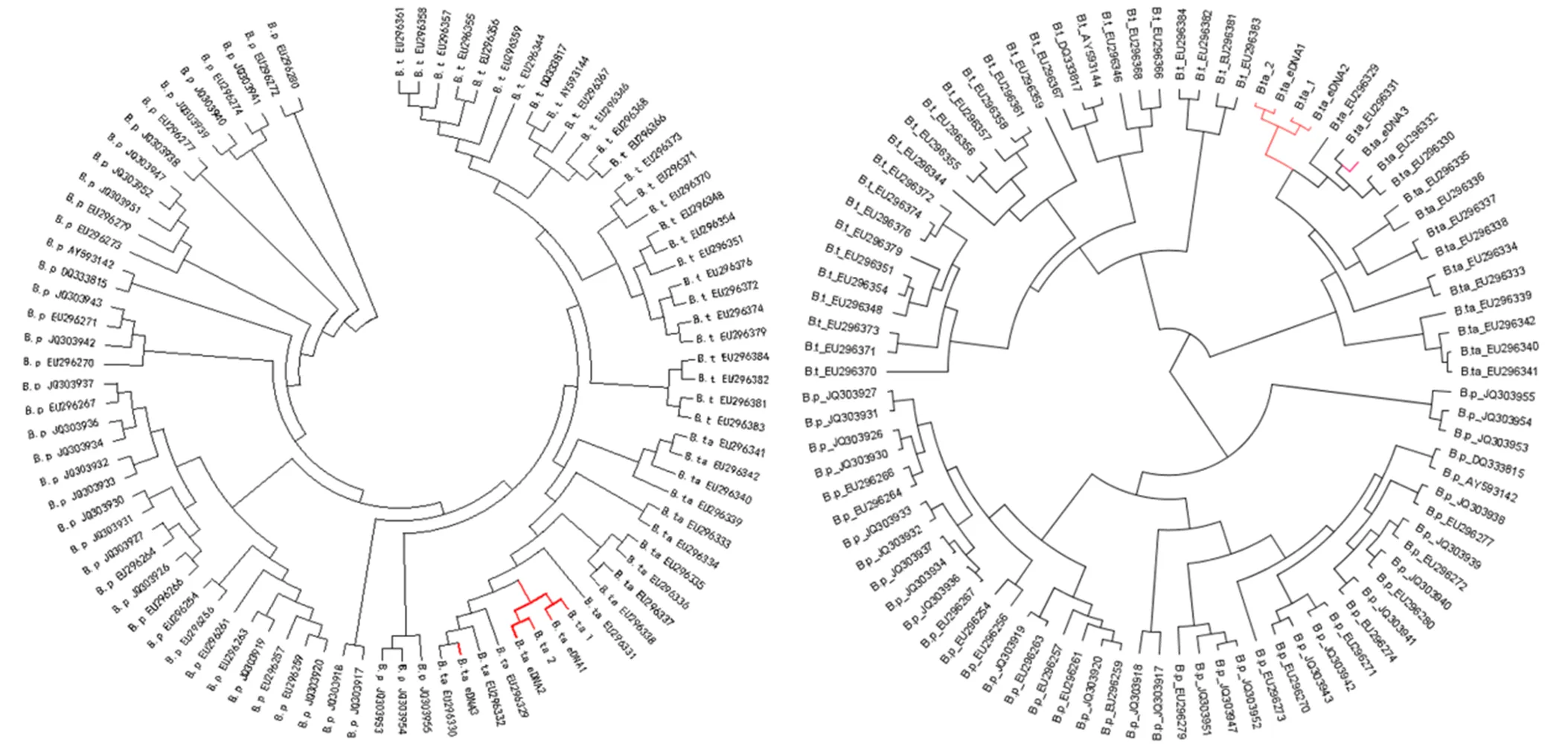
(a) (b)
Figure 5 Molecular Phylogenetic analysis by Maximum Likelihood method (a) and Bayes method (b)
3 讨论与结论
3.1 水样采集时间的确定
在自然水体环境中eDNA的获取成功与否与目标物种在对应水域的存在与否是直接相关的[8,12-13]。本研究前期调研发现,山溪鲵属在7月左右发现率高,理论来讲进入旱期后由于代谢加快,eDNA会有一定的获取量,然而2020年的整个暑期雨季持续时间长,在很长一段时间的调研中只发现了本研究采集到的这一次,因此不排除雨季引起的洪水将山溪鲵属物种冲到大河流域中,发生自然灾害性的死亡,使其数量急剧减少。因此采集自然环境水样,应选择雨量少且干燥炎热的季节。
3.2 滤膜的不同保存方法对eDNA获取存在一定的影响
大部分关于eDNA分析流程的研究中会对水样过滤方式及eDNA提取方式进行比较[8,15,25-26,37],很少有对滤膜保存方式进行比较的研究,本研究首次对常用的滤膜保存方式进行了对比,在同一种过滤及eDNA提取方式的前提下,酒精浸泡法所获得的eDNA浓度最高,质量也最好(818 ng·μL-1),其次为锡箔纸包裹法(655 ng·μL-1)和唾液收集管收集法(252 ng·μL-1)。Goldberg的研究中应用eDNA技术分析了淡水中山蛙和大鲵的分布情况,该研究是首次将过滤后的滤膜放入95%乙醇中冷冻保存的,且成功提取到了这些物种的eDNA[8]。其他使用酒精浸泡滤膜的类似研究中,所过滤的水质都为淡水资源[11,21,25],因此可以认为酒精直接浸泡法适用于保存过滤了淡水的滤膜。而锡箔纸包裹的类似研究中,涉及到的水样为海水[12]或本身水质偏酸[24]的水域。唾液收集管收集滤膜的研究只有陈治等[22]的研究中出现过,其过滤的是近海水域的海水,在该研究中认为唾液收集管收集的滤膜样本能更好的获得eDNA,这与本研究结果相反,这可能与研究对应的水质不同有关。综合以上推测水质清澈的水域过滤后将滤膜用95%酒精浸泡的贮存方法有助于eDNA的获取。
3.3 影响eDNA进行PCR扩增的主要因素
对成功获取的eDNA进行PCR扩增时,发现前人的研究有的用不同型号的Taq 酶[15,20,26,29],有的直接用PCR Mix[8],两种方法都成功扩增了目的条带。由于使用PCR Mix可将操作过程最少的暴露在试验空气中,有助于目的条带的扩增,因此本研究也采用了Mix混合液,且同时将两种不同的Mix做了比较,发现高保真Mix能更好的扩增出水样eDNA的目的条带,因此扩增过程中酶或Mix的使用在eDNA的分析过程中也是至关重要的。
3.4 线粒体Cytb基因及系统发育分析技术在本研究中的重要性
线粒体DNA的高拷贝数使其广泛应用于eDNA分析中,尤其是Cytb基因[6,10,12,19],不仅如此,Cytb基因还被认为是解决系统发育分析类问题中比较可信的研究之一[38]。已有一些研究在山溪鲵属物种中通过Cytb基因进行了系统发育分析, Fu等的研究中应用Cytb基因分别构建了我国西藏地区以及西南地区山溪鲵属物种的系统发育树,对现存的山溪鲵属进行了分类[39-40];也有专门针对西藏山溪鲵、太白山溪鲵和无斑山溪鲵这些特定种的系统发育分析,且都是应用Cytb基因探究了这些山溪鲵种群在不同地域或气候条件下的群体分类以及群体分化时间[38,41-44]。以上研究都是通过阳性DNA扩增的目标基因进行聚类分析,且结果都可信。本研究将eDNA扩增的Cytb序列进行系统发育分析,也同样得到了可信的物种归属判定。
通过本研究得出后期在野外水体环境中进行山溪鲵属鉴定的流程:在山溪鲵活动高峰期(干旱少雨季)进行水样的采集,使用0.45 μm 孔径的硝酸纤维素滤膜(CN)过滤之后用酒精浸泡法保存滤膜,在2周以内使用DNeasy blood and tissue kit (Qiagen, Germany)进行DNA的提取,采用高保真Mix进行PCR扩增,将阳性扩增送去测序,成功测得的序列与已有的三种山溪鲵属的序列进行聚类分析,以便明确采集到的水体中可能存在的山溪鲵属物种。
[1] 金义文, 刘炯宇, 江建平, 等. 中药材羌活鱼基原考订[J]. 中药材, 2005, 28(9): 838-841.
[2] 张佑民, 刘兴连, 孙长铭. 陕西水生野生动植物[M]. 西安: 西安地图出版社, 2006.
[3] 黄棨通, 龚大洁, 张海军, 等. 我国山溪鲵属分布及保护对策[J]. 野生动物学报, 2017, 38(4): 682-688.
[4] 赵阿妮. 黑河湿地保护与开发利用研究[D]. 杨凌: 西北农林科技大学, 2005.
[5] 刘睿翀. 陕西黑河流域地表水与地下水耦合模拟研究[D]. 西安: 长安大学, 2015.
[6] FICETOLA G F, MIAUD C, POMPANON F, et al. Species detection using environmental DNA from water samples[J]. Biol Lett, 2008, 4(4): 423-425.
[7] DEJEAN T, VALENTINI A, DUPARC A, et al. Persistence of environmental DNA in freshwater ecosystems[J]. PLoS One, 2011, 6(8): e23398.
[8] GOLDBERG C S, PILLIOD D S, ARKLE R S, et al. Molecular detection of vertebrates in stream water: a demonstration using Rocky Mountain tailed frogs and Idaho giant salamanders[J]. PLoS One, 2011, 6(7): e22746.
[9] BIGGS J, EWALD N, VALENTINI A, et al. Using eDNA to develop a national citizen science-based monitoring programme for the great crested newt ()[J]. Biol Conserv , 2015, 183: 19-28.
[10] JERDE C L, MAHON A R, CHADDERTON W L, et al. “Sight-unseen” detection of rare aquatic species using environmental DNA[J]. Conserv Lett , 2011, 4(2): 150-157.
[11] GOLDBERG C S, SEPULVEDA A, RAY A, et al. Environmental DNA as a new method for early detection of New Zealand mudsnails ()[J]. Freshw Sci , 2013, 32(3): 792-800.
[13] EICHMILLER J J, BAJER P G, SORENSEN P W. The relationship between the distribution of common carp and their environmental DNA in a small lake[J]. PLoS One, 2014, 9(11): e112611.
[14] SECONDI J, DEJEAN T, VALENTINI A, et al. Detection of a global aquatic invasive amphibian, Xenopus laevis, using environmental DNA[J]. Amphibia-Reptilia, 2016, 37(1): 131-136.
[15] GEERTS A N, BOETS P, VAN DEN HEEDE S, et al. A search for standardized protocols to detect alien invasive crayfish based on environmental DNA (eDNA): a lab and field evaluation[J]. Ecol Indic , 2018, 84: 564-572.
[16] CLUSA L, GARCÍA-VÁZQUEZ E. A simple, rapid method for detecting seven common invasive fish species in Europe from environmental DNA[J]. Aquat Conserv : Mar Freshw Ecosyst , 2018, 28(3): 619-629.
[17] THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples[J]. PLoS One, 2012, 7(8): e41732.
[18] 徐念, 常剑波. 长江中下游干流环境DNA样本鱼类物种检测的初步研究[J]. 水生态学杂志, 2016, 37(5): 49-55.
[19] YAMANAKA H, MINAMOTO T. The use of environmental DNA of fishes as an efficient method of determining habitat connectivity[J]. Ecol Indic, 2016, 62: 147-153.
[20] 张爱菊, 郝雅宾, 郭爱环, 等. 基于高通量测序技术的鱼类环境DNA研究中通用引物的筛选验证[J]. 浙江农业学报, 2019, 31(10): 1615-1623.
[21] 吴昀晟, 唐永凯, 李建林, 等. 环境DNA在长江江豚监测中的应用[J]. 中国水产科学, 2019, 26(1): 124-132.
[22] 陈治, 宋娜, 源利文, 等. 舟山近海水样环境DNA获取方法的建立[J]. 水生生物学报, 2020, 44(1): 50-58.
[23] TAKAHARA T, MINAMOTO T, YAMANAKA H, et al. Estimation of fish biomass using environmental DNA[J]. PLoS One, 2012, 7(4): e35868.
[24] TSUJI S, YAMANAKA H, MINAMOTO T. Effects of water pH and proteinase K treatment on the yield of environmental DNA from water samples[J]. Limnology, 2017, 18(1): 1-7.
[25] HINLO R, GLEESON D, LINTERMANS M, et al. Methods to maximise recovery of environmental DNA from water samples[J]. PLoS One, 2017, 12(6): e0179251.
[26] DEINER K, DE WALSER J C, MÄCHLER E, et al. Choice of capture and extraction methods affect detection of freshwater biodiversity from environmental DNA[J]. Biol Conserv , 2015, 183: 53-63.
[27] 费梁, 叶昌媛, 江建平. 中国两栖动物彩色图鉴[M]. 成都: 四川科学技术出版社, 2010.
[28] 赵虎, 王启军, 姜维, 等. 太白山溪鲵形态特征测量与分析研究[J]. 基因组学与应用生物学, 2017, 36(2): 622-629.
[29] 姜维, 王启军, 邓捷, 等. 以川陕哲罗鲑为目标物种的水样环境DNA分析流程的优化[J]. 应用生态学报, 2016, 27(7): 2372-2378.
[30] TAKAHARA T, MINAMOTO T, DOI H. Using environmental DNA to estimate the distribution of an invasive fish species in ponds[J]. PLoS One, 2013, 8(2): e56584.
[31] 马红英. 利用DNA组学技术对晋江马起源的研究[D]. 北京: 中国农业大学, 2019.
[32] PILLIOD D S, GOLDBERG C S, ARKLE R S, et al. Estimating occupancy and abundance of stream amphibians using environmental DNA from filtered water samples[J]. Can J Fish Aquat Sci, 2013, 70(8): 1123-1130.
[33] GOLDBERG C S, SEPULVEDA A, RAY A, et al. Environmental DNA as a new method for early detection of New Zealand mudsnails ()[J]. Freshw Sci , 2013, 32(3): 792-800.
[34] DRUMMOND A J, RAMBAUT A. BEAST: Bayesian evolutionary analysis by sampling trees[J]. BMC Evol Biol, 2007, 7: 214.
[35] DRUMMOND A J, SUCHARD M A, XIE D, et al. Bayesian phylogenetics with BEAUti and the BEAST 1.7[J]. Mol Biol Evol, 2012, 29(8): 1969-1973.
[36] KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Mol Biol Evol, 2016, 33(7): 1870-1874.
[37] MINAMOTO T, NAKA T, MOJI K, et al. Techniques for the practical collection of environmental DNA: filter selection, preservation, and extraction[J]. Limnology, 2016, 17(1): 23-32.
[38] 梁佳轩. 基于Cytb和D-loop区序列分析太白山溪鲵的谱系地理学[D]. 西安: 陕西师范大学, 2010.
[39] FU J Z, WANG Y Z, ZENG X M, et al. Genetic diversity of eastern Batrachuperus (Caudata: Hynobiidae)[J]. Copeia, 2001, 2001(4): 1100-1107.
[40] FU J Z, ZENG X M. How many species are in the genus Batrachuperus? A phylogeographical analysis of the stream salamanders (Family Hynobiidae) from southwestern China[J]. Mol Ecol , 2008, 17(6): 1469-1488.
[41] LU B, ZHENG Y C, MURPHY R W, et al. Coalescence patterns of endemic Tibetan species of stream salamanders (Hynobiidae:)[J]. Mol Ecol, 2012, 21(13): 3308-3324.
[42] 王贵虎, 丁义晶, 闫季泽, 等. 秦岭地区北方山溪鲵的遗传多样性和种群分化[J]. 西北大学学报(自然科学版), 2013, 43(6): 915-920.
[43] 丁义晶, 王贵虎, 陈振宁, 等. 青海省发现两栖纲小鲵科无斑山溪鲵[J]. 动物学杂志, 2014, 49(3): 428-431.
[44] HUANG Z S, YU F L, GONG H S, et al. Phylogeographical structure and demographic expansion in the endemic alpine stream salamander (Hynobiidae:) of the Qinling Mountains[J]. Sci Rep, 2017, 7: 1871.
[DOI] 10.13610/j.cnki.1672-352x.20211105.016
Establishment of analysis method for environment DNA offrom water samples
MA Hongying, ZHANG Han, DENG Jie, ZHAO Hu, KONG Fei, JIANG Wei, ZHANG Hongxing, WANG Qijun
(Shaanxi Key Laboratory for Animal Conservation, Shaanxi Institute of Zoology, Xi’an 710032)
This study established the method of environmental DNA (eDNA) analysis ofthrough a series of optimization, to provide a theoretical basis for resource investigation and protection ofin future research. This study takes() as the research object to filter water from the aquaculture tank, and genus-specific primer used for the amplification of Cytb gene, afterthat phylogenetic tree was conducted according to sequence analysis to determine the belonging of target species in water. The main results as follows: (1) Three membrane storage methods (like membrane directly socked in alcohol, membrane wrapped in tinfoil and membrane collected with saliva collection tube) have been compared, found that membrane directly socked in alcohol can increase the eDNA yield compared with the other; (2) The PCR amplification efficiency of high-fidelity Mix is higher than ordinary in eDNA analysis; (3) Classification of those species that amplified using genus-specific primer were conducted on a phylogenetic analysis. [Conclusion]This study is the first to establish an optimal method for obtaining eDNA offrom water samples, which provides reference for eDNA extraction of other species who lived in similar waters and have less genetically diverse.
eDNA;;phylogenetic analysis
S931.3
A
1672-352X (2021)05-0784-06
H. Using environmental DNA to estimate the distribution of an invasive fish species in ponds[J]. PLoS One, 2013, 8(2): e56584.
2021-11-8 9:59:16
[URL] https://kns.cnki.net/kcms/detail/34.1162.S.20211105.1129.032.html
2021-03-01
陕西省科学院博士启动专项:“利用水样eDNA分析研究周至黑河水域山溪鲵的资源分布”(2020k-19)资助。
马红英,博士,助理研究员。E-mail:mhying7916@163.com
通信作者:王启军,副研究员。E-mail:wqjab@126.com
