ITGB1基因表达与鹅肥肝形成的关系
2021-12-04张宜辉耿拓宇龚道清
顾 旺,张宜辉,张 军,耿拓宇,龚道清
基因表达与鹅肥肝形成的关系
顾 旺,张宜辉,张 军,耿拓宇,龚道清*
(扬州大学动物科学与技术学院,扬州 225009)
为探究基因与鹅肥肝形成的关系,选取30只70日龄健康朗德公鹅,随机分为填饲组与对照组并进行24 d填饲。运用基因序列进行生物信息学分析,采用RT-qPCR技术检测不同填饲阶段(填饲12 d与24 d)鹅肝脏中基因的表达水平,同时,使用不同剂量的脂肪肝形成相关因子(胰岛素、葡萄糖、油酸、亚油酸和棕榈酸)处理鹅原代肝细胞,检测基因的表达水平。结果发现:全长CDS共2 418 bp,编码805个氨基酸,与鸭的同源性高于98%,可编码1种亲水性蛋白;与对照相比,填饲12 d与24 d时,鹅肝脏中基因的表达均显著上调(<0.05);0.5 mmol·L-1油酸、亚油酸和棕榈酸均能显著地诱导鹅原代肝细胞中基因上调(<0.05);200 mmol·L-1葡萄糖能极显著地诱导鹅原代肝细胞中基因上调(<0.01);此外,0.2 mmol·L-1胰岛素也能在一定程度上有诱导其表达上调的趋势(= 0.098)。结果表明,基因表达上调与鹅肥肝形成密切相关,受脂肪肝形成相关因子诱导,提示基因表达上调可能会促进鹅肥肝的形成。
鹅;;肥肝;基因表达
自然界中,部分鸟类迁徙时肝脏发生膨大易于储存物质能量以供消耗利用,朗德鹅由灰雁驯化而来继承了这一特性[1]。鹅脂肪肝(肥肝)是由填饲生产的一种高营养水禽食品,受到消费者青睐[2]。研究发现,鹅肥肝与人、鼠的非酒精性脂肪肝不同,会发生脂肪变性膨大但无病理损伤[3],这能为非酒精性脂肪肝病的研究与治疗提供一定的参考。目前,对鹅肥肝形成的基本过程已有初步认识,胰岛素抵抗是其发生的中心环节,糖脂合成与代谢发生紊乱,脂肪合成、脂蛋白运输及脂肪酸β氧化间平衡受到破坏,脂肪迅速沉积形成肥肝[4]。然而,相关分子机制仍有待进一步阐明,“多重打击”学说提供了新的见解[5]。此外,鹅肥肝无损伤可能与补体系统成分及肿瘤坏死因子(tumor necrosis factor α,TNF-α)表达受到抑制有关[6-7]。整合素β1基因(integrin β1,)编码二聚体跨膜蛋白,可作为膜受体介导细胞与细胞及细胞外基质(extracellular matrix,ECM)间的相互作用参与众多生物学过程[8-9],主要通过细胞粘附和调节信号转导两种途径调控组织修复、免疫反应、细胞增殖生长和迁移等[10-12]。同时在肥胖等过程中,ECM控制着脂肪组织的分化、迁移、修复、存活和发育,其重塑可能是脂肪组织健康扩散所必需的[13]。因此,推测基因可能通过细胞与ECM间的作用影响鹅肥肝的增殖、修复和免疫等过程,参与鹅肥肝形成。
目前,基因与鹅肥肝形成的关系及作用鲜见报道。因此本研究以灰羽朗德公鹅为试验对象,对基因进行简单的生物信息学分析,并检测其在肝脏与原代肝细胞中的表达水平,初探其与鹅肥肝形成的关系,进一步明晰其作用机制。
1 材料与方法
1.1 试验动物与样本采集
选取70日龄灰羽朗德公鹅30只,均重(3.72 ± 0.19)kg,均分为对照组和填饲组,单只单笼。对照鹅自由采食,填饲鹅常规填饲24 d,于淮安笠诚畜禽养殖公司进行。填饲料为拌入1 %植物油和1 %食盐的熟玉米。预饲期3 d,每天总填饲量分别为100 g、200 g和300 g;常规填饲第1~7 天,每天总填饲量为400 g;填饲第8~14 天,每天总填饲量为600 g;填饲第15~24 天,每天总填饲量为800 g。填饲12 d与24 d时,各组选取6只屠宰,采集肝脏放入液氮转运后–70 ℃保存。
1.2 主要试剂
高糖基础培养基购自美国GIBCO公司;葡萄糖购自北京索莱宝科技有限公司;胰岛素、油酸钠、亚油酸钠和棕榈酸钾购自美国SIGMA公司;TRNzol Universal Reagent试剂购自天根生化科技(北京)有限公司;HisScriptQ RT SuperMix for qPCR和Vazyme AceQ qPCR SYBR Green Master Mix试剂购自南京诺唯赞生物科技有限公司。
1.3 种蛋孵化与原代肝细胞的分离培养及相关因子处理
朗德鹅种蛋来源于扬州瑞农股份有限公司,常规孵化(温度38 ℃,湿度65 %~70 %),孵化22 d时用于原代肝细胞的分离培养[14]。用基础培养基、胎牛血清、青霉素、链霉素、EGF和2 % BSA配制完全培养基悬浮肝细胞,并接种至12孔板中,于培养箱中培养。
细胞培养12 h后,经PBS洗涤后用于因子处理。在原有高糖完全培养基上,分别添加0.05、0.1和0.2 mmol·L-1胰岛素,100、200 mmol·L-1葡萄糖,0.25、0.5 mmol·L-1油酸钠、亚油酸钠、棕榈酸钾,并进行细胞培养。对照组用高糖完全培养基培养,由基础培养基、胎牛血清、青霉素、链霉素、表皮生长因子和2 %牛血清白蛋白组成。试验均重复3次。细胞处理后再培养14 h用于RNA提取。
1.4 生物信息学分析
根据课题组前期肝脏转录组测序结果,在NCBI上将朗德鹅基因序列与GenBank鹅物种()序列进行比对;用MEGA软件将鹅基因编码区序列(coding sequence,CDS)与多个物种(鸭、鸡、火鸡、人、小鼠、大鼠、猪、牛、绵羊和兔)进行同源性比对,并构建系统发生树;在ExPASy上进行蛋白质亲疏水性分析;利用PRABI对氨基酸序列进行二级结构分析;利用SWISS-MODEL预测其蛋白三级结构。

表1 引物信息表
1.5 ITGB1基因mRNA表达水平的检测
根据 GenBank 序列,设计引物,由南京金唯智生物公司合成(见表1)。遵循试剂说明书提取肝脏和肝细胞的 RNA,反转录成 cDNA,并在CFX定量仪中反应(20 μL体系:Mix 10 μL,上下游引物各0.4 μL,无酶水7.2 μL,cDNA 2 μL)。反应条件:95 ℃ 5 min,95 ℃ 10 s和60 ℃ 34 s,40个循环。试验结果采用 ΔT值表示,以2-ΔΔCT法计算分析。
1.6 统计分析
试验结果均用平均值±标准误(Mean ± SEM)表示,并进行-test检验分析,确定差异显著性,< 0.05表示差异显著(*),< 0.01表示差异极显著(**),0.05 << 0.01表示差异存在显著趋势。
2 结果与分析
2.1 ITGB1基因序列的分析
依据课题组前期填饲朗德鹅肝脏转录组的测序结果,获得基因序列,并发现其表达显著上调。经比对发现,朗德鹅基因测序序列与NCBI上鹅(Anser cygnoides domesticus)序列的比对率为99.63 %,说明测序准确。鹅基因CDS共2 418 bp,编码805个氨基酸(见图1),与鸭、鸡、火鸡、人、小鼠、大鼠、猪、牛、绵羊和兔核苷酸水平上的同源性分别为98.61 %、90.14 %、90.77 %、68.89 %、68.39 %、66.12 %、64.16 %、64.76 %、64.57 %和75.65 %,而氨基酸水平上的同源性则分别为99.63 %、97.45 %、97.70 %、86.41 %、86.62 %、86.41 %、87.60 %、87.34 %、87.60 %和86.02 %(见表2)。通过系统发生树(Bootstrap consensus tree)发现,鹅与鸭聚为一类,再与鸡与火鸡汇为一类,同时鼠类与家禽类较为接近汇为一类;人与牛、羊反刍动物汇为一类再和家禽类、鼠类汇为一大类;此外,猪、兔汇为一类再与其他一大类汇总(见图2)。体现了鸟类与哺乳动物进化的区别,进一步证实基因测序的准确。

图1 鹅ITGB1基因CDS核酸与氨基酸序列图
Figure 1 The coding sequence and the predicted amino acid sequence of goose1 gene
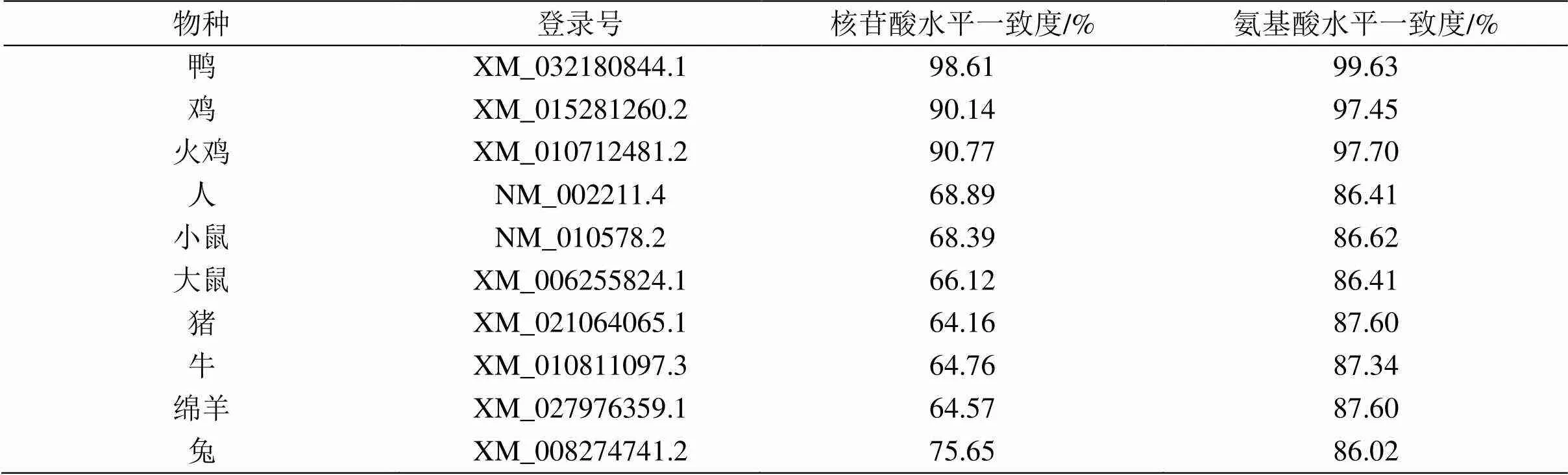
表2 鹅ITGB1基因与其他物种在核苷酸水平上的同源性

图2 基于核苷酸序列分析的ITGB1基因系统发生树
Figure 2 The phylogenetic tree ofgene based on analysis of nucleotide sequences

图3 鹅ITGB1蛋白疏水性分布图
Figure 3 The putative hydrophobicity of ITGB1 protein
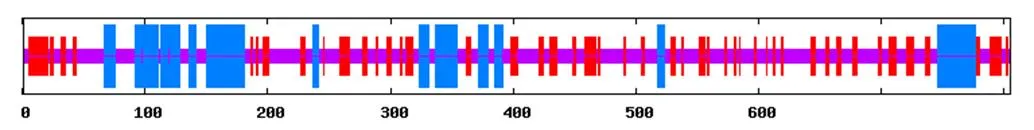
图4 鹅ITGB1蛋白的二级结构
Figure 4 The secondary structure of goose ITGB1 protein
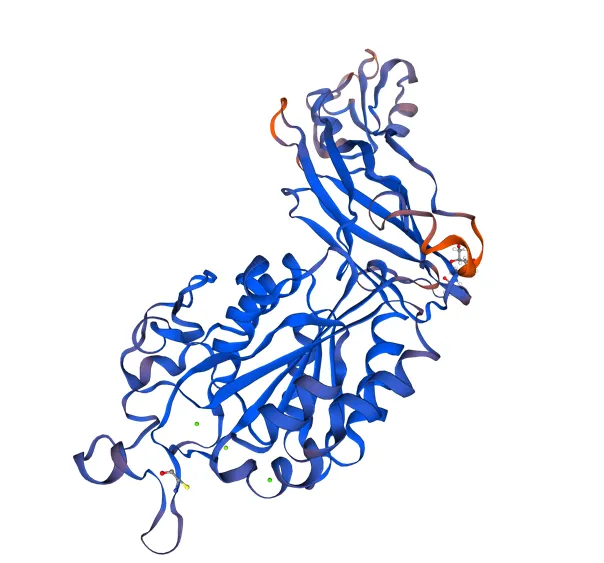
图5 鹅ITGB1蛋白的三级结构
Figure 5 The putative tertiary structure of goose ITGB1 protein
在ExPASy上进行蛋白质亲疏水性预测,发现疏水性最高为4.500,最低为–4.500,且低分值区域(score< 0)比高分值区域(score> 1.5)多,由此推测,ITGB1蛋白是一种亲水性蛋白(见图3);在PRABI上进行氨基酸序列二级结构预测,发现具有α螺旋(Alpha helix:20.75 %)、延长片段(Extended strand:23.60 %)和无规卷曲(Random coil:55.65 %)3种结构(见图4);在SWISS-MODEL上预测其蛋白三级结构(见图5)。
2.2 ITGB1基因在填饲鹅肝脏中的表达
经12 d填饲后,填饲鹅肝脏平均重量为352.19 g,明显高于对照鹅肝脏重量116.23 g(< 0.01);同样24 d填饲后,填饲鹅肝脏平均重量为830.19 g,明显高于对照鹅肝脏重量102.95 g(< 0.01),表明填饲后肝脏脂肪显著沉积(见表3)。本研究通过RT-qPCR技术检测试验鹅肝脏中基因的表达水平,发现与对照相比,填饲12 d、24 d时鹅肝脏中基因的表达均显著升高(< 0.05,见图6)。

表3 不同填饲时间的鹅肝重
注:同列**表示差异极显著(< 0.01)。

1.对照鹅肝脏;2.填饲鹅肝脏。*表示P< 0.05,**表示P< 0.01。下同。
Figure 6 Expression ofgene in livers of overfed geese
2.3 ITGB1基因在脂肪肝形成相关因子处理的鹅原代肝细胞中的表达
胰岛素抵抗是鹅肥肝形成过程的中心环节,糖脂代谢也参与其中[4]。为探究基因表达变化与鹅肥肝形成的内在联系,本研究采用不同剂量的葡萄糖、胰岛素和脂肪酸因子处理鹅原代肝细胞,并通过RT-qPCR检测其基因的表达水平。发现与对照相比,0.2 mmol·L-1胰岛素有诱导基因表达上调的趋势(= 0.098),见图7(A);200 mmol·L-1葡萄糖可极显著诱导基因表达上调(< 0.01),见图7(B)。0.5 mmol·L-1油酸、亚油酸和棕榈酸同样显著诱导基因表达上调,且0.25 mmol·L-1亚油酸处理也显著诱导基因表达上调(< 0.05),见图7(C)。

图7 ITGB1基因在脂肪肝形成相关因子处理的鹅原代肝细胞中的表达
Figure 7 Expression ofgene in primary hepatocytes goose treated with fatty liver formation related factors
3 讨论与结论
不同于其他动物,鹅肥肝是生理性脂肪肝。在鹅填饲过程中,肠道消化利用了大量的玉米并迅速转化为脂肪沉积在肝脏中,且肝脏脂肪变性时无病理损伤发生。这说明适度过量的营养物质有利于肥肝的形成,且鹅肝脏本身能够耐受脂肪细胞增殖、变性与沉积。肝脏脂肪变性及堆积在朗德鹅中更为重要,肌肉和皮下脂肪组织中则较不发达[1],这可能与肝脏脂肪细胞增殖的增强有关,其中卵泡抑素样蛋白5基因可能发挥了一定作用[15-16]。同时,鹅肥肝耐受损伤的能力可能与肝脏补体系统,炎症因子受到抑制等有关,也可能是肠道内乳酸菌丰度增加提供了保护作用,且肠道组织结构及屏障功能保持着稳态[17]。基因编码整合素蛋白,可作为膜受体介导细胞与细胞及细胞外基质间的相互作用,对细胞周围微环境作出反应,与细胞增殖、组织修复和免疫反应等密切相关。整合素与多种生长因子和细胞因子受体也有着广泛的相互作用(如生长因子受体和肝细胞生长因子受体等),从而促进细胞增殖[8-9]。整合素β1可能是一种氧敏感分子,在皮肤表面伤口愈合过程中促进角质形成细胞的迁移,而肠道上皮创伤修复也与其有关[18-19]。此外,钱涛报道血管生成素样蛋白7能上调基因的表达,促进巨噬细胞迁移,提示可能参与免疫[20]。因此,推测基因可能通过细胞增殖、介导免疫和组织修复来参与鹅肥肝形成。基因与鹅肥肝形成的关系及作用机制尚鲜见报道,且前期通过填饲鹅肝脏转录组测序分析筛选到基因表达上调,基于此开展本研究。
本研究通过与NCBI上基因数据的比对及与其他物种(鸭、鸡、火鸡、人、小鼠、大鼠、猪、牛、绵羊和兔)的同源性比较,验证了该基因测序的准确;并通过核苷酸和氨基酸序列分析,简述了其基本蛋白结构与功能;再者通过RT-qPCR技术检测填饲鹅肝脏及原代肝细胞中基因的表达情况,揭示胰岛素、葡萄糖、脂肪酸与基因表达的内在联系。结果显示,填饲鹅基因测序准确,全长CDS共2 418 bp,805个氨基酸,与鸭的同源性高达98 %,可编码1种亲水性蛋白;填饲整个过程中鹅肝脏中基因表达显著上调,可能是该基因的高表达有利于增强细胞与ECM间的生物学作用从而促进肝细胞的增殖,并加剧脂肪快速沉 积[13];同时在组织修复及免疫反应中具有一定的生物学效应[21],这可能也为肝脂肪细胞的剧烈扩散提供有益的保护作用从而抑制炎症和细胞凋亡等损伤,但有待进一步证实。有趣的是,适宜浓度的胰岛素、葡萄糖、脂肪酸总体上也诱导鹅原代肝细胞中基因的表达,这与本研究肝脏中的结果具有一致性。鹅肥肝的主要营养成分包含油酸、亚油酸及棕榈酸等,而加强糖脂代谢及胰岛素抵抗是其形成的基本条件[4]。糖脂代谢紊乱可能会伴随着的信号传递强度的改变[22],说明脂肪肝形成相关因子对有显著的刺激作用,加强的表达与功能,促进肝细胞增殖。因此,的表达上调可能是鹅肥肝形成因素诱导的结果,脂肪肝形成与的表达成正向关系。但促进鹅肝脏脂肪细胞增殖的具体机制以及对各因子诱导效应的反馈有待进一步的研究。
本研究发现在鹅肥肝形成过程中,基因在肝脏中表达显著上调,并受到胰岛素、葡萄糖、脂肪酸因子的诱导,可能会促进鹅肥肝的形成,为深入研究基因在鹅肥肝形成中的作用机制奠定基础。
[1] DAVAIL S, GUY G, ANDRÉ J, et al. Metabolism in two breeds of geese with moderate or large overfeeding induced liver-steatosis[J]. Comp Biochem Physiol A Mol Integr Physiol, 2000, 126(1): 91-99.
[2] 王来娣, 邵丹, 龚道清. 鹅肥肝生产性能及品质影响因素的研究进展[J]. 中国畜牧兽医, 2012, 39(6): 218-221.
[3] 柳序, 郭松长, 刘耀文, 等. 鹅肝脏脂肪变性和鹅肥肝形成的分子保护机制研究进展[J]. 动物营养学报, 2018, 30(7): 2453-2458.
[4] 柳序, 刘耀文, 匡佑华, 等. 鹅肥肝的形成及主要影响因素的研究进展[J]. 经济动物学报, 2019, 23(4): 234-239.
[5] 华爽, 吕明慧, 刘倩颖, 等. 糖脂代谢病的发病机制: 多重打击学说[J]. 世界中医药, 2019, 14(3): 638-644.
[6] LIU L, ZHAO X, WANG Q, et al. Prosteatotic and protective components in a unique model of fatty liver: gut microbiota and suppressed complement system[J]. Sci Rep, 2016, 6: 31763.
[7] GENG T, YANG B, LI F, et al. Identification of protective components that prevent the exacerbation of goose fatty liver: characterization, expression and regulation of adiponectin receptors[J]. Comp Biochem Physiol B Biochem Mol Biol, 2016, 194/195: 32-38.
[8] BLANDIN A F, RENNER G, LEHMANN M, et al. β1 Integrins as therapeutic targets to disrupt hallmarks of cancer[J]. Front Pharmacol, 2015, 6: 279.
[9] BRIZZI M F, TARONE G, DEFILIPPI P. Extracellular matrix, integrins, and growth factors as tailors of the stem cell niche[J]. Curr Opin Cell Biol, 2012, 24(5): 645-651.
[10] 闵伟杰. ITGB1促进胶质瘤细胞增殖的作用和机制研究[D]. 上海:第二军医大学, 2016.
[11] LANGEREIS J D. Neutrophil integrin affinity regulation in adhesion, migration, and bacterial clearance[J]. Cell Adh Migr, 2013, 7(6): 476-481.
[12] AKSORN N, CHANVORACHOTE P. Integrin as a molecular target for anti-cancer approaches in lung cancer[J]. Anticancer Res, 2019, 39(2): 541-548.
[13] RUIZ-OJEDA F J, MÉNDEZ-GUTIÉRREZ A, AGUILERA C M, et al. Extracellular matrix remodeling of adipose tissue in obesity and metabolic diseases[J]. Int J Mol Sci, 2019, 20(19): 4888.
[14] 洪胜辉, 张军, 邵丹, 等. 鹅肝细胞的高纯分离及培养[J]. 中国兽医学报, 2012, 32(9): 1378-1380.
[15] LI S, BROWN M S, GOLDSTEIN J L. Bifurcation of insulin signaling pathway in rat liver: mTORC1 required for stimulation of lipogenesis, but not inhibition of gluconeogenesis[J]. PNAS, 2010, 107(8): 3441-3446.
[16] 刘立东, 温康, 顾旺, 等. 卵泡抑素样蛋白5基因在鹅肥肝形成中表达调控的研究[J]. 中国家禽, 2019, 41(13): 6-10.
[17] 顾旺. 鹅肥肝形成过程中肠道结构的适应性变化及转录组分析[D]. 扬州:扬州大学, 2019.
[18] TANG D, YAN T T, ZHANG J H, et al. Notch1 signaling contributes to hypoxia-induced high expression of integrin β1 in keratinocyte migration[J]. Sci Rep, 2017, 7: 43926.
[19] SWAIN S D, GRIFKA-WALK H N, GRIPENTROG J, et al. Slug and snail have differential effects in directing colonic epithelial wound healing and partially mediate the restitutive effects of butyrate[J]. Am J Physiol Gastrointest Liver Physiol, 2019, 317(4): G531-G544.
[20] 钱涛. 血管生成素样蛋白7的促炎功能研究[D]. 武汉:华中农业大学, 2016.
[21] KOIVISTO L, HEINO J, HÄKKINEN L, et al. Integrins in wound healing[J]. Adv Wound Care, 2014, 3(12): 762-783.
[22] 王利微, 杨海鹏, 蔡雁. 血清与胎盘组织中PPAR、FABP-4、ITGB1对妊娠女性子痫前期发病影响[J]. 临床军医杂志, 2018, 46(07): 763-766.
The relationship betweengene expression and the formation of goose fatty liver
GU Wang, ZHANG Yihui, ZHANG Jun, GENG Tuoyu, GONG Daoqing
(College of Animal Science and Technology, Yangzhou University, Yangzhou 225009)
To explore the relationship betweengene and goose fatty liver formation, thirty healthy 70-day-old male Landes geese were randomly divided into a control group and an overfed group for 24 days of overfeeding. The sequence ofgene was used for bioinformatics analysis, and its expression levels in the liver of geese at different overfeeding stages (12 and 24 days) were measured by RT-qPCR. Meanwhile, different doses of fatty liver formation related factors (insulin, glucose, oleic acid, linoleic acid, and palmitic acid) were used to treat goose primary hepatocytes to detect the expression levels ofgene. The results showed that the total length of CDS is 2 418 bp, encoding 805 amino acids (which is a hydrophilic protein) and the homology between goose and duck is higher than 98 %. The expression ofgene in goose fatty liver was significantly up-regulated (<0.05) after 12 and 24 days of overfeeding compared with the control group. And 0.5 mmol·L-1oleic acid, linoleic acid and palmitic acid could significantly induce the up-regulated expression ofgene in goose primary hepatocytes (< 0.05). Moreover, 200 mmol·L-1glucose could also extremely significantly induce the up-regulated expression ofgene in goose primary hepatocytes (< 0.01). Beside, 0.2 mmol·L-1insulin could also somehow have tend to induce the up-regulated expression ofgene (= 0.098). The results indicated that the up-regulation ofgene was closely related to the formation of goose fatty liver and could be induced by factors related to fatty liver formation, which suggested that up-regulation ofgene expression may promote the formation of goose fatty liver.
goose;; fatty liver; gene expression
S835
A
1672-352X (2021)05-0771-06
10.13610/j.cnki.1672-352x.20211105.015
2021-11-8 9:27:10
[URL] https://kns.cnki.net/kcms/detail/34.1162.S.20211105.1129.030.html
2020-06-13
国家自然科学基金项目(31672405,31972546)资助。
顾 旺,硕士研究生。E-mail:414031651@qq.com
通信作者:龚道清,博士,教授,博士生导师。E-mail:yzgong@163.com
