G-四联体与聚合酶链式反应联用可视化检测沙门氏菌
2021-12-03刘健慧耿凤珍张先舟檀建新
刘健慧,耿凤珍,张先舟,高 浩,李 聪,高 洁,檀建新,*
(1.河北农业大学食品科技学院,河北 保定 071000;2.河北大学附属医院,河北 保定 071000)
沙门氏菌(Salmonella)是常见的食源性致病菌,可通过污染食品进入肠道,经大量繁殖后引起呕吐、腹痛、腹泻。全世界每年由沙门氏菌导致的患病人数高达9 380万,死亡15.5万 人[1-4]。其中,因食用被其污染的畜产品致病的人数占沙门氏菌病例的75%,是爆发公共健康问题的主要原因[5-7]。近年来,抗生素的滥用、细菌自身耐药性的提升等都对食品安全问题构成了威胁[8-9]。
食源性病原菌因少量存在于食品中而易被背景微生物区系所掩盖,难以识别[10]。从食品中高效准确识别和鉴定出病原菌是防范食品安全事件发生的前提。目前沙门氏菌的检测方法有多种,各有优缺点。如:常规检测法包括预富集、选择性富集、选择性培养基上培养和生化测试等步骤,虽特异性高,但耗时长,且对操作人员要求较高[11]。免疫学检测法虽操作相对简单、灵敏,但抗体的制备繁琐、价格昂贵且不易得到[12]。随着核酸等温扩增技术的完善[13],环介导等温扩增技术[14]、滚环扩增技术[15]、跨越式滚环扩增技术[16]等均已用于致病菌的检测,这些方法方便快捷、特异灵敏、不需复杂设备,但因其引物设计复杂、成本高,特别是由于气溶胶污染容易出现假阳性等弊端,在一定程度上限制了其应用。核酸适配体[17]检测法,具有特异、灵敏的优点,但适配体结构受环境因素影响大,与目标物不易结合成牢固的复合体,技术上仍有待完善。本实验采用稳定性和实用性强的聚合酶链式反应(polymerase chain reaction,PCR)技术[18],同时引入G-四联体可视化检测概念,可达到较其他检测方法相近的直观、定量的检测效果[19]。
G-四联体是富含鸟嘌呤(G)的DNA或RNA序列串联重复形成的非规范的四联结构,在真核端粒中过度表达,起到增殖或生长抑制作用[20-22]。Gellert等[23]通过X射线证明由4 个鸟嘌呤形成的共平面四分体,可通过氢键和碱基堆积作用形成稳定的G-四联体,其结构有平行、反平行和混合式3 种形式[24]。稳定的G-四联体可与氯高铁血红素(Hemin)结合,形成具有过氧化物酶活性的模拟酶(DNAzyme),催化H2O2与2,2’-联氮-双(3-乙基-苯并噻唑琳-6-磺酸)二胺盐(2,2’-diazo-bis(3-ethylbenzothiazolyn-6-sulfonic acid) diamine salt,ABTS)由无色变为绿色[25],基于这一原理,已实现对金属离子[26-27]、核酸分子[28]等的信号放大检测。由于这些检测体系中最终信号的强弱与G-四联体的量呈正比,因此,将G-四联体序列(或其互补序列)偶联到用于特异性识别目标核酸序列上,通过高效扩增目标序列,可实现G-四联体的大量富集而将检测信号放大,并用于目标序列的检测。本研究将PCR的特异性、灵敏性与G-四联体的DNAzyme酶学活性、生物传感器特性相结合,以沙门氏菌invH基因为靶基因,设计并筛选出一对5’端偶联G-四联体反向互补序列的PCR扩增引物,通过对靶基因特异性扩增得到大量带有G-四联体的PCR产物,并形成G-四联体结构,发挥DNAzyme活性,建立了针对沙门氏菌的G-四联体与PCR联用的可视化检测方法,以期为食源性致病菌的灵敏、快速、直观检测提供一种新策略和思路。
1 材料与方法
1.1 材料与试剂
本实验所用菌株如表1所示,均由河北农业大学生物工程实验室提供;引物由金唯智生物科技有限公司合成;细菌基因组DNA提取试剂盒、DNA纯化回收试剂盒天根生化科技有限公司;2×Es TaqMaster Mix(Dye)北京康为世纪生物科技有限公司;100 bp DNA ladder宝生物工程(大连)有限公司;琼脂糖凝胶回收试剂盒、琼脂糖 北京全式金生物技术有限公司;Hemin、过氧化氢、二甲基亚砜 北京Solarbio公司;ABTS 美国Sigma公司;其他主要试剂均为国产分析纯。

表1 本研究所用菌种Table 1 Strains used in this study
1.2 仪器与设备
MTH-012型旋涡混合仪 海门其林贝尔仪器制造有限公司;070-851型PCR仪 德国Biometra公司;DYY-10 C型电泳仪 北京市六一仪器厂;JY04S-3E型凝胶成像分析系统 北京科普尔科技发展有限公司;K5500型超微量核酸测量仪 北京凯奥科技发展有限公司;iMark酶标仪 美国Bio-Rad公司。
1.3 方法
1.3.1 细菌基因组提取
取1 mL过夜培养菌液,提取细菌基因组DNA,具体操作方法见细菌基因组DNA提取试剂盒说明书。用核酸测量仪对其DNA浓度及纯度进行测定。
1.3.2 引物设计
根据PCR引物设计原则设计出用于特异性识别和扩增invH序列的上下游引物,上游引物(H-F)序列为:5’-GAAAGAGCAACTGGCCAACG-3’,下游引物(H-R)序列为:5’-ATGCTTGAGCTGATTGC GC-3’,并在引物的5’端外加一段G-四联体的反向互补序列,修饰后的上游引物(G-F)序列为:5’-CCCACC CACCCACCC GAAAGAGCAACTGGCCAACG-3’,修饰后的下游引物(G-R)序列为:5’-CCCACCCA CCCACCC ATGCTTGAGCTGATTGCGC-3’,其中5’-CCCACCCACCCACCC-3’为G-四联体序列(G3T)3G3的反向互补,其在K+离子存在条件下形成平行G-四联体结构,并且随着富G序列的不断延长,其稳定性无明显变化,可视化反应较明显[29]。
1.3.3 PCR反应体系与条件优化
以沙门氏菌基因组DNA为模板,用H-F/R作引物PCR扩增目标序列,胶回收,获得的PCR产物经核酸蛋白仪测定浓度后作为后续实验的模板。
以G-F/R为引物,进行G-四联体与PCR扩增联用可视化检测体系的优化。首先,在无模板PCR体系中,观测1、2、3、4、5、6、8、10 μmol/L引物浓度下引物二聚体形成及对G-四联体显色反应的影响;之后进行引物浓度与模板质量浓度之间的优化;确定最适引物浓度后再进行不同循环数与模板质量浓度的PCR。引物浓度梯度设置为2、3、4、6 μmol/L;循环数梯度设置为20、23、25、28、30 个循环。
各优化组经过可视化判断,最终确定PCR反应体系为:50 μL 2×Es TaqMaster Mix(Dye),4 μL上、下游修饰引物G-F/R(4 μmol/L),4 μL DNA模板,用无菌ddH2O补至100 μL。PCR扩增条件为:95 ℃预变性5 min,95 ℃变性30 s,65 ℃退火30 s,72 ℃延伸15 s,72 ℃延伸5 min,30 个循环。PCR产物用2%的琼脂糖凝胶电泳(140 V、40 min)鉴定,利用凝胶成像系统对结果观察分析。
1.3.4 G-四联体与PCR联用的可视化反应体系优化
按照DNA纯化回收试剂盒步骤回收的PCR产物中加入30 μL K+缓冲液(40 mmol/L KCl、20 mmol/L Tris-HCl pH 7.4、0.05% Triton-X)与15 μL不同终浓度Hemin工作液充分混匀,终浓度梯度设置为2、4、6、8、10 μmol/L。在PCR仪中95 ℃加热使PCR产物变性,变性时间梯度设置为1、3、5、8、10 min。之后以2 ℃/s的速率从95 ℃降温至4 ℃,并于4 ℃孵育,完成G-四联体折叠,孵育时间梯度设置为5、10、20、30、40 min。最终加入50 μL的ABTS(1.28 mmol/L)和H2O2(1.28 mmol/L)进行显色反应。室温显色5 min后,酶标仪测量421 nm处的吸光度,选择最优的可视化结果进行后续实验。
1.3.5 G-四联体与PCR联用可视化的特异性检测
为验证构建方法的特异性,分别对表1中16 株菌的基因组DNA进行提取,其中包括7 株沙门氏菌和9 株非特异性菌株。根据优化实验得出的最佳反应条件和最优反应体系进行G-四联体与PCR扩增方法联用检测,测定421 nm的吸光度,验证构建的可视化检测方法检测沙门氏菌的特异性。
1.3.6 G-四联体与PCR联用可视化的灵敏度检测
过夜培养的甲型副伤寒沙门氏菌,取1 mL进行基因组DNA的提取,之后将所提取的核酸进行梯度稀释并测其核酸含量,按照已经优化好的条件进行PCR扩增,再利用纯化产物进行G-四联体显色反应,421 nm波长处比色测定,以检测该方法的灵敏度。
1.3.7 G-四联体与PCR联用可视化检测法对人工污染牛奶样品中沙门氏菌的检出限
取过夜培养的沙门氏菌,0.85%的生理盐水进行10 倍梯度稀释,每个稀释度各取1 mL进行菌落计数。用上述各稀释度菌液5 mL接种于45 mL灭菌的牛奶中,均质后各取1 mL提取基因组DNA,作为检测的模板,对G-四联体与PCR扩增联用的人工污染牛奶进行检出限分析。
1.3.8 G-四联体与PCR联用可视化检测法的实际应用与评价
为检验G-四联体与PCR联用方法在实际样品中的检测效果,在当地各大超市及市场随机购买30 份容易被沙门氏菌污染的样品,包括散装牛奶10 种、鲜鸡蛋10 种、鲜肉10 种。根据上述反应程序,以30 份实际样品提取的DNA进行G-四联体与PCR联用可视化检测实验,同时按GB 4789.4—2016《食品微生物学检验 沙门氏菌检验》方法检测,其结果作为对照。
1.4 数据处理
数据的处理计算及标准曲线的绘制均采用Microsoft Excel 2010软件分析。
2 结果与分析
2.1 PCR反应体系与反应条件优化
2.1.1 无目标模板时引物浓度对G-四联体与PCR联用的可视化显色效果影响
与常规PCR引物相比,本研究在引物的5’端额外加了G-四联体的反向互补序列,使得引物长度增加且结构复杂,可能更易于二聚体的形成,引起非特异性扩增,甚至影响G-四联体结构,从而造成背景信号增大,干扰检测结果。为排除这种可能性,把引物稀释成10、8、6、5、4、3、2、1 μmol/L系列浓度,对其进行G-四联体与PCR联用的可视化显色观察和检测。由图1A可知,不同引物浓度下,无模板PCR体系经扩增后未产生目的条带,而引物二聚体的增加与引物浓度呈正比;由图1B可知,不同引物浓度下,引物二聚体的存在对于显色的影响用肉眼难以区分,421 nm波长处吸光度较空白略有增加,但不同引物浓度间差异不明显。因此,当PCR体系中无目标模板DNA时,引物浓度高低对G-四联体与PCR联用的可视化显色效果无明显影响。
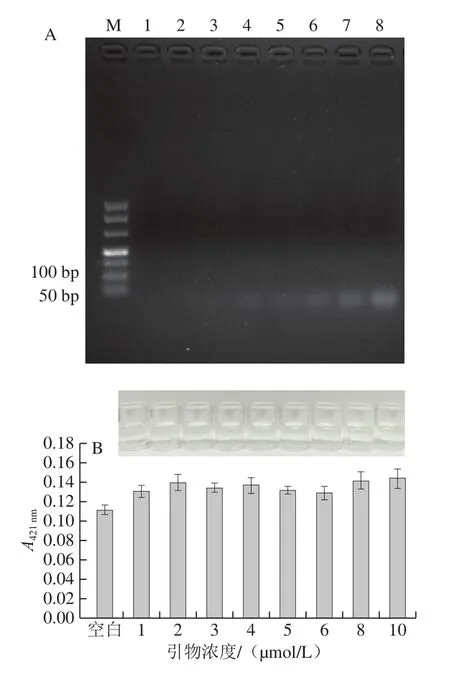
图1 无靶标模板PCR体系中引物浓度对G-四联体显色反应的影响Fig.1 Effects of different primer concentrations in PCR system without target templates on the chromogenic reaction of G-quadruplex
2.1.2 PCR体系中引物浓度对模板质量浓度的优化
由PCR扩增原理知,当模板质量浓度固定时,在一定范围内随着引物浓度的增加,PCR产物也会增多,反之亦然。因此,在DNA扩增过程中,引物浓度与模板量之间存在着相互作用关系,找到两者之间相对合适的浓度对于确定PCR检测到的模板最低量至关重要。图2显示,当引物浓度达到4 μmol/L时,模板检出限为500 fg/μL,且不再随着引物浓度的增加而改变,因此选用引物浓度4 μmol/L作为后续实验基础。
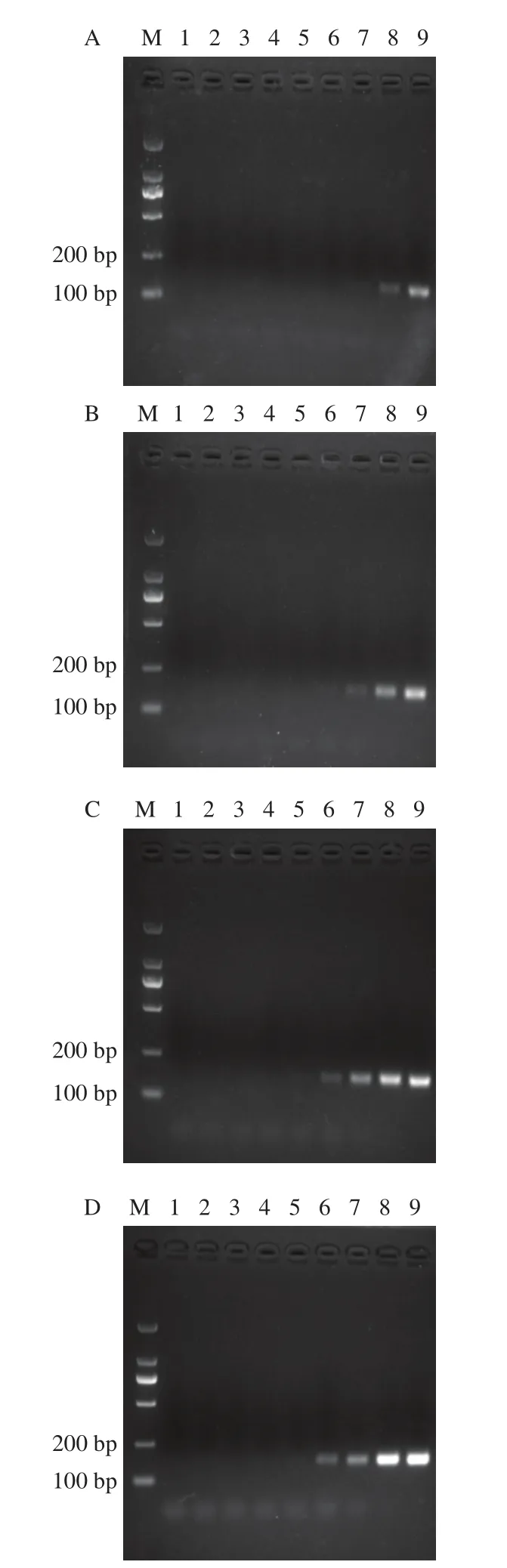
图2 引物浓度对模板质量浓度优化电泳图Fig.2 Optimization of ratio of primer concentration to template concentration
2.1.3 PCR扩增循环数的优化
循环数优化也是G-四联体与PCR联用可视化检测方法构建中的重要环节,循环数的多少直接影响PCR产物及G-四联体的生成量。循环数少会影响灵敏度,循环数过多,又会导致不同模板质量浓度的PCR扩增产物之间差异不明显,所以找到一个适合的循环数非常重要。在引物浓度设定为4 μmol/L的基础上,可通过循环数与模板质量浓度之间的合适条件确定循环数。
阴性对照以无菌ddH2O替代模板,图3显示,随着模板质量浓度的降低,A421nm比色结果与阴性对照之间的差异越来越小。不同的循环数比较,28、30 个循环对模板的检出效率远高于20、23、25 个循环,检出限更低,且30 个循环可视化效果最为明显,故选择30 个循环进行后续实验。
2.2 G-四联体与PCR扩增联用可视化检测显色条件的优化
2.2.1 Hemin终浓度的优化
Hemin浓度会直接影响与G-四联体结合后的复合体催化活性的强弱。将Hemin在检测体系内的终浓度设置为2、4、6、8、10 μmol/L进行孵育,以此探究检测体系中的最适Hemin的浓度。由图4A可以看出,随着Hemin浓度逐渐增大,体系吸光度逐渐增大,当浓度到达6 μmol/L以后,检测体系的吸光度几乎达到饱和,趋于平稳,故选择Hemin的终浓度为6 μmol/L。
2.2.2 95 ℃变性时间的优化
带有G-四联体互补序列的上下游引物经过PCR扩增后,含有G-四联体序列的双链不断积累,在互补碱基之间的氢键、碱基堆积力等分子作用力下会形成稳定的DNA双螺旋结构,影响G-四联体形成。高温可以引起双链DNA变性成单链,而富G序列在单链时才会组装形成特有的结构,最终达到G-四联体的显色效果。因此,使双链DNA充分变性,是G-四联体形成的前提。由图4B可知,当变性时间为1 min时,就能够出现肉眼可见的颜色变化,说明双链DNA遭到破坏同时形成了G-四联体;随着95 ℃变性时间的延长,显色体系吸光度逐渐增大,且在8 min时达到了峰值,之后呈现较为平稳的状态,吸光度不再有较大的波动,说明当温度达到95 ℃且变性8 min后,该体系中的双链达到全部解链的状态,因此选用8 min的变性时间进行后续实验。
2.2.3 4 ℃孵育时间的优化
PCR扩增产物中的双链最大程度的变性为单链,可保证G-四联体的形成量达到最多,那么从95 ℃到4 ℃的急速降温可使高温下变性成的单链DNA尽量维持其单链状态,又为G-四联体保持稳定打下基础。由图4C可知,当4 ℃孵育时间达到5 min时就已经有了肉眼可辨的颜色变化,当孵育时间达到10 min时,吸光度达到了最大,而后续随着孵育时间的延长,反而吸光度趋于下降。表明在10 min时,DNA复性成双链的速率还较低,且单链DNA与Hemin发生结合的几率大,吸光度高。而随着4 ℃温育时间的延长,单链自身恢复双螺旋结构的速率加快,与Hemin的复合效率变低,使得吸光度出现下降趋势。所以选用4 ℃孵育时间10 min进行后续的显色实验。
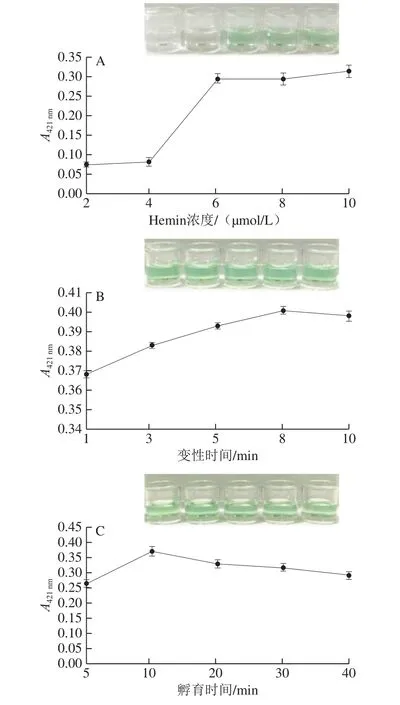
图4 Hemin浓度(A)、95 ℃变性时间(B)和4 ℃孵育时间(C)的优化Fig.4 Optimization of hemin concentration (A), denaturation (95 ℃)time (B) and incubation (4 ℃) time (C)
2.3 G-四联体与PCR联用可视化检测沙门氏菌的特异性
为评价该方法的特异性,分别对表1中16 株菌的基因组DNA进行提取,根据优化实验得出的最佳反应条件和最优反应体系进行PCR扩增和G-四联体显色,在保证其他菌株的DNA浓度远大于沙门氏菌DNA浓度且其他条件相同下,验证构建的可视化检测方法对沙门氏菌的特异性。由图5可知,只有含目标菌基因组DNA(沙门氏菌基因组DNA)的反应体系发生了明显的颜色变化,其他含非目标菌基因组DNA的反应体系均未变色,证明G-四联体与PCR联用可视化检测法对沙门氏菌具有高度的特异性。
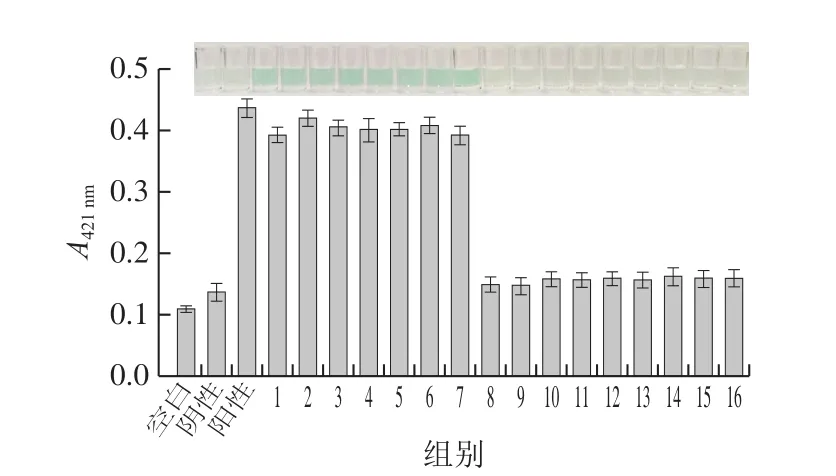
图5 G-四联体与PCR扩增联用可视化检测方法的特异性Fig.5 Specificity of the visual detection method
2.4 G-四联体与PCR联用可视化检测灵敏度的确定
取1 mL培养过夜的沙门氏菌进行基因组DNA的提取,之后将所提取的核酸进行梯度稀释并测其核酸含量,得到0.07、0.7、9.7、79.9、771.6 ng/μL的模板样品,在优化的反应条件和反应体系下进行G-四联体与PCR联用可视化检测,以确定用沙门氏菌基因组作模板时该方法的检出限。由图6A可知,空白对照颜色无明显变化,且吸光度随着沙门氏菌基因组DNA浓度的增加,颜色也越来越深。由图6B可见,模板质量浓度对数(lgC)与吸光度之间存在良好的线性关系,线性回归方程为y=0.129 9x+0.217 9,R2=0.994 5,灵敏度达到0.07 ng/μL,说明构建的G-四联体与PCR联用可视化检测方法的灵敏度效果好,足以对沙门氏菌属进行有效鉴别和定量检测。
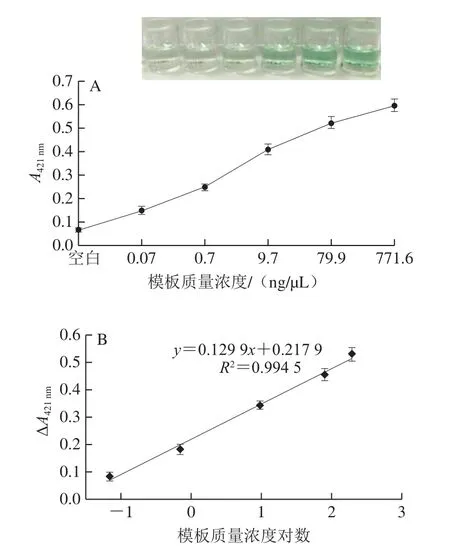
图6 G-四联体与PCR扩增联用可视化检测方法的灵敏度Fig.6 Sensitivity of the visual detection method
2.5 G-四联体与PCR联用可视化检测法对人工污染牛奶样品中沙门氏菌的检出限分析
利用G-四联体与PCR扩增联用可视化检测方法对沙门氏菌标准菌株污染的牛奶进行检测,其中人工污染的牛奶中菌浓度为1.2×105~1.2×101CFU/mL,由图7可知,当污染牛奶中的菌浓度为1.2×105~1.2×102CFU/mL时,显色较为明显,检出限为1.2×102CFU/mL,因此所建立的G-四联体与PCR扩增联用可视化检测方法便捷有效,可应用于实际检测中。但此次检测灵敏度相比于纯菌的检出限高,可能是由牛奶中蛋白含量高降低了基因组DNA的提取效率所致。

图7 G-四联体与PCR扩增联用可视化检测人工污染沙门氏菌的牛奶样品Fig.7 Application of the visual detection method to artificially contaminated samples
2.6 G-四联体与PCR联用可视化的实际应用与评价
对市场采集的30 份样品分别采用可视化检测与国标检测,结果如表2所示,国标检测方法检测沙门氏菌阳性2 例,采用G-四联体与PCR联用可视化检测得到相同的检测效果,检测准确率高达100%,说明本实验构建的方法可实现对市售样品的准确检测,具有实际应用潜力。
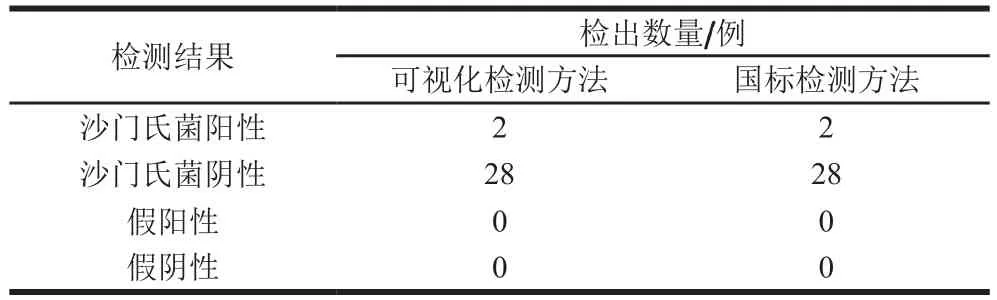
表2 实际样品检测效果的评价Table 2 Evaluation of the visual detection method for actual samples in comparison with the national standard method
3 讨论与结论
目前,关于PCR与G-四联体反应相结合的方法,已有对单核增生李斯特菌[30]、幽门螺杆菌[31]、乙肝病毒[32]的检测报道。但其研究或达不到定量效果;或只在引物的一端加入G-四联体的互补序列,导致反应循环数多且目标产物低;或是设计了复杂的催化信标探针,增加了操作难度。本实验将PCR扩增与G-四联体联用建立的可视化的沙门氏菌检测方法,具有如下特点,一是利用PCR的特异性识别和高效扩增目标DNA的特点,既可以通过引物与沙门氏菌特异性基因片段识别保证检测方法的特异性,又可以通过DNA的高效扩增积累大量的与扩增产物相连的G-四联体序列,增强灵敏度。二是利用G-四联体/Hemin的DNAzyme高效催化活性,催化H2O2将无色的ABTS2-氧化为绿色的ABTS*-。通过上述过程,可将两次放大的信号转变成肉眼可见的颜色变化,通过酶标仪可实现对沙门氏菌的高效、灵敏的定量检测,同时也可以通过裸眼观察颜色变化做出半定量或定性分析,避免了PCR凝胶电泳过程及EB污染等问题。
G-四联体作为一种模拟酶,与蛋白质结构的酶相比,具有较高化学稳定性、结构简单便于合成、易于修饰改良性状等特点,如何巧妙地利用G-四联体的过氧化物酶活性和生物传感器特性,是当前人们关注的焦点之一[33]。要实现PCR扩增和G-四联体联用可视化检测沙门氏菌,关键在于如何获得大量G-四联体展现其酶学特性,进而提高灵敏度。本研究主要采取如下措施:1)在设计引物时选择了随着富G序列的不断的延长,其稳定性无变化,可视化反应明显的平行G-四联体结构,提高反应的灵敏度[29];2)引物浓度与PCR扩增循环数是决定最终生成带有G-四联体序列的扩增产物量的关键因素,引物浓度低或扩增循环数少,生成的PCR产物及附带的G-四联体的量少、显色浅,影响灵敏度。循环数过高,虽然灵敏度提高了,但是过高的扩增结果将影响显色的梯度,掩盖了模板浓度(菌体浓度)的差异。因此,根据本研究结果选择4 μmol/L及以上浓度,扩增循环数在30左右,即可获得较高的灵敏度和较好的显色梯度效果。3)实验发现在引物上同时标记G-四联体的互补序列,无目标模板条件下扩增后不会造成本底信号的明显增加,也不影响有目标模板时PCR正常扩增,同时能在较少的循环数下,实现产物的大量积累(如相同循环数下获得的G-四联体的量理论上至少是不对称PCR法的2 倍)[34],得到较好的可视梯度变化。4)与通过不对称PCR及核酸外切酶酶切等方式获取单链DNA相比[35-36],本实验直接将PCR产物热变性,且在变性前加入Hemin工作液,使DNA双链解旋成富G单链DNA过程与Hemin结合形成G-四联体同时进行,减少了操作步骤和成本,缩短了检测时间。
总之,通过上述实验设计和一系列优化策略,建立了G-四联体与PCR联用可视化检测沙门氏菌的方法,该方法特异性强、灵敏度高、稳定性好、操作简便、易于观察、成本低,适合实地定性检测或借助酶标仪实现高通量检测,为食源性致病菌在核酸检测方面提供了新策略和思路。但实样检测与纯菌样品检测相比,由于DNA提取效率降低,将影响灵敏度;另外,针对不同实际样品,可能需要摸索和建立目标病原菌的菌数对数、提取的基因组浓度与吸光度之间的函数关系曲线,才能实现有效准确地检测。
