茶树COBRA基因家族全基因组鉴定及表达分析
2021-11-29艾安涛李燕丽章文益吕立堂
艾安涛,李燕丽,2,章文益,吕立堂,3*
(1.贵州大学茶学院,贵州 贵阳 550025;2.湄潭县茶产业发展中心,贵州 湄潭 564100;3.贵州大学农业生物工程研究院/生命科学学院,山地植物资源保护与种质创新省部共建教育部重点实验室,贵州 贵阳 550025)
茶树(Camelliasinensis(L.)O.Ktze.)是我国重要的经济作物之一,在“十三五”期间为脱贫攻坚做出了极大的贡献,“十四五”更将起着重要的作用。
茶树的生长发育,会受到干旱、高低温等不同的逆境胁迫,COBRA基因在茶树的生长发育和逆境响应中有着很重要的作用。COBRA基因是由Schindelman[1]在研究拟南芥的根发育时发现的,经研究COBRA基因作为一类GPI锚定蛋白,主要作用于细胞伸长、纤维素沉积的过程[2],COBRA蛋白的结构极为保守,主要包含 N 端信号肽、碳水化合物结合域、富含半胱氨酸的 CCVS 结构域和C末端 GPI(Glycosyl phosphatidyl inositol)蛋白的ω-位点[3]。COBRA基因对由根、茎、叶和其他营养器官的结构组织组成的细胞壁成分的生物合成至关重要[4]。李家洋[5]对COBRA编码蛋白BC1进行克隆鉴定,结果表明BC1基因通过降低细胞壁厚度和增加纤维素含量,在机械组织细胞壁的生物合成中起着重要的作用;在拟南芥中研究发现AtCOBL4参与次生细胞壁纤维素合成[6],AtCOBL9与根毛发育有关[7],以上研究结果表明COBRA基因广泛存在于植物中,并且与植物细胞壁、纤维素等有关。
目前为止,在高粱、亚麻、冬枣、毛果杨中鉴定出COBRA基因的家族成员,分别有10、24、10、14个。自茶树全基因组测序完成之后,关于茶树基因家族的鉴定和表达模式研究迅速开展,但是关于茶树COBRA基因家族的研究还未见报道。本试验通过对茶树基因组数据进行分析,从茶树基因组中鉴定到COBRA基因,并对该基因家族成员的蛋白理化性质、亚细胞定位、进化关系、保守结构域、染色体定位等进行生物信息学分析,同时分析了茶树COBRA家族成员在聚乙二醇(PEG)诱导的干旱胁迫、茉莉酸甲酯处理、盐胁迫、冷胁迫处理中的转录组数据,最后对茶树进行茉莉酸甲酯处理,基于荧光定量PCR技术的表达水平验证,为茶树COBRA家族基因机制和抗逆响应中的功能奠定基础。
1 材料与方法
1.1 试验材料
贵州大学茶学院种质资源圃中2年生‘福鼎大白茶’茶树为试验材料,移栽至人工气候室(光照:时间14 h,温度25℃,相对湿度65%,光照强度10000 Lux;黑暗:时间10 h,温度20℃,相对湿度65%)15 d,用于胁迫表达模式分析。
在茶树基因组数据库TPIA(http://tpia.teaplant.org/)、植物基因组数据库(https://plants.ensembl.org/index.html)、拟南芥基因组数据库(https://www.arabidopsis.org/)中下载茶树、烟草、拟南芥的全基因组数据,包括基因组注释、CDS、染色体定位、蛋白结构等信息,用于茶树COBRA基因家族的生物信息学分析。
1.2 茶树COBRA基因家族的筛选与分析
首先从拟南芥基因组数据库得到拟南芥COBRA基因家族,以拟南芥COBRA基因序列作为参考,在茶树基因组数据库得到的茶树基因组注释,利用COBRA基因特有的保守结构域模型进行BLASTP比对,设定阈值为E<1e-5,获得假设CsCOBRA基因。再用NCBI-CDD数据库(https://www.ncbi.nlm.nih.gov/)分析CsCOBRA基因的编码蛋白特征,确保CsCOBRA基因仅有其COBRA结构域。对获得序列整理并去除重复项,最后得到茶树COBRA基因家族成员。根据下载的茶树基因组数据库的perl脚本对茶树COBRA蛋白质的染色体、启动子、终止子、外显子、氨基酸长度进行分析,并用http://web.expasy.org/compute_pi/和https://www.genscript.com/tools/wolf-psort在线鉴定其相对分子质量、等电点和亚细胞定位。
1.3 茶树COBRA基因蛋白保守结构域分析
基于茶树COBRA蛋白序列结构,在 NCBI-CDD数据库(https://www.ncbi.nlm.nih.gov/)提取茶树COBRA蛋白序列中相关保守结构域,利用ClustalW程序进行多序列比对,并利用在线软件TBtools绘制出结构域图。
1.4 茶树COBRA基因结构、蛋白保守基序分析
将茶树COBRA基因组DNA序列和CDS 序列提交至 GSDS 2.0(http://gsds.cbi.pku.edu.cn/)进行基因结构分析,通过MEME(http://meme-suite.org/)进行保守结构域分析,motif数目设置为15个,motif 宽度范围设置为6~50氨基酸,并利用在线软件TBtool绘制出保守基序图。
1.5 茶树COBRA基因家族顺式作用原件分析
用TBtool软件从基因组序列中获取起始密码子上游2000 bp基因序列,并用plant CARE软件(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)分析其顺式作用原件,确定茶树COBRA基因家族顺式作用原件,结果用Excel制作表格。
1.6 茶树COBRA系统进化分析
根据ClustalW对下载的拟南芥、烟草与茶树的COBRA蛋白序列进行多序列比对,比对结果利用MEGA X软件构建进化树模型,再利用MEGA10.0的临近法(neighbor joining,NJ)分析,Boostrap参数设为1000,其他参数为系统默认值,构建系统进化树。
1.7 茶树COBRA染色体定位及分布
基于茶树基因组数据库TPIA(http://tpia.teaplant.org/),利用 HMM 3.0 软件中的 perl 脚本从基因组信息中获取CsCOBRA基因在染色体上的起始位置信息,利用TBtools软件基于基因注释信息对同源关系进行可视化。
1.8 茶树COBRA表达模式分析
为分析茶树COBRA基因基因家族的表达情况,从茶树基因组TPIA数据库(http://tpia.teaplant.org)下载它们在茶树8个组织(花、茎、根、顶芽、嫩叶、成熟叶、老叶、果实)及茉莉酸甲酯处理、冷胁迫、盐胁迫、PEG 诱导的干旱胁迫后的转录组数据(TPM值),同时鉴定差异表达基因,结果使用TBtools软件制作热图,最后选用Adobe Illustrator CS6软件加工、处理图片。
1.9 实时荧光定量PCR分析
基于转录组数据筛选到的CsCOBRA基因序列,利用IDT(https://sg.idtdna.com/pages)在线设计RT-qPCR引物(表1),引物序列由北京擎科生物科技有限公司合成。采用CTAB法,提取茉莉酸甲酯处理的福鼎大白茶总RNA,利用1.0%琼脂糖凝胶电泳和酸浓度检测仪检测其质量和浓度,利用天根Fastking gDNA Dispelling RT SuperMix kit将RNA反转录为cDNA。RT-qPCR反应体系:10 μL SYBR Green qPCR Mix,上下游引物各0.4 μL,cDNA模板1.5 μL,补加ddH2O至20 μL。反应程序:95℃预变性3 min;95℃变性10 s,60℃退火20 s,72℃延伸30 s,反应进行40个循环,以茶树CsGAPDH为内参基因。利用2-ΔΔCT算法[8]计算基因的相对表达量,利用Excel软件绘制柱状图。

表1 荧光定量PCR引物序列
2 结果与分析
2.1 茶树COBRA基因家族成员鉴定及理化性质预测
通过拟南芥、茶树基因组数据BLAST比对出来的假设CsCOBRA基因提交到NCBI-CDD在线网站进行保守域的结构验证,最终得到了16个茶树COBRA基因,并根据基因在染色体的位置及其同源关系将其命名,进一步对茶树COBRA基因编码的蛋白质进行理化性质分析(表2)。结果显示:茶树COBRA基因分布于1、2、7、9、11、13号染色体,外显子数为2~9个,编码区长度在889(CsCOBRA09)~8742(CsCOBRA08),相对分子质量最小的是24807.70(CsCOBRA10),最大的是74813.12(CsCOBRA12),等电点为5.43(CsCOBRA14)~9.14(CsCOBRA09),经WOLF-PSORT在线软件预测蛋白亚细胞定位,结果显示除CsCOBRA09定位在细胞外,其余茶树COBRA基因都是定位在细胞膜上。
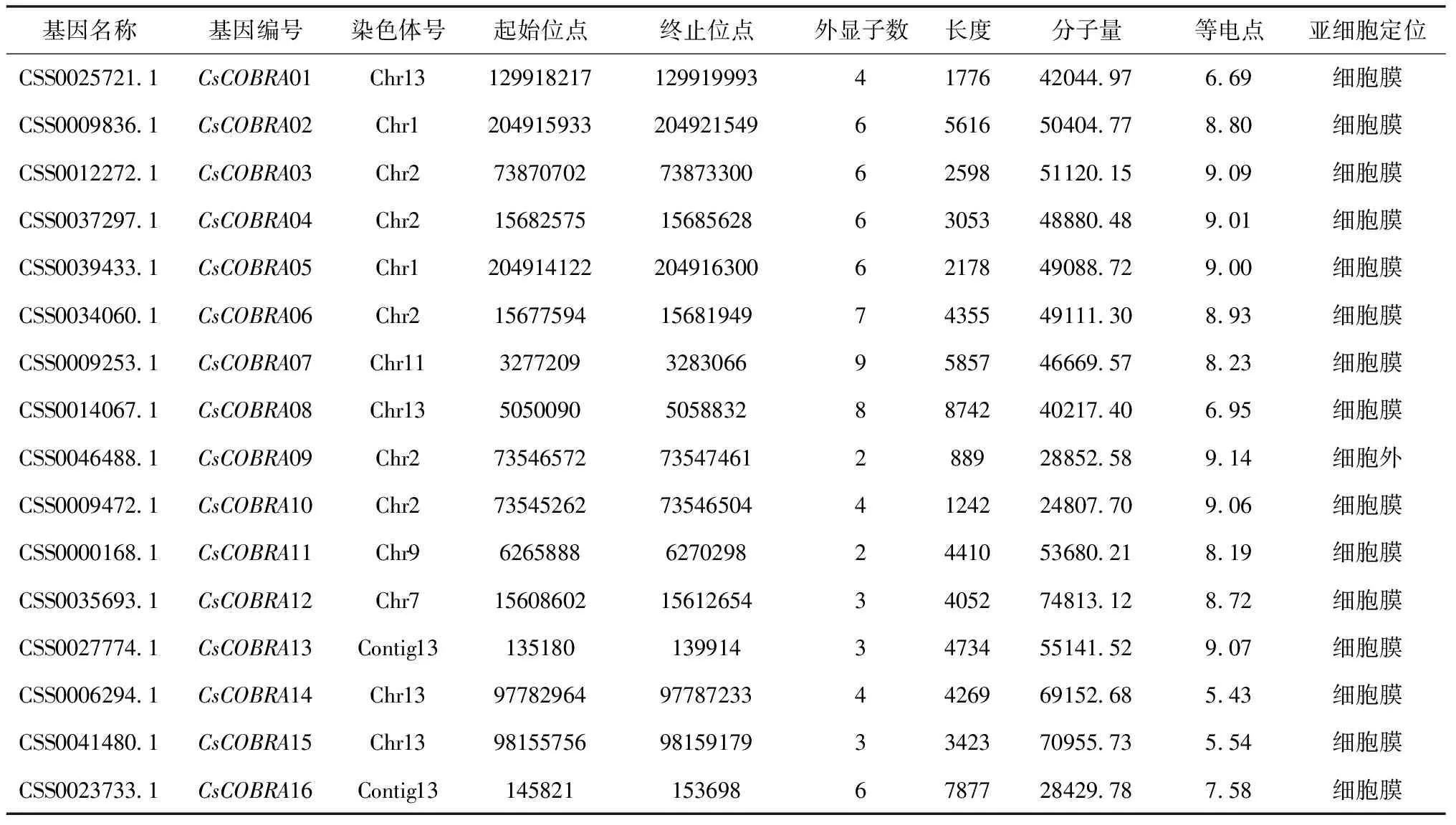
表2 茶树COBRA基因家族基因的特征
2.2 茶树COBRA基因蛋白保守结构域分析
根据茶树COBRA蛋白序列结构,在NCBI-CDD数据库提取相关保守结构域,并制作保守结构域图(图1)。结果显示除CsCOBRA09外,每个茶树COBRA基因仅有一个保守结构域COBRA,说明得到的16个茶树COBRA基因有很高的保守结构域。
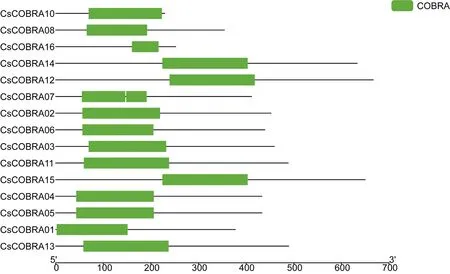
图1 茶树COBRA基因蛋白保守结构域图
2.3 茶树COBRA基因结构、蛋白保守基序分析
根据茶树COBRA基因组 DNA 序列和 CDS序列提交至 GSDS 2.0的结果进行基因结构分析,通过 MEME进行保守结构域分析,利用TBtools绘制基因结构、蛋白保守基序(图2)。据图2A显示,CsCOBRA02、CsCOBRA03、CsCOBRA05、CsCOBRA11、CsCOBRA12、CsCOBRA13有部分区段不能翻译蛋白质,外显子有2(CsCOBRA11)~9个(CsCOBRA07);据图2B的基序显示,茶树COBRA家族有一定的保守性,至少有15个保守基序(Motif)以上,由图可知CsCOBRA的基序个数由3个(CsCOBRA16)到11个(CsCOBRA12、CsCOBRA14、CsCOBRA15),Motif1~Motif15广泛分布在茶树COBRA基因中,CsCOBRA02~CsCOBRA05拥有相同的保守基序,CsCOBRA12、CsCOBRA14、CsCOBRA15也拥有相同的保守基序,Motif1除了CsCOBRA09之外都存在,Motif4、Motif6除了CsCOBRA09、CsCOBRA16不存在之外在其他CsCOBRA都有,Motif2、Motif5、Motif7、Motif8、Motif12分布于茶树COBRA13条基因中,结果说明Motif1、Motif4、Motif6、Motif2、Motif5、Motif7、Motif8、Motif12在茶树COBRA基因中发挥着重要的作用。
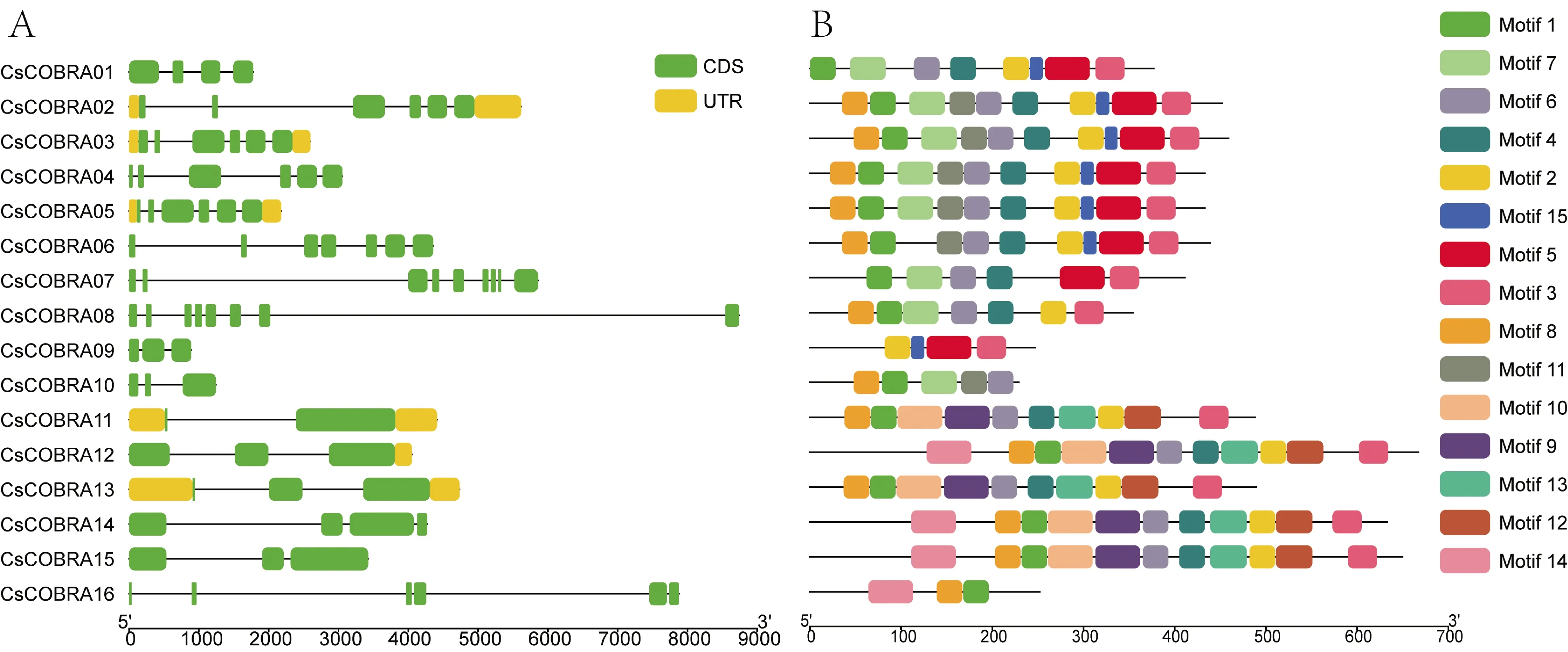
图2 茶树COBRA蛋白的基因结构、保守基序分析
2.4 茶树COBRA基因家族顺式作用原件分析
将鉴定到的16条CsCOBRA基因利用plant CARE软件确定顺式作用原件(表3),结果表明:有81%(13/16)的CsCOBRA具有光相应原件,说明大多数CsCOBRA家族成员与光反应有关;大多CsCOBRA基因还含有脱落酸、赤霉素、生长素、水杨酸、茉莉酸甲酯等激素类顺式作用原件,除此之外还有脱水、低温、盐、干旱、冷胁迫的顺式作用原件,说明CsCOBRA基因广泛参与了茶树生长发育和响应非生物胁迫作用。
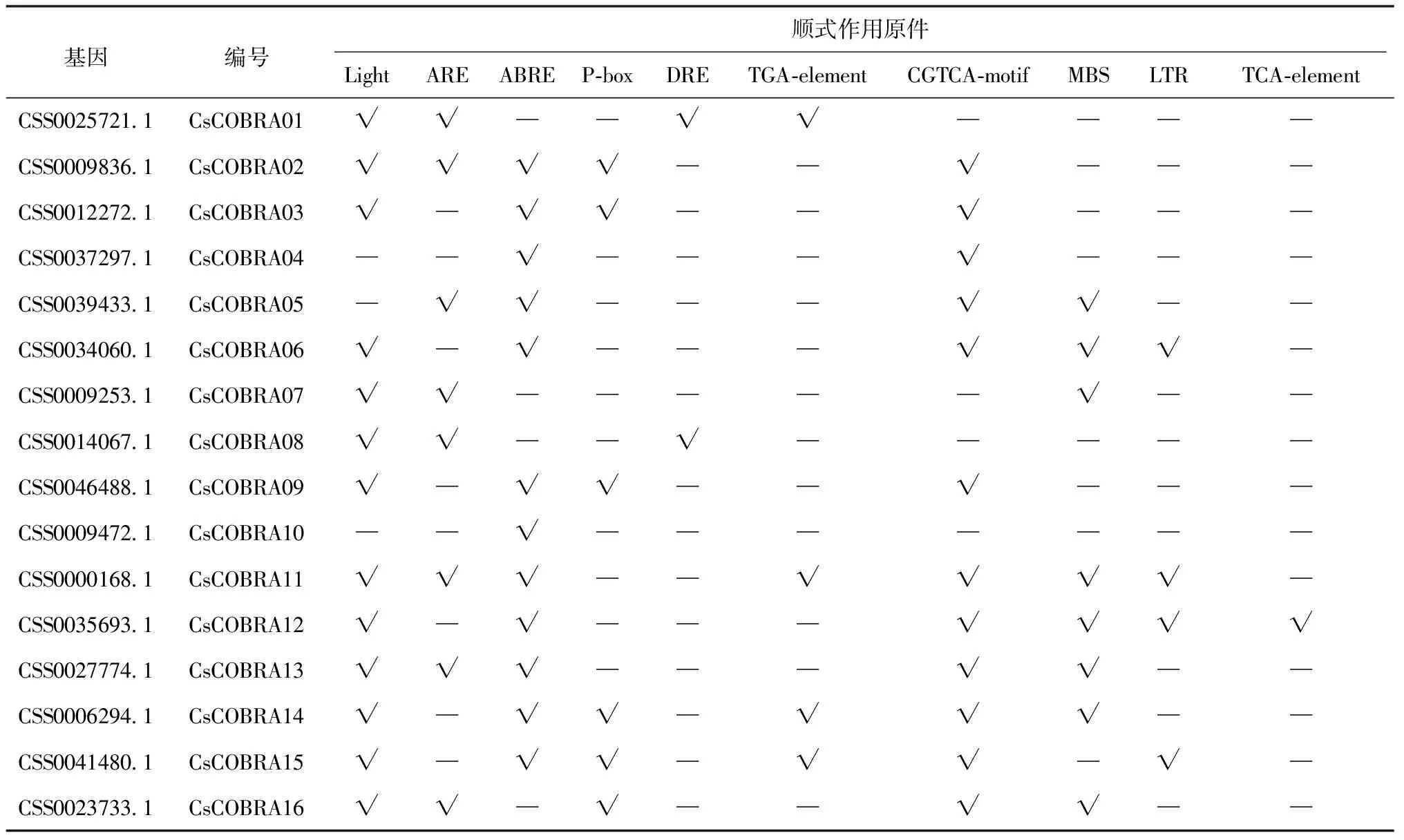
表3 茶树COBRA顺式作用元件分析
2.5 茶树COBRA蛋白序列系统进化分析
为了研究茶树COBRA基因家族成员的分类,以 11个拟南芥COBRA蛋白和14个烟草COBRA蛋白的蛋白序列作为参考,与筛选得到的16个茶树COBRA基因蛋白进行多序列比对并构建进化树(图3)。结果发现拟南芥、烟草、茶树COBRA基因在5个亚家族中都有分布,茶树COBRA基因和拟南芥、烟草同源性很高,COBRA基因物种分化非常保守,3个物种的COBRA基因可能具有相似的生物学功能。根据拟南芥和烟草的同源基因分类,将拟南芥、烟草、茶树COBRA基因家族分为5个亚家族,茶树COBRA在GroupⅠ~GroupⅤ亚家族分别有3、2、5、3、3个成员。
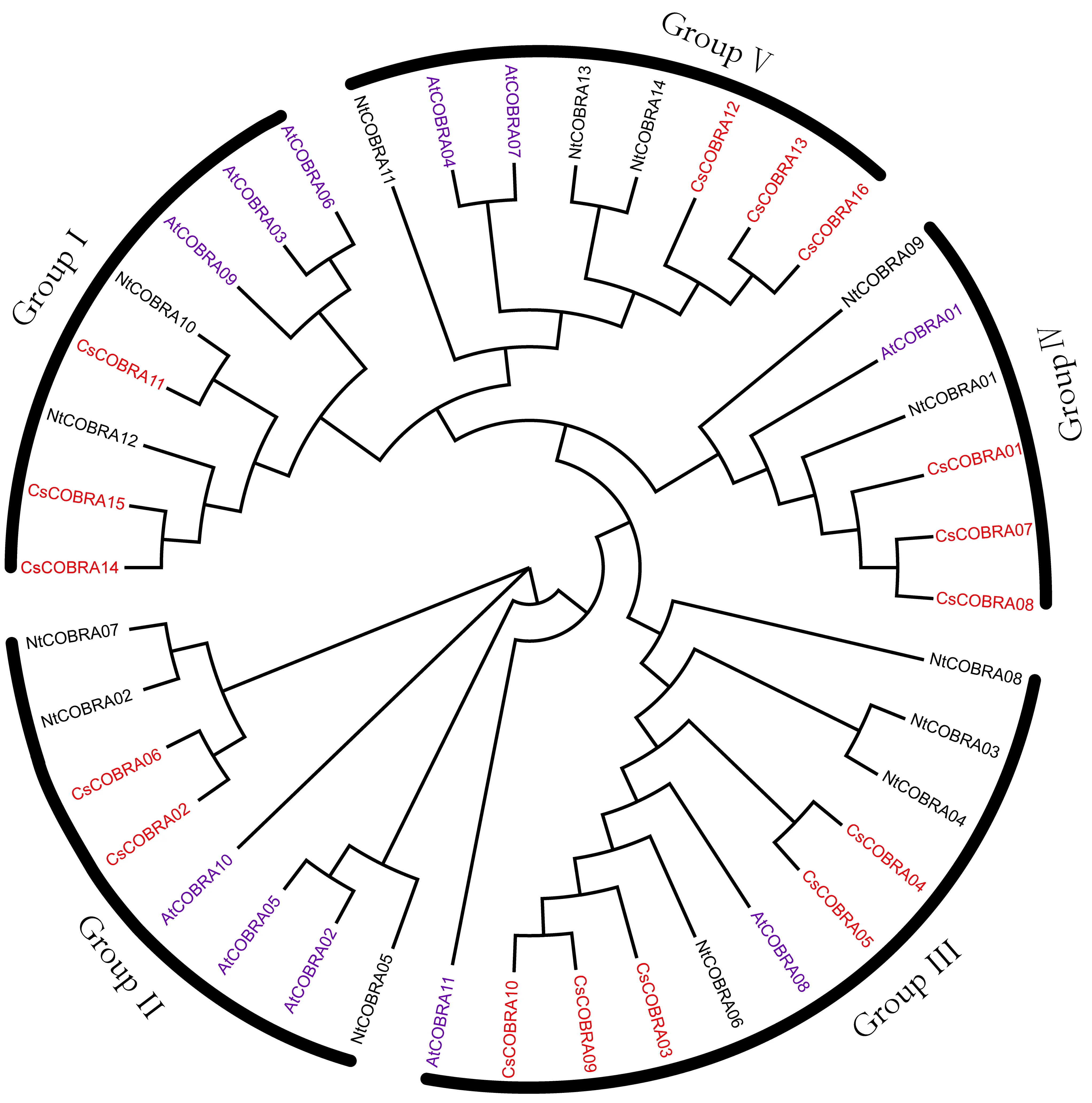
图3 茶树、烟草和拟南芥COBRA基因家族的进化分析
2.6 茶树COBRA染色体定位及分布
为了解茶树COBRA染色体定位分布情况,将鉴定到的16条CsCOBRA基因与茶树基因组数据库TPIA比对分析,获取CsCOBRA基因在染色体上的起始位置信息,利用TBtools软件绘制染色体定位(图4)。结果发现除了CsCOBRA13、CsCOBRA16不能定位到染色体外,其余CsCOBRA基因不均匀的分布在Chr1、Chr2、Chr7、Chr9、Chr11、Chr13号染色体上,其中CsCOBRA12、CsCOBRA11、CsCOBRA07分别定位到染色体Chr7、Chr9、Chr11上,CsCOBRA02、CsCOBRA05定位到染色体Chr1,CsCOBRA03、CsCOBRA04、CsCOBRA06、CsCOBRA09、CsCOBRA10定位到染色体Chr2,CsCOBRA01、CsCOBRA08、CsCOBRA14、CsCOBRA15定位到染色体Chr13上。
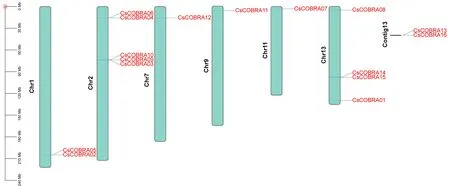
图4 茶树COBRA 基因的染色体位点
2.7 茶树COBRA表达模式分析
为了解茶树COBRA基因基因家族的表达情况,从TPIA数据库下载它们在茶树8个组织(花、根、茎、嫩叶、成熟叶、老叶、顶芽、果实)的转录组数据,结果使用TBtools软件制作热图(图5)。在不同组织热图中结果显示CsCOBRA01~CsCOBRA16基因在茶树各部位都有表达,其中CsCOBRA02、CsCOBRA11、CsCOBRA13、CsCOBRA16、CsCOBRA06的表达量较高,CsCOBRA04、CsCOBRA08、CsCOBRA01、CsCOBRA07、CsCOBRA05、CsCOBRA14、CsCOBRA15的表达量较低,CsCOBRA02、CsCOBRA11在根和茎的表达量是最高的,其次CsCOBRA13、CsCOBRA16表达量相同且在花中最高,CsCOBRA03、CsCOBRA09、CsCOBRA10表达量相同且在茎中最高,CsCOBRA01、CsCOBRA07、CsCOBRA08和CsCOBRA14、CsCOBRA15表达量相同在各部位表达量都比较低,由此可见不同CsCOBRA基因参与到茶树不同部位的生长过程。
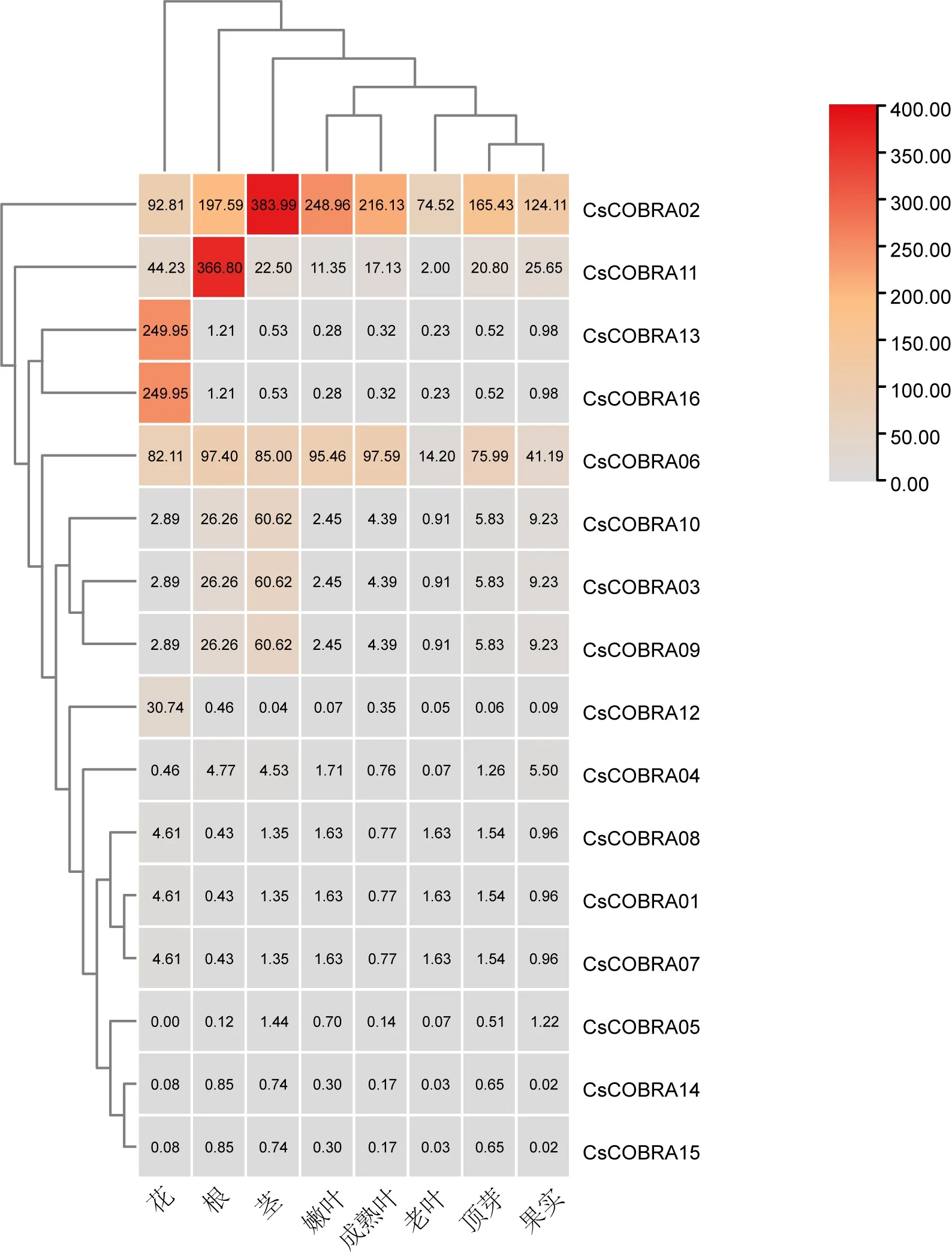
图5 茶树COBRA基因在不同组织中表达模式
本研究对茶树CsCOBRA基因在茉莉酸甲酯(0、12、24、48 h)、冷胁迫处理(CK、CK1、CK3)、盐胁迫(0、24、48、72 h)、干旱胁迫(0、24、48、72 h)4种胁迫的转录组数据进行整理,绘制热图(图6)。在茉莉酸甲酯处理后,CsCOBRA02的表达量逐渐升高,CsCOBRA06、CsCOBRA11在12、24 h的表达量降低,后逐渐升高,其余基因随着时间的推移而表达量没有明显的变化,而且表达量都比较低;在冷胁迫下,CsCOBRA02、CsCOBRA06的表达量逐渐升高,CsCOBRA11的表达量稍微提高,CsCOBRA01、CsCOBRA02、CsCOBRA04、CsCOBRA05、CsCOBRA07、CsCOBRA08、CsCOBRA09、CsCOBRA10、CsCOBRA12、CsCOBRA13、CsCOBRA14、CsCOBRA15的表达量受到冷胁迫的影响都很低,有一些甚至不表达;在盐胁迫下CsCOBRA02、CsCOBRA06、CsCOBRA11的表达量较高,特别是CsCOBRA06基因,其余基因检测到的表达量都很低;在干旱胁迫下CsCOBRA02、CsCOBRA06、CsCOBRA11的表达量较高。由上可知,CsCOBRA02、CsCOBRA06、CsCOBRA11在不同的胁迫处理下,表达量都相对较高,除了这3种基因外,其余基因在胁迫处理下的表达量都很低,有一些甚至不表达。
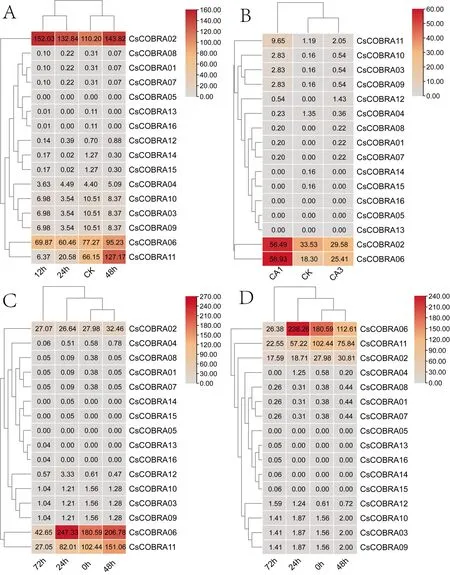
图6 茶树COBRA基因在茉莉酸甲酯、冷、盐、和干旱胁迫下的表达分析
2.8 实时荧光定量PCR分析
为验证转录组测序结果的可靠性,本研究根据基因在8个组织(花、茎、根、顶芽、嫩叶、成熟叶、老叶、果实)及4种胁迫(茉莉酸甲酯处理、冷胁迫、盐胁迫、PEG 诱导的干旱胁迫)的转录组数据,选取3个CsCOBRA基因(CsCOBRA02、CsCOBRA06、CsCOBRA11),对福鼎大白茶树进行茉莉酸甲酯处理,采用荧光定量PCR技术对CsCOBRA02、CsCOBRA06、CsCOBRA11的表达情况进行了检测(表4,图7)。
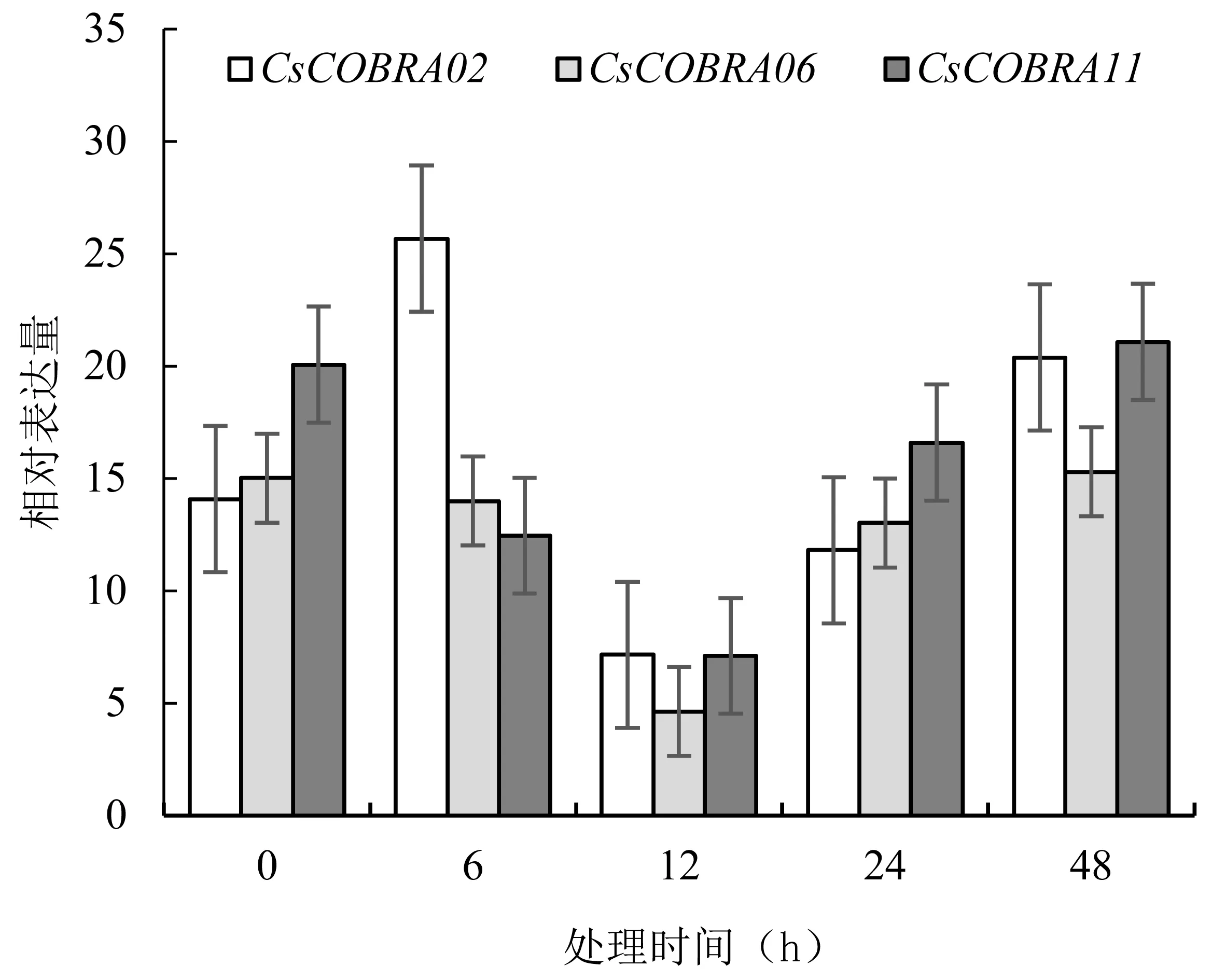
图7 RT-PCR分析CsCOBR基因在茉莉酸甲酯处理下的表达分析
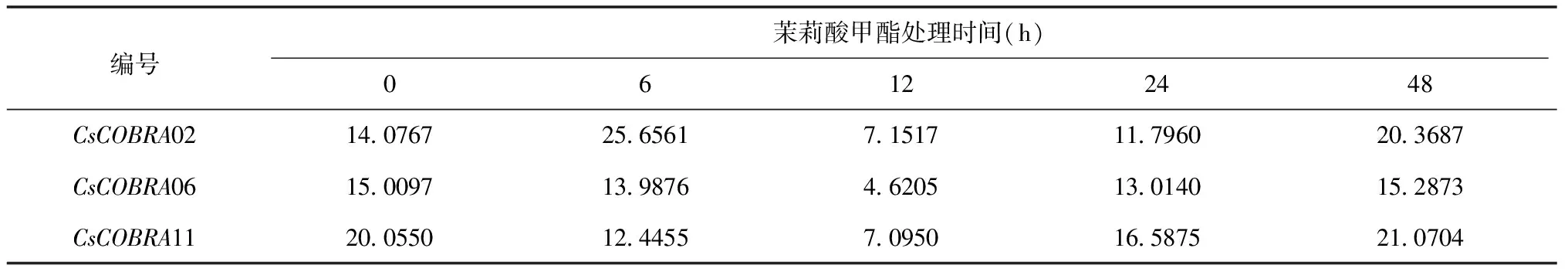
表4 RT-PCR分析CsCOBR基因在茉莉酸甲酯处理下的表达分析
结果显示:茉莉酸甲酯处理0~48 h,CsCOBRA02在6 h时表达量最高,之后表达量由高到低,最后升高;CsCOBRA06和CsCOBRA11的表达量大致走向由高到低,而后继续升高。在12 h时CsCOBRA02、CsCOBRA06、CsCOBRA11表达量最低,为7.1517、4.6205、7.0950。RT-PCR荧光定量分析与转录组茉莉酸甲酯处理数据表达量有所差异,但是表达趋势基本一致。
3 结论
COBRA基因家族的研究在高粱[9]、亚麻[10]、冬枣[11]、毛果杨[12]、棉花[13]、玉米[14]等物种中鉴定出相关成员,家族成员数量分别是10、24、10、14、9、19个。本研究鉴别茶树COBRA基因家族总共有16个成员,与高粱、亚麻、冬枣、毛果杨、棉花、玉米鉴定出来的家族成员数量没有太大的差异,说明鉴定结果有一定的可靠性。根据理化性质分析,相对分子质量24807.70~74813.12,等电点为5.43~9.14,亚细胞定位显示CsCOBRA基因几乎都是定位在细胞膜上;CsCOBRA基因几乎只有一个保守结构域COBRA,说明茶树COBRA基因非常保守;CsCOBRA基因的蛋白结构、保守基序(motif)也是均匀分布;茶树COBRA基因与拟南芥、烟草的多序列比对结果表明,茶树COBRA基因在5个亚家族中都有分布,拟南芥、烟草、茶树的COBRA基因同源性很高;茶树COBRA基因均匀分布在各染色体上;8个组织和茉莉酸甲酯处理、冷胁迫、盐胁迫、PEG 诱导的干旱胁迫后的转录组数据得到热图,CsCOBRA02、CsCOBRA06、CsCOBRA11在不同的胁迫处理下,表达量都很高,所以将CsCOBRA02、CsCOBRA06、CsCOBRA11设计特异性引物,在不同时间的茉莉酸甲酯处理下荧光定量PCR分析,结果说明3个CsCOBRA基因表达量趋势大致与转录组数据一致。
COBRA基因编码一种糖磷脂酰肌醇锚定蛋白,控制细胞壁、纤维素微纤丝的正确定位和细胞的定向伸长,对植物器官的形态发生起重要作用,与植物的寿命、体积、高度、细胞壁的机械强度有关[15]。茶树是多年生植物,是我国重要的经济作物。茶树的生长发育,会受到环境和人为的影响,研究结果显示CsCOBRA基因可能与茶树的细胞壁、纤维素,还有寿命、机械强度有关,间接影响茶叶的产量。目前对茶树COBRA基因家族的研究还未见报道,本研究对茶树COBRA基因进行鉴定分析,将为深入了解茶树COBRA基因奠定基础。
