高丹草低氢氰酸含量性状主效QTL PA7-2的精细定位
2021-11-09吴国芳于肖夏杨东升卢倩倩李景伟李佳奇
吴国芳, 于 卓, 于肖夏, 杨东升, 卢倩倩, 李景伟, 李佳奇
(内蒙古农业大学农学院, 内蒙古 呼和浩特 010019)
高丹草是高粱(Sorghumbicolor)与苏丹草(Sorghumsudanense)种间杂交育成的一年生重要饲用牧草之一,具有杂种优势强、营养价值高、分蘖数多,再生性好、耐盐碱、可多次刈割利用等优良特性[1-4],但是茎叶中含有一定量的氢氰酸含量,家畜采食过量会引起中毒现象[5-6],生产上亟待培育低氢氰酸含量(每公斤鲜草≤15 mg)的高丹草新品种。
数量性状QTL(Quantitative trait locus,QTL)定位分为初步定位和精细定位。初步定位到的QTL区域,精度一般>10 cM,难以确定QTL的具体位置,并且不能判断该区域是含有1个主效QTL还是多个微效QTL[7]。精细定位是在初步定位的基础上,针对主效QTL区域,构建遗传背景相同的次级分离群体,在已构建遗传图谱的基础上探索新的分子标记,对现有的图谱进行补充,增大分子标记的密度,将目标基因位点控制在一个更精确的区间内,精度一般在1~5 cM[8]。群体分离分析法(Bulked segregation analysis,BSA)是与分子标记相结合快速筛选目标性状相关标记的方法,通常与简单重复序列(Simple sequence repeats,SSR)分子标记结合应用,即基于BSA-SSR方法,借助紧密连锁的分子标记和表型数据进行主效QTL定位[9-11]。通过主效QTL位点的两个侧翼标记筛选等位基因重组株QIRs(Quantitative trait locus (QTL) isogenic recombinants),选出与表型性状相关的基因位点,并进行选择性基因分型、表型鉴定和精细定位[12,13],这是从正向遗传学角度发掘数量性状主效基因、功能解析及标记辅助育种的重要途径[14-17]。目前在水稻、小麦、玉米和大豆等作物上已有相关研究报道[18-21],而在饲草上迄今尚未见有报道。
本课题组前期通过研究高丹草高密度分子遗传图谱构建、氢氰酸含量性状的QTL定位发现,氢氰酸含量性状是由多基因调控的数量性状,受遗传因子和环境因素的影响[5-6]。其中,房永雨等[22]定位了控制氢氰酸含量性状的2个微效QTL cn1和cn2,位于高丹草5号和7号染色体上,遗传贡献率分别为7.4%和8.6%;于肖夏等[23]定位了1个低氢氰酸含量主效QTLPA7-1,其位于7号染色体,遗传贡献率为25.3%;吴国芳等[5]在多个环境下定位了控制氢氰酸含量性状的16个QTLs,它们分布在1,2,4,6,7,8和10号染色体上,遗传贡献率范围为0.87%~39.9%,其中贡献率大于20%的主效QTLs共4个,与低氢氰酸含量性状相关的有3个,分布在4、6和7号染色体上,在2个环境中表现稳定的QTL有9个,在4个环境中找到一个新的表现稳定的主效QTLPA7-2,遗传贡献率为33.1%。
本试验采用BSA-SSR方法,以高丹草F2分离群体和其亲本为材料,从中选出等位基因重组株QIRs群体,并套袋自交获得F3群体,通过鉴定F3群体低氢氰酸含量基因型得到重组株后,重点对主效QTLPA7-2进行精细定位研究,以期为高丹草低氢氰酸含量性状候选基因挖掘、功能解析及分子标记辅助育种奠定基础。
1 材料与方法
1.1 试验材料与种植
材料为散穗高粱×红壳苏丹草的F2分离群体1 200个单株、F3分离群体500个单株及其亲本。分别于2019年4月22日与2020年5月24日种植在内蒙古农业大学农场,行距为50 cm,株距为20 cm,试验地位于呼和浩特市赛罕区,土壤为沙壤土,pH=7.8~8.2,肥力中等(N为60.5 mg·kg-1,P为8.5 mg·kg-1,K为100.6 mg·kg-1)[5]。生长期间根据生长状况及时施肥、灌水,确保植株对养分和水分的需求。
1.2 氢氰酸含量测定
在高丹草拔节期,株高约为100 cm时,田间剪取F2和F3分离群体单株及亲本的新鲜茎叶,按单株用微型粉碎器粉碎混匀,每个样品分别称取1.0 g,进行蒸馏。采用汪建飞等[24]提出的异烟酸-吡唑啉酮显色测定法(λmax=636.5 nm)测定样品中的氢氰酸含量,每个样品重复测定3次。
1.3 DNA提取及BSA基因池建立
用天根公司生产的植物基因组DNA提取试剂盒提取高丹草F2、F3代低氢氰酸含量(<15 mg·kg-1,简称低氰)和高氢氰酸含量(>300 mg·kg-1,简称高氰)的极端单株各10株及母本散穗高粱(高氰)和父本红壳苏丹草(低氰)的DNA。从极端株中各吸取5 μL的DNA进行等量混合,形成低氰基因池和高氰基因池,用于筛选与低氢氰酸含量性状相关的SSR标记。
1.4 SSR引物设计及PCR扩增
根据高粱基因组EST序列,借助SSR Hunter软件查找SSR位点,利用Primer 5软件设计出SSR引物序列[25],委托南京金瑞斯生物科技有限公司合成。
PCR反应体系总体积为20 μL,其中含Mg2+的10×buffer 2.0 μL,2.5 mM的dNTP 1.5 μL,5 U·μL-1的Taq-poLymerase 0.2 μL,10 ng·μL-1上下游引物各1 μL,模板DNA 2 μL,ddH2O 12.3 μL。
扩增程序为循环前94℃预变性5 min,94℃变性30 s,56℃退火30 s,72℃延伸1 min,38个循环;循环后72℃延伸10 min,4℃保存。PCR反应结束后,加入5.0 μL变性剂,95℃变性5 min,迅速取出在冰上冷却,置-20℃条件下保存备用。
1.5 等位基因重组株(QIRs)筛选
QTL等位基因重组株(QTL isogenic recombinants,QIRs)是指在检测到的QTLs中,只在1个QTL位点是杂合的,目标区域会发生大量重组,其他QTLs位点是纯合的单株[26]。本试验用课题组前期得到的PA7-2的两个侧翼标记(B4195-500和Xtxp31-600)对1200个F2分离群体进行筛选,选出PA7-2区域为杂合的单株,构成QIRs群体。
1.6 遗传连锁图谱构建与QTL定位
1.6.1遗传连锁图谱构建 采用Join map 4.0遗传作图软件,设置LOD阈值≥3.0条件下,对高丹草F3代群体分离单株基因组DNA经PCR扩增获得的SSR多态性标记及课题组前期得到的QTLPA7-2侧翼的5个SSR标记(B4195-500、Xtxp183-950-Xtxp121-470-Xtxp321-220和Xtxp31-600)进行遗传作图,用Map chart 2.5软件绘制出高丹草的新连锁群图谱。
1.6.2QTL定位 采用区间作图法(Interval mapping,IM),Map QTL 6.0分析软件,在扫描步长为1.0 cM、α=0.001的水平上,利用置换检测(Permutation test)检验法做1 000次重复,将假阳性错误概率限制在5%,估算QTL范围内的LOD≥3.0阈值,对F3群体单株的低氢氰酸含量测定值进行QTL定位分析,检测出与低氢氰酸含量相关的QTLPA7-2。
1.7 主效QTL PA7-2精细定位
为了验证高丹草低氢氰酸含量主效QTLPA7-2的稳定性及保证精细定位的准确性,从F2群体中选取了在PA7-2处为杂合,PA7-1处为纯合,并且在侧翼标记区域的标记表型分别为低氢氰酸含量和高氢氰酸含量的极端重组株各5株套袋自交获得F3种子,从每个极端单株中选取颗粒饱满的50粒种子(共500粒种子),于2020年5月10日在内蒙古农业大学农场试验地种植形成500个单株的F3分离群体。
在精细定位区间内进一步开发SSR标记以缩小定位区间。通过高粱基因组序列信息查找与氢氰酸性状相关的基因序列,然后利用网站(http://archive.gramene.org/markers/)信息设计了12对特异SSR引物(表1),对F3精细定位群体进行PCR扩增,得到SSR多态性标记,建立遗传图谱并进行主效QTLPA7-2精细定位[27]。进而利用与QTLPA7-2紧密连锁的SSR标记对F3精细定位群体单株中的30个QIRs株进行精细定位。极端个体挑选的原则为:低氢氰酸含量显著小于隐性对照CK1(10.99 mg·kg-1),高氢氰酸含量显著大于杂合型对照CK2(134.37 mg·kg-1);高氢氰酸含量极显著大于显性对照CK3(323.40 mg·kg-1)。从每个对照中随机选取20个植株,测定其氢氰酸含量的平均值和标准差,将这些值与极端重组的值进行比较,确定关键重组位点[27]。

表1 12对特异SSR引物序列Table 1 The nucleotide sequences of 12 pairs of specific SSR primers combinations
1.8 数据处理与统计
用Excel 2007软件进行数据处理,利用SPSS 25.0统计软件进行氢氰酸含量性状表型正态分布分析。
2 结果与分析
2.1 氢氰酸含量性状分布特征
在株高约100 cm时母本散穗高粱茎叶鲜草氢氰酸含量均极显著地高于父本红壳苏丹草,其杂种F2代群体1 200个单株的氢氰酸含量性状分离明显,大多数个体介于双亲之间,少部分个体呈超亲现象,且测定值频率分布符合正态特征(图1),偏度和峰度均小于1(表2),表明高丹草氢氰酸含量性状呈数量性状遗传,可用于QTL定位分析。
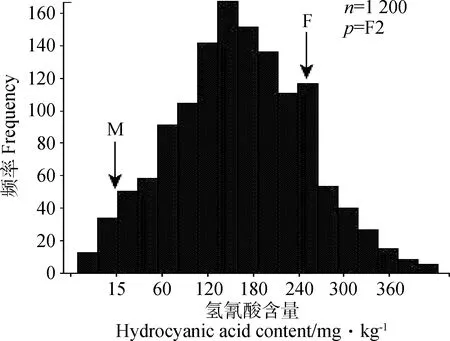
图1 高丹草F2群体氢氰酸含量测定值频率分布图Fig. 1 Frequency distribution of hydrocyanic acid contents in F2 populations of sorghum-sudangrass hybridF:♀散穗高粱;M:♂红壳苏丹草;n为群体大小;p为群体类型;箭头表示各亲本氢氰酸含量值F:Female parent scattered ear sorghum;M:Male parent red hull sudangrass;n is the population size;p is population type. Arrows represent the hydrocyanic acid content values of parents
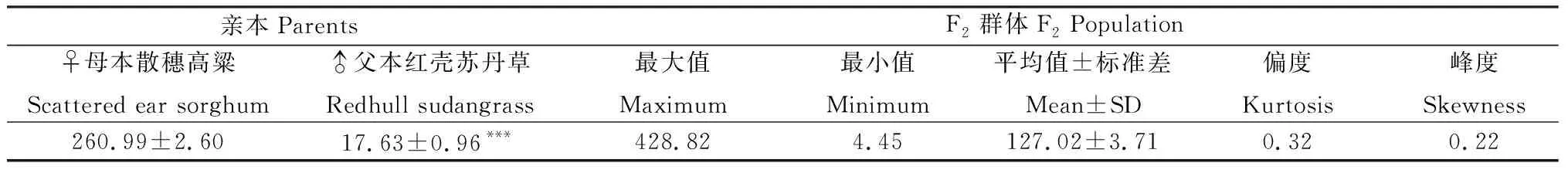
表2 高丹草F2群体及其亲本氢氰酸含量性状表型变异Table 2 Phenotypic variation of hydrocyanic acid content in F2 population and its parents of sorghum-sudangrass hybrid 单位:mg·kg-1
2.2 重组株(QIRs)筛选
利用定位获得的QTLs紧密连锁的侧翼标记,以低氢氰酸含量主效QTLPA7-2(B4195-500、Xtxp31-600)区域为杂合的单株进行筛选,从1200个单株的F2大群体中筛选到121个重组株形成了PA7-2的QIRs群体,该群体呈偏正态分布,大部分单株氢氰酸含量≤60 mg·kg-1,比低氰父本红壳苏丹草的含量高,极显著低于母本散穗高粱(260 mg·kg-1),表明筛选出的重组株大部分属于低氢氰酸含量,且是杂合的(图2和表3)。
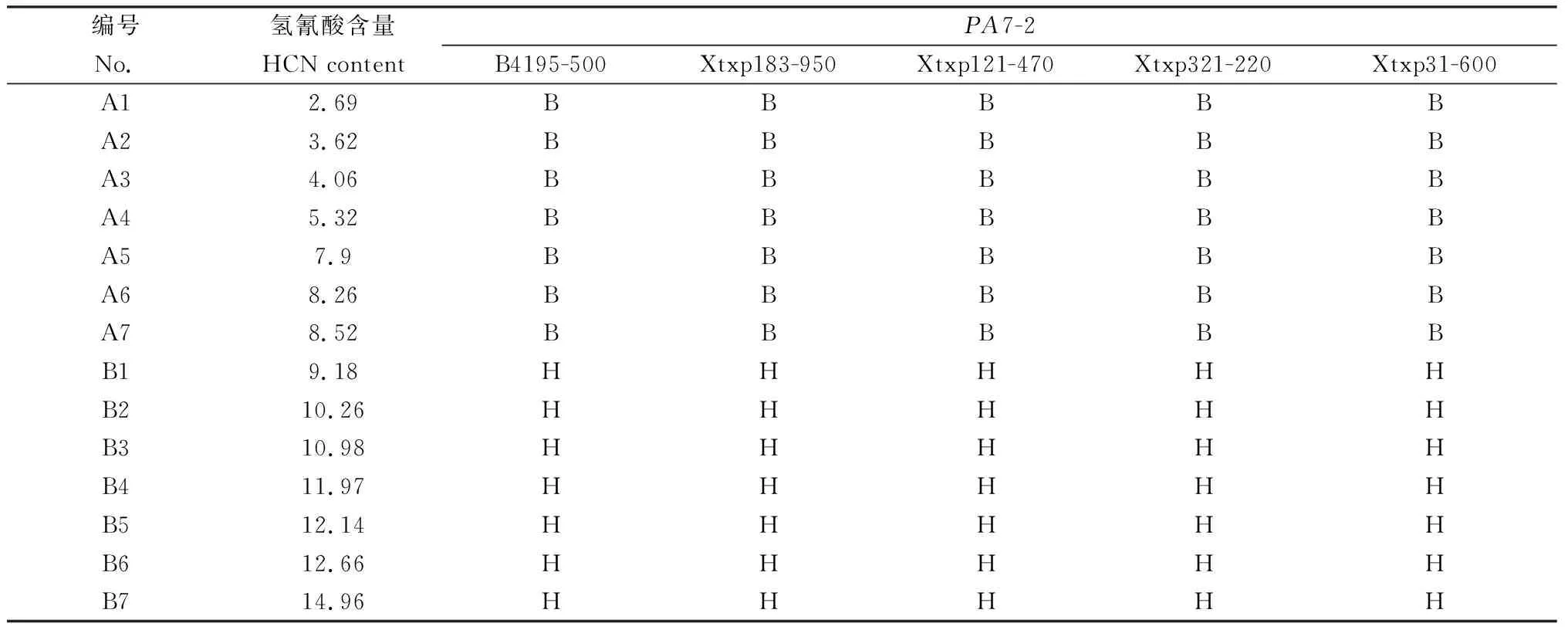
表3 主效QTL PA7-2区域的部分低氰QIRs的获得Table 3 Partial low-cyanide QIRs obtainment in the major QTL PA7-2 region
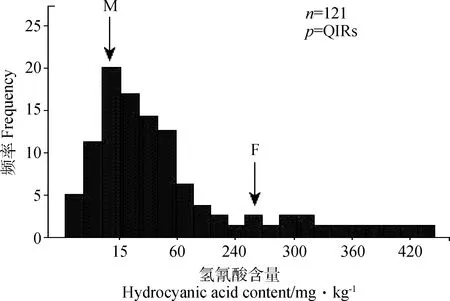
图2 高丹草F2代QIRs株氢氰酸含量测定值频率分布图Fig. 2 Frequency distribution of hydrocyanic acid contents in F2 generation quantitative trait locus isogenic recombinants of sorghum-sudangrass hybridF:♀散穗高粱;M:♂红壳苏丹草;n为群体大小;p为群体类型;箭头表示双亲的氢氰酸含量值F:Female parent scattered ear sorghum;M:Male parent red hull sudangrass;N is the population size;P is population type. Arrows represent the hydrocyanic acid content values of each parent
2.3 F3精细定位QIRs群体的建立
从QIRs中获得的极端单株(低氰植株≤15 mg·kg-1,高氰植株>260 mg·kg-1)经套袋自交后,得到F3分离群体,用QTLPA7-2的侧翼标记(B4195-500、Xtxp183-950、Xtxp121-470、Xtxp321-220和Xtxp31-600)筛选该群体,得到130个重组株构成了PA7-2的精细定位群体,其氢氰酸含量亦呈正态性分布,大多数重组株的氢氰酸含量<60 mg·kg-1,为低氰杂合型,表明该群体用于主效QTLPA7-2的精细定位是可行的(图3,表4)。
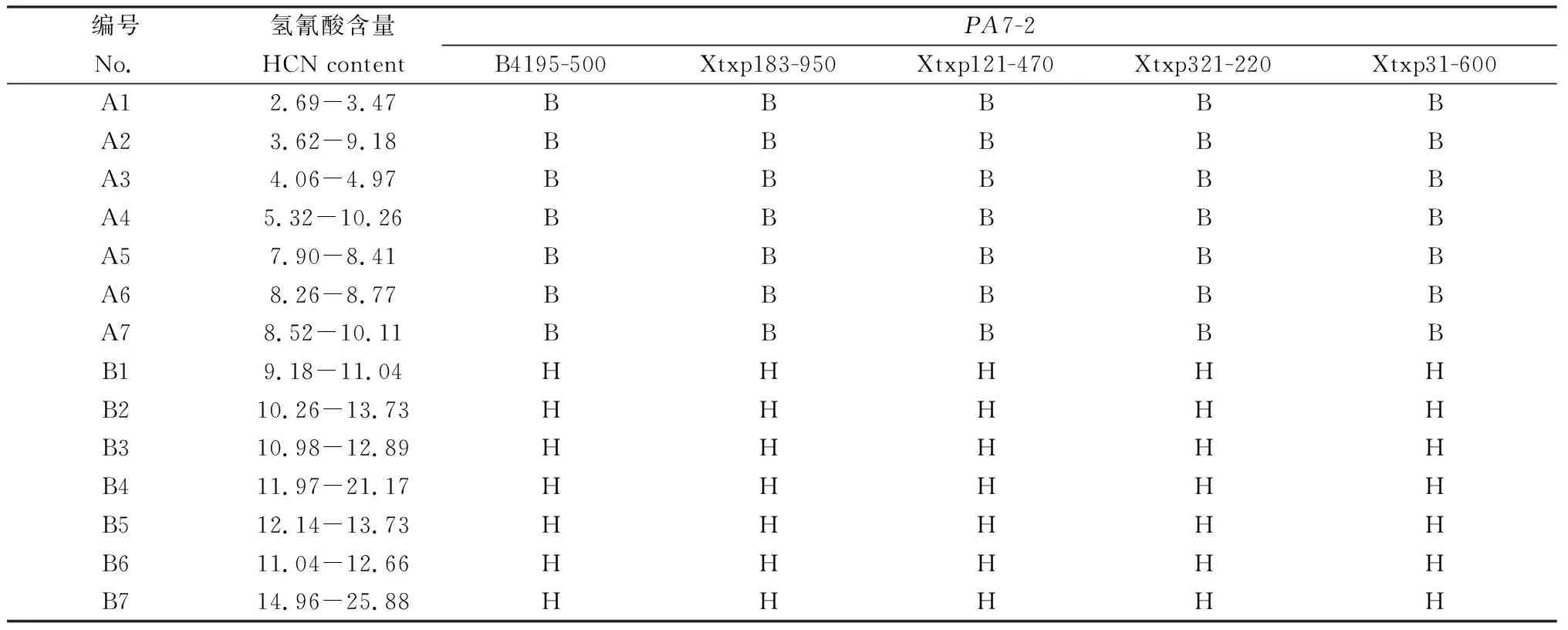
表4 高丹草的F3代部分QIRs氢氰酸含量性状分离Table 4 Segregation of hydrocyanic acid content characters of partial quantitative trait locus isogenic recombinants in F3 generation of sorghum-sudangrass hybrid
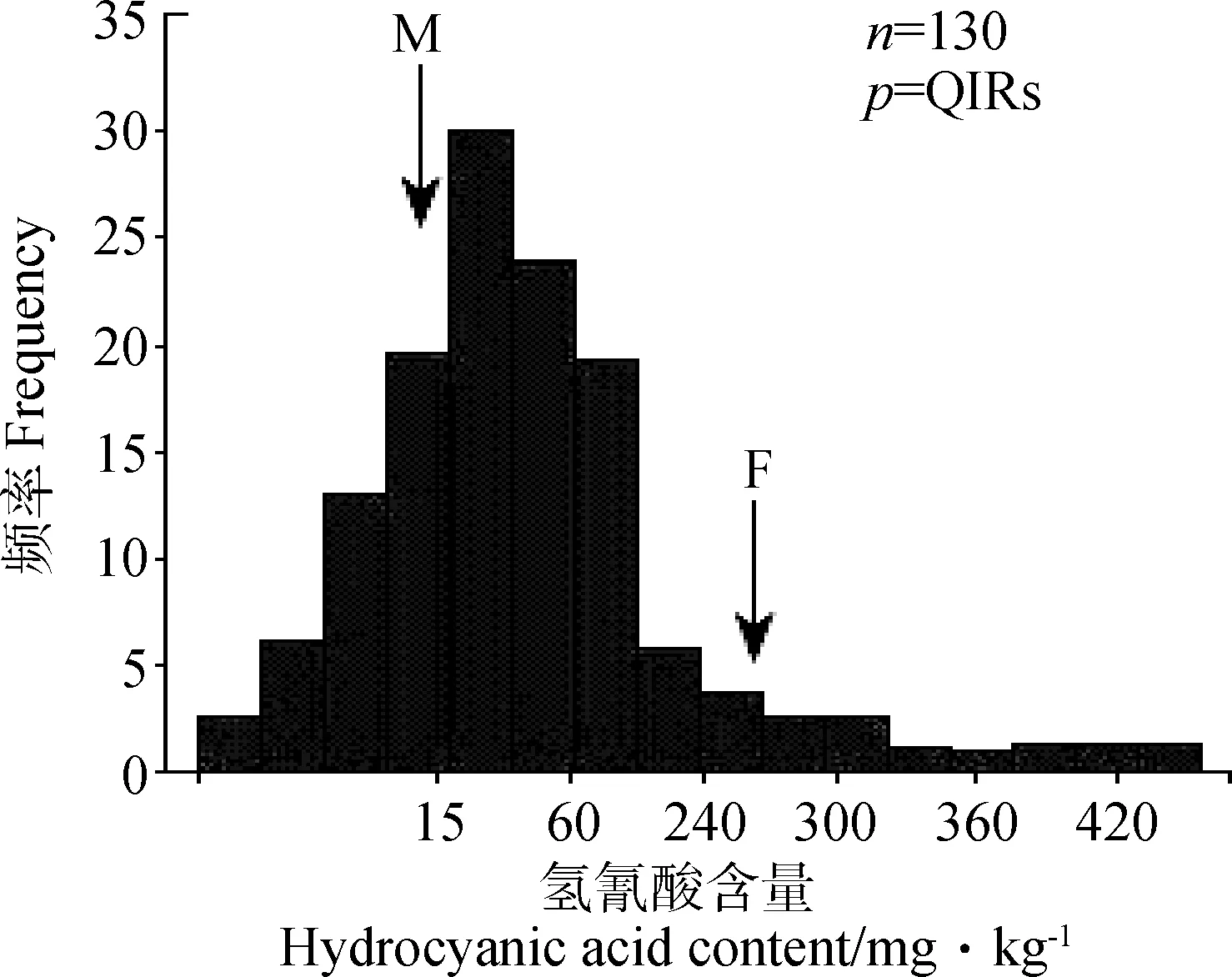
图3 高丹草F3代QIRs株氢氰酸含量测定值频率分布图Fig. 3 Frequency distribution of hydrocyanic acid contents in F3 generation quantitative trait locus isogenic recombinants of sorgum-sudangrass hybridF:♀散穗高粱;M:♂红壳苏丹草;n为群体大小;p为群体类型;箭头表示亲本氢氰酸含量值F:female parent scattered ear sorghum;M:male parent red hull sudangrass;n is the population size;p is population type. Arrows represent the hydrocyanic acid content values of each parent
2.4 QTL PA7-2在遗传图谱上标记区间的确定
利用新设计的12对SSR特异性引物对130个F3QIRs株精细定位群体及其亲本基因组DNA进行PCR扩增,共得到43个多态性SSR标记,加上PA7-2区域的5个标记构建了含有48个连锁标记的遗传连锁图谱,其全长为230.7 cM,各标记间平均距离为4.81 cM(图4)。利用该连锁图谱对130个重组单株的氢氰酸含量测定值进行QTL定位发现,QTLPA7-2位于标记Sobic.8 g1-600和XM00242-400之间,其低氢氰酸含量性状的遗传贡献率为48.2%(表5),表明PA7-2在F3群体中未丢失,可在物理图谱上进行精细定位。

表5 F3精细定位群体氢氰酸含量性状QTL定位Table 5 The QTL of hydrocyanic acid contents identified by QIRs population of F3 fine-mapping generation
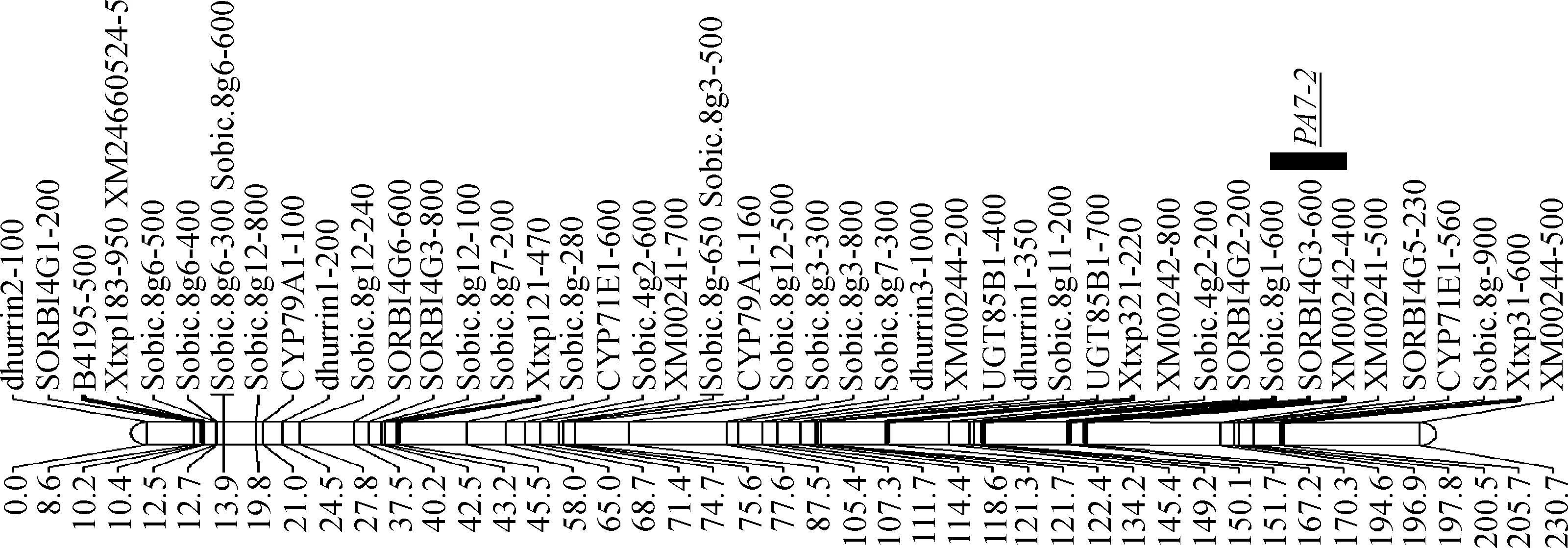
图4 QTL PA7-2的连锁标记图谱及定位区间的确定Fig.4 Linkage map and QTL mapping results of fine mapping population in sorghum-sudangrass hybrid在连锁群的左侧显示标记位置,右侧显示标记名称。黑色矩形代表检测到的QTL。The marker position was shown on the left side of the linkage group,and the marker name was shown on the right side. The black rectangle represents the QTL detected.
2.5 QTL PA7-2的精细定位
利用与QTLPA7-2紧密连锁的7个SSR标记建立了物理图谱(图5a),并与氢氰酸含量性状相关的对照组(低氰CK1、中氰CK2和高氰CK3的QIR株)进行比较,把从F3代130个QIRs株中选出的30个QIRs单株划分目标区域,按低氢氰酸含量和高氢氰酸含量性状的基因型分成12组(G1-G12,图5b)。进而用植株的平均氢氰酸含量数据与对照组比对确认,在标记Sobic.8 g1-600和XM00242-400之间为13个低氢氰酸含量重组株G1-G6。其中G1-G4共12个QIRs株的氢氰酸含量范围为4.06~10.11 mg·kg-1,显著小于隐性对照组CK1的氢氰酸含量,据此可将低氢氰酸含量PA7-2区域确定到Sobic.8 g1-600标记的下游,该区域只含1个SSR标记(SORBI4G3-600),表明该标记为低氢氰酸含量性状紧密连锁分子标记。而G7-G12的QIRs株的氢氰酸含量显著增高(107.51~478.57 mg·kg-1),逐渐接近或超过CK3的氢氰酸含量,为高氢氰酸含量区段,位于标记XM00242-400的下游。
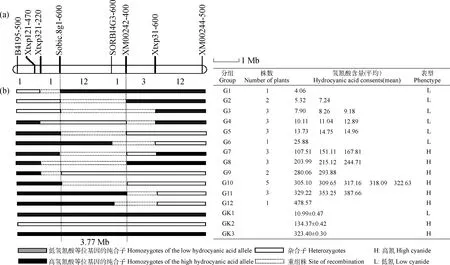
图5 QTL PA7-2精细定位Fig. 5 Fine-mapping of QTL PA7-2a:连锁群上PA7-2区域的物理图,相邻标记间重组株的数量在图a下方显示。b通过分析30个重组株的基因型和表型,将PA7-2基因座缩小为Sobic.8 g1-600和XM00242-400;通过分析13个低氢氰酸含量重组株的基因型和表型,将PA7-2基因座确定在Sobic.8 g1-600和XM00242-400标记之间的3.77 Mb区域;灰色矩形CK1为红壳苏丹草低氢氰酸等位基因纯合子,白色矩形CK2为杂合子,黑色矩形CK3为散穗高粱高氢氰酸等位基因纯合子,白色点状矩形为重组位点。在右边的表格中,表示分组、各组重组株的数量和基因型类别。每个CK的平均值±SD(随机选择的20个F3植株)a:A high-resolution physical map of the PA7-2 region on chromosome;The number of recombinants between adjacent markers are indicated below the map. b The PA7-2 locus was narrowed down to Sobic.8 g1-600 and XM00242-400 by analyzing the genotypes and phenotypes of the 30 recombinants;The PA7-2 locus was delimited to a 3.77 Mb region between the Sobic.8 g1-600 and XM00242-400 markers by analyzing the genotypes and phenotypes of the 13 QIRs of low cyanide;Gray rectangles CK1 indicate the homozygotes with the low cyanide allele of ‘Red hull sudangrass’,white rectangles CK2 represent the heterozygotes,black rectangles CK3 indicate the homozygotes with the high cyanide allele of ‘Scattered ear sorghum’,and white dotted rectangles represent the site of recombination. In the table on the right,group genotype category,the number of recombinants in each group;Mean values ± SD (n = 20 randomly chosen plants) are shown for each CK
经与高粱基因组比对,最终将低氢氰酸含量主效QTLPA7-2缩小在SSR标记Sobic.8 g1-600和XM00242-400之间,这2个标记正好处于高粱8号染色体区段的51.415 Mb~55.182 Mb(53 912 597-57 862 260 bp)之间,该区域为3.77 Mb。
3 讨论
氢氰酸是一种有毒的化合物,在植物中以生氰糖苷的形式存在,家畜采食过量会引起中毒现象[5-6],因此,培育低氰高丹草品种尤为必要。目前,对低氢氰酸含量性状的研究甚少,本研究选在氢氰酸含量上有极显著差异的亲本进行杂交、自交获得F2代分离群体,结合表型与分子标记基因型进行重组株筛选,从中获得低氰重组单株,为明确低氰性状遗传提供作图材料。
QTL定位可将目标性状基因定位在遗传图谱上的相对位置,而精细定位是将目标性状基因精确到某一染色体上的实际物理位置,是对植物数量性状目标基因图位克隆、候选基因挖掘及功能解析的主要途径[27]。在作物上,有不少学者进行了重要数量性状的QTL精细定位研究,如代资举等[28]利用近等基因系将玉米雄穗分枝数qTBN5精细定位至13.2 Mb的物理区间,金迪等[29]利用小麦F2群体将抑制芒长的QTL B2精细定位至4.84 Mb(471.28~476.12 Mb)物理区间。本试验以高丹草F3代QIRs群体首次将高丹草低氢氰酸含量主效QTLPA7-2精细定位至3.77 Mb(51.415 Mb~55.182 Mb)物理区间,这说明精细定位群体的选择对定位结果有一定影响,即不同的作物种类其定位群体类型可以不同。另外,本试验为了降低或消除遗传背景对精细定位结果的影响,直接利用QTLPA7-2的SSR侧翼标记对高丹草F2和F3群体进行筛选,选取了QTLPA7-2区段为杂合的单株构成QIRs,而基因组的其余部分均与亲本相同,可对复杂数量性状进行遗传分析,屏蔽了非重组单株对PA7-2的影响。为了确保精细定位的准确性,在F3精细定位群体中只选取了具有极端表型的单株进行精细定位,如在红壳苏丹草杂合类型的单株中,仅挑选氢氰酸含量小于红壳苏丹草单株的植株(≤15 mg·kg-1);在散穗高粱杂合类型的单株中,仅挑选氢氰酸含量高于散穗高粱的植株,不仅可以减小遗传背景的影响,而且还具有省时、减少工作量的优点。
用BSA混池法筛选与作物目标性状相关的分子标记,能够克服没有创建近等基因系NILs(Near isogenic lines)的限制[30],但该方法需要考虑建池单株数量和分子标记的图谱密度。研究表明,若两个亲本的遗传背景较一致时,建池单株数至少需要有2株;若亲本的遗传背景差异较大,则需要增加单株数量;而且建池单株数量还与供试材料的基因组大小有关,若基因组太大(如小麦17 Gb),建池的单株数不宜太多,否则因多态性过高会影响试验结果;此外,需根据所选分子标记图谱密度的大小来确定建池株数,密度高(标记间密度<5 cM)可以减少株数,密度低(标记间密度>10 cM)可增加株数,但一般不超过20株[31-32]。本试验所用亲本材料散穗高粱和红壳苏丹草均为二倍体高粱属的两个不同种,遗传背景具一定的相似性,现已查明高粱基因组为750 Mb,基因组相对较小[33]。据此,我们选用F3代高氰和低氰的QIRs群体各10个单株建立低氰DNA基因池和高氰DNA基因池,筛选出12对特异性SSR引物,对高丹草杂种F3代130个QIRs株的基因组DNA进行PCR扩增得到SSR多态性标记48个,建立了密度较高的遗传连锁图谱,并精细定位了主效QTLPA7-2,因此,BSA法是快速寻找与低氢氰酸含量性状相关标记的有效方法。
4 结论
以高丹草F2和F3群体QIRs为材料,利用BSA-SSR技术首次将低氢氰酸含量性状主效QTLPA7-2精细定位至3.77 Mb的物理区间,并确定了与其紧密连锁的SSR标记SORBI4G3-600,为高丹草低氢氰酸含量性状PA7-2候选基因挖掘及分子标记辅助育种等研究奠定了基础。
