环境DNA技术在水生态领域应用研究进展
2021-11-09赵彦伟陈家琪董丽麻晓梅白洁田凯
赵彦伟,陈家琪,董丽,麻晓梅,白洁,田凯
(北京师范大学环境学院,水环境模拟国家重点实验室,北京 100875)
对生物资源与生态系统进行全面系统的调查评估,是生态环境研究的重点,也是保护全球生物多样性的重要举措。基于形态学鉴定的传统生物监测方法对样本的完整性和鉴定人员的技术要求高,尤其对于隐蔽性强、生活习性难以掌握、形态变化大的物种,鉴定难度较大,这些均为生物监测和物种保护工作的开展带来了困难。因此,需要一种快速高效、侵入性低且受物种自身影响小的技术来解决当前的生物监测问题。
环境DNA(Environmental DNA,eDNA)作为当前重要的技术手段之一,为环境生态的相关研究提供了新思路与新方法,其推动着物种鉴定工作由形态学识别向分子生物学过渡。任何物种都有自己特有的DNA 序列,皮肤、粪便、唾液、配子、分泌物等都会携带着自身的DNA 片段,其脱落到环境中后,即可通过一系列分子生物学操作流程检测到环境中物种的存在信息[1],这即是eDNA 技术的基本原理。eDNA 技术直接从环境样品中提取DNA,无需对任何物种进行分离,可将生物监测对环境带来的影响降到最低[2−3]。涉及eDNA的研究最早开始于20世纪80年代,经过近年的发展,该技术的研究逐步深入,采集范围逐步扩大,尤其在水生态领域得到了广泛的应用。
相较于其他的生物鉴别技术,eDNA 是一项新技术领域,因此受到较多学者的关注。马鸿娟等[4]、陈炼等[5]、张辉等[6]对eDNA 技术进行了较为全面的综述,对该技术的发展历程、通用操作流程及其在生物多样性研究中的应用进行了系统的介绍。本文在这些综述基础上,吸收了近年eDNA 技术在水生态领域的最新研究成果,强调了水生态研究中采样环境、目标物种变化情况下不同技术环节的特点与比较,重点总结了eDNA 技术在水生态领域的应用,分析了该技术应用的局限性,并根据水生态系统特点提出了相应改进措施,对eDNA技术未来发展趋势进行了展望。
1 eDNA技术的操作方法
eDNA 技术主要包括样品采集、DNA 提取、PCR扩增及测序、生物信息学分析。在不同的研究中,采样环境、目标物种等重要因素会有差异,从而使不同的技术环节方法有所变化。
在水环境应用中,为减少外界环境因子对水体中遗留的生物DNA 的影响,样品采集时不宜对水体造成过大的扰动,通常采集研究区表层水即可[7]。样品采集过程中要格外注意外来污染或交叉污染,必须严格佩戴一次性手套并及时更换,以免给最后的物种识别过程带来干扰。在采集平行样本的同时设计空白样本[8],如将一瓶加盖的蒸馏水保存在冰上,以检查现场工作期间的污染。样品采集后要立刻冷藏防止水体中生物DNA 降解,并在最短的时间内进行初步处理。
样品采集后,采用滤膜法和沉淀法均可以达到浓缩样品提取DNA 的目的,且两种方法各有优势,沉淀法所需水样明显少于滤膜法,但滤膜法可获得更高浓度的DNA,当前滤膜法中的抽滤方式应用较多。DNA 的提取方法包括物理法、化学法、生物法等,也有研究选择相应的DNA 试剂盒提取水环境中的DNA。有研究表明,应根据目标物种选取合适的DNA 提取方法,以保证提取到的DNA 浓度最佳,如提取真核生物DNA 时采用抽滤法与DNeasy Blood &Tissue Kit试剂盒相结合的方法检出率更高[9]。
PCR 扩增首先需要进行引物的设计。引物设计需依不同的研究目标而定,如果是对研究区进行全面的生物多样性评价,则要选择通用性引物,以保证此引物扩增到的物种序列最全;如果是对特定物种进行监测评估,则需根据目标物种特点设计特异性引物。之后进行PCR 扩增,扩增后产物经琼脂糖凝胶电泳检测,若检测结果符合要求,即可进行上机测序。
测序得到的原始序列需经过低质量过滤、序列拼接、去除嵌合体等一系列质控过程,得到的高质量有效序列将按照一定的序列相似性进行OTU聚类(OTU相当于传统生物监测中的“种”),在每个OTU 中选取代表序列(通常为最长序列)与相应的参考数据库比对进行物种的注释分析,不同数据库的鉴定范围通常不同(表1),需根据目标物种选择合适的数据库。以OTU聚类和物种注释结果为基础,可得到区域内的物种组成与分布、群落间结构差异等信息,还可进一步挖掘物种之间的进化关系,探究不同环境因子对物种群落结构的影响等。

表1 常见基因数据库及鉴定范围Table 1 Common genetic databases and appraisal scope
2 eDNA技术在水生态研究中的应用
2.1 生物多样性监测
2.1.1 物种的识别与监测
生物多样性是衡量某一区域物种丰富程度的重要指标,是环境生态学领域的重要基础性工作,生物多样性监测的难点在于快速准确地识别物种,eDNA技术因可以解决这一问题,而在浮游动物、藻类、鱼类等生物类群的监测中得到了应用(表2)。其中,鱼类是 eDNA 监测的重要类群之一,THOMSEN 等[18]发现eDNA 技术可监测到形态学监测方法识别不到的鱼类;赵梦迪[19]、SIGSGAARD 等[20]、杨海乐等[21]将 eDNA技术与形态学监测方法对鱼类的监测结果进行了比较,证明了eDNA 技术较高的覆盖度和用于渔业生物多样性监测的可行性。利用eDNA 技术,宋伦等[22]、LIU 等[23]、岳一鸿等[24]对藻类进行了监测,唐晟凯等[25]、胡愈炘等[26]对浮游生物进行了监测,这些研究证实了利用eDNA 技术对水生态系统中的物种进行识别与监测,全面评估区域物种资源状况的潜力。
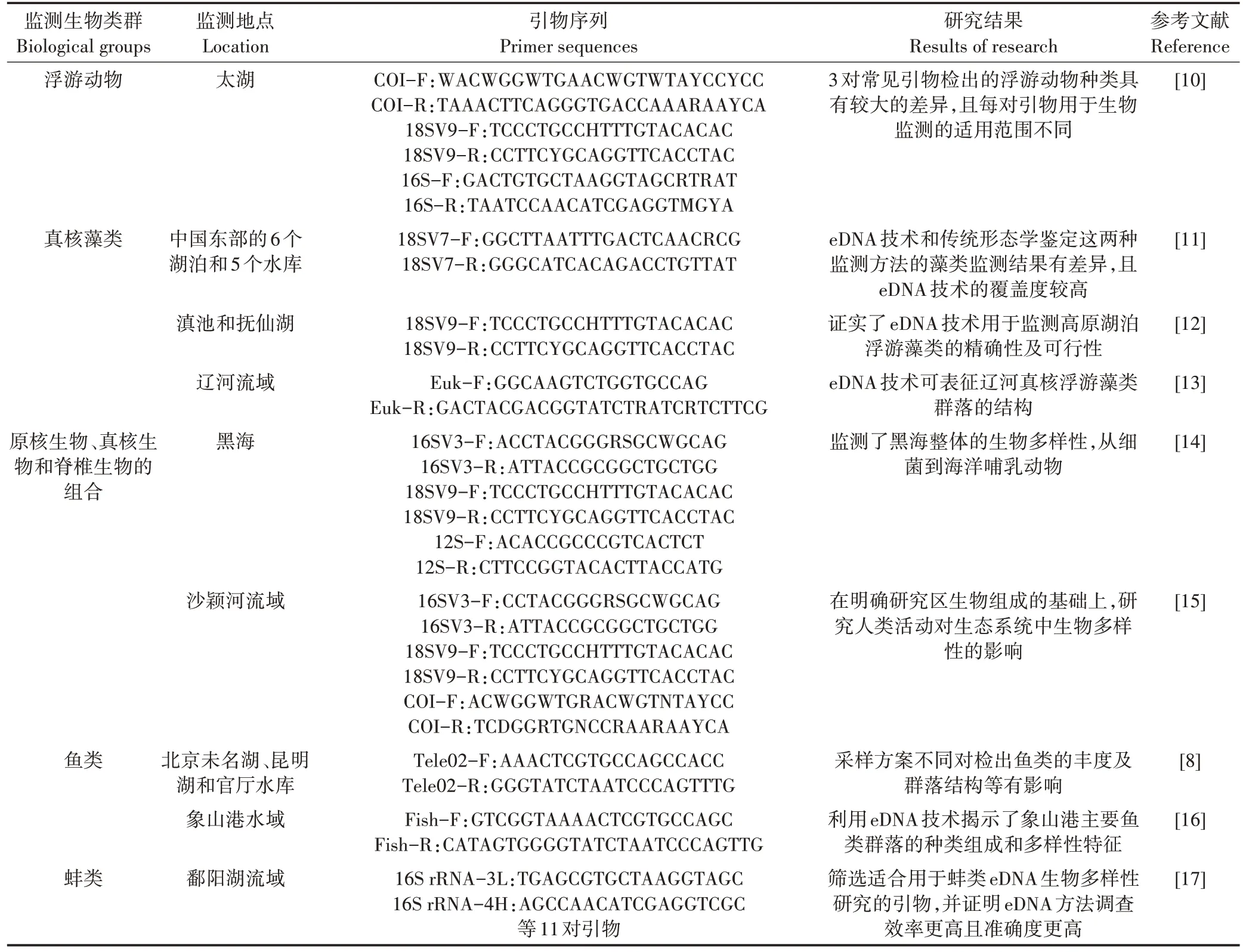
表2 eDNA技术用于水生态监测的应用实例Table 2 Application examples of eDNAinaquatic ecosystem monitoring
2.1.2 濒危物种和入侵物种监测
濒危物种保护和外来入侵物种防治是维持生态系统平衡、保护全球生物多样性的重要举措。一般情况下,入侵物种早期阶段和濒危物种的物种数量低,利用传统的形态学监测方法难以及时发现,这增加了珍稀物种灭绝和外来物种大规模繁殖的风险,影响到生态系统结构的稳定性。尽早掌握濒危物种和入侵物种早期阶段的分布范围、演变趋势等基本信息是监测工作的关键[27−29],已有研究利用eDNA 技术完成了对典型入侵物种克氏原螯虾的监测[30],还有研究利用eDNA 技术检出了全球第四只斑鳖。利用eDNA 技术还实现了对美国牛蛙[31]、亚洲鲤鱼[32]、斑马贻贝[33]、蓝腮太阳鱼[34]等入侵物种,江豚[35]、欧白鲑[36]、野生娃娃鱼[37]、澳洲麦氏鲈[38]、长鳍金枪鱼[39]等濒危物种的监测,这些证实了eDNA 技术在入侵物种的及时监测预警、珍稀物种的追踪研究等方面的应用价值与潜力。
2.2 生物量评估
已有研究表明,某一物种释放的eDNA 浓度和其生物量呈正相关关系[40],此结论为生物资源的综合评估提供了一个新的思路。孙晶莹等[41]选取太湖流域常见的5 种浮游动物为研究对象,利用eDNA 技术对其开展了生物量监测研究,发现在选择合适引物的基础上,eDNA 技术可实现对浮游动物的半定量检测。李苗等[42−43]在建立优化中国对虾(Fenneropenaeus chinensis)eDNA 操作流程的基础上,进一步对eDNA在水体中的存留时间进行了研究,这些研究对中国对虾进行定量的生物量评估至关重要。秦传新等[44]分别综述了实验室可控环境和野外环境条件下利用eDNA 技术进行生物量评估的研究进展,并提出要更多地关注影响eDNA 释放、迁移、降解的因素,进而为生物量评估提供重要的依据。尽管目前已有众多针对鱼类、两栖动物等物种生物量估算的研究,但由于不同物种释放eDNA 的浓度和速度不尽相同,想要实现跨类群的生物量评估还需要eDNA 技术更深入的发展。
2.3 生物食性分析
以往的食性分析多基于形态学鉴定、同位素追踪等方法[45],对濒危性、夜行性以及生活习性难以掌握的生物测定难度较大。近年来eDNA 技术被广泛应用于动物食性的分析,其最大的优势在于能进行快速精准的测定,有利于精确解析动物食物网的关系和生态系统的功能。周天成等[46]利用基因测序技术分析了一种海洋贝类——塔形马蹄螺(Tectus pyramis)的食物组成并推测其属于沉积物碎屑食性生物,测序结果共获得41 个OTU,分属11 个门类,其中有孔虫、真菌、后生动物是其消化道中存在的重要类群。除此之外,eDNA 技术还被用于多种海洋生物的食性研究,如刺参、龙虾、方星格虫等[47]。
食性分析是生态学研究的重点,也是了解水生生物与环境关系及捕食者与被捕者关系的基础,是对已有食物链研究的重要补充[48],对研究区内食物网结构的重建和生态调控具有重要意义。但世界上绝大多数生物的食性分析还处于较为粗略的定性描述阶段,eDNA 技术将会成为人类更全面明确生物食性、研究食物链/网结构的有利工具。
2.4 生态风险评估
生态风险评估是生态毒理学研究中的重要且基础内容,传统生态毒理学方法强调对化学物质暴露过程的评估,忽视了对群落结构及生态系统基本功能的影响,评估结果具有一定的应用局限性。ZHANG[49]认为eDNA 技术开拓了生态毒理学研究的新时代,该技术既可解决当前生态风险评估存在的问题,又可扩展未来生态毒理学研究的领域和方法。YANG 等[50]、LI 等[51]利用eDNA 技术监测了水体中的原核生物、真核生物等生物群落结构,评估了其与pH、总氮、总磷等环境因子的关系;XIE 等[52]利用eDNA 技术评估了漏油对沿海沉积物中细菌、原生动物、后生动物群落的影响;YANG 等[53]探究了铜的不同浓度对湿地沉积物中原核生物和真核生物多样性、群落结构以及生物间关系的影响。这些研究推动着eDNA 技术在生态系统监测、污染诊断和生态修复等方面更深入的应用。
2.5 生物流行病的预测与预防
疾病生态学是用生态学观点研究环境、宿主和病原体之间关系及其发展规律的新学科,其能明确疾病进化和生态之间的相互作用,预测疾病爆发的方式和地点,指导相关部门的管理决策,有效预防生物流行病的传播。当前,eDNA 技术已用于研究水体中的寄生虫疾病[54]。CARRARO 等[55−56]、GOMES 等[57]通过研究证实了eDNA 技术用于预测预防水体寄生虫爆发的潜力。其中CARRARO 等[55]以瑞士Wigger 分水岭为研究区,以对本地鲑鱼物种有极大影响的增生性疾病(Proliferative kidney disease,PKD)为研究对象,绘制了其病原体F.sultane的DNA 浓度分布图,并建立了研究水传播疾病的流行病学和疾病动力学模型;同时利用eDNA 技术对整个河流上游网络中引发鲑鱼疾病的固着无脊椎动物F.sultane及其寄生虫T.bryo⁃salmonae的分布和丰度进行了估计,通过这两种生物的生物量变化来预测PKD在研究区内的传染状况[56]。这项研究开启了利用eDNA 技术估计目标物种在研究区内分布情况及有效预测预防流行病传播的新模式,而传统的调查方法迄今还无法做到这一点。
3 eDNA技术应用的局限性与改进
和传统生物监测方法相比,eDNA 技术具有非入侵性、高灵敏度、不受环境条件限制等优势,但也存在一系列有待完善的问题,在实际应用过程中存在局限,还不能完全取代传统的调查方法,这些问题制约着eDNA技术的应用。
3.1 缺乏标准化的操作流程
选择效果最佳、最经济便捷的操作流程与方案一直是科研人员关注的焦点,随着eDNA 技术的不断发展及研究团队的壮大,eDNA 技术实验方案也在不断增加。已有研究表明,不同的DNA 捕获与提取方案[9,58]会对最终的监测结果产生影响。但因受采样环境、采样范围、监测目标物种等多重因素的限制,当前缺乏一套通用的eDNA 技术标准化操作流程。研究者在研究过程中采取的操作方法不尽相同,导致研究结果的对比性差,存在一些争议[7,28]。今后应加强eDNA 技术在水生态研究中应用的普适性研究,使eDNA 技术能更广泛地应用于水生生态监测中。需要综合考虑不同类型水生生物的生活特征,对eDNA 技术的整个操作流程进行优化,针对不同目标物种与研究目的,明确与细化采样、提取、PCR扩增、分析等的操作指南与技术规范,从而建立一个规范统一的操作方案与流程,以推动eDNA技术更广泛的应用与发展。
3.2 监测精度及准确性有待提高
eDNA 在水体中释放的浓度及其分布受到水动力条件[59]、水体温度及光照条件[60]、季节变化、生物活动等多种因素的影响,还与人类活动干扰强度密切相关,且eDNA 会随着时间和各环境因子的改变而发生降解。当前研究大多未考虑外界环境条件变化对DNA 分布的影响[5],使得监测结果与实际情况存在一定的误差。近年来国内外学者针对这一问题进行了有效探索,主要集中在季节变化对特定生物eDNA 的影响[35],水体温度及营养条件与eDNA 降解之间的关系[61]等方面,但这些研究具有个案特征,并不能作为普遍的规律指导eDNA 的广泛应用,eDNA 时空尺度研究的精确度还有待提升。为此,需要研究者们在模拟并建立外界干扰和物种分布之间对应关系的基础上[7],积极探究影响eDNA 产生和降解速率的环境因素,进而更好地为生物量评估和生物流行病的预测预防等相关工作的开展提供理论基础。
3.3 假阳性和假阴性错误无法避免
假阳性和假阴性错误是基于研究区目标物种的DNA 是否被检出以及目标物种是否真实存在而产生的,主要受技术敏感性、引物偏倚性等因素的影响[35]。在实际操作中,假阳性和假阴性错误的出现可能会带来较大的损失,尤其是在濒危物种和入侵物种的监测中,物种的误判对生态系统的影响是巨大甚至是毁灭性的。为了尽可能减少假阳性和假阴性错误的出现,需提高引物的通用性,严格控制实验操作过程的准确性,设置阳性和阴性对照以保证实验结果的可靠性,对实验中的外来污染和交叉污染进行实时监控,还可以结合其他鉴定方法进行对比分析,校核实验结果,从而提高eDNA技术用于生物监测的有效性和可靠性。
3.4 物种参考数据库需要完善
基因测序序列和物种参考数据库的比对是保障eDNA 技术应用准确性的关键环节。主要在于,物种参考数据库的完整性和质量直接影响着特异性DNA条形码的可用性,进而影响生物多样性的监测结果,更决定着运用eDNA 技术进行物种鉴定的可靠性[62−63]。数据库的不完整可能会导致大量的基因序列无法被注释,造成海量遗传信息的遗失。近年来,NCBI、SILVA、Greengenes、PR2 等一些常用的公共数据库的数据在不断更新和完善,也有研究者在积极建立研究区的本土生物基因库,如:太湖浮游生物基因库[64−65]、中国的底栖动物条形码数据库[66]等,但数据库的容量依旧不足。为保证结果有效性和数据可用性,同时提高基因序列注释到种水平的概率,可从两个方面改进:一是建立本土基因库,加强全球资源和成果共享;二是完善和优选现有数据库,建立一个生物类群更多、基因序列更全的公共数据库。
3.5 eDNA丰度和生物体数量的关系需要进一步明确
eDNA 技术只能从序列数量推断OTU 的相对丰度,虽然可利用实时荧光定量PCR 等方法对特定DNA 进行绝对定量,但对于还原形态学方法中的物种绝对丰度或生物量的预期,仍然相差甚远。目前多采用多引物法或仅对单一类群的特异基因测序结果进行分析,以弥补eDNA 监测技术还原度低的问题。近年来,物种的基因拷贝数、基因组大小、生物体积大小等因素逐渐被纳入到矫正因子的参数选择中[67],也有研究者在探索不依赖于形态学分类注释的描述群落结构和多样性的eDNA 监测和数据分析方法[68−69],这进一步丰富了eDNA 与生物体对应关系的研究体系。
4 总结与展望
在水生态监测研究中,传统的形态学生物监测方法受耗时长、难度大、成本高等因素的限制,无法实现对水生生物的精准监测和识别。eDNA 技术目前虽然也存在着流程不规范统一、数据库不完善等问题,但其具有的省时高效、灵敏度高、环境友好等优势,正推动着该技术成为生物多样性监测和评估的一个强有力工具。因此,eDNA 技术和传统监测方法的互相补充才能为全球大规模、高效率的生物监测提供技术支撑。
eDNA 技术是分子生态学发展的重要产物,它将极大地提升人类从生态系统中获取数据的能力,未来关于eDNA 的进一步发展将涉及技术和应用两个层面。技术方面,在解决技术流程、监测精度、水生生物种数据库等问题的同时,应加强新兴测序技术及平台的研发,将基因测序结果和监督式机器学习进行有效结合,实现水生态监测的智能化,不断提高监测效率,使更大批量的测序成为可能,还应将eDNA 技术引入水生生物的食物网/链研究中,丰富eDNA研究的技术体系。应用方面,一是在稳步推进eDNA 技术应用于水域生物多样性与水生态监测等常规研究的同时,还应拓展eDNA 技术在水生态系统的研究领域,加强该技术在水域食物链/网重构、流域营养盐控制、流域生态调控与管理中的应用;二是要积极开展试点性研究,尝试将eDNA 技术引入国家水环境与水生态监测体系,编制并完善与eDNA 技术应用及实施相关技术的指南、导则与方法,进而实现eDNA 技术在水生态环境监测领域更广泛的应用。
