低蛋白饲粮对山羊肝脏转录组的影响
2021-11-06朱雯汤莹莹孙昕旸周明张子军陈兴勇
朱雯 汤莹莹 孙昕旸 周明 张子军 陈兴勇
(安徽农业大学动物科技学院,合肥 230036)
近年来,畜牧业的快速发展增加了畜禽饲料原料的需求量。与之相比,我国粮食产量涨幅相对较低。饲料资源尤其是优质蛋白质饲料较为紧缺,如大豆每年进口8.0×107t,中美贸易战和新发展格局下,蛋白资源紧缺威胁到饲料粮安全,非常规蛋白资源开发、低蛋白平衡日粮等成为问题解决方向[1]。然而,大部分研究表明,低蛋白饲粮会影响反刍动物生产性能[2-3]。肝脏是调节糖脂代谢的重要器官,瘤胃发酵以及饲粮来源的各种营养素均通过门静脉进入肝脏[4]。由此可见,肝脏功能对反刍动物生产性能的影响起着至关重要的作用。目前,低蛋白日粮在肝脏中代谢调控机制仍不明确,现代组学技术发展迅速,可有效阐明这一问题。
转录水平的调控是动物生命活动调节的重要调控形式,转录组测序是了解动物基因总体表达情况的强大工具。目前高通量转录组测序技术已经应用在营养和环境对反刍动物生长性能调控的影响机制研究中[5-6]。一大批涉及营养物质吸收及信号转导的基因得到鉴定,对于研究动物生长性能调控的分子机制起到很大的推动作用。
团队前期研究结果发现,饲粮粗蛋白水平较NRC推荐水平低2.80个百分点时显著降低安徽白山羊的生长与生产性能[7]。本研究采用转录组测序(RNA-Seq)技术,分析低蛋白饲粮影响肝脏基因的表达特性,重点挖掘营养素代谢相关基因,以期揭示低蛋白饲粮影响动物生产性能的分子机制,为低蛋白饲粮的营养调控提供理论依据。
1 材料与方法
1.1 材料
安徽白山羊购自六安绿洁畜牧有限公司,RNA提取试剂盒、RNA纯化试剂盒均购自 QIAGEN公司。
1.2 方法
1.2.1 试验动物、日粮和样品采集 试验前对羊只进行检疫,预试期进行驱虫、防疫、定期消毒,保证羊舍干净。试验羊单栏饲喂,每天06:30和18:30各饲喂1次,确保槽内约10%的剩料。试验期内所有羊只自由饮水和采食。试验设计和动物饲养管理同本课题组前期研究报道[7]。简述如下:本试验采用单因子随机分组设计,选取24只体重接近(16.0±1.35)kg的健康安徽白山羊公羊(3-4月龄),随机分为2组,分别饲喂正常蛋白质水平(14.8%)饲粮(对照组,CK组)和80%蛋白水平(12.0%)饲粮(低蛋白组,LCP组)。基础饲粮参照NRC(2007)[8],体重20 kg、日增重150 g/d的公羊营养需要量自行配制。试验饲粮为全混合颗粒饲料,直径为2.5 mm,长度为8 mm,试验饲粮组成及营养水平见表1。试验结束后,每组随机抽取3只羊屠宰,宰前禁食24 h,禁水2 h。屠宰后立即取肝脏组织放入无酶冻存管,保存于液氮备用。本实验通过安徽农业大学科学伦理委员会批准(批准号SYDWP20190600601)。

表1 试验饲粮组成及营养水平(干物质基础)Table 1 Ingredients and chemical composition of the experimental diets(dry matter basis)
1.2.2 RNA提取及转录组测序 通过Trizol法进行山羊肝脏组织总RNA提取,分别使用NanoPhotometer spectrophotometer和Qubit 2.0检 测提 取RNA的纯度和浓度,使用Aglient2100检测RNA的完整性。所有样品RNA提取并检测合格后,用干冰保存并送至广州基迪奥生物公司,应用Illumina Novaseq6000高通量测序平台进行测序分析。
1.2.3 测序数据分析 对测序所获得的原始数据,按照基迪奥生物公司的数据处理方法,进行原始数据的过滤,获得高质量的clean reads。然后通过HISAT2软件进行序列对比,获得山羊肝脏样本特异序列信息,并进行基因组定位分析。
1.2.4 差异表达基因(DEGs)的筛选以及功能富集分析 以FDR<0.05 且 |log2FC|>1为阈值进行差异表达基因的筛选,其中Fold Changes(FC)代表不同处理之间基因表达的差异。结合GO(gene ontology)和KEGG(kyoto encyclopedia of genes and genomes)数据库对筛选获得的差异表达基因进行功能注释、GO及KEGG分析,P<0.05 的通路为相关基因富集到的差异生物学通路。
1.2.5 基因的蛋白质互作网络分析 采用STRING数据库的蛋白质互作信息构建低蛋白饲粮调控相关差异表达基因的蛋白质互作网络。
1.2.6 差异表达基因的验证 为了对转录组测序的结果进行验证,随机挑选5个差异表达基因,利用Primer5.0软件设计扩增引物(表2),采用qRT-PCR的方法,对其表达水平进行验证。将各处理的样品总 RNA 反转录合成 cDNA,并以此为模板,以 β-actin为内参,进行 qRT-PCR反应。qRT-PCR的反应体系、反应程序以及基因相对表达量的计算均参照Li等[5]方法进行。

表2 qRT-PCR反应所用引物Table 2 Primers used for qRT-PCR
2 结果
2.1 测序质量分析
采用高通量测序平台,对低蛋白饲粮处理及对照组的肝脏样品进行了RNA测序。结果显示,共获得46 719 458-62 169 938条reads,测序数据去除接头以及低质量数据后分别获得7 007 918 700-9 325 490 700个高质量数据(clean reads),所有样品过滤后高质量序列数占原始下机序列的比例均大于95%,表明6个样品构建的文库质量较好;同时Q30的数值均大于90%,说明本次测序的质量非常可靠(表3)。
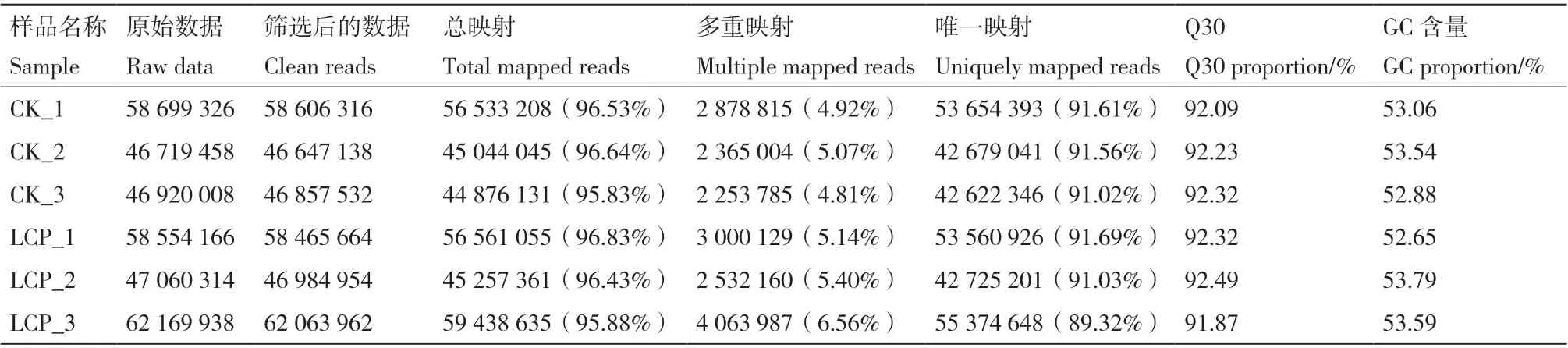
表3 各样品测序质量Table 3 Sequencing data quality of each sample
进一步对样品重复间数据的相关性分析,结果(图1)表明,重复之间的皮尔森相关系数均大于0.8,最高则达到了0.965 6,说明数据之间的重现性较好,可用于下一步的分析。
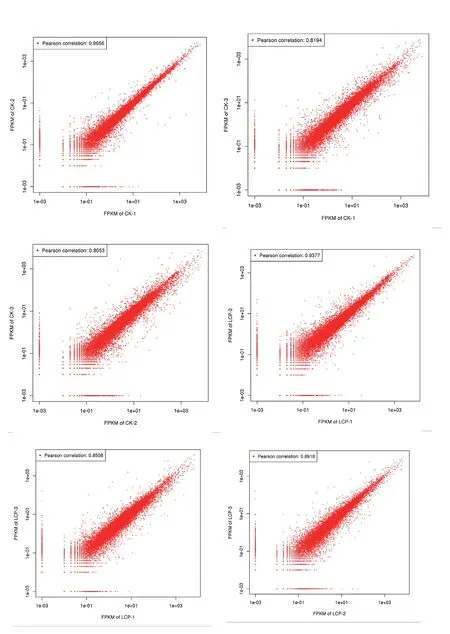
图1 样品重复之间的相关性分析Fig.1 Pearson correlation analysis between the repeated samples
2.2 差异表达基因统计分析
将低蛋白饲粮组和对照组样品的转录组数据进行对比分析,以FDR < 0.05 以及|log2FC| > 1为筛选条件,筛选差异表达基因。结果表明,低蛋白饲粮处理后,差异表达基因的数量达到了62个,其中,上调49个,下调13个。上调基因包含与蛋白质水解相关基因丝氨酸蛋白酶35(PRSS33)和PCOLCE2;能量代谢相关基因重组人碳酸酐酶14(CA14)和MDF1;脂肪酸代谢相关基因人源全长重组蛋白(ACSBG1)、脂肪酸合成酶(FASN)、酰基辅酶A硫酯酶(acyl-coenzyme A thioesterase 4)和LOC102184912。下调基因包含与糖代谢相关基因丙酮酸羧化酶(PC)和LOC108635023;胰岛素代谢相关基因胰岛素受体底物(IRS2)。
2.3 qRT-PCR验证
为了验证RNA测序结果的可靠性,随机选取了PC、FASN、促生长因子(IGF1)、IRS2以及CYP4A18五个基因进行qRT-PCR扩增,并与其测序结果进行对比(图2),从图2可以看出,所选择的5个基因在两种方法中表达趋势一致,说明本研究RNA-Seq测序结果较为可靠。

图2 RNA-Seq数据(A)与qRT-PCR(B)基因表达水平的比较Fig.2 Comparison and verification of the quantitative results for selected genes from the RNA-Seq(A)and qRT-PCR analyses(B)
2.4 差异表达基因的GO功能分析
为了进一步了解差异表达基因的功能,通过GO数据库对筛选到的差异表达基因进行基因功能富集分类,包括细胞组分(cellular component,CC)、生物过程(biological process,BP)以及分子功能(molecular function,MF)3个方面(图3)。在BP过程中,差异表达基因所富集的GO的数目为21;MF及CC中分别富集到8和13个GO条目。在BP功能中,两组间的差异表达基因在细胞过程(cellular process)和单机体进程(single-organism process)富集到的差异基因数目最多。在CC组分中,细胞器(organelle)和细胞结构(cell part)中所占比例最大。在MF功能中,差异基因在结合功能(binding)中所占的比例最高,催化活性(catalytic activity)次之。
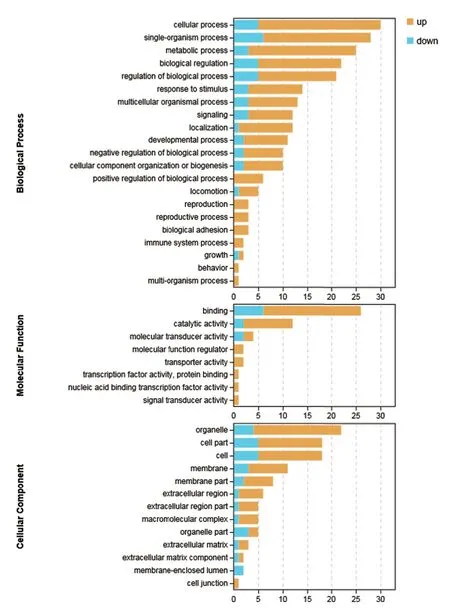
图3 差异表达基因的GO分析Fig.3 GO analysis of differentially expressed gene
在分子功能方面,上调的差异表达基因富集到3个GO条目(表4),其中,结合(binding)基因数目最多(20,40.8%),其次为催化活性(catalytic activity)基因(10,20.4%);在生物过程方面,上调的差异表达基因富集到13个GO条目(表5),其中细胞进程(cellular process)基因数目最多(25,51.0%),其次为单体进程(single-organism process)基因(22,44.9%)和催化进程基因(22,44.9%)。

表4 上调差异表达基因的GO功能注释分析(分子功能)Table 4 GO functional annotation of up-regulated expressed genes(molecular function)

表5 上调差异表达基因的 GO 功能注释分析(生物过程)Table 5 GO functional annotation of up-regulated expressed genes(biological process)
在分子功能方面,下调的差异表达基因富集到3个GO条目(表6),其中,结合(binding)基因数目最多(6,46.2%),其次为催化活性(catalytic activity)基因(2,15.4%);在生物过程方面,下调的差异表达基因富集到13个GO条目(表7),其中单机体进程(single-organism process)基因数目最多(6,46.2%),其次为生物调节(biological regulation)基因(5,38.5%)和细胞进程(cellular process)基因(5,38.5%)。

表6 下调差异表达基因的GO功能注释分析(分子功能)Table 6 GO functional annotation of down-regulated expressed genes(molecular function)

表7 下调差异表达基因的 GO 功能注释分析(生物过程)Table 7 GO functional annotation of down-regulated expressed genes(biological process)
2.5 差异基因KEGG通路注释
通过对差异表达基因进行KEGG通路分析(P≤ 0.05),共获得23条差异信号通路(图4),文中列举富集后最显著的前5个通路以做示例说明(表8),包括:富集因子最高的为味觉传导(taste transduction),脂肪酸合成(fatty acid biosynthesis),胰岛素信号通路(insulin signaling pathway),视黄醇代谢(retinol metabolism)和癌症转录失调(transcriptional misregulation in cancers)等。部分差异基因富集到与营养素代谢相关的脂肪酸合成以及胰岛素信号通路中。如上调基因acyl-coenzyme A thioesterase 4和FASN富集到脂肪酸合成通路;上调基因ACSBG1和细胞色素(CYP4A1)富集到PPAR信号转导通路;上调基因FASN和蛋白磷酸酶1调节亚基3B(PPP1R3B)以及下调基因IRS2富集到胰岛素信号通路。
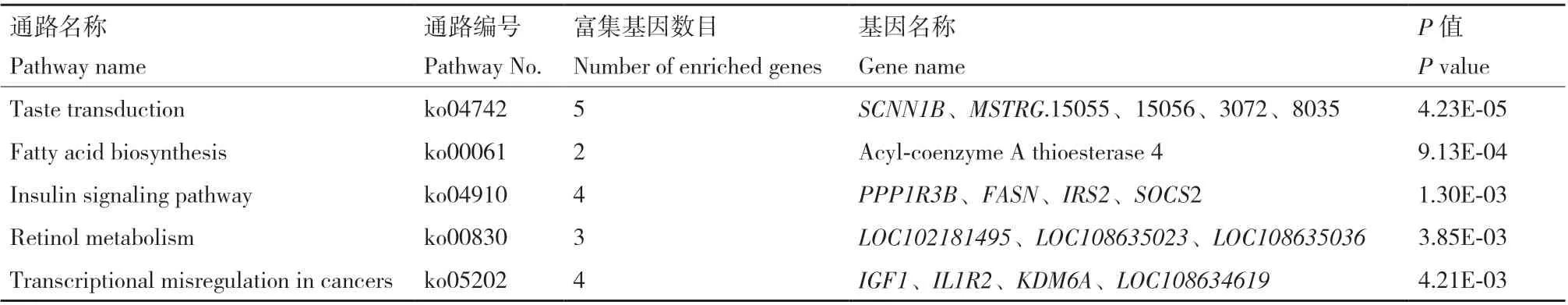
表8 低蛋白日粮饲喂条件下山羊肝脏组织富集显著性差异的前5条通路Table 8 Top 5 pathways enriched from the differential expressed liver genes of low crude protein fed goat
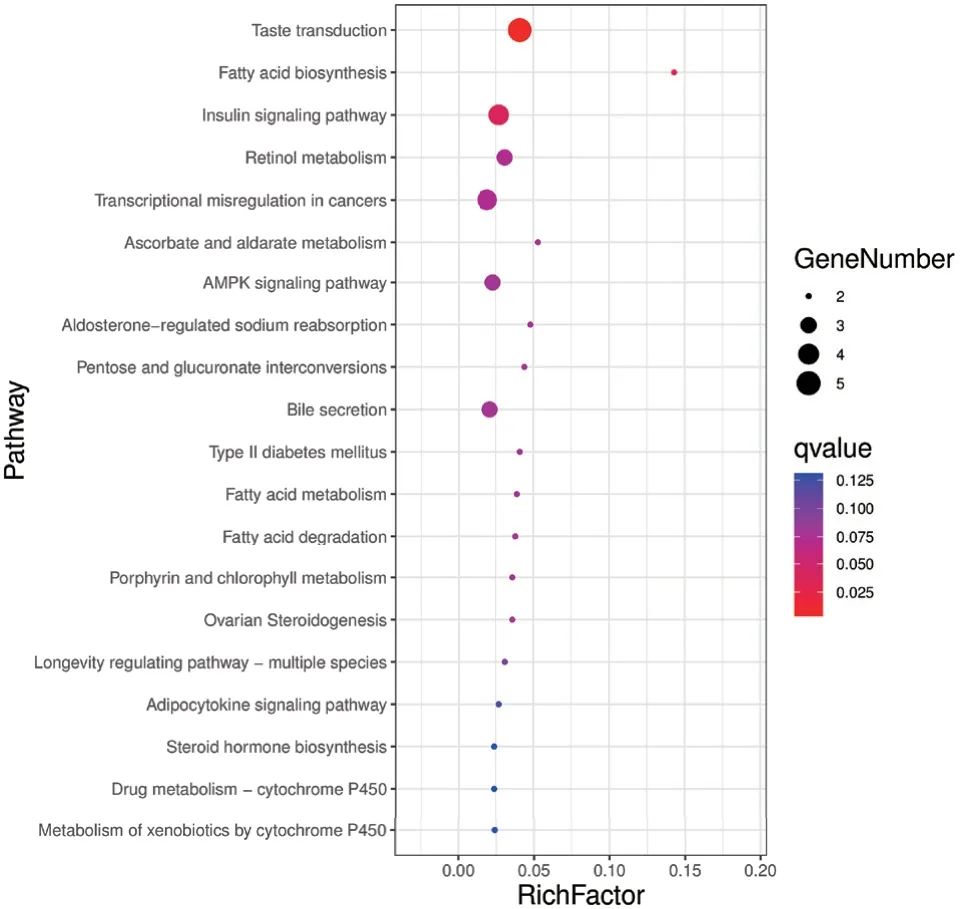
图4 差异表达基因的KEGG分析Fig. 4 KEGG analysis of differentially expressed genes
2.6 基因的蛋白互作网络分析
蛋白质互作网络分析结果发现(图5),在62个差异表达基因中,有29个基因之间发生互作关系。越靠近互作图中心的位置,表示该基因可能发挥的作用越强。由图中可知,IGF1,胰岛素样生长因子结合蛋白1(IGFBP1),胰岛素样生长因子酸不稳定亚 基(IGFALS),LOC108634620,LOC108634720,LOC108634721,C102182562,细胞因子信号转导抑制因子(SOCS2),以及组蛋白去甲基化酶(KDM6A)与其它基因有较强的互作关系。
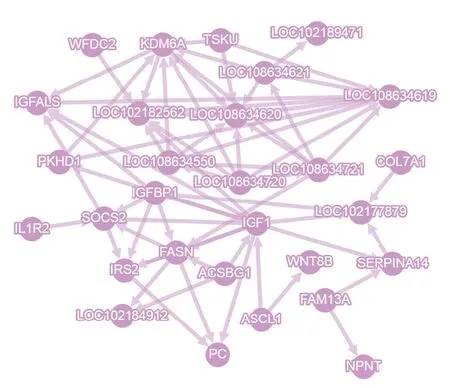
图5 差异表达基因的PPI分析Fig.5 PPI analysis of differentially expressed genes
3 讨论
RNA-Seq技术,主要研究某一时间段内特定细胞在一种功能状态下转录出的所有RNA的总和,是研究基因表达图谱以及蛋白质生物信息功能的重要方法[9]。本研究通过RNA-Seq技术对低蛋白饲粮和正常组饲粮饲喂山羊肝脏组织进行了差异表达基因筛选,并对其中的5个基因进行了qRT-PCR验证,验证结果与测序结果一致,表明测序结果可靠。本研究中,低蛋白饲粮显著降低了PC和IGFBP1表达量,增加IGFALS,IGF1和IRS2基因表达量。丙酮酸羧化酶是调节肝脏糖异生的限速酶[10]。IGF1可促进组织摄取葡萄糖,调节糖原异生和糖酵解,促进蛋白质和脂肪合成[11]。IRS2是胰岛素信号传递的重要介质,在血糖和胰岛素敏感性的调控中发挥着重要作用[12]。IGFALS几乎由肝脏产生并分泌到血液循环中,以高浓度存于血清中[13],其可与IGF-1形成复合物稳定血清中IGF-1的半衰期[14]。IGFBP1是胰岛素样生长因子结合蛋白家族的成员之一,可负向调控胰岛素信号通路[15]。课题组前期研究发现,在低蛋白饲粮饲喂条件下,肉羊血清葡萄糖浓度显著下降[7]。可见,低蛋白日粮可正向调控胰岛素信号通路,抑制肝脏糖异生,最终影响肉羊血糖浓度。
本研究结合 GO数据库对差异基因主要参与的生物学过程进行了分类,发现差异表达基因主要参与了结合和催化活性等生物学过程,表明饲粮蛋白水平对肝脏的生理和代谢有较大的影响。研究发现,低蛋白饲粮通过提高猪[16]和羊[17]骨骼肌细胞中FASN基因表达,增加骨骼肌肌间脂肪的含量。与前人研究结果相似,本研究结合KEGG富集分析,筛选出23条差异信号通路,其中2个上调表达基因(酰基辅酶A硫酯酶和FASN)参与脂肪酸合同通路。碳水化合物反应原件(ChoRE)是脂类合成的关键转录因子,可被高糖激活,诱导下游基因脂肪酸合成酶的转录和表达[18]。FASN是脂肪酸从头合成的关键酶,可直接与ChoRE启动子结合,促进葡萄糖转化为脂类[19]。因此,在山羊肝脏中FASN表达量的上升提示低蛋白饲粮可增加肝脏脂肪的合成。
过氧化物酶体增殖物活化受体(peroxisome porliferator-activated receptor,PPAR)属 II 型核受体超家族成员,是一类由配体激活的核转录因子,是调节脂质代谢、脂肪生成、胰岛素敏感、炎症反应、细胞生长和分化的重要因子[20]。KEGG分析得到PPAR信号通路的显著富集,提示机体能量平衡受影响。PPAR目前发现有3种亚型,PPARα,PPARβ(δ)和 PPARγ,其在不同组织中表达。PPARα 主要在脂肪酸氧化较高的组织中表达,如肝脏、心脏、肌肉和棕色脂肪组织等,参与脂肪酸代谢、炎症反应等[21]。上调基因CYP4A1和ACSBG1基因富集于PPAR信号通路。CYP4A1 参与环境化合物在体内的代谢过程,受PPARα调控,是脂肪酸β酸氧化过程中的关键酶。ACSBG1可促进细胞增殖,诱导细胞凋亡[22]。本研究中,CYP4A1和ACSBG1基因表达量在低蛋白饲粮组山羊肝脏组织中均上调,表明低蛋白饲粮可促进山羊肝脏细胞的增殖,诱导肝脏脂肪酸氧化,而过度的脂质氧化会对肝脏造成直接的毒性作用。因此,低蛋白饲粮对肝脏健康不利。通过PPI互作图发现KDM6A与其它基因有较强的互作关系。KDM6A在包括转录在内的多种生物进程中发挥非常重要的作用[23],KDM6A基因敲除会影响细胞分化[24]。本研究中,低蛋白饲粮显著降低山羊肝脏组织中KDM6A基因的表达,同样提示低蛋白饲粮对肝脏组织发育不利。
4 结论
在低蛋白饲粮饲喂条件下,山羊肝脏组织大量的基因发生了显著的差异表达,部分基因与蛋白质代谢、能量及糖脂代谢相关。这些差异表达基因的功能主要集中于肝脏胰岛素信号通路和脂质代谢调控。低蛋白饲粮促进肝脏脂肪酸的合成和脂质过氧化,抑制肝脏糖异生,不利于肝脏组织发育。
