铜绿假单胞菌对鲎素耐药前后的差异表达基因及SNP变化研究
2021-11-06洪军卫夏怡吉冰洁叶延欣程天赐
洪军 卫夏怡 吉冰洁 叶延欣 程天赐
(河南城建学院生命科学与工程学院,平顶山 467036)
铜绿假单胞菌(Pseudomonas aeruginosa,PA)作为一种人畜共患条件性致病菌,通常感染肺部、尿道、烧伤等部位,已成为医院和养殖厂等感染的三大致病菌之一。由于对抗生素的多重耐药性,在临床抗感染治疗中有着特殊重要的地位。该菌极易形成生物膜,单独使用抗生素的治疗效果并不理想。目前对铜绿假单胞菌感染的防治是医学界迫切需要解决的问题,对其耐药性产生的研究为预防和治疗由铜绿假单胞菌引起的感染具有重要的指导意义。
鲎素(Tachyplesin I)是1988年由Nakamura等[1]从中国鲎血细胞的酸性提取物中分离出来的一种具有17个氨基酸残基的典型环状β-折叠结构的阳离子抗菌肽。已研究证明鲎素具有广谱抗菌活性,可开发成抗菌药物、抗肿瘤药物、免疫调节剂和饲料添加剂等。2019年12月份国家农业农村部发布194号公告指出:自2020年元旦起,我国饲料中全面禁止添加抗生素,减少滥用抗生素造成的危害,维护动物源食品安全和公共卫生安全。因此寻找一些新型抗菌剂替代抗生素已成为新时代绿色环保养殖行业的重中之重。作为抗生素替代品的侯选开发抗菌肽之一,除了作为抗菌药物使用外,也可以作为新型功能性添加剂应用到饲料中。然而,已有研究报道表明一些细菌(尤其人类病原菌)已对部分抗菌肽产生了耐药性[2-3]。本课题组在实验室条件下通过长期连续增高浓度鲎素诱导后,铜绿假单胞菌CGMCC 1.2620和ATCC 27853菌株均能对鲎素产生不同程度的耐药性[4-5],但是铜绿假单胞菌耐药前后能引起哪些基因的差异表达以及是否引起SNP和sRNA的变化,以及sRNA是否能调控这些差异表达基因尚不清楚。
随着高通量测序技术的迅速发展,细菌转录组学的研究可帮助人们探究细菌耐药前后发生差异表达的基因以及筛选出具有调控作用的非编码RNA。sRNA(small RNA)是一类广泛存在于原核生物基因组中能被转录但不编码蛋白质的一类RNA 分子,长度为50-300 nt的核苷酸,位于基因组2%-12%的基因间区域。有研究表明,细菌为适应各种不同的外界环境,通过较为复杂的基因调控网络来对其做出反应,而其中sRNA发挥了重要的作用[6-7],并揭示sRNA与细菌耐药性密切相关[8]。通过转录组学研究手段筛选细菌中存在的sRNA并研究sRNA所发挥的功能,对揭示细菌耐药基因转录后调控具有重要意义。Miller等[9]表明铜绿假单胞菌中存在着一系列sRNA,其中Rsm W的过度表达及Rsm Z的表达水平降低均有助于增强生物膜的形成,而Rsm Z的过度表达则会抑制生物膜的形成。刘翠翠[10]利用RNA-Seq技术对福氏志贺菌的耐药株中差异表达的sRNA进行了GO富集分析和KEGG Pathway分析,为揭示福氏志贺菌sRNA与其耐药性的关系奠定基础。Zhang等[11]表明在环境适应过程中,细菌利用sRNAs来抑制或激活其蛋白质组的大部分表达,并且已经鉴定出了两个sRNAs(Sr0161和ErsA)主要是与负责碳青霉烯类抗生素摄取的主要孔蛋白OprD mRNA相互作用。这两个sRNAs与oprD的5'UTR碱基配对导致细菌对美罗培南的耐药性增加。Sr0161在没有Hfq的情况下,正调控PagL的表达,降低其促炎特性并导致多粘菌素抗性。通过RNASeq技术等转录组学研究手段可筛选出对细菌外排泵系统、生物被膜形成等具有调控作用的sRNA及靶基因,从RNA水平揭示了sRNA对细菌耐药性形成的影响。
SNP(single nucleotide polymorphism)主要是指基因组在核苷酸水平上发生变异引起的DNA序列多态性,包括单个碱基转换、颠换以及单个碱基插入和缺失等。在基因组DNA中,任何碱基都可能发生突变,因此SNP可存在于基因的任意序列中,可能位于非编码序列中,也可能位于基因序列中。张栋等[12]采用直接测序法从P0代尼罗罗非鱼的β2m基因组序列中筛选出30个SNP,获取其位置信息,并利用Popgen32和picc -calc软件分析其遗传参数,结果表明24个SNPs的基因型和等位基因频率与无乳链球菌的耐感/易感态显著相关。
本研究通过对铜绿假单胞菌野生株CGMCC1.2620(PA1.2620)和 高 抗 鲎 素 突 变 株CGMCC1.2620-99(PA-99)进行转录组测序,在转录组水平上通过对差异表达基因以及差异表达的sRNA的靶基因进行功能富集和SNP分析,找到与抗药相关的差异表达的sRNA基因及其相关通路信息以及SNP变化,为进一步探讨铜绿假单胞菌对鲎素的耐药性机制提供理论依据。
1 材料与方法
1.1 材料
实验菌株:铜绿假单胞菌CGMCC1.2620(PA1.2620)由中国微生物菌种保藏中心(CGMCC)提供。耐鲎素突变株铜绿假单胞菌PA-99是由PA1.2620菌株在本实验室长期连续暴露的增高鲎素浓度选择压力下转接99代获得的突变株。鲎素对PA1.2620和PA-99菌株的最小抑制浓度值分别为10 μg/mL和150 μg/mL。
抗菌肽:鲎素(tachyplesin I,含有两个二硫键的环行肽)由吉尔生化(上海)有限公司合成,纯度为95%以上。
1.2 方法
1.2.1 测序样品制备及转录组测序 PA1.2620原始菌株和PA-99突变株在30℃恒温摇床上180 r/min培养12 h后,按OD600=0.2的浓度转接到新鲜的MHB培养基中,并生长至OD600=1.0时开始收集。用细菌总RNA纯化试剂盒提取和纯化总RNA(Sangon Biotech Co.,Ltd.,Shanghai,China)。为 了 确 保总RNA能满足转录组测序分析的质量,分别采用Nanodrop、Qubit 2.0、Agilent 2100、电泳方法检测RNA样品的纯度、浓度、完整性和是否有基因组DNA污染等。每个样品3个重复。总RNA样本检测合格后,委托百迈客生物科技有限公司对样品进行cDNA文库构建和有参转录组测序分析;测序平台Illumina HiSeq2500。
1.2.2 基因表达量及差异表达基因功能分析 利用DESeq软件进行差异表达分析,基因表达量的计算使用RPKM(Reads Per Kilobase of transcript per Million fragments mapped)法。将Fold Change≥2且FDR<0.01作为筛选差异表达基因标准,即log2FC≥1 是上调表达,log2FC≤1下调表达[13]。得到差异表达基因后,对差异表达基因进行功能注释、KEGG Pathway和GO功能分析[14-16]。利用KEGG和Gene Ontology数据库找出与整个基因组背景相比在显著差异表达基因中q-Value≤0.05的显著性富集的Pathway及GO条目。
1.2.3 RT-qPCR验证 用RT-qPCR来验证转录组测序检测结果的准确性。分别从PA-99突变株和原始菌株PA1.2620中选择5个靶基因进行RT-qPCR分析。每个基因设3个重复。此外,使用KAPA SYBR FAST qPCR试剂盒在ABI7900实时荧光定量PCR仪上进行RT-qPCR测定。利用Primer Premier 5软件根据铜绿假单胞菌基因组序列设计特异性引物。反应条件:95℃ 2 min,94℃ 20 s,60℃ 20 s,72℃ 30 s,40个循环。以16S rRNA为内参,基因的相对表达水平用2-ΔΔCt方法定量[17]。
1.2.4 差异表达sRNA及靶基因的分析 利用miRDeep2 软件将测序Read比对到基因组上后,看那段区域是否能形成发夹结构,且Read位于臂上,得到一个分值,筛选后得到预测结果,用的软件是miRDeep2。筛选出PA1.2620与PA-99差异表达的sRNA;对筛选出来的差异sRNA靶基因数据进行处理,找出差异表达的基因,并对其进行GO功能和KEGG通路富集分析。
1.2.5 差异表达基因和sRNA靶基因的SNP分析 基于各样品Reads与参考基因组序列的Bowtie比对结果,使用GATK软件识别测序样品与参考基因组间的单碱基错配,查找基因区潜在的单核苷酸多态性SNP位点。
在有参转录组测序的结果上进行比对,对实验中所用对照组与处理组的各个样品组内发生的SNP进行比对统计,并对其类型和数目统计结果进行分析。通过百迈客云平台的基于基因ID过滤GFF信息小工具,在铜绿假单胞菌的gff文件中提取得到基因对应的位置信息。将SNP的位置信息与基因对应的位置信息比对,可以得到差异基因中产生SNP的数目和类型。所得数据主要通过百迈客云平台分析。
2 结果
2.1 测序数据及表达量分析
对原始测序数据进行过滤和质量控制,之后对两个样品的转录组测序数据进行评估,包括与参考基因组的比对(比对效率都在87.65%及以上)、rRNA统计、测序随机性评价以及Reads在参考基因组上的分布等,这些分析结果表明该测序结果较为理想。在铜绿假单胞菌6 318个基因中,PA-99与PA1.2620菌株的表达差异的基因有1 459个,其中上调表达的基因为672个,下调表达的基因为787个。其中log2FC值上调表达倍数为3.0以上有35个基因,下调表达倍数为3.0以上基因有58个,且下调倍数log2FC大于11为28个基因。此结果表明,铜绿假单胞菌对鲎素产生耐药性导致许多基因发生差异表达。
2.2 PA1.2620与PA-99菌株差异表达基因的功能富集分析
2.2.1 差异表达基因的GO功能富集分析 由GO富集的前20个GO条目图1可知,在分子功能方面发生富集的亚类主要有:核苷酸结合(nucleotide binding,18)、甲酸脱氢酶(NAD+)活性(formate dehydrogenase(NAD+)activity,6)、错 配DNA结合(mismatched DNA binding,4)、受体结合(receptor binding,3)等等;在生物过程发生主要富集的GO条目有:细胞呼吸(cellular respiration,4)、错配修 复(mismatch repair,4)、细 胞 溶 解(cytolysis,4)等;在细胞组分主要发生富集的GO 条目是细胞膜的组成部分(integral component of membrane,158)。最为富集的前3条GO Term依次为:integral component of membrane、nucleotide binding、formate dehydrogenase(NAD+)activity。在GO功能研究中,我们重点关注GO富集的前20个GO条目。
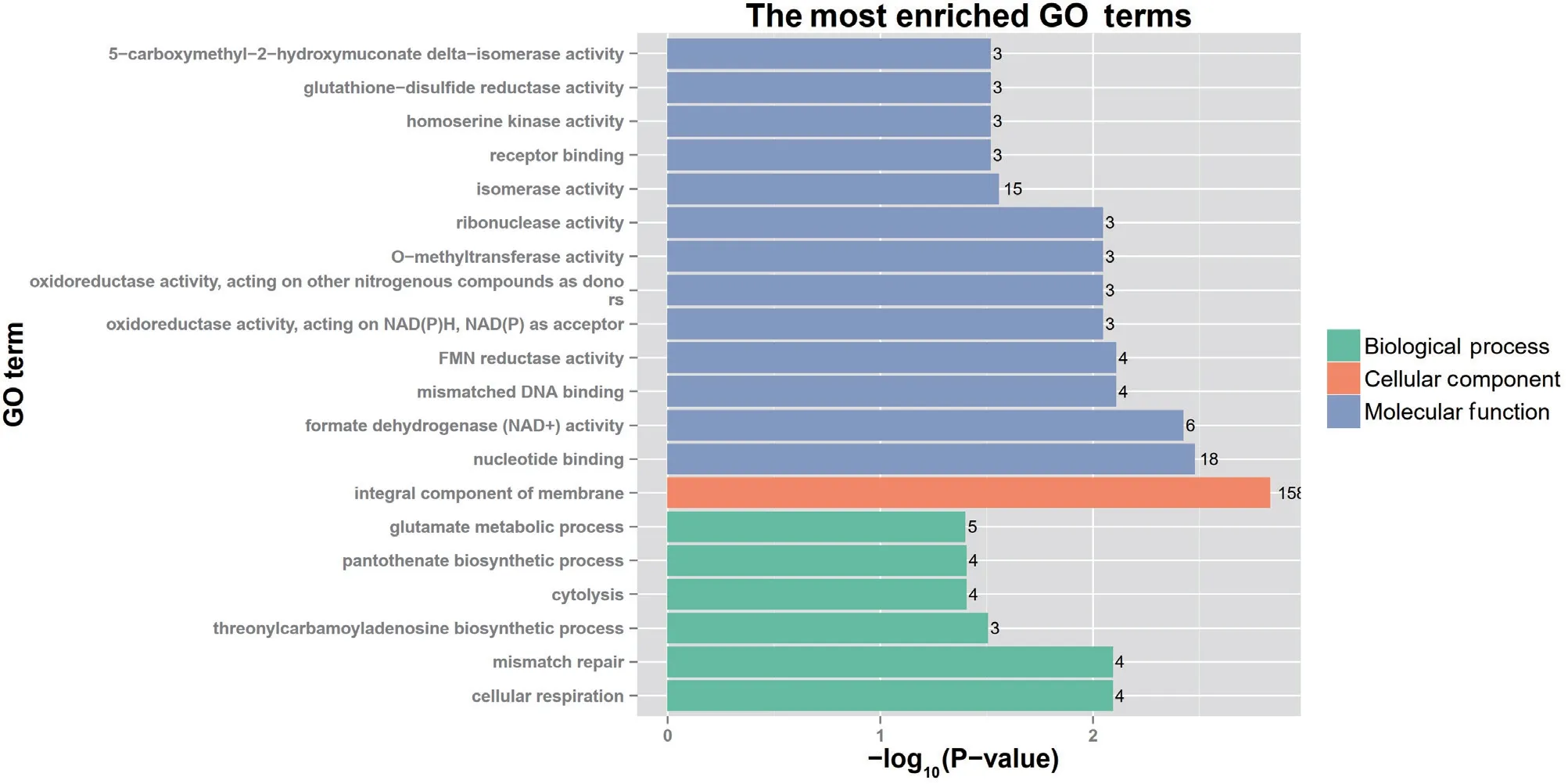
图 1 PA1.2620 与 PA-99 菌株差异基因的 GO 富集图Fig. 1 GO enrichment analysis of differentially expressed genes between PA1.2620 vs. PA-99 strains.
2.2.2 KEGG通路富集分析 根据PA1.2620与PA-99菌株差异表达基因的KEGG结果可知,此次分析将395个基因注释到KEGG的98条通路上,涉及到各种氨基酸、脂质、能量、糖类等代谢(266个基因,最多)、ABC转运(36个基因)、双组分系统(43个基因)、细菌分泌系统(16个基因)、细菌趋化性(16个基因)、氨酰生物合成(14个基因)等通路。由图2富集的前20个通路可知,无显著富集的通路。其中细菌趋化性(bacterial chemotaxis)、氨酰生物合成(aminoacyl-tRNA biosynthesis)通路中所涉及的基因通常与抗药性密切相关。但Toluene degradation、Chlorocycchexane and chiorobenzene degradgfion、Phosphonate and phosphinate melaboisn、Novobiocin biosynthesis、Aminobenzoate degradation这些通路需要我们重点关注。

图2 PA1.2620与PA-99菌株差异基因的KEGG富集图Fig.2 KEGG pathway enrichment analysis of differentially expressed genes between PA1.2620 vs. PA-99 strains
2.3 RT-qPCR验证
为了验证转录组测序及后续SNP和sRNA靶基因等分析的结果,根据文献报道挑选了疑似耐药性的ParR、phoB、PA3573和opr86差异表达基因以及上调表达量最高的PA0912基因进行RT-qPCR测定。如图3所示,所有选择的差异表达基因在RNA-Seq和RT-qPCR结果之间显示一致的表达模式,但是整体而言RT-qPCR数值较RNA-Seq表达量数值偏高。

图3 RT-qPCR验证结果Fig. 3 RT-qPCR validation results
2.4 SNP分析
2.4.1 SNP突变类型统计 对PA-99突变株各样品对比PA1.2620样品筛选出SNP位点数目、转换类型比例、颠换类型比例以及杂合型SNP位点比例进行统计,如表1所示。以及对比分析得到各个处理样品组的SNP类型进行数据统计如图4所示。根据图和表可知:相对于对照基因组来讲,各个样品组间发生的SNP类型频率相对一致,在其中发生转换类型的频率远高于颠换类型,在转换之中数量最多的为A/G,其次是C/T。

表1 SNP位点统计表Table 1 Statistical table of SNP sites
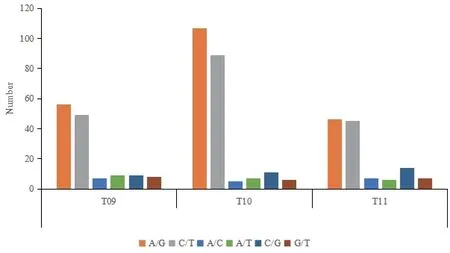
图4 PA-99突变株样品的SNP突变类型分布图Fig. 4 Distribution of SNP mutation types in PA-99 mutant samples
2.4.2 差异表达基因上产生的SNP分析 以PA1.2620菌株作为对照组,将PA-99组菌株与其对比结果汇总在一起查找差异表达基因的SNP。由PA1.2620与PA-99菌株的差异表达基因上产生的SNP可知,在1 459个差异表达基因中,共有SNP发生的基因数目为43个(25个上调和18个下调基因),其中新基因是10个如Novel_244、Novel_137、Novel_28等,差异表达值最大的基因是未知功能基因(ID为Novel_28基因)。对这些发生SNP基因过滤掉仅在一个生物学重复中发生的碱基突变,在这43个差异表达基因中SNP为PA-99菌株3个重复组共同发生的基因数目为22个(12个表达上调和10个表达下调基因)如表2所示,此结果也表明不仅不同处理组间存在SNP变化,而且重复组内也存在SNP变化的差别。这22个发生SNP的基因中包括5个新基因,7个假定蛋白基因,10个已知功能基因,SNP主要发生在基因表达量log2FC值在1-3倍之间。Novel_745基因的差异表达值最大(上调表达log2FC=2.58倍以上),是一种未知功能蛋白,但仅发生了一个碱基突变。这些发生SNP突变的已知基因,如pagL 基因是脂质A脱酰基酶,是参与脂质A代谢过程功能,下调表达log2FC=2.3倍以下;opr86是一个编码外膜蛋白基因;PmrB双组分调节系统信号传导激酶,与脂多糖的修饰有关。

表2 PA1.2620与PA-99菌株差异表达基因的SNP信息Table 2 SNP analysis of differentially expressed genes in PA1.2620 vs. PA-99 strains
2.5 PA1.2620与PA-99菌株差异表达sRNA分析
2.5.1 差异表达sRNA及其靶基因数目分析 由表3和图5可知:按照筛选差异表达基因的标准,筛选到PA1.2620与PA-99菌株的差异表达sRNA共有11个,其中1个sRNA上调表达,10个下调表达,且均是未知功能sRNA。通过对其靶基因进行筛选,筛选到的靶基因总数为5 840个,其中差异表达靶基因为1 247个。每个sRNA所对应的靶基因个数差别较大,其中sRNA Novel_583有968个靶基因包括223差异表达靶基因,而Novel_33有25个靶基因,仅存在4个差异表达靶基因。这些结果表明,sRNA所对应的靶基因越多,调控的基因功能越多,且存在不同sRNA基因调控同一靶基因的现象。因其存在不同sRNA有相同的靶基因,过滤掉重复后其靶基因总数为4 027个,其中差异表达靶基因为863个。随后重点关注这些sRNA的差异表达靶基因。

图5 PA1.2620与PA-99菌株差异表达的sRNA靶基因数目统计图Fig.5 Statistical diagram of the number of sRNA target genes of differentially expressed genes in PA1.2620 vs. PA-99 strains

表3 PA1.2620与PA-99菌株差异表达sRNATable 3 sRNA of differentially expressed genes in PA1.2620 vs. PA-99 strains
2.5.2 sRNA差异表达靶基因GO功能富集分析 根据PA1.2620与PA-99的 差 异 表 达sRNA靶 基 因的GO富集前20个GO Term可知(图6),在分子功能大类下主要发生富集的GO Term有:高丝氨酸激酶活性(homoserine kinase activity,3)、5-羧甲基-2-羟基黏液酸δ-异构酶活性(5-carboxymethyl-2-hydroxymuconate delta-isomerase activity,3)、转录因子结合(transcription factor binding,8)等等;在生物学过程大类下主要发生富集的GO Term有:组氨酸生物合成(histidine biosynthetic process,7)、苏氨酸氨基甲酰胺腺苷生物合成过程(threonylcarbamoyladenosine biosynthetic process,3)、细胞蛋白质代谢过程(cellular protein metabolic process,13)等。最富集的前5个GO Term依次为:组氨酸生物合成(histidine biosynthetic process)、高丝氨酸激酶活性(homoserine kinase activity)、5-carboxymethyl-2-hydroxymuconate delta-isomerase activity(3)、threonylcarbamoyladenosine biosynthetic process、transcription factor binding,均为极显著富集水平。

图6 PA1.2620与PA-99菌株表达差异sRNA靶基因的GO富集图Fig.6 GO enrichment of sRNA target genes of DEGs in PA1.2620 vs. PA-99 strains
2.5.3 差异sRNA靶基因的KEGG富集分析 通过KEGG富集图7可知,没有显著富集的通路。但是Biosynthesis of unsaturated fatty acids、Phosphotransferase system(PTS)、Degradation of aromatic compounds、Carbon metabolism等通路所涉及的差异表达基因富集较可靠。
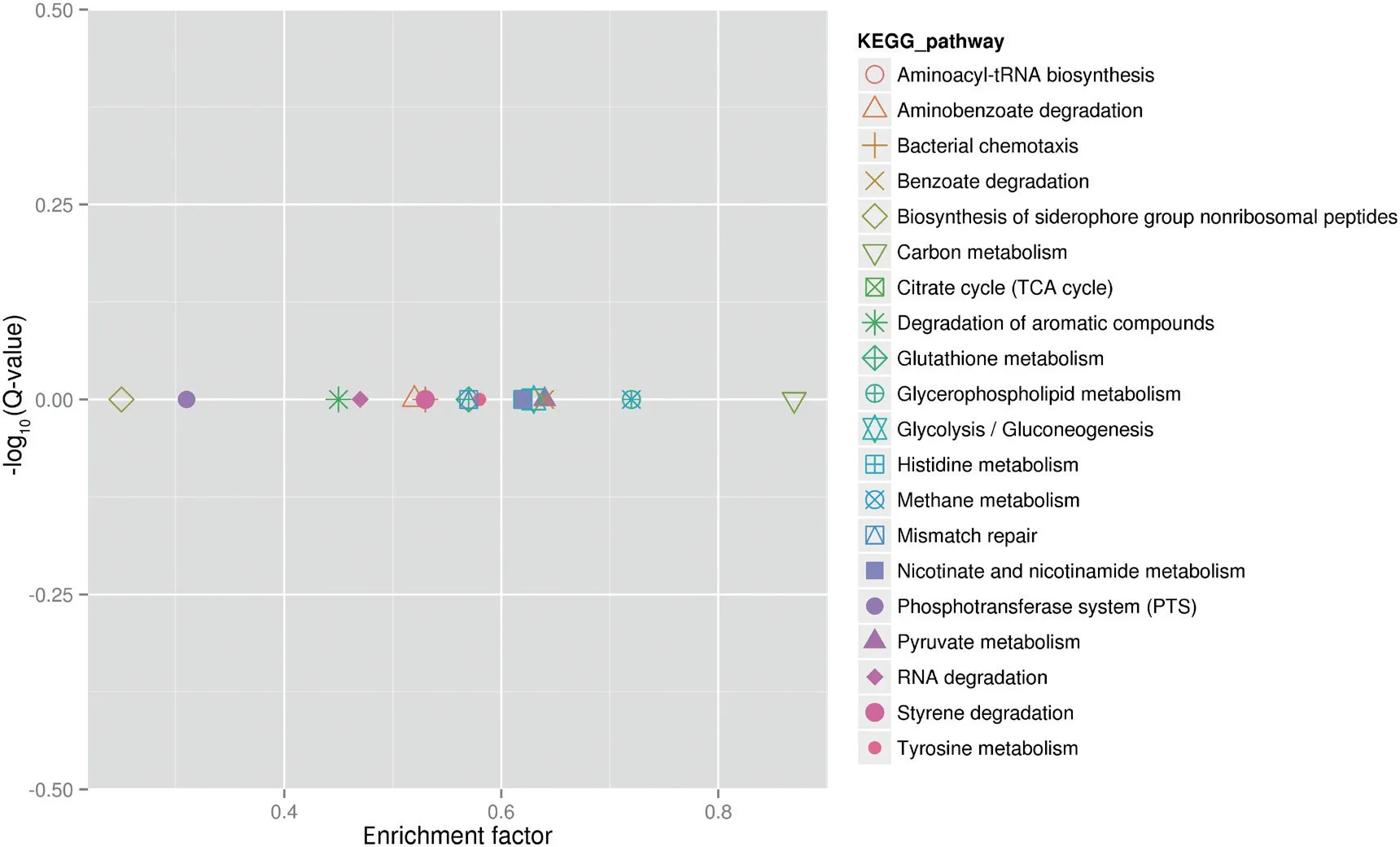
图7 PA1.2620与PA-99菌株表达差异sRNA靶基因的KEGG富集图Fig.7 KEGG enrichment of differentially expressed sRNA target genes in PA1.2620 vs. PA-99 strains
2.5.4 差异表达sRNA靶基因的SNP分析 对PA1.2620与PA-99菌株的差异表达的11个sRNA发生的SNP的数据为0个。对其差异表达sRNA的863个差异表达靶基因上发生的SNP的数据进行分析,共有22个靶基因发生SNP变化,其中在PA-99菌株的3个生物学重复中两者或两者以上共同发生的有10个靶基因(表4)。sRNA靶基因发生SNP变化的较少,且多发生在较低差异表达靶基因,仅有pvdL基因有2个位点突变,其余9个基因均发生一个位点突变。这些SNP的变化主要涉及的sRNA所作用的靶基因除了3个靶点新基因外,还涉及到编码外膜蛋白基因opr86;双组分调节系统信号传导激酶pmrB,该双组分调控系统调控PA3552-PA3559 脂多糖修饰操纵子,该操纵子是阳离子抗菌肽和多粘菌素B阳离子抗菌肽耐药所必需的;以及编码肽合成酶基因pvdL,主要参与铜绿假单胞菌铁载体合成和脂类生物学合成过程,参与催化活性的分子功能。sRNA如何参与这些靶基因的功能有待进一步研究及验证。

表4 sRNA相关靶基因的SNP变化分析Table 4 SNP analysis of sRNA target genes
3 讨论
通过对耐药菌株中差异表达基因的分析,有助于研究者们从众多差异表达的基因中筛选出与细菌耐药性密切相关的基因及其可能参与的代谢途径,可为揭示细菌耐药性机制提供新的思路,同时为新型抗耐药菌药物的研究奠定理论基础。sRNA是一类新发现的基因表达调控因子,通过与靶mRNA或靶蛋白配对,从而调控细胞的生理功能以应对各种环境变化,尤其在细菌耐药性的产生方面至关重要。这些差异表达的sRNA主要是通过作用于相应的靶mRNA,进而发挥其调控作用。在基因组水平上,由单个核苷酸变异引起的DNA序列多态性即称为SNP,SNP可进行复杂性状的关联分析,从而揭示相关性状的遗传机理。基因突变产生的耐药性是一个长期积累的过程,不同于转录组,基因组较稳定,不易发生突变,在经过特殊的诱导处理后可产生定向的突变以应对环境中的选择性压力。这些突变可能是菌株生存环境变化后为适应新环境做出的一些应对调整,可能与耐药性、毒力等相关基因的特异性突变无关。对于差异表达基因、sRNA所对应靶基因与相应SNP进行分析,可从转录组和基因组层面来预测铜绿假单胞菌对鲎素耐药性产生的原因,从而对于耐药性机制的预测作为相互补充。因此,本研究基于转录组测序的基础上,对差异表达基因的SNP和sRNA的靶基因进行了预测分析,以期为铜绿假单胞菌对鲎素的耐药性的产生提供一定的理论支持。
本研究通过对高抗鲎素铜绿假单胞菌PA-99菌株与PA1.2620敏感菌株进行转录组测序,在转录组水平上通过对差异表达基因及sRNA靶基因进行富集分析,发现这些基因大多与氨基酸、糖类等物质的代谢通路、DNA转录、金属离子的转运以及代谢、细菌趋化性、双组分调控系统等通路有关以及涉及到细胞膜组成部分、核苷酸结合及酶的活性等功能。对于差异表达基因和sRNA靶基因的SNP变化,主要涉及到编码脂质A脱酰基酶、外膜蛋白、冷休克蛋白、铁代谢、以及与脂多糖修饰相关等已知基因和一些编码的假定蛋白、部分编码的新基因有关,这些靶基因的SNP变化是否与抗药性相关有待进一步验证。这些结果表明sRNA在调控铜绿假单胞菌对鲎素的抗药性方面也发挥了重要作用。
典型的双组分调控系统由组氨酸激酶和相关的应答调控蛋白组成,组氨酸激酶感受外界环境变化后调节应答蛋白的磷酸化水平,而后对一系列应答基因进行调控。双组分调控系统在铜绿假单胞菌的致病性、毒力、生物膜形成和耐药性等方面起着重要作用[18]。PmrA-PmrB双组分调控系统在革兰氏阴性菌对多黏菌素B耐药过程中起重要作用[19]。本研究发现耐鲎素铜绿假单胞菌PA-99菌株中双组分调控系统有43个差异表达基因,包括一些受调控的抗性基因,如pmrA-pmrB、反应调节因子PhoP(基因ID:Novel_194)。PhoA编码的碱性磷酸酶(基因ID:Novel_498,499)和PA-99突变体中的两个成分反应调节因子PhoB;并预测到sRNA靶基因中有29个差异表达基因,且仅编码修饰脂多糖PmrB基因的SNP发生碱基突变。此结果表明,双组分调控系统在铜绿假单胞菌对鲎素的耐药中同样发挥作用,且sRNA也有可能参与调节双组分系统表达。
氨酰-tRNA生物合成(aminoacyl-tRNA biosynthesis)的功能是将氨基酸与含有相应反密码子的tRNAs精确匹配。除了在蛋白质合成和某些细菌革兰氏阳性菌肽聚糖交联或膜磷脂修饰中发挥重要作用外,氨酰基-tRNA生物合成还直接参与抗生素生物合成和耐药性[20]。例如,MprF蛋白通过催化细胞内膜脂的氨基酰化来调控细胞壁对阳离子抗菌肽的通透性,从而介导抗菌肽的耐药性[21]。膜脂的氨酰化通过调控细胞膜双分子层的净负电荷,其方式依赖于氨酰化 tRNA底物。在本研究中,氨酰基-tRNA生物合成通路在高抗鲎素铜绿假单胞菌PA-99菌株中有14个差异表达基因以及预测的sRNA靶基因中有10个差异表达基因在此通路中,但这些基因无SNP发生变化。这个结果表明铜绿假单胞菌对鲎素抗药性也涉及到此通路。
生物膜形成的耐药机制在铜绿假单胞菌中起着关键作用。铜绿单胞菌生物膜的形成途径包括群体感应、细菌分泌系统和化学感觉系统等。在转录组数据中,我们发现有许多与生物形成相关的差异表达基因及部分sRNA靶基因,如鞭毛组装(ko02040)、细菌分泌系统(ko03070)和细菌趋化(ko02030)通路,这些通路涉及基因之前被认为与生物膜形成途径有关。在前期研究中发现,PA-99突变体比原始菌株PA1.2620更容易形成生物膜,胞外多糖含量也高于原始菌株[5]。已有学者探讨了sRNA在某些细菌的铁代谢、糖代谢以及氨基酸代谢中所发挥的调控作用[22]。针对铁代谢,许多细菌都会进化出自身特殊的铁调控系统来限制铁的摄取。Fur作为一种较为重要的铁调控蛋白参与到sRNA对铁的调节过程中。在本研究中,sRNA的靶基因vreR是与铁代谢相关基因,也发生了SNP碱基突变。葡萄糖、氨基酸等是细菌的能源物质,在细菌耐药过程中也是受到了严格的调控。有研究指出细菌可以通过sRNA调节群体感应系统,进而调控毒力产生、抗菌药物耐药等过程。但是,相对于大肠杆菌、沙门氏菌等细菌,关于铜绿假单胞菌的sRNA调控耐药机制的相关报道相对来说较少,这其中的机制尚不清楚。
4 结论
铜绿假单胞菌对鲎素的耐药性导致许多基因发生差异表达,且有些差异表达基因受到sRNA的表达调控,并有极个别相对低差异表达基因的SNP发生碱基突变,这些突变基因主要与膜蛋白修饰、铁代谢和编码的假定蛋白等有关。尽管本研究未涉及这些SNP突变基因的功能验证,但在文献中已有报道这些已知基因是与抗药性相关,且已选择5个代表性差异表达基因通过荧光定量证实差异表达基因的表达趋势与转录组测序表达量基本一致。
