藏、汉族EPAS1基因启动子区DNA甲基化的差异研究*
2021-10-22李小薇肖军雷慧芬白晓莹孟方园李翠莹
李小薇 肖军 雷慧芬 白晓莹 孟方园 李翠莹
我国高原面积约占国土面积1/4,因低压低氧、干燥寒冷等不仅限制其发展,而且严重威胁进入高原环境工作及旅游等人员的身心健康。世居平原人员首次进入高原后会发生红细胞(RBC)数目、血红蛋白(Hb)浓度增加等生理改变,以改善组织供氧。但是进入高原若产生过多的红细胞则使血液粘度加大,进一步加重组织缺氧,不利于高原习服。近年来,多项研究发现EPAS1、EGLN1等低氧诱导因子相关基因的序列变异在高原低氧适应中具有重要作用,且与藏族人相对低Hb浓度密切相关[1,2]。然而关于EPAS1基因DNA甲基化、组蛋白乙酰化等表观遗传学变化在高原低氧习服与适应中的研究较少。因此本研究拟在世居高原藏族、平原汉族及久居高原汉族中比较EPAS1基因DNA甲基化水平的差异,并分析其在高原低氧习服与适应的可能作用。
材料与方法
1 样本来源 抽取世居高原藏族、世居平原汉族及久居高原汉族(>10年)各10名志愿者抗凝血标本,志愿者全部为19~50周岁健康男性,平原汉族组19~44周岁(平均年龄2 7.8 0±8.0 8),久居高原汉族组24~50周岁(平均年龄33.30±8.11),世居高原藏族21~50周岁(平均年龄31.10±7.84)。所有志愿者均签署知情同意书,并通过调查,排除相互之间的亲缘关系。本研究经中国人民解放军空军特色医学中心(原空军总医院)伦理委员会批准(第2017-08-YJ02)。
2 试剂和仪器
2.1 试剂:D N A提取试剂盒(Bio TeKe Corpration),DNA染料(BioTeKe Corpration),琼脂糖(BIOWEST,REGΜLAR (AGAROSE,G-10)),NaHSO3(Zymo),EpiTYPER™Reagent Kit(Agena,Inc),总RNA提取试剂盒(天根生化,北京),Quant cDNA第一链合成试剂盒(天根生化,北京),SuperReal PreMix Plus (SYBR Green)(天根生化,北京)。
2.2 仪器:低速离心机(湖南湘仪实验室仪器开发有限公司),核酸自动提取仪(Bio TeKe Corpration),分光光度计(Nano Drop 2000,Thermo),凝胶电泳成像仪(Major science),ABI veriti-384 PCR仪(ABI),384-well SpectroCHIP®bioarray芯片(Agena,Inc),Mass ARRAY Nanodispenser 点样机(Agena,Inc),MassARRAY Analyzer 4.0质谱仪(Agena, Inc),普通PCR仪(伯乐T 1 0 0),实时荧光定量PCR仪(Thermo Fisher)。
3 实验方法
3.1 DNA提取:采用DNA提取试剂盒,取适量抗凝外周血,参照操作说明书提取基因组DNA。通过OD检测及凝胶电泳检测DNA浓度及纯度;DNA总量:3~5 μg,浓度大于50 ng/μL。
3.2 甲基化引物设计与合成:通过美国国家生物信息中心(NCBI)获取EPAS1基因序列(序列号NC_000002),按甲基化常规设计方法,截取基因转录起始位点的上游5 000 bp至下游1 000 bp序列,并预测CpG岛,针对CpG岛区进行方案设计。确定目的序列输入到EpiDesigner软件进行引物设计。根据软件运行结果,选择并确定合适的引物序列,合成带有T7 RNA聚合酶启动子序列的引物,5'端引物序列-aggaagagagGGGAATTAGATTGATTTTTTTAATTTT G,3'端引物序列-cagtaatacgactcactatagggagaaggctA ACCTCTACCCTAACCCTAACCC。扩增片段大小为600 bp,设计引物位置距离转录起始点-783 bp~-183 bp。
3.3 NaHSO3处理待检测DNA样品:使用3.6M亚硫酸盐,作用DNA模板1.5 h,将样本DNA中没有甲基化的C全部转化为U(相当于DNA中的T)。使用3.2设计的一对带有T7启动子序列的特殊引物扩增样本,得到带有T7 RNA聚合酶启动子序列的600 bp扩增产物。
3.4 Agena MassArray系统进行甲基化检测:在体外转录体系中,利用T7 RNA聚合酶,将扩增产物转录为RNA片段。在上步操作体系中,利用RNase A能够特异性识别并切割RNA中U 3'端的特性,将RNA片段切割成携带有CpG位点的小片段。使用Agena MassArray®飞行质谱分析系统检测产物,检测结果使用MassArray EpiTYPER™ 1.3软件(Agena,Inc)获取原始数据和圆点图,根据含G峰和含A峰的面积比较,计算出待检样本甲基化及非甲基化程度,属于两者相对定量比值。检测步骤包括PCR扩增反应,SAP反应,T切/RNase A 消化反应,树脂纯化,芯片点样,Massarray分析及数据输出。
3.5 总RNA提取及cDNA合成:采用血液总RNA提取试剂盒,取适量抗凝外周血,参照操作说明书提取总RNA。同时,采用Quant cDNA第一链合成试剂盒进行cDNA合成,所得产物保存在-20℃冰箱。
3.6EPAS1基因mRNA表达引物设计、合成及qRTPCR扩增:通过美国国家生物信息中心(NCBI)获取EPAS1基因RNA序列(序列号NC_000002),Primer Premier 5引物设计软件设计EPAS1基因mRNA表达引物,F-5'ACGCCACCCAGTACCAGGA-3',R-5'AATGAGGGCCCGAGCAGC-3'。采用SuperReal PreMix Plus(SYBR Green)试剂盒参照试剂说明书检测不同分组中EPAS1基因mRNA表达量。
4 统计学处理 采用SPSS 16.0进行不同分组人群中EPAS1基因启动子区DNA甲基化程度的差异分析,符合正态分布进行独立样本t检验,不符合正态分布采用非参数Mann-Whitney U检验,P<0.05表示差异有统计学意义。
结 果
本项目选择E P A S 1基因转录起始位点G上游783 bp~183 bp启动子区域,包含59个CpG位点的甲基化区域,检测结果包含37个CpG位点甲基化程度。运用Agena MassArray核酸质谱定量检测平原汉族、久居高原汉族及高原藏族3组DNA甲基化程度的差异,选择甲基化程度>2.5%的10个位点分析发现平原汉族、久居高原汉族及高原藏族部分CpG位点甲基化程度存在显著差异(表1)。EPAS1基因启动子区整体DNA甲基化程度分别为平原汉族组8.87%±10.31%、久居高原组7.17%±10.12%及高原藏族组9.32%±13.07%,3组人群中均无统计学差异(P>0.05,图1)。
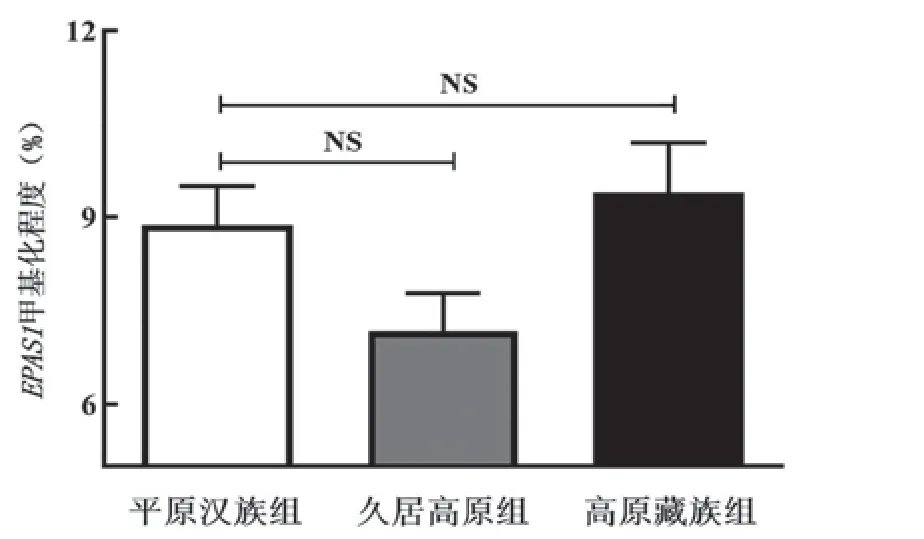
图1 3组人群中EPAS1基因启动子区DNA甲基化程度

表1 3组人群中EPAS1基因启动子区甲基化程度的差异
关于表观遗传学中DNA甲基化的研究普遍认为,基因启动子区DNA甲基化发挥基因表达抑制的作用。本研究分析发现,久居高原汉族EPAS1基因表达量显著高于平原汉族,世居高原藏族略高于平原汉族(图2)。
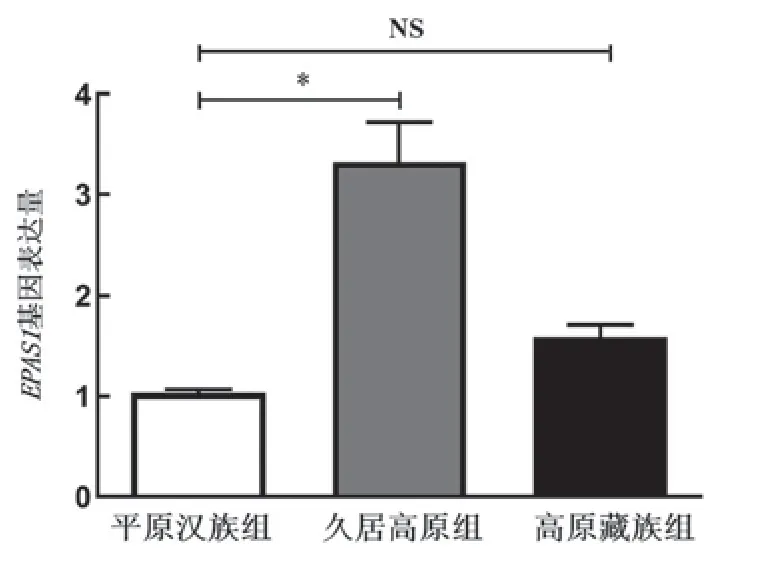
图2 3组人群中EPAS1基因表达量的比较久居高原组vs平原汉族组,*P<0.05
有研究发现CpG位点甲基化与特定基因序列突变相关[3]。因此,研究者在3组人群中分析了EPAS1基因rs13419896、rs1868092及rs4953354位点序列突变与DNA甲基化的关系,结果发现3个位点序列突变与DNA甲基化程度不存在统计学差异(P>0.05,图3)。
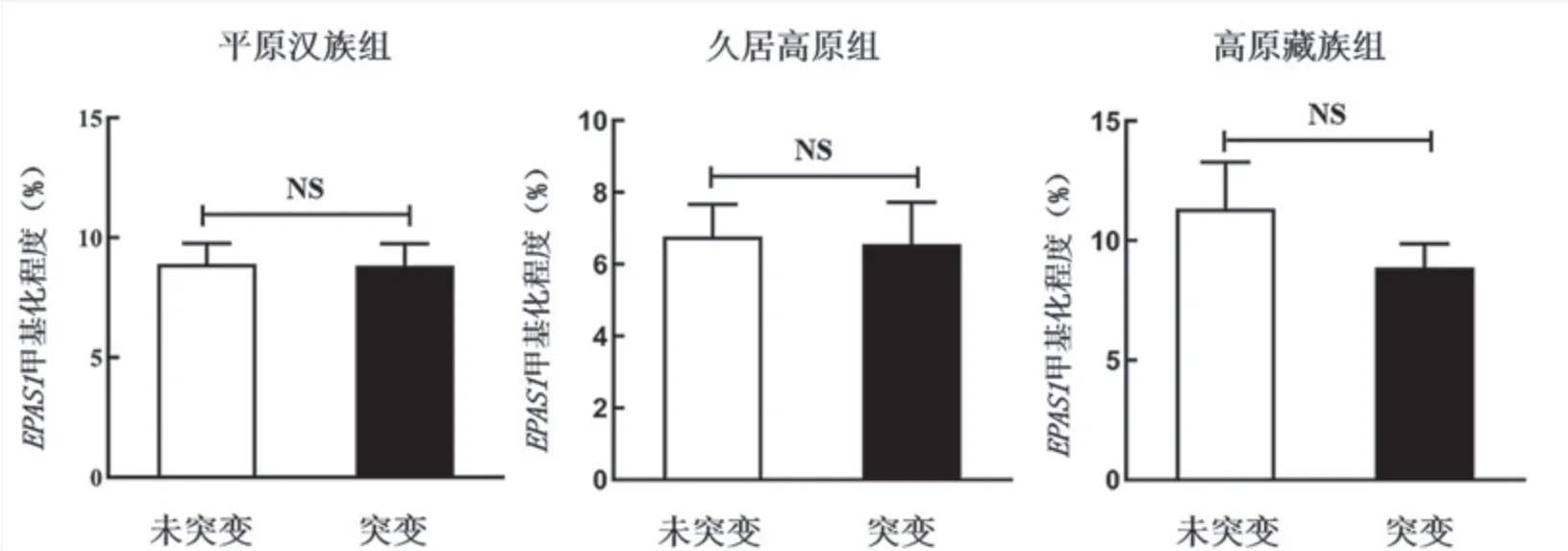
图3 3组人群中EPAS1基因序列突变与启动子区DNA甲基化的分析
讨 论
随着基因组学及表观基因组学的不断发展,人们对遗传学的认识和研究越来越深入,关于人类发育及肿瘤等疾病的调控机制除了包括基因序列变异等遗传学改变外,还包括DNA甲基化、组蛋白修饰等表观遗传学的作用。据报道,DNA甲基化与低氧诱导因子(HIF)共同参与高原低氧转录调节[4]。有研究发现低氧反应元件(HRE)与HIF-1结合位点存在多个CpG岛,若某些特殊CpG位点发生甲基化,则会抑制DNA与各类转录因子结合,提示HIF-1结合的HRE相关基因调控对DNA甲基化水平具有一定的敏感性[5]。近年已有多项研究发现,高原低氧诱导因子相关基因EPAS1、EGLN1等序列变异及mRNA表达与高原适应密切相关,但关于EPAS1基因启动子区DNA甲基化与高原低氧适应的研究较少[1,2]。因此,本研究对比了世居平原汉族、久居高原汉族及世居高原藏族3组中EPAS1基因DNA甲基化水平的差异,并分析EPAS1基因DNA甲基化程度对高原低氧适应的作用。
本研究采用Agena MassArray核酸质谱定量分析3组人群中EPAS1基因启动子区DNA甲基化差异,结果发现世居高原藏族、世居平原汉族及久居高原汉族中仅部分CpG位点甲基化程度存在统计学差异,启动子区整体甲基化程度无统计学差异,久居高原汉族低于平原汉族,与已有研究一致。如国外一项研究发现生活在安第斯山脉的高海拔(4 388米)人群EPAS1基因DNA甲基化水平低于生活在低海拔(0米)地区的同种人群,且甲基化程度与移居高原的居住年限呈负相关,因此研究者提出DNA甲基化对于高海拔地区的适应过程发挥重要作用[6]。
DNA甲基化特征在不同种族人群中存在差异[7]。本研究发现高原藏族人群的甲基化程度高于其他2组汉族人群,这与国外的研究不一致,但主要原因是国外的研究是在同一人群不同海拔高度。本文中高原藏族与平原汉族属于不同的人群,可能不同人群本身具有甲基化水平的差异,但是饮食、压力及运动等环境因素的作用引起了不一致结果。正如在埃塞俄比亚适应高海拔地区的遗传结构中发现,高原藏族特有的与高原适应相关基因序列变异在埃塞俄比亚高原适应中无明显相关,同时在埃塞俄比亚高海拔与低海拔人群中未发现HIF相关基因的DNA甲基化差异[8]。
国内一项关于高原低氧适应DNA甲基化谱的研究发现,世居高原藏族与移居平原藏族之间存在4 2 1个差异甲基化基因,排名前5位的差异甲基化基因包括5-羟色胺受体5B(HTR 5BP)、肌球蛋白1 C(M Y O 1 C)、富含亮氨酸的重复序列3 4(LRRC34)、含有144个家族的卷曲螺旋结构域N末端样(CCDC144NL)及核糖核酸酶H1假基因1(RNASEH1P1)等;移居高原汉族与平原汉族之间存在55个差异甲基化基因,排名前5的差异甲基化基因包括受体家族11亚家族H成员2(OR11H2)、HBS 1样翻译GTP酶(HBS 1L)、嗅觉受体家族4亚家族K成员15(OR4K15)及T细胞受体β常数2(TRBC2)。国内在高海拔及低海拔地区的藏族与汉族DNA甲基化谱的研究中未展示与低氧诱导因子相关基因的甲基化差异[9]。
国内外研究证实,世代生存在高海拔地区的人类在适应高原缺氧环境的过程中表观遗传因素发挥了一定作用,但机制仍不清楚[6,8-9]。多项研究发现,关于癌症形成机制中表观遗传因素具有重要作用,比如缺氧诱导因子HIF与表观遗传机制共同作用,对癌细胞缺氧做出积极反应[10-12];在结直肠癌的研究中发现,EPAS1基因是高甲基化的[13]。这些研究强调了DNA甲基化在介导细胞应对缺氧反应中发挥重要作用,这也支持人类可通过表观遗传机制适应高海拔缺氧环境的观点。本研究发现,久居高原汉族EPAS1基因启动子区DNA甲基化水平低于平原汉族,而mRNA表达量显著高于平原汉族,意味着平原汉族进入高原后可能通过DNA甲基化的改变影响EPAS1基因mRNA表达来适应高原低氧环境;而藏族EPAS1基因DNA甲基化水平高于汉族,但mRNA表达量仍高于平原汉族,这与汉族中结果不一致,藏族世代居住高海拔地区,其适应高原的机理更复杂,DNA甲基化以何种机制参与其中,仍需进一步研究。
综上所述,本研究发现久居高原汉族EPAS1基因DNA甲基化程度低于平原汉族,DNA甲基化程度低可促进EPAS1基因mRNA表达,结果表明汉族人群移居高原后可通过DNA甲基化的改变影响EPAS1基因表达以适应高原缺氧环境。高原藏族EPAS1基因DNA甲基化程度高于平原汉族及久居高原汉族,这与EPAS1基因mRNA表达水平不一致,可能跟不同人群及环境因素有关系,通过其他未知途径参与高原适应。根据本研究发现,下一步可进行同一人群不同海拔高度全基因组DNA甲基化研究,这些基因和通路在高原缺氧适应过程中可能会发生DNA甲基化变化。
利益冲突所有作者均声明不存在利益冲突
