基于瘤内异质性分割的软组织肿瘤MRI影像组学辅助诊断研究
2021-10-13周晓娅尚圣捷王颖妮董越刘冠宇罗娅红蒋西然
周晓娅,尚圣捷,王颖妮,董越,刘冠宇,罗娅红,蒋西然
1. 济宁医学院 基础医学院,山东 济宁 272067;2. 中国医科大学 智能医学学院,辽宁 沈阳 110122;3. 辽宁省肿瘤医院(中国医科大学肿瘤医院)医学影像科,辽宁 沈阳 110042
引言
软组织肿瘤起源于人体结缔和滑膜等软组织,在人体内分布广泛,其中四肢部位发病最为常见。恶性软组织肿瘤的发病率较低,仅占成人恶性肿瘤的1%[1],但患者死亡率却高达20%~30%,2019年美国新增软组织肿瘤患者中,死亡率约40%[2]。因此,对软组织肿瘤进行早期临床诊断和制定精准治疗方案对患者的康复和预后至关重要[3]。
磁共振成像(Magnetic Resonance Imaging,MRI)技术利用静磁场和射频磁场获得人体组织影像,具有软组织分辨率高及多方位成像的优点,是当前软组织肿瘤检测的主要方法之一[4],已成为临床上对软组织瘤进行术前评估的重要影像学工具[5]。然而,由于软组织瘤具有明显异质性并存在多种亚型,在临床诊断中容易发生误诊或漏诊,影响治疗时机[6]。因此,当前亟需一种针对MRI影像的软组织肿瘤精准辅助分析方法。近年来,影像组学技术发展迅速,通过从临床影像中高通量获取高维度影像特征信息,筛选有诊断价值的定量化特征建立相应计算机预测模型[7],能够对多种肿瘤疾病进行有效评估和辅助诊断[8-11]。然而,当前针对软组织肿瘤的计算机影像组学分析研究较少,可能是由于软组织瘤发病率较低,因此数据收集存在困难。前期Corino等[12]针对软组织肿瘤PET/CT影像初步建立了计算机分析模型,虽然该研究只纳入了19例患者,缺乏临床可靠性,但该研究提示我们计算机辅助分析方法对软组织肿瘤具有一定的辅助诊断潜力。Wang等[13]则进一步通过影像组学分析提取出了软组织肿瘤MRI影像中有价值的特征信息建立预测模型,表明影像组学特征对肿瘤的术前分级有一定的预测价值。然而,由于软组织肿瘤本身存在明显的肿瘤异质性,肿瘤细胞在瘤内聚集分布不均匀,对影像组学模型的建立和效果产生较大影响[14]。因此,本研究中,我们针对软组织瘤内异质性的问题进行了有针对性的探索,分析了不同瘤内亚区及肿瘤完整区域的影像组学预测价值,并建立双序列MRI融合辅助诊断模型。
1 材料与方法
1.1 患者情况
本研究经中国医科大学辽宁省肿瘤医院伦理委员会批准,收集了2017年6月至2019年4月在医院就诊的75例软组织肿瘤患者的MRI影像数据。患者信息如表1所示。数据研究标准为:① 患者术前进行CE-T1和T1WI MRI扫描;② 经病理学确诊为软组织肿瘤。排除标准为:① 患者除软组织肿瘤以外还患有其他肿瘤疾病;② MRI数据采集前有过放化疗史;③ MRI数据不全或影像模糊。全部患者按照2:1随机分为训练集和测试集。

表1 本研究纳入患者的临床信息
1.2 MRI影像扫描
软组织肿瘤患者MRI影像通过Verio 3.0 Tesla 扫描仪(Siemens,德国)扫描获得。MRI扫描参数为Tra T1WI(TR/TE=514.0 ms/14.0 ms),Tra enhanced T1WI(TR/TE=514.0 ms/14.0 ms);层厚:5.0 mm;视野:350.0 mm×250.0 mm;矩阵:360×448。以3.0 mL/s速度静脉注射剂量0.1 mmol/kg造影剂钆喷酸葡胺。扫描所得MRI影像数据采用DICOM图片格式储存于辽宁肿瘤医院PACS。
1.3 肿瘤区域勾画及瘤内亚区分割
由具有10年工作经验的影像科医生使用ITK-SNAP v3.6将收集的MRI影像数据勾画出软组织肿瘤病灶区域(Region of Interest,ROI),包括肿瘤及其边缘区域,并输出保存为NII格式。在手工分割软组织肿瘤病灶过程中,如遇肿瘤边缘不清晰等难以确定的病例时,会协同骨软科及影像科专家共同会诊后决定分割区域。医生分割肿瘤病灶区域时不知晓患者的病理诊断结果。在每个MRI序列影像ROI区域内,采用无监督k-means聚类算法,依据轮廓系数参数来选取最优k值,从而将ROI分割为两个子区域(高亮度亚区和低亮度亚区),采用欧氏距离作为距离度量,两个对象的距离越近,其相似度就越大,从而聚类具有相似成像模式的肿瘤体素。
1.4 影像组学特征提取及筛选
通过Python v3.6软件编程,使用开源Pyradiomics包( 网 址 :https://pyradiomics.readthedocs.io) 对 MRI影像组学特征进行提取,包括一阶统计量特征、形态学特征、纹理特征和滤波特征。提取得到的特征通过R语言glmnet包运行最大相关最小冗余(Max-Relevance and Min-Redundancy,mRMR)算法对特征进行相关性排序筛选[15],通过Wilcoxon秩和检验方法计算特征P值。
1.5 影像组学模型建立及验证
通过Matlab(2016a版本)编程建立k-最近邻(K-Nearest Neighbor,KNN)机器学习分类器模型,为评估所构建计算机模型鉴别软组织肿瘤良恶性的能力,通过Matlab编程绘制工作特性曲线(Receiver Operating Characteristic,ROC),由最大约登指数计算得到ROC曲线的最佳阈值[16],计算ROC曲线下面积(Area Under Curve,AUC)、准确率(Accuracy,ACC)、特异性(Specificity,SPE)和敏感性(Sensitivity,SEN)作为模型衡量指标。使用R语言v3.6 RMS软件包绘制诺莫图模型。全部算法运行的计算机平台硬件配置为:3.7 GHz Intel i7-8700K CPU 和 64 GB 3000 MHz DDR4 RAM。
2 结果
图1所示为本研究采集的软组织肿瘤患者双序列MRI影像及医生手工勾画的ROI,图中黄色线条表示影像科医生沿着软组织肿瘤区域的边缘进行手工勾画的痕迹。

图1 本研究使用的软组织瘤MRI影像及肿瘤ROI勾画
2.1 瘤内亚区分割
为了探讨软组织肿瘤内部异质性区域的预测价值,本文采用k-means无监督聚类方法进行瘤内分割,通过枚举,令k从2到9,为避免局部最优解,在每个k值上重复运行数次k-means,并同时计算k的平均轮廓系数,最后选取轮廓系数最大的值对应的k为2作为最终的集群数目(图2)。
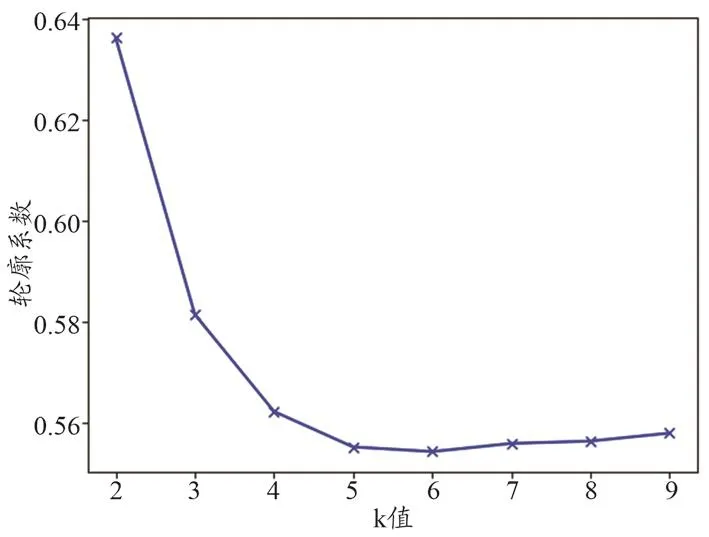
图2 对75例软组织肿瘤患者的进行k-means瘤内分割时,不同的k取值对应的轮廓系数的值
研究对T1WI和CE-T1序列的整体ROI、低亮度亚区和高亮度亚区分别进行了影像特征提取和分析。图3所示为对软组织瘤 T1-CE MRI(图 3a~b)和 T1WI MRI(图3c~d)进行瘤内分割的结果。表2列出了针对软组织肿瘤完整肿瘤区域以及肿瘤内亚区分别建立的KNN模型预测结果,并相应地绘制出如图4所示的基于训练集和测试集的ROC曲线。可见,T1WI序列和CE-T1序列的高亮度亚区的预测效果都比较好,AUC分别达到0.753(SEN=0.533,SPE=0.800)和 0.818(SEN=0.667,SPE=0.867)。

图3 瘤内亚区分割的结果示例

图4 KNN分类器对CE-T1和T1WI MRI影像绘制的ROC曲线

表2 肿瘤完整区域及瘤内分割亚区预测结果
2.2 MRI影像组学特征分析
本研究通过对CE-T1序列和T1WI序列中肿瘤区域及瘤内高亮度亚区分别提取影像组学特征并筛选后,最终获得5个诊断价值最高的特征,其中2个属于Exponential特征,3个属于Log特征。表3列出了各特征的单独预测AUC值及P值。

表3 CE-T1和T1WI MRI序列中高亮度亚区内筛选获得的影像组学特征
2.3 诺模图模型的构建及验证
图5a所示为本研究使用筛选得到的5个影像组学特征绘制的诺模图风险预测模型。软组织肿瘤患者的恶性风险的计算方法为:由患者T1增强和T1平扫MRI序列影像计算得到的分数在模型总分数轴上的对应位置竖直向下做一条直线并与总风险轴相交,从而获得总风险值。图5b和图5c表明,模型的校正曲线在训练集和测试集上均贴近经过原点的45°斜线,表明本研究建立的模型预测结果与真实结果具有良好的一致性。图5d和图5e为模型在训练集和测试集上的ROC曲线,预测AUC值分别为 0.865(SPE=0.763,SEN=0.763) 和 0.856(SPE=0.867,SEN=0.800),表明模型具有较低的误诊率和漏诊率。

图5 用于软组织瘤良恶性鉴别的诺模图模型的建立及验证
图6所示为本研究建立模型的决策曲线分析结果,横轴和纵轴分别为模型的阈值概率和净获益率,图中黑色直线为假定软组织肿瘤患者均患有良性瘤,蓝色曲线为假定全部患者均患有恶性瘤,本研究所构建模型对应为红色曲线。当阈值概率在0.10~0.86时,本研究建立的模型对软组织肿瘤患者有良好获益。

图6 本研究建立影像组学模型的DCA结果
3 讨论
随着医学影像人工智能技术的快速发展,对肿瘤疾病影像建立计算机分析方法以辅助医生进行临床决策已经成为智能医学领域新的发展趋势[17]。本研究针对软组织肿瘤异质性问题,进行了基于瘤内亚区分割的影像组学分析。本研究从T1CE 和T1WI MRI的整体ROI以及瘤内高亮度和低亮度亚区分别提取影像组学特征建立预测模型,结果表明在两种MRI序列影像中,瘤内高亮度亚区均具有最高的诊断效果(表2)。最终筛选得到的5个影像组学特征中,有3个来自CE-T1序列,2个来自T1WI序列。本研究筛选出的特征都属于滤波后的高维特征,这可以部分解释临床医生通过肉眼观察CE-T1和T1W TMRI影像对软组织肿瘤进行诊断中存在较高的恶性瘤漏诊率[6],这是因为模型筛选到的诊断能力强的特征均存在于高维度空间,人眼难以对其进行识别和洞察。本研究获得的5个特征中,有3个属于纹理特征类,这与前期发表的软组织肿瘤影像学分析报道具有一致性。Xu等[18]和Vallières等[19]分别在软组织肿瘤FDG-PET影像中发现纹理特征具有较高的诊断价值。由于纹理特征往往体现肿瘤细胞的异质性分布特性,因此本研究结果提示我们肿瘤异质性是反应软组织肿瘤自身特性的重要指标,表明软组织肿瘤MRI影像中的瘤内高亮度区域内包含更多的潜在诊断信息,值得引起相关研究者的关注。本研究获得的融合模型对软组织瘤良恶性的鉴别效果明显优于近年发表的两篇前期报道,可能的原因是本研究采用T1增强和T1平扫序列进行特征级融合建模,能够获得来自增强和平扫两个序列的互补信息。此外,本研究纳入的患者数量相对较多,提高了影像组学模型的可靠性。而前期研究报道仅使用了单一MRI序列,且患者数量太少,分别仅纳入了19例[12]和35例[7]患者。
为了进一步建立有临床应用价值的辅助诊断模型,本研究建立了近年使用较为广泛的可视化诺模图模型[20]。基于CE-T1和T1WI序列MRI影像特征构建的融合诺模图模型对软组织肿瘤进行预测,测试集AUC=0.856,可表明该模型具有较高的诊断能力。校正曲线结果表明诺模图模型的预测结果与真实值接近(图4),模型的可靠性较好。决策曲线分析表明,对于医生或患者,使用本研究构建的诺模图模型方法来鉴别恶性软组织瘤在概率阈值为0.10~0.86时会显著获益,这说明诺模图模型具有良好的潜在临床应用价值。
本研究存在一定的局限性:① 由于软组织肿瘤属于罕见病,发病率较低,因此本研究纳入的患者数量相对较少,我们在今后的研究中会通过与多家医疗机构共同合作,扩大数据量,纳入更多MRI序列并对比不同序列的诊断价值,验证模型的鲁棒性;② 本研究纳入的软组织瘤患者样本中,良性和恶性瘤患者比例不均衡,可能存在一定的偏倚性,虽然已使用smote方法进行均衡,数据分布的不平衡性仍不能完全消除,可能会影响所构建模型的灵敏度和特异性;③ 由于本院软组织瘤患者临床指标特别是血清学标志物检测不全面,因此本研究没有纳入临床指标;④ 本研究对肿瘤ROI的勾画为医生手工操作,较为费时耗力,今后应引入深度学习自动分割技术,以减少勾画工作量;⑤ 由于目前条件有限,本研究后期会根据影像常规检查与病理诊断结果计算医生的漏诊率和误诊率,更好地与影像组学模型进行对比说明。
综上,本研究对软组织瘤患者的T1增强和T1平扫MRI序列中的肿瘤区域及瘤内高亮度和低亮度亚区的影像组学预测价值进行了对比分析,并建立了融合诺莫图模型。结果表明基于瘤内区域分割的影像组学融合分析方法适合于辅助医师对软组织肿瘤进行良恶性鉴别,具有良好的潜在临床应用价值。
