区域生长全卷积神经网络交互分割肝脏CT图像
2021-10-10张丽娟李东明王晓坤
张丽娟, 章 润, 李东明, 李 阳, 王晓坤
(1. 长春工业大学 计算机科学与工程学院,吉林 长春 130012;2. 吉林农业大学 信息技术学院,吉林 长春 130118;3. 空军航空大学 航空作战勤务学院,吉林 长春 130022)
1 引 言
CT是无侵害性的器官体外成像手段,由于其成像速度较快、分辨力较高、效果较好,已经成为临床医生进行医疗诊断不可或缺的重要手段。在肝脏疾病的诊断中,可视化技术与医学图像分析占有主导地位。通过对肝脏CT图像进行分割,提取出肝脏组织并获得相应的特征信息,医生可以很直观地了解患者肝脏内部的详细情况,对诊断及下一步治疗计划的制定起到关键作用。
半自动分割方法[1]使用浅层图像特征和用户交互,以边界框或涂鸦的形式进行交互以执行分割。早期的区域相似性分割[2]、图切割[3]、归一化切割[4]、测地线[5]和其他基于图形的随机游走[6]已应用于2D分割,在某些情况下还应用于3D分割。当要分割的区域相当均匀并且可以通过低层次特征(例如边缘、相邻像素之间的强度差和其他局部信息)准确捕获其特征时,半自动方法可以提供良好的分割效果;但在更复杂的环境中,由于浅层特征被限制,这些方法需要大量的用户交互才能提供足够质量的细分结果。最近,具有编码-解码架构的全卷积神经网络(FCN)[7]在全自动分割领域获得最优秀结果,能有效完成2D[8-10]和3D[11-12]分割任务。尽管已证明这些方法及其后续方法[13-15]可以在公开的数据集上提供最优秀的性能,但它们并未提供用户交互的手段。这方面限制了其在临床环境中的适用性,因为在临床环境中,精度要比速度更优先考虑。
计算机视觉界最近提出各种新颖的方法:将深度学习方法加上用户交互形成半自动分割。基于深度学习的半自动分割方法可以提供准确的结果,同时为用户提供了一种自然的交互方式。全卷积神经网络不再像以前的方法那样依赖于浅层特征,而是能够捕捉更复杂的模式,并学习“对象性”的概念,从而获得更好的结果。Xu[16]等人将用户提供的正点击和负点击转换为两个欧几里得距离图,然后将其与图像的RGB通道连接起来构成输入数据训练FCN网络,通过组合几种随机采样策略来模拟用户的点击模式。由Wang[17]等人提出的DeepIGeoS使用深度学习并将用户涂鸦的测地距离变换图像作为额外的FCNN通道来进行交互式分割。然而,对于看不见的对象类,分割性能可能会恶化。Can[18]等人提出另一种基于深度学习的方法,基于初始涂鸦使用随机游走[6]方法得到初始分割图像,来监督全卷积神经网络,将注释所需时间大幅缩短。Sakinis[19]等人提出一种基于深度学习的半自动分割方法通过前景和背景点击获得高斯图,作为额外的通道进行交互式分割,并可对分割结果进行调整,对分割有差错的地方点击交互以提高分割精度。本文提出一种基于一维卷积神经网络改进的区域生长法,使用点击交互产生初始分割,然后将其与图像的灰度通道连接起来构成图像/初始分割对。相比较其他方法的交互信息,将点击交互产生的初始分割作为交互信息更为精确,以此获得更好的性能。
2 算法流程
区域生长法(Region Growing)是从一组代表不同生长区域的种子像素开始,将种子像素邻域里符合生长准则的像素合并到种子像素所代表的生长区域中,并将新添加的像素作为新的种子像素继续合并,直到找不到符合条件的新像素为止。本文对该方法的生长准则进行了改进,并只需选取一两个生长种子点即可完成分割,算法的流程如图1所示。首先对腹部CT图像序列集进行预处理,只提取含有肝脏的切片;采用Window-Leveling算法将原始CT图像转换为灰度图像,突出肝脏区域;通过直方图均衡化处理增加对比度,进行中值滤波算子卷积计算,平滑图像,抑制噪声。然后计算像素在不同算子下的梯度值,将其作为该像素的特征,形成像素特征向量,训练神经网络,该网络以一对像素特征向量为输入,以两像素的关联度系数为输出。为每一对像素特征向量设置标签,如果两像素来自同一区域,同为肝脏内区域或同为肝脏外10像素距离区域,标记为1,其他标记为0。最后将训练好的神经网络模型作为区域生长算法的生长准则,手动选取一种子点产生分割结果。

图1 改进区域生长算法流程Fig.1 Algorithm steps
通过上述流程,得到改进后的区域生长法, 在图像肝脏区域用鼠标选取一点作为交互信息得到初步分割结果。在训练区域生长全卷积神经网络时,通过求取肝脏区域的中心位置来模拟用户交互,得到区域生长法分割结果;然后将分割结果与图像的灰度通道连接起来构成图像/初始分割对,一同输入U-net[8]网络中,将该网络称为区域生长全卷积网络(Region-growing Fully Convolutional Neural Network,Rg-FCNN),如图2所示。
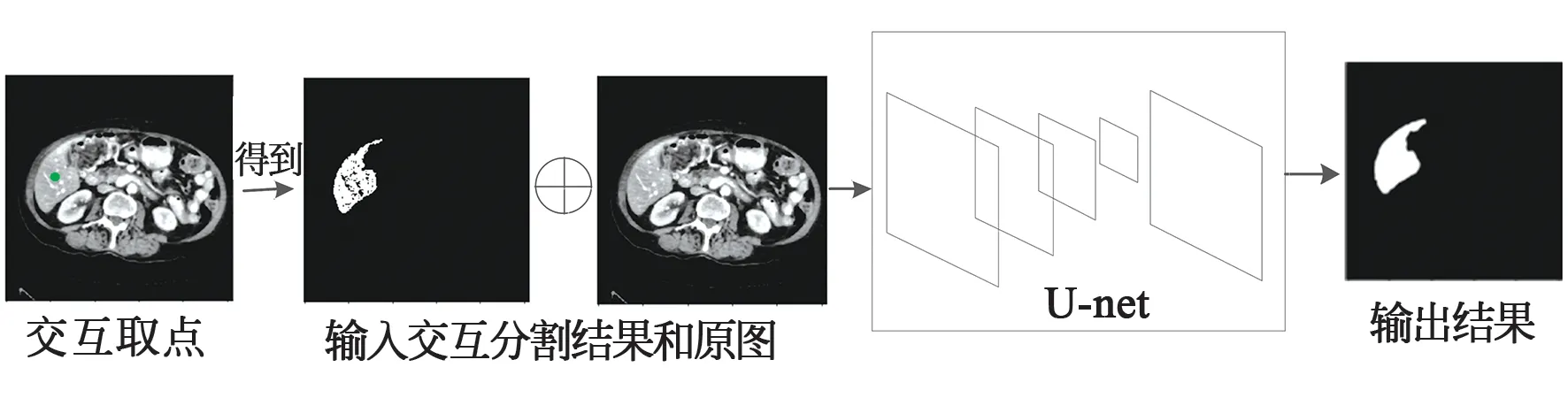
图2 区域生长全卷积神经网络Fig.2 Region-growing fully convolutional neural network
3 图像预处理
3.1 切片提取
实验所用数据是BCV腹部挑战数据集,是由大肠癌化疗试验和一项回顾性腹疝研究中随机选择30例腹部CT扫描组成,包括原始CT图像和对应的分割标签。标签数据由两名经验丰富的专业人员手工分割标记,并由放射科医生使用MIPAV软件进行体积验证。数据为NIFTI格式的腹部CT图像序列集,切片层数范围从85~198,切片厚度范围为2.5~5.0 mm,每层切片大小是512×512,每一个像素包含一个12位的CT值,单位为HU(Hounsfield Unit)。
本文腹部CT图像序列集共有13个腹部器官,只需要提取含有肝脏的切片。在提取切片时,利用的是训练集中的分割标签图像。在标签图像中,专业人员已经将13个腹部器官与数字一一对应,其中肝脏对应的数字为6。由于CT成像原理,肝脏切片的序列号是连续的,中间不会出现间隔。因此只要在标签图像序列集中查找最早出现数字6的序列号start和最后出现的序列号end,即可完成切片提取。start序号后几张和end序号前几张,肝脏区域可能会比较小,可根据需要适当调整序号区间,例如[start+5:end-5]。
3.2 图像预处理
提取的腹部CT图像包含了肝脏、胃、脾等多个器官,且各器官的边缘模糊,如果强制将图像灰度化,则图像的分辨度不高,采用图像增强方法,很难突出所要研究的关键部位。在CT图像中,不同器官、组织的密度是在一个固定的值域,提取肝脏区域,只需将肝脏的CT值区间映射到灰度区间即可。本文采用Window-Leveling(W/L)窗口算法[20],实现肝脏CT图像到灰度图像的转换。在W/L窗口算法中,假设图中某像素点的CT值为f(x),将需要处理的CT值区间一般称为窗口,区间长度称为窗口宽度(Window Width,WW),区间的中心称为窗口窗位(Window level,WL),则经过窗宽窗位预处理后该像素点的值g(i)为:
(1)
其中:min=WL-0.5 WW,max=WL+0.5 WW,肝脏组织的CT值通常位于50~250之间,取WW=200,WL=150,将大小在50~250之间的CT值映射到[0,255]的灰度值区间,小于50映射为0,大于250映射为255,得到CT图像的灰度图。对转换后的图像进行直方图均衡化来增加对比度,通过中值滤波消除孤立的噪声点。
图3是对图像进行预处理的实例,其中图3(a)是原始的CT切片图像,图3(b)是CT图像转化后的灰度图像,图3(c)是最终预处理后图像。

图3 CT图像预处理Fig.3 CT image preprocessing
4 改进区域生长法
4.1 训练数据的提取
由于人体腹部CT图像纹理复杂,结构间对比度小,边缘不明显。本文选取了多种微分算子对图像进行边缘检测,得到每个像素对应的算子处理后的梯度值作为该像素的特征,形成像素特征向量。在宏观上,进行多种边缘检测是为了综合考虑各种算子处理结果,消除噪声及局部因素的影响,提高肝脏边界的准确性。结合区域生长算法的特性,肝脏边界一旦确定,肝脏区域内的种子点生长就会被限制在边界内,从而完成肝脏分割。在微观像素上,通过多种边缘检测算子处理增加了像素的特征,在判断待测点是否合并到种子像素所代表的生长区域中时,除了像素值,有更多的特征维度可以去比较。图4是多种算子进行边缘检测实例。其中图4(a)是边缘检测对象,图4(b)是进行Sobel算子滤波后结果,图4(c)是进行Robert算子滤波后结果,图4(d)是进行Canny算子滤波后结果。

图4 各算子边缘检测Fig.4 Edge detection of each operator
将各微分算子处理后的结果图像作为原图像通道数叠加到一起,则对于图像上每个位置上的像素点而言,不再只是单通道灰度图像的灰度值,而是一个一维向量,称为像素特征向量:
f=[α1,α2,α3...α8],
(2)
向量的长度为图像叠加后的通道数目,α1为原图的灰度值,后面的值分别对应各算子滤波后结果。本文使用神经网络作为区域生长法的生长规则,不同于传统的AlexNet[21]、VGGNet[22]图像分类网络,预测输入的图像为哪一类,本文的神经网络用于判别输入的一对像素是否为一类,称该网络为区域生长神经网络(Region-growing Neural Network, Rg-net)。因此在准备训练数据阶段,需要随机选取一对像素点进行组合配对,并同时标注标签,1表示是同一类,0表示不是同一类。首先限定取值区域:利用原图对应的肝脏分割标签,求肝脏边界向外10像素曼哈顿距离(Manhattan Distance)以内区域:
disf(p(x1,y1),P(x2,x2))=
|x1-x2|+|y1-y2|<10,
(3)
区域包含两部分:肝脏内部区域和肝脏外10像素距离区域。在区域内任意选取两像素组合配对,形成神经网络的一个输入样本X:
Xi=[f1,f2],
(4)
对应的输出标签Y:
(5)
4.2 区域生长神经网络
区域生长神经网络(Rg-net)架构如图5所示,类似于卷积神经网络,先进行卷积层,然后通过flatten层将二维输入一维化,过渡到全连接层,最后通过sigmoid激活函数输出常数概率值。但是网络的输入有所不同, 因此本文对卷积层进行了改动,使用的是一维卷积。一维卷积层也叫时域卷积,常用于在一维输入信号上进行邻域滤波。对输入8*2的向量进行1*1核长的一维卷积相当于对其进行卷积核为1*2的二维卷积。卷积核步长为1,即每次卷积,卷积核都对应向量的一整行,相邻行之间互相独立,不进行交叉合并。并通过增加卷积核的数目来提升输出向量的维度,增加解的空间。
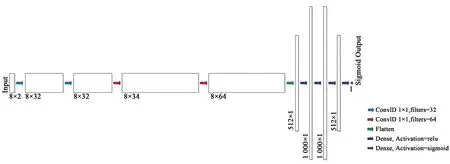
图5 区域生长神经网络Fig.5 Region-growing neural network

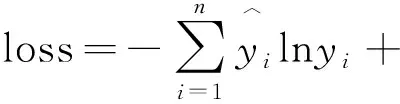
(6)
图像分割前,要先进行多种微分算子边缘检测,方便提取像素特征向量。在使用区域生长法判断种子像素邻域里的像素是否合并到种子像素所代表的生长区域中时,将种子像素和待测像素的像素特征向量输入Rg-net网络,得出两像素关联度。在判断时,还要设置一个阈值T:输出大于阈值T时,待测点符合生长准则,合并到种子像素所代表的生长区域中;小于阈值T时,不符合生长准则,不合并。图6是本文算法在不同阈值T下初分割结果。图6(a)为待分割图像,随着阈值T减小,被合并到生长区域的像素就越多,肝脏区域内空洞逐渐减少,如图6(b~d)所示。但当阈值过低时,区域生长趋势会突破肝脏边界,如图6(e~f)所示。为了确保算法分割的稳定性和统一性,本文阈值T统一选取为0.95。
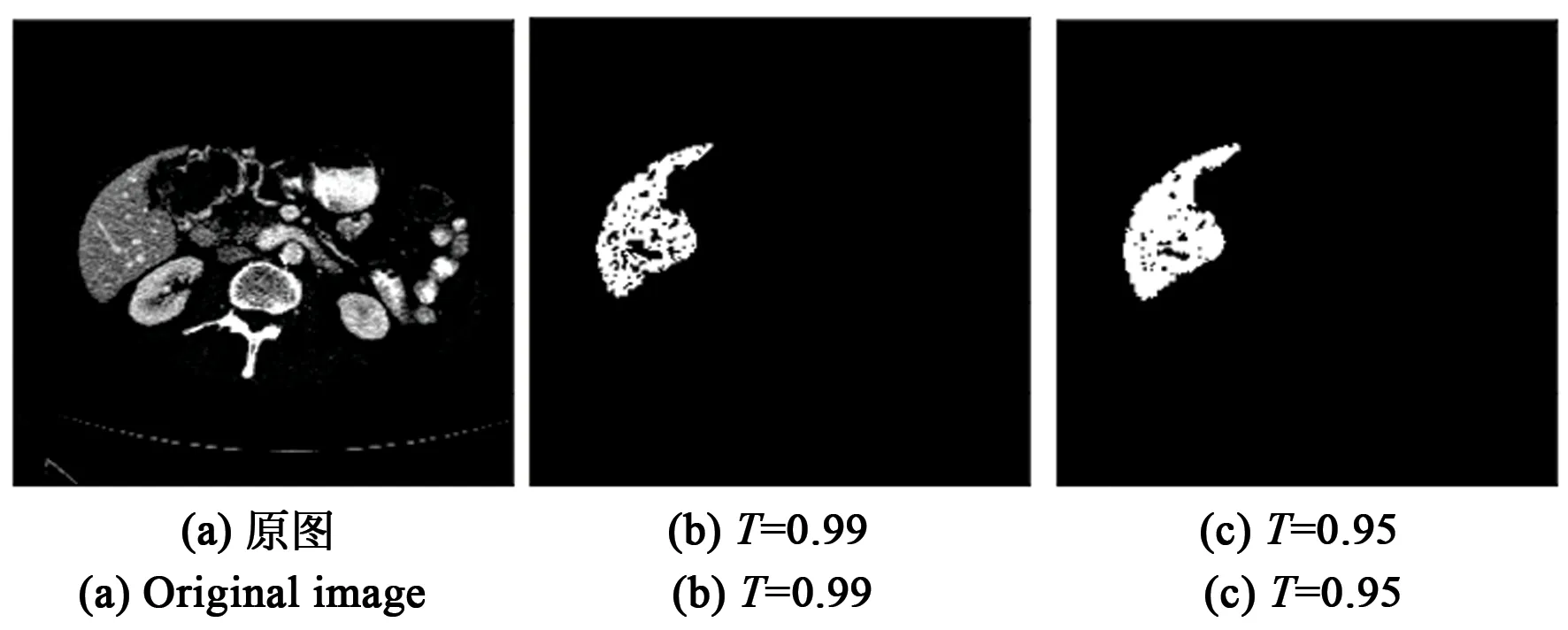
图6 不同阈值T初分割情况Fig.6 Image segmentation results with different thresholds
5 区域生长全卷积神经网络
本文的区域生长全卷积神经网络(Rg-FCNN)以U-net网络模型为基础,通过最大池化层进行下采样,每经过一个池化层就是一个尺度,包括原图共有5个尺度。通过卷积层提取特征,使用same卷积,保持卷积前后图像尺寸不变。通过双线性插值进行上采样,每上采样一次,就和特征提取部分对应的尺度进行融合。残差网络[23]是一类特殊的卷积网络,它通过残差连接而解决了梯度反传问题,即当神经网络层级非常深时,梯度仍然能有效传回输入端。图7为本文中残差模块的结构,残差块的输出结合了输入信息与内部卷积运算的输出信息,这种残差连接保证深度模型至少不低于浅层网络的准确度。
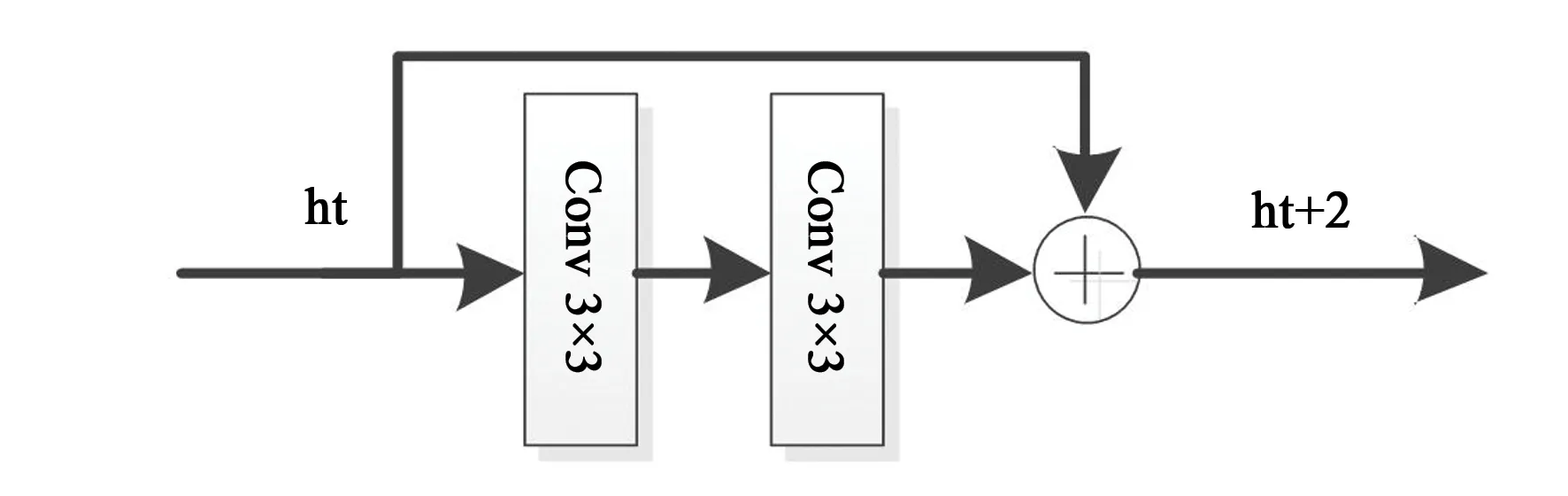
图7 残差块结构Fig.7 Structure of residual block
在下采样卷积层中分别加入不同数目的残差模块,如图8所示。网络输出层使用Sigmoid激活函数,
(7)
Sigmoid函数将输出值z映射到[0,1],表示概率分布的有效实数空间。
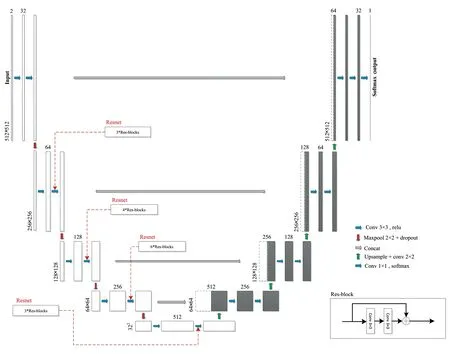
图8 区域生长全卷积神经网络架构Fig.8 Region-growing fully convolutional neural network architecture
从30例CT序列集中提取500张切片,并对所有的切片使用上节提出的改进区域生长法进行交互式分割。由于数据量大,人工手动交互是不可行的。利用肝脏CT图像对应的分割标签,通过求取分割标签里肝脏区域的中心位置来模拟用户交互,比较符合人们在交互选点时选择中心位置的习惯。同时为避免中心位置像素点是极端点情况,将中心像素8邻域内像素均命为种子像素。得到初始分割结果,并和原图叠加在一起作为Rg-FCNN网络的输入数据,随机打乱顺序后,按照3∶1∶1的比例分为训练集、验证集和测试集。
选择合适的训练次数,避免因为训练次数太多而导致过拟合,训练次数太少而欠拟合。将训练集和验证集输入网络中训练200次,记录训练过程中每轮(epoch)的训练损失值和精度,以及验证集损失和精度,如图9、图10所示。观察图9、

图9 训练和验证损失值Fig.9 Trainin and validation loss
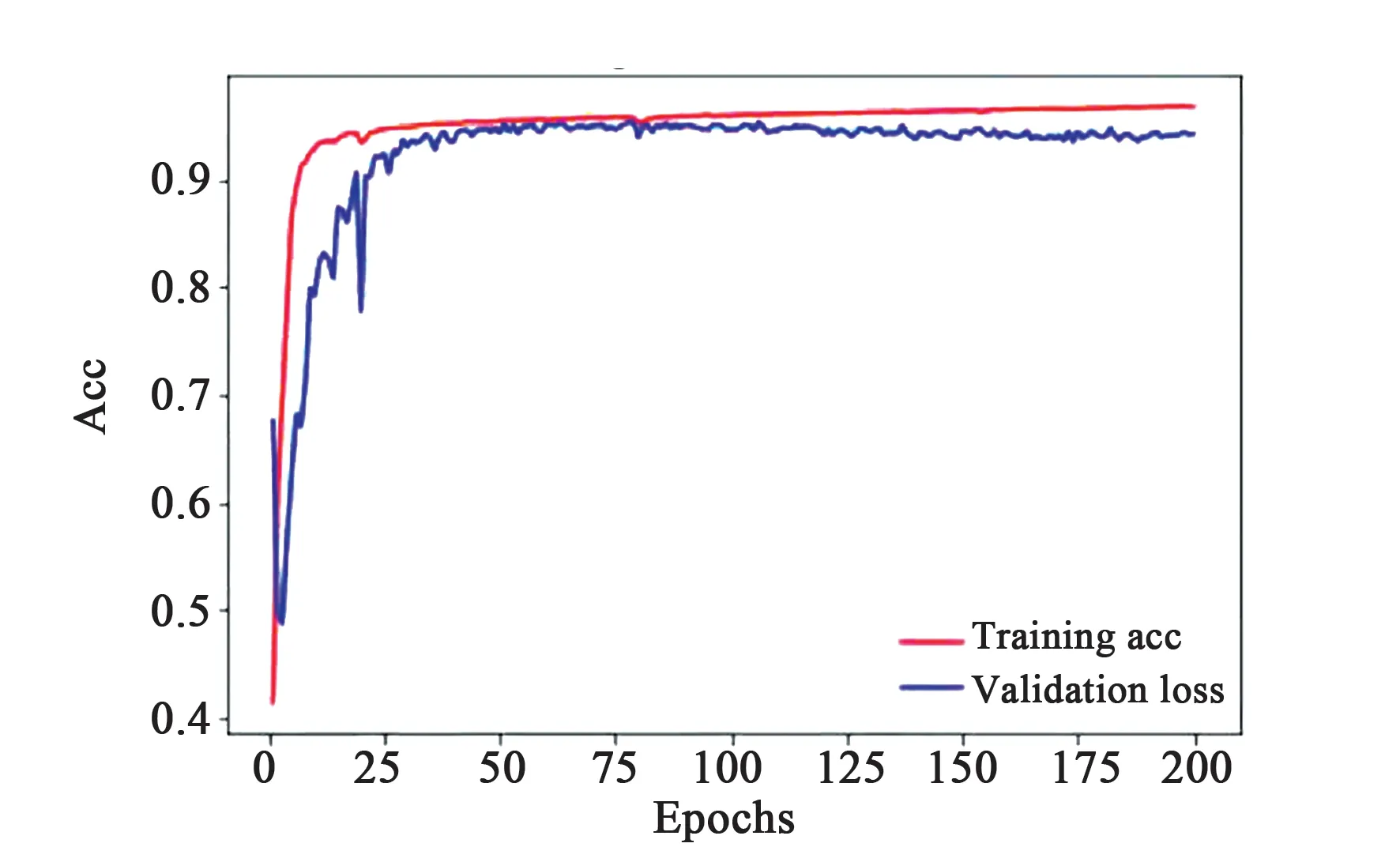
图10 训练和验证的评估值Fig.10 Training and validation acc
图10,epochs从0到75,训练集和验证集随着训练次数增加,损失值减小,精度提高;epochs从75到200,训练集损失值逐渐减小,精度提高,而验证集逐渐增加,精度降低,网络过拟合。因此选择训练次数为75的效果最好。
6 实验结果与分析
6.1 实验结果
本文实验环境:Ubuntu16.04 LTS 64位操作系统,CPU Intel Xeon E5 64核心,GPU NVIDIA GeForce GTX 1080 Ti 11G * 4,基于深度学习Keras框架搭载U-ode网络模型,Keras后端使用tensorflow,CUDA 8.0 GPU运算平台及cuDNN7.5深度学习GPU加速库,完成区域生长全卷积神经网络的训练和测试。
图11是使用本文改进的区域生长算法和Rg-FCNN进行肝脏分割的3个案例,其中图11(a)是经过预处理后的肝脏图像,图中绿点由鼠标点击形成,作为种子像素点;图11(b)是肝脏最终分割结果的二值图像;图11(c)是使用改进的区域生长算法得到的分割结果;图11(d)是图11(a)和图11(c)通道连接构成图像/初始分割对,输入Rg-FCNN网络得到的分割结果。

图11 实验结果。(a)预处理后的肝脏图像;(b)肝脏最终分割结果的二值图像;(c)改进的区域生长算法得到的分割结果;(d)图(a)和图(c)通过连接构成图像/初始分割对输入Rg-FCNN网络得到的分割结果。Fig.11 Experimental results. (a)Preprocessed liver images; (b) Binary images of the find segmentation result of liver; (c) Segmentation results by the improved region growth algorithm; (d) Channels of Fig.(a) and Fig.(c) are connected to form an image/ initial segmentation pair, the segmentation results are obtained by inputting the Rg-FCNN network.
6.2 对比分析
为了进一步对本文区域生长全卷积网络算法进行验证,将本文算法和U-net[8]网络、Deep Interactive[16]算法的实验分割结果进行比较分析,其中U-net网络架构使用的是本文加入残差块后的U-net网络模型。图12是各算法进行肝脏分割的4个实例,其中图12(a)是经过预处理后的肝脏图像,图12(b)是肝脏图像对应的分割标签;图12(c)是U-net网络分割结果,图12(d)是Deep Interactive网络分割结果,图12(e)是本文Rg-FCNN网络分割结果。

图12 不同算法实验结果。(a)预处理后的肝脏图像;(b)肝脏图像对应的分割标签;(c)U-net网络分割结果;(d)Deep lnterative 网络分割结果;(e)本文Rg-FCNN分割结果。Fig.12 Experimental results of different algorithms. (a)Preprocessed liver images; (b)Corresponding segmentation labels of the liver images; (c) Segmentation vosnlts of U-net network; (d) Segmentation results of Deep Interactive network; (e) Segmentation resnlts of Rg-FCNN network of this paper.
观察U-net网络分割结果,在肝脏区域外有白色小块,明显不属于肝脏区域,出现了过分割,这主要是由于腹部CT图像内部结构对比度低,患者之间差异性大。而Deep Interactive网络和本文网络加入了交互信息,使网络更容易定位到肝脏区域的位置。二者的分割结果均有效完成了肝脏分割,与分割标签基本吻合。
为了进一步对比各网络对肝脏图像分割效果,对分割结果进行定量分析。目前,已经有许多评估方法被应用于医学图像的分割中。但是,由于在肝脏的分割研究中没有统一的、标准的可用数据集,使得肝脏的分割并没有统一的评测标准。当前比较常用的评估分割结果的方法是差异实验法,即将计算机分割结果与正确的分割结果相比较。本文将通过Dice系数(Dice Similarity Coefficient)、像素准确率(Pixel Accuracy,PA)、类别平均像素准确率(Mean Pixel Accuracy,MPA)、平均交并比(Mean Intersection over Union,MIoU),4个指标来评估分割结果。其计算都是建立在混淆矩阵(Confusion Matrix)的基础上,混淆矩阵如表1。

表1 混淆矩阵Tab.1 Confusion matrix
Dice系数常被用来评价分割结果的准确性,取值范围在0~1之间,表示计算机分割结果与真实结果之间的重合程度,Dice= 0说明两分割结果完全没有重叠,分割效果最差;Dice=1则说明两个集合完全一致,分割效果最好。
(8)
像素准确率指被正确预测的肝脏像素和被正确预测的非肝脏像素在全部像素中所占的比例,即对角线预测的全部为正确值,公式为:
(9)
平均像素准确率是肝脏像素准确率和非肝脏像素准确率的平均值,对于二分类问题N=2,公式为:
(10)
交并比是语义分割的标准度量参数,是两个集合的交集和并集的比值,平均交并比是交并比的一种升级形式,是肝脏与非肝脏交并比的平均值。公式为:
(11)
表2为各算法对测试集100张图片肝脏分割结果平均指标,图13、14为实验结果Dice、PA指标箱型图。

表2 肝脏分割结果指标数据Tab.2 Index data of liver segmentation results (%)
观察图13和图14,本文Rsg-FCNN网络分割结果指标:Dice系数平均达到96.69%, PA达到99.62%,均明显高于U-net网络和Deep interactive网络指标。
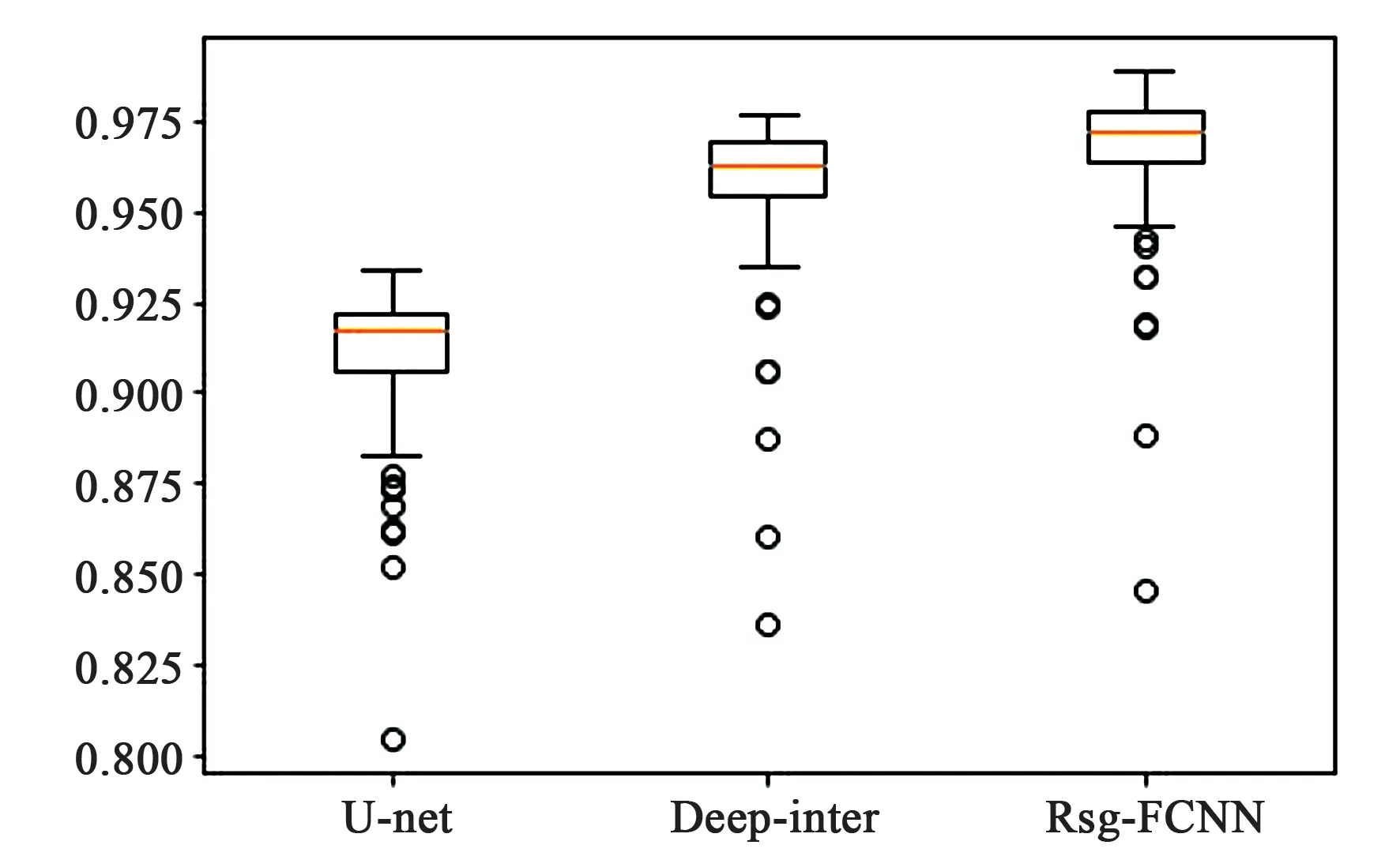
图13 实验结果Dice指标Fig.13 Dice index
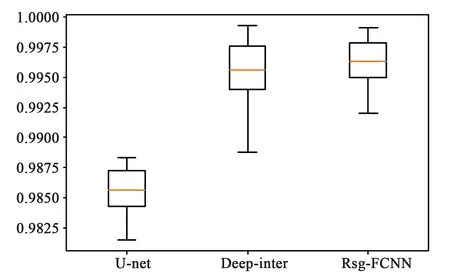
图14 实验结果PA指标Fig.14 PA index
7 结 论
本文通过Rg-net网络统筹考虑像素的灰度值、不同梯度值等低层次信息作为生长规则,提高了算法的稳定性。并将分割结果作为交互信息与原图灰度通道连接在一起,形成Rg-FCNN交互式分割网络。通过实验结果及评价指标可知,本文Rg-FCNN网络交互方式操作简单,分割结果边缘更精细,与肝脏区域边界基本吻合,Dice指标平均达到96.69%,PA平均达到99.62%。
