下一代测序技术在食源性致病微生物风险识别和溯源中的应用
2021-10-10刘云哲张喜悦王君玮
刘云哲,王 琳,张喜悦,邹 明,王君玮,赵 格
(1.中国动物卫生与流行病学中心致病微生物监测室,农业农村部畜禽产品质量安全风险评估实验室(青岛),山东青岛 266032;2.青岛农业大学动物医学院,山东青岛 266109)
近年来,食品中微生物含量超标导致的食源性致病微生物(主要指食源性致病菌)污染问题层出不穷。世界卫生组织(World Health Organization,WHO)调研数据[1]显示,全球每年因摄入被食源性致病菌污染的食物而罹患疾病的人数高达6 亿,死亡人数约42 万,死亡病例中5 岁以下儿童占比较高,达到了29.76%。在我国,每年约有1/6 的人因食用被食源性致病菌污染的食品而患病[2]。
美国疾病预防控制中心(Centers for Disease Control and Prevention,CDC)相关调查[3]表明,近几年美国沙门氏菌感染率日渐增长,在食源性致病菌中居于首位。据我国相关报道[4],沙门氏菌是引发微生物性食源性疾病的首位致病菌,而猪肉是首位归因食品。实际上,可引发食源性疾病的常见致病菌还有大肠杆菌、单增李斯特菌等。此外还可能存在一些未知病原,这些病原往往难以通过常规检测手段鉴定,因而导致相关部门不能对即将暴发的食品安全事件迅速作出反应。目前,诸如聚合酶链式反应(PCR)等分子生物学技术往往只能针对纯化后的某一种细菌的特征性靶点开展检测[5],而下一代测序技术在快速检测食源性致病菌和预防食源性疫情传播,尤其是在发现未知食源性病原体方面拥有巨大潜力[6]。因此,为更好地对食源性致病菌进行监测,采用更高效准确、灵敏特异的微生物检测技术变得尤为重要。
1 技术简介
下一代测序技术是一次性可对几十万到几亿的DNA 分子进行序列测定的高通量技术,正广泛应用于生命科学各个领域。与传统微生物检测技术相比,下一代测序技术操作过程已经实现了自动化或者半自动化,可以迅速精确地获得样品中的微生物基因组信息,且无需对样品进行前增菌培养便可对其中的多种微生物同时进行分析。理想的下一代测序技术应该具有迅速、准确、花费低且易操作等优点[7]。
目前,下一代测序技术主要指二代测序和三代测序技术。二代测序技术因其高通量、高效率、高准确性及低成本等优势逐渐普遍被应用于不同领域[8-13]。微生物的全基因组测序(whole genome sequencing,WGS)技术在二代测序中应用较为广泛,它能检测样品中各遗传物质的相关性,从而追溯疫病暴发源头。2011 年,以PacBio等研发的SMRT 测序技术和Oxford 研发的Nanopore Technologies 为代表的三代测序技术应时而生[14-16]。三代测序技术面世后,微生物的宏基因组测序(metagenomic next-generation sequencing,mNGS)有了更好的发展,特别是二代测序和三代测序技术联合使用,实现了微生物宏基因组或者全基因组既高效又准确的测序。mNGS的优势在于能发现新病原体和以前未发现的病原体,而且能在未找到导致疾病暴发病原体的前提下对疫情进行初步诊断,这为及时检测暴发疾病,开展溯源研究和风险评估,以及鉴定新的、难以发现的病原体提供了巨大帮助。
2 技术优势
目前对于食品微生物检测,传统检测方法主要是先对致病菌分离培养,然后再依据国标GB 4789 系列的生化鉴定方法进行鉴定[17]。传统鉴定微生物分子分型技术包括扩增片段长度多态性(AFLP)[20]、限制性片段长度多态性(RFLP)[21]、脉冲场凝胶电泳(PFGE)[22]、多位点序列分型(MLST)[23]、多位点可变数串联重复分析(MLVA)[24]等。这些常规技术常被用于食源性疾病暴发的病原基因型鉴定,很长一段时间里它们作为主要工具和金标准应用于食源性致病菌的监测识别和溯源追踪,但这些技术同样普遍存在耗时长、检测通量不高、准确性较低等弊端,而且操作过程繁琐[25-26]。
近些年发展起来的mNGS 技术,同样可应用于食源性致病菌检测[18-19]。由于mNGS 技术可以针对采集的样品一次性获取所含有的微生物基因组和丰度信息,研究致病菌和样本中其他菌类的相关性,特别是不会错过传统培养难以获得的微生物信息,因此具有巨大潜力。
目前PFGE 等技术分辨率有限,特别是在区分属于同一细菌克隆的菌株时;而WGS 技术则可以准确检测难以培养的生物体,包括难以培养的细菌和厌氧菌[27]。此外WGS 技术可以取代目前基于PCR 的毒素检测,如最近对耐甲氧西林金黄色葡萄球菌(MRSA)的检测证明[28],WGS 抗菌表型和基因型表现出一致性,证明了WGS 可用于指导治疗,并可用于发现新耐药机制。
2012 年,在一次沙门氏菌疫情暴发中,PFGE无法将被污染的样品与未被污染的同类食品区分开来;而WGS 技术不仅可以精确确定致病菌的物种亚型,而且可以检测其对所有药物的耐药性[29]。Joensen 等[30-31]在应用WGS 技术的基础上,建立了一个能够及时准确识别产毒素大肠杆菌菌株的分型和监测体系。Dallman 等[33]通过WGS 分析方法发现了产志贺毒素大肠杆菌病例之间的聚集性和关联性,而且还发现了一些之前未发现的规模不大但分布较广泛的聚集性疾病。对2007—2016 年加拿大散发的沙门氏菌病病例研究[33]发现,应用WGS方法检测出来的数据与流行病学调查结果相一致,而常规PFGE 方法则无法对以上不同的暴发事件进行准确区分。
由上可见,下一代测序技术与传统病原微生物检测技术相比,具有快速准确、分辨率高等优势,在病原微生物鉴别诊断中发挥着越来越重要的作用。表1 列举了二代和三代测序的特点[7]。
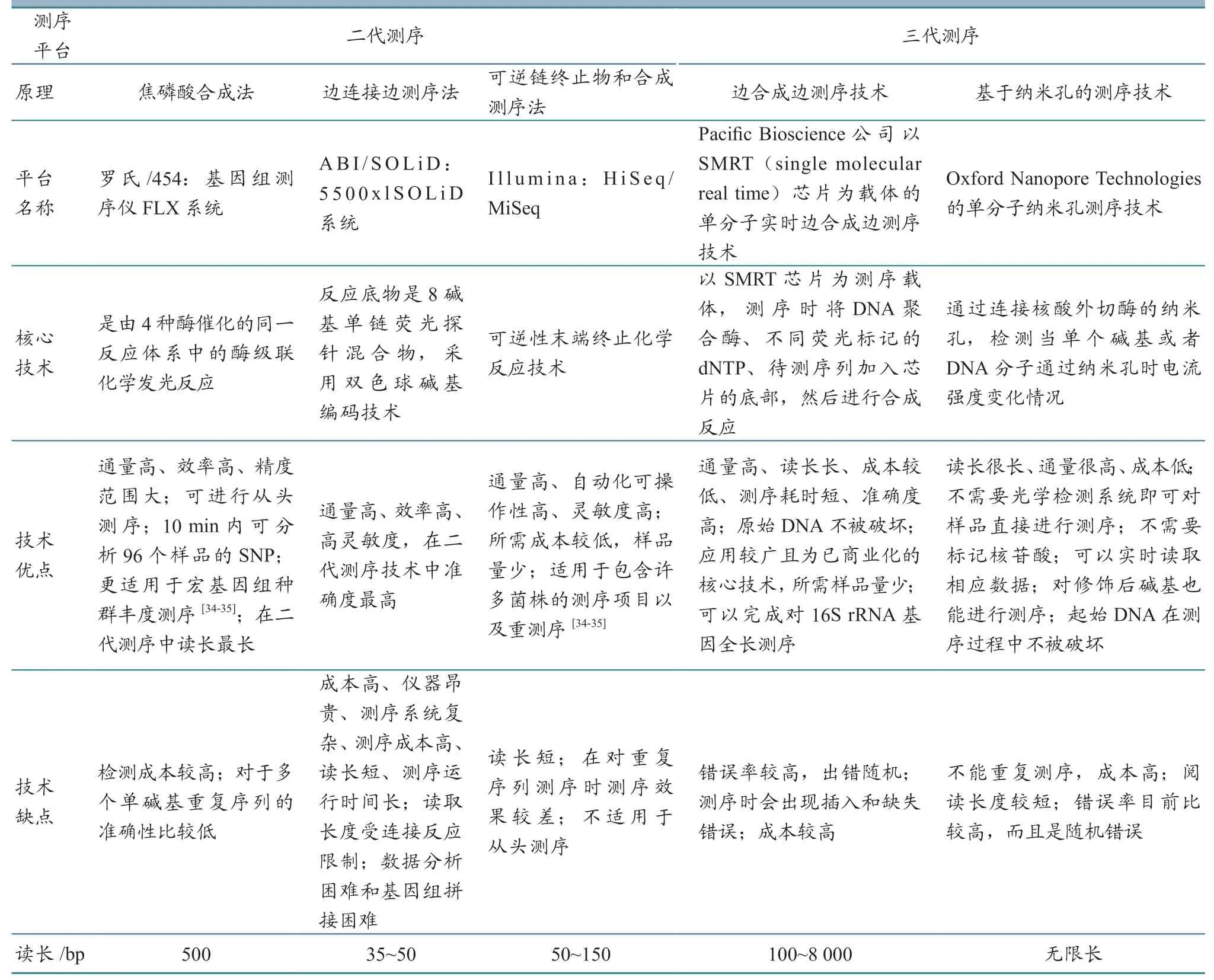
表1 主要的下一代测序技术比较
下一代测序技术以其明显的优势成为近年来研究应用的热点,但它也存在劣势:低丰度的致病菌在样本量不足时容易被其他序列信息掩盖,同时该技术也不能获得菌株进行下游试验研究。所以,下一代测序数据在现阶段更多的是作为补充,但随着微生物组学的发展和配套生物信息方法学技术的进一步开发,它也将成为食源性致病菌基因组数据平台中不可或缺的一部分[36]。
3 在食源性致病菌风险识别中的应用
3.1 生鲜动物产品
Edirmanasinghe 等[37]在对来自禽肉、屠宰场的家畜胴体和人类的肠炎沙门氏菌分离株进行WGS 分析和表型特征鉴定后,证明了肠炎沙门氏菌分离株在家畜、零售家禽和人类之间的传播。Zagdoun 等[38]将Illumina MiSeq 测序方法应用于猪肉火腿的微生物细菌多样性分析调查,发现在其制作成火腿过程中(尤其是切片步骤)比生肉具有更高的微生物污染风险。
针对2014 年5—9 月在英国暴发的肠炎沙门氏菌病,研究人员对来自英国和欧洲其他国家病例的分离株进行了WGS 分析,证实英国的疫情与一家德国生产商的鸡蛋有关[39];2014 年6 月,研究人员使用下一代文库制备和Illumina HiSeq 2500 测序技术检测发现,法国6 起沙门氏菌病的暴发与1起奥地利沙门氏菌病有关,而法国和奥地利肠炎沙门氏菌病的暴发则与上述德国生产商供应的鸡蛋有关[39]。WGS 以前曾被用于沙门氏菌的前瞻性监测[40]以及多国沙门氏菌疫情暴发监测,但在这里,它首次被用于实时鉴定多国沙门氏菌暴发,并及时告知相关部门应采用的公共卫生控制措施[41]。
2015 年5 月—2018 年10 月,研究人员在欧盟和欧洲经济区调查了一起大型肠炎沙门氏菌疫情的多国暴发事件[42-44],他们使用WGS 进行了一项病例对照研究,包括对从登记人口中随机抽样的确诊/疑似病例以及2016 年10 月在食品机构进食的病例进行食品追溯对照研究,发现来自波兰的鸡蛋是感染媒介;通过系统发育分析,在人类病例中发现了两株肠炎沙门氏菌,随后在波兰鸡蛋的主要生产场所中发现了同样的肠炎沙门氏菌株,从而证实疫情来源。可见,在鸡蛋这种微生物污染较低的产品中,下一代测序技术在致病菌风险识别和监测方面发挥了很大优势。
目前,我国尚无利用下一代测序技术进行食源性疾病暴发后病原溯源的相关报道,但已有研究采用WGS 技术分析畜产品中微生物组成。有学者[45]在我国西藏和俄罗斯采集自然发酵的牛奶,应用高通量技术检测发现来自不同地域的牛奶样品中微生物群落具有明显差异。另有学者[46]运用基于16S rDNA 基因的高通量测序(HTS)技术对冷藏(4 ℃)猪肉的细菌微生物群落等进行调查,发现假单胞菌、不动杆菌和发光细菌在冷藏猪肉中占优势,并且这些分类群可加速冷藏猪肉腐败。
3.2 生鲜植物产品
下一代测序技术同时也已应用于生鲜植物产品危害因子监测。如沙门氏菌污染的番茄已多次造成食源性疾病暴发,Ottesen 等[47]利用MiSeq DNA 测序及mNGS 技术成功从番茄样本中鉴别出沙门氏菌;Leonard[48]等从菠菜中识别到产志贺毒素大肠杆菌;Andreevskaya 等[49]运用SOLiD 测序技术对豆类乳杆菌进行WGS 分析,发现其腐败功能与丙酮酸代谢途径异常有关,同时发现了该菌的特有基因。
2014 年9 月,美国CDC 收集了从全美病人、食品及环境中分离到的所有单增李斯特菌株,运用WGS 分析发现几名患者的分离菌株与食用豆芽有关[50],在运用了WGS 分析后的3 年内,共监测识别和解决了多达14 起的单增李斯特菌小规模暴发事件[50-51]。2014 年3 月,美国监管机构发现被单增李斯特菌污染的生菜,仅使用PFGE 很难建立与同期患者分离株的联系,然而应用WGS 分析表明来自俄亥俄州一名患者的分离株与生菜分离株高度相关[52]。
4 在食源性致病菌风险溯源中的应用
4.1 大肠杆菌
2011 年5 月,大肠杆菌O104:H4 型在德国暴发并且迅速蔓延。流行病学调查[53]显示,埃及进口的豆芽是此次疫情暴发的源头;传统微生物学方法[54]显示,此次暴发菌株的亚型和表型特征均相同;WGS 分析[55]发现,引起这起严重疫病暴发的菌株可能是通过水平转移的方式,获得了编码志贺毒素的相关致病因子。2011 年在对德国暴发的产志贺毒素大肠杆菌(O104:H4 型)腹泻粪便样品研究中,已经证实WGS 可应用于食源性疾病暴发后病原体鉴别[56]。Underwood 等[57]选取一项早期大肠杆菌(O157:H7 型)暴发研究中患者及动物样品,利用WGS 进行比较分析及致病菌鉴别,证明5 种亚型致病菌在感染人类前广泛分布于该农场区域,传染源是动物和环境。
Saltykova 等[58]通过计算机模拟宏基因组学数据,将碎肉分离株与产志贺毒素大肠杆菌(STEC)暴发期间收集的人源分离株联系起来,证明mNGS可用于食源性疾病暴发的溯源追踪。Schaeffer[59]利用三代测序技术对大肠杆菌其他7 株分离自非洲的大肠杆菌以及4 株属于其他血清型的大肠杆菌进行WGS 分析,数据显示导致德国疾病暴发的大肠杆菌与其他大肠杆菌菌株不同,其属于大肠杆菌另一个分支。
4.2 沙门氏菌
2009 年7 月,用于制作意大利腊肠的胡椒被沙门氏菌污染而引起食源性疾病暴发,Bakker等[60]选取暴发时的分离菌株和暴发前的菌株,经检测后发现这些菌株具有完全相同的PFGE 图谱,无法识别出暴发菌株;而通过wgSNP(基于全基因组的单核苷酸多态性分型)分析,不仅能够对本次暴发的相关菌株进行准确识别,而且还监测到之前一些未发现的引起小型暴发的菌株。这充分表明wgSNP 分子分型技术在暴发致病菌的风险识别和溯源分析上比传统PFGE 技术具有更高的分辨力和准确度。
Ashton 等[61]应用二代测序技术中的Illumina HiSeq 平台对2013 年暴发的沙门氏菌病进行了WGS 分析,证明了在蛋黄酱中分离到的鼠伤寒沙门氏菌是导致此次疾病暴发的病原菌。Hoffmann等[62]利用WGS 技术查明了造成金枪鱼刮板被污染的400 多种肠炎沙门氏菌亚种的来源。
4.3 单增李斯特菌
Ruppitsch 等[63]对单增李斯特菌血清型代表菌株和暴发菌株进行wgMLST(全基因组多位点序列分型)分析方法应用比较,结果显示wgMLST技术可以准确区分不同暴发的菌株以及与暴发相关或不相关的菌株。同时,wgMLST 技术可对MLST和PFGE 技术分型结果一致而流行病学却并不相关的菌株加以区分。Gilmour 等[64]运用WGS 技术成功鉴别出2008 年加拿大食源性疾病暴发的致病菌为李斯特菌。
mNGS 在检测受污染冰淇淋中的单增李斯特菌方面具有优势,并有研究[65]证明其可以将目前追溯所需时间减少一半。可见,下一代测序技术不仅可以发现菌株,还能精确了解病原菌的传播源头,明确传播途径等,甚至能够精确区分疾病大流行中不同暴发事件的先后顺序,同时还能发现其中隐藏的小疾病的聚集性暴发,这一点传统分子分型方法难以做到,而这些特点在疾病追溯和预防控制中至关重要。
5 展望
目前二代测序技术占据主流市场,相信在不远的未来三代测序技术会逐渐普及到各行各业。届时,四代、五代等更先进的测序技术也将应运而生,甚至成为未来实验室分析以及食品中食源性致病菌的常规检测手段。二代和三代测序技术各有其优势及缺点,因此在实际检测时应选择合适方法,甚至2 种及以上方法结合使用。希望未来下一代测序技术能与更多新兴方法相结合,使其不仅可应用于食源性致病菌的检测识别及溯源,还能为更好地推动食品安全事业发展作出贡献。
