盐胁迫下垂穗披碱草叶片转录组差异表达分析
2021-09-28梁书荣赵政文
梁书荣 赵政文

摘要:为探究垂穗披碱草在盐胁迫下的分子响应机制,将200 mmol/L NaCl处理0、6、12、24、36 h的垂穗披碱草叶片进行转录组测序。结果表明,共获得转录本序列数量为462 235条,经过注释分析获得单基因序列数量229 597条,这些差异基因涉及氨基酸的合成和代谢、淀粉和蔗糖代谢、类黄酮生物合成、碳代谢和植物激素信号传导等途径,可作为研究垂穗披碱草盐胁迫响应主要研究机制。荧光定量PCR(qRT-PCR)验证表明,挑选的7个差异表达基因的表达趋势与测序结果相一致,说明测序结果准确可靠。本研究测序结果为垂穗披碱草的基因功能分析、分子标记开发和遗传多样性研究提供了理论参考。
关键词:垂穗披碱草;盐胁迫;转录组;基因差异表达
中图分类号: S184 文献标志码: A
文章编号:1002-1302(2021)18-0065-07
收稿日期:2021-06-20
基金项目:国家现代牧草产业技术体系建设专项(编号:CARS-34)。
作者简介:梁书荣(1980—),女,河南郑州人,硕士,讲师,研究方向为植物生理学。E-mail:lsrong2001@163.com。
我国北方大部分地区为干旱半干旱区,拥有着大面积不同程度的盐碱地[1],因此盐分成为影响牧草在北方种植生长的主要因素之一。垂穗披碱草(Elymus nutans G.)属于禾本科小麦族披碱草属,为多年生丛生草本植物,具有一定的抗旱、耐寒能力,其野生类型广泛分布于我国的东北、华北、西北和西南等地区[2-4]。垂穗披碱草草质柔软、适口性好,在生态保护、草场植被恢复和人工草地等方面也具有重要作用[5-6]。非生物因素盐胁迫在一定程度上影响垂穗披碱草的推广应用,因此,本研究通过对盐胁迫下垂穗披碱草不同时间段的转录组测序,阐述垂穗披碱草响应盐胁迫的分子过程,为垂穗披碱草耐盐功能基因的挖掘、分子标记开发以及培育耐盐新品种提供借鉴。
盐逆境条件下对植物产生的危害主要包括渗透胁迫、离子毒害和氧化胁迫等,严重时直接导致植物死亡[7-8]。植物在盐胁迫环境下通过调节自身的基因表达、信号传导、能量代谢及蛋白质合成,响应盐胁迫带来的危害以提高自身的耐盐能力[9-10]。近几年,众多学者通过转录组测序技术(RNA-Seq)在分子生物学层面揭示基因表达调控,更好地阐释植物的抗逆境能力。第2代测序技术已经在黑麦草[11]、羊草[12]、狗牙根[13]以及金冕草[14]研究中得到应用,通过获得干旱、盐胁迫下差异基因并进行表达分析,阐述耐性响应分子机制。目前,垂穗披碱草响应盐胁迫的研究主要在生理生化方面,而通过2代测序技术阐述胁迫下分子响应机制少有报道。本研究采用第2代高通量测序技术分析盐胁迫环境下垂穗披碱草叶片转录组信息,筛选获得的差异基因进行分析、验证,为深入研究垂穗披碱草对盐胁迫的分子响应机制和筛选耐盐功能基因提供理论依据。
1 材料与方法
1.1 材料与生长条件
以垂穗披碱草为试验材料,将籽粒饱满种子消毒后放入培养皿中,并置于温度26 ℃光照8 h和温度16 ℃ 16 h黑暗的光照培养箱内进行种子萌发。当种子萌发且幼苗长到7~8 cm时,移栽至装有蛭石与珍珠岩按体积比3 ∶1混合带有小孔的花盆(20 cm×16 cm×13 cm)中,每盆3~5株置于植物工厂温室。用1/2 Hoagland 营养液浇灌,每3 d换1次,待幼苗长到4叶期时挑选长势一致、大小均匀的幼苗小心移栽到装有1/2 Hoagland营养液的三角瓶中恢复3 d,然后在含有200 mmol/L NaCl的 1/2 Hoagland 营养液处理下胁迫共设5个处理,分别为CK:0 h、A:6 h、B:12 h、C:24 h、D:36 h。各处理至少10株幼苗,3次重复,对胁迫0、6、12、24、36 h的垂穗披碱草叶片取样,经液氮处理后,放置于 -80 ℃ 超低温冰箱保存备用。
1.2 RNA-Seq样品检测和文库构建
使用全式金植物RNA提取试剂盒(ER301-01)来提取植物样品的 RNA,分别提取垂穗披碱草对照组CK和处理组A、B、C、D叶片中的RNA。所提取的RNA使用NanoDropND2000分光光度计检查总RNA的浓度和质量。经提取合格的RNA送至测序公司进一步确认质量,通过检测合格后的RNA使用带有Oligo(dT)磁珠富集真核生物mRNA。富集完成后将mRNA打断成短片段,以mRNA为模板进行反转录合成cDNA。通过AMPure XP beads对双链cDNA进行片段大小选择,最后进行PCR扩增以构建cDNA文库。
1.3 Illumina测序及测序数据的分析
1.3.1 测序原始数据质量评估与测序数据质控
文库质检合格后,高通量测序平台上机测序。经 CASAVA 软件通过序列碱基识别后转换为原始序列(sequenced reads)即raw datas。为保证信息分析质量,使用Trimmomatic软件(v0.33)去除含有接头(adapter)的reads;去除无法确定碱基信息且比例大于10%的reads;再去除低質量reads(质量值Q≤20的碱基数占整条read的50%以上的reads),最后得到整洁序列(clean datas),后期分析都基于clean datas,其结果以FASTQ文件格式存储[15]。
1.3.2 转录本拼接
采用Trinity对clean reads进行拼接[16]。使用Trinity常用软件Corset进行转录本分析,根据转录本间shared reads将转录本聚合为许多cluster,在与不同样本间的转录本表达水平及H-Cluster算法相结合,把样本之间有差异表达的转录本从原cluster分离,建立新的cluster,最终每个cluster被定义为“Gene”。
1.3.3 基因功能注释及差异表达富集分析
为了获取研究的垂穗披碱草基因序列与本物种基因的功能信息,进行7个公共数据库的基因功能注释,包括Nr、Nt、SwissProt、InterPro、COG、GO和KEGG数据库。使用RSEM软件[17]以Trinity拼接得到的转录组作为参考序列(ref),将每个样品的clean reads对ref做mapping。对于有生物学重复的样品,采用DESeq进行分析,筛选阈值为p-adjusted<0.05筛选差异基因。GO富集分析方法为GOseq[18],此方法基于Wallenius non-central超几何分布。生物体内差异基因行使不同的生物学功能,通过Pathway富集分析能确定显著差异表达基因参与的最主要代谢途径和信号接受、传导途径。KEGG作为Pathway相关的主要公共数据库,不仅提供了所有可能的代谢途径,而且全面注解了相关酶催化反应,是分析生物体内代谢和代谢网络研究主要工具。
1.4 PCR转录组数据验证
为验证垂穗披碱草测序结果的准确性,随机选择7个差异表达基因进行qRT-PCR 分析。采用TransScript One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒,将提取对照组和盐胁迫处理组叶片的总RNA反转录合成cDNA,采用Primer Premier 5 软件设计荧光定量引物(表1),并进行qRT-PCR 分析。具体方法参照TransStart Top Green qPCR SuperMix说明书,对每一样品进行3次重复,使用2-ΔΔCT相对定量法定量计算相对表达量。
2 结果与分析
2.1 测序数据分析
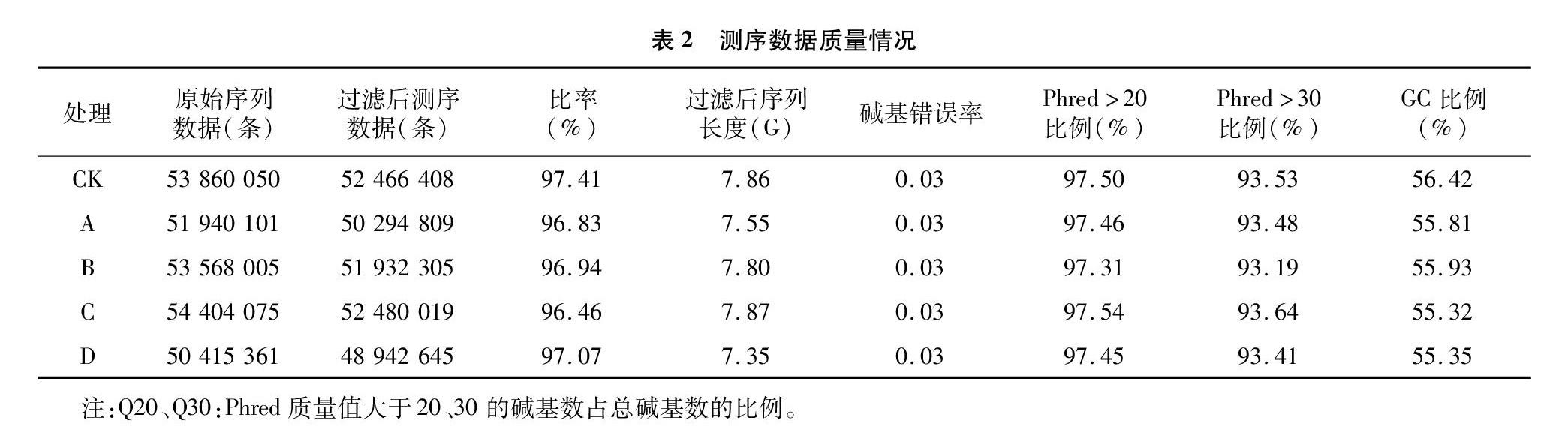
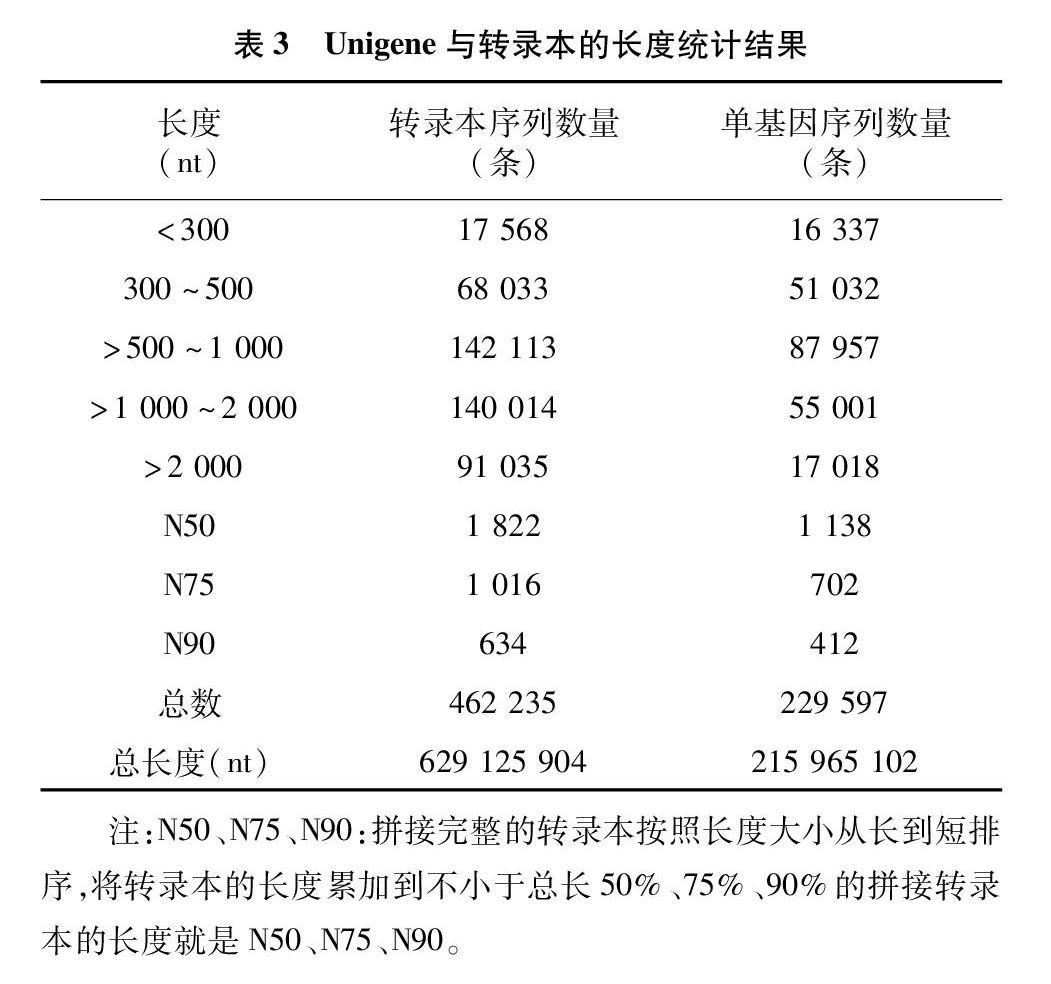
本研究通过第2代高通量测序技术进行转录组测序,对15个样本测序结果(表2),分为5个组,对照组CK和处理组A、B、C、D的Raw Reads分别在53 860 050、51 940 101、53 568 005、54 404 075和 50 415 361 条。经过质控,去除接头、低质量的序列后,得到clean reads都在52 466 408、50 294 809、51 932 305、52 480 019 和48 942 645条。各样本得到的clean reads的量占raw reads的比例都大于96%,过滤后序列长度都在7.00 G左右,且Q20和Q30分别都在97%和93%,GC含量在55.32%~56.42%,说明测序质量较高,达到建库要求。数据经Trinity拼接后,获得转录本数量为462 235条(表3),总长度为629 125 904碱基(nt),N50、N75、N90分别是1 822、1 016、634 bp,>2 000 bp的转录本数量为91 035条,<300 bp的转录本数量为17 568条;Unigene共229 597条,总长度是215 965 102 nt,N50、N75和N90分别是1 138、702、412 bp,>2 000 bp的转录本数量为17 018条,<300 bp的转录本数量为 16 337 条,拼接组装统计结果比较完整,测序结果较好。
2.2 差异基因功能注释与表达分析
对预测的Unigene进行基因注释(图1),将Unigene的序列分别与7个主要的生物信息数据库,如NR、KO、NT等进行基因信息注释比对,获得了229 597条注释信息,其中,获得最多的注释信息是NR数据库,占全部基因的58.36%;最少注释信息是KO数据库,占18.38%(图1)。在浓度为 200 mmol/L NaCl胁迫下,垂穗披碱草经6、12、24、36 h 胁迫后与对照CK的样本unigene进行差异表达基因分析。垂穗披碱草经6 h胁迫后与对照CK相比,差异基因有7 865个(图2-a),其中上调基因有4 808个,下调基因有3 057个;12 h胁迫后与对照CK相比,有13 681个差异基因,其中有8 060个上调表达基因,有5 621个下调表达基因(图2-b);24 h胁迫后与对照CK相比有6 934个差异基因,其中上调表达基因有3 589个,下调表达基因有3 345个(图2-c);盐胁迫36 h后与对照CK相比有6 633个差异表达基因,其中上调为3 485个基因,下调表达基因有3 148个(图2-d)。
2.3 差异表达基因的GO功能注释
GO(gene ontology,GO) 是基因功能注释分类标准化体系,GO分为分子功能(molecular function)、生物过程(biological process)和细胞组成(cellular component) 3个Ontology。基于GO数据库将对照组与处理组的Unigenes进行注释和分析,分析结果前30个富集最显著的GO term(图3)。分类注释结果表明,在生物过程分类中,注释数量最多的是碳水化合物代谢过程,其次是响应胁迫过程,最低的为糖蛋白分解代谢过程。在细胞组分分类注释中,结果前30个富集最显著共有6个,依次为叶绿体、质、质体基质、叶绿体基质、核糖核蛋白复合物和转录因子复合物。在分子功能中显著性最高的为水解酶活性(水解O-糖基化合物),数量最低的为天冬酰胺酶活性。
2.4 差异表达基因的KEGG功能分析
KEGG(Kyoto Encyclopedia of Genes and Genomes,KEGG)是系统分析基因信息的数据库,是一个整体网络进行研究基因功能。以對照组和处理组之间的差异基因进行KEGG代谢通路分析,获得5 191个差异基因,注释到108个代谢通路。通过进一步统计分析的代谢通路,筛选出盐胁迫下垂穗披碱草前20条富集最显著的pathway(图4),主要有次生代谢物的生物合成、碳代谢、氨基酸的生物合成、淀粉和蔗糖代谢、内质网中的蛋白质加工、糖酵解/糖异生和氨基酸的代谢等。其中,差异基因注释到pathway数量最多的是次生代谢物的生物合成,差异表达基因为683个;其次是碳代谢通路途径,差异表达基因为194个。从次生代谢物的生物合成、淀粉和蔗糖代谢、糖酵解/糖异生、泛醌和其他萜类化合物-醌生物合成、卟啉和叶绿素代谢、类黄酮生物合成、氨基酸的生物合成及植物激素信号传导等代谢通路中筛选出与垂穗披碱草响应盐胁迫的候选基因。
2.5 差异基因及qRT-PCR验证
为了证实转录组数据的可靠性,选取7个差异表达基因,其中,包括上调4个基因和下调3个基因,利用qRT-PCR仪以Actin为内参进行对照组和处理组基因的表达量分析。通过与RNA-Seq数据结果比较分析,线性回归分析得到正相关系数(r=0.852 5),表明这些选择的7个差异基因在转录组数据中的表达与qRT-PCR结果吻合良好(图5)。
3 讨论与结论
植物在整个生长发育过程中不断受到外界环境因素的制约和影响,当外界环境盐分高于植物耐受阈值时,会对植物造成不良影响甚至死亡。高浓度盐分对植物造成2种主要的胁迫影响:渗透胁迫(由水分利用效率低引起)和离子毒害(主要是Na+)(由细胞内离子稳态紊乱引起)[19]。植物为适应盐胁迫条件会响应调节生理生化变化形成复杂的防御机制,包括维持细胞膜的完整性、清除活性氧 (ROS)、渗透调节和离子跨膜转运,以及恢复细胞离子平衡,都是致力于降低盐度造成的渗透性或离子损伤[9,19]。
盐胁迫下植物体内会发生一系列生化反应,导致植物体内积累大量的活性氧,这些活性氧大多具有高活性和强度性,会引起蛋白氧化、DNA损伤及脂类的过氧化,对细胞结构产生巨大影响[20-22]。植物为了清除活性氧带来的损伤,植物细胞或细胞器如叶绿体、线粒体和过氧化物酶体会启动活性氧清除抗氧化防御系统。该系统的主要成分包括一些能清除活性氧的酶类,如过氧化氢酶、超氧化物歧化酶、过氧化物酶、抗坏血酸过氧化物酶等[22-23]。植物体内次生代谢物质类黄酮是调控植物生长和抗逆性过程的主要物质,具有抗氧化和ROS清除功能。研究表明,植物体内类黄酮在3个关键酶苯丙氨酸解氨酶基因(PAL)、肉桂酸羟化酶基因(4CL)和4-4-香豆酰辅酶A连接酶基因(C4H)作用下由苯丙氨酸生成中间产物4-香豆酰CoA,再由查尔酮合成酶基因(CHS)催化生成查尔酮[24]。李爽等研究盐胁迫下中亚滨藜通过提高类黄酮含量,降低根系中的丙二醛(MDA)和过氧化氢(H2O2)含量,缓解了盐胁迫对幼苗的影响提高了耐盐性[25]。在盐胁迫下垂穗披碱草类黄酮生物合成途径和苯丙氨酸、酪氨酸和色氨酸生物合成途径及次生代谢物的生物合成途径,在KEGG pathway富集分析中呈现显著性,说明垂穗披碱草为抵御盐胁迫危害时产生了次生代谢产物类黄酮。
脱落酸(abscisic acid,简称ABA)作为一种植物激素调控植物的生长而且在植物抵抗环境胁迫中起着重要的作用,具有调节植物细胞内水分平衡和抗渗透胁迫的功能[13,26]。研究表明,盐胁迫下拟南芥在外源ABA处理下诱导激活大量的ABA合成基因,其抗渗透胁迫能力增强,而ABA缺失突变体的耐盐性显著下降,其渗透能力比野生植株敏感[27]。在ABA响应盐胁迫调控途径中,ABA将识别到的响应盐胁迫信号传递给下游转录因子(如MYB、WRKY、NAC、bZIP等),转录因子再与下游的响应胁迫基因顺式作用元件相结合识别,最终使得响应盐胁迫基因差异表达植株耐盐性增强[28-29]。Wang等研究表明,盐胁迫下拟南芥种子萌发过程中,响应盐胁迫的ABA合成和信号传导相关基因直接负调控MYB1转录因子[30]。唐立郦等报道了在转基因紫花苜蓿中过表达GsWRKY20能够减少盐对苜蓿细胞内离子平衡的影响,从而提高苜蓿耐盐性[31]。众多研究也发现,盐胁迫下紫花苜蓿、草地早熟禾、狗牙根等,植物体内NAC转录因子受盐胁迫的诱导显著表达[32]。通过对盐胁迫下垂穗披碱草差异表达基因进行KEGG pathway富集分析中发现,与植物激素信号传导相关的所有基因共104条。
在植物中,SOS途径即关于调控植物体内离子平衡和植物响应盐胁迫信号传导途径逐渐被重视。SOS信号传导途径包括3个SOS家族关键基因(如SOS1、SOS2、SOS3),SOS1、SOS2基因是植物耐盐性的关键基因,研究发现拟南芥SOS1和SOS2突变体对高浓度的Na+和低浓度的K+敏感性较强[33-34]。SOS1功能的核心区域N端有12个膜转运的结构域,具有高度的疏水性,核心区域与细菌、真菌的质膜Na+/H+反转运蛋白有相似性[35]。SOS2基因编码一个丝氨酸/苏氨酸型的蛋白激酶,N端的催化激活结构域類似于AMPK激酶,植物正常情况下其表达量低,盐胁迫处理后植物根系内SOS2表达量迅速升高[36]。SOS3基因编码一个具有EF手型结构的与酵母钙调磷酸酶B亚基的Ca2+结合蛋白,植物细胞受到高浓度的Na+胁迫时,细胞膜上的受体将信号传递给细胞内,使得Ca2+浓度增加,SOS3活性激活与SOS2形成一种复合体(即SOS3/SOS2复合体),使得SOS2蛋白激酶激活。SOS3/SOS2复合体一方面通过磷酸化细胞膜上的Na+/H+反向转运体SOS1,将细胞内过多的Na+排出细胞膜外;另一方面还可以激活液泡膜上的Na+/H+反向转运体AtNAX1使其将细胞内过多的Na+区隔化储存起来[33,35]。二者结合共同调节细胞质内的盐浓度,使细胞内离子稳态平衡,SOS途径在植物抗盐胁迫方面起着重要的作用。垂穗披碱草在盐胁迫下的差异基因KEGG pathway分析中,与甘氨酸、丝氨酸和苏氨酸代谢途径相关的共76条,在前20条KEGG代谢通路富集分析中呈显著变化,并且注释到8个上调基因参与了垂穗披碱草的盐胁迫。
通过测序数据分析得到,Raw Reads经质控,分别得到7.86G(CK)、7.55G(A)、7.80G(B)、7.87G(C)、7.35G(D)的clean bases。通过差异基因筛选共得到,胁迫后6 h时差异基因有7 865个,上调基因有4 808个,下调基因有3 057个;12 h胁迫后有13 681个差异基因,其中有8 060个上调表达基因,有5 621个下调表达基因;24 h胁迫后有6 934个差异基因,上调表达基因有3 589个,下调表达基因有3 345个;盐胁迫36 h后有6 633个差异表达基因,上调为3 485个基因,下调表达基因有3 148个。通过KEGG pathway分析,与叶绿素代谢、类黄酮生物合成、丝氨酸和苏氨酸代谢、碳代谢和植物激素信号传导等可以作为分析垂穗披碱草响应盐胁迫机制研究的主要内容,揭示垂穗披碱草叶片响应盐胁迫的分子机制。
参考文献:
[1]王佳丽,黄贤金,钟太洋,等. 盐碱地可持续利用研究综述[J]. 地理学报,2011,66(5):673-684.
[2]王 沛,陈玖红,王 平,等. 披碱草属植物抗逆性研究现状和存在的问题[J]. 草业学报,2019,28(5):151-162.
[3]赵永强,张俊超,赵旭红,等. 垂穗披碱草落粒性评价及农艺性状的相关性分析[J]. 草业科学,2017,34(8):1711-1720.
[4]祁 娟,罗 琰,王 沛,等. 碱胁迫对超干处理垂穗披碱草种子萌发及幼苗生长的影响[J]. 中国草地学报,2017,39(1):79-84.
[5]张典业,张丽静,黄德君,等. 高寒地区垂穗披碱草种质资源综合评价[J]. 草地学报,2015,23(6):1239-1246.
[6]刘春华,苏加楷,黄文惠.禾本科牧草耐盐性的研究[J]. 中国草地,1992,14(6):12-17,22.
[7]Liu J L,Wu N.Biomass production of switchgrass in saline-alkali land[J]. Advanced Materials Research,2014,1008/1009:93-96.
[8]李志丹,干友民,泽 柏,等. 牧草改良盐渍化土壤理化性质研究进展[J]. 草业科学,2004,21(6):17-21.
[9]Parvaiz A,Satyawati S.Salt stress and Phyto-biochemical responses of plants-a review[J]. Plant,Soil and Environment,2008,54(3):89-99.
[10]Yang Y Q,Guo Y.Unraveling salt stress signaling in plants[J]. Journal of Integrative Plant Biology,2018,60(9):796-804.
[11]Farrell J D,Byrne S,Paina C,et al. De novo assembly of the perennial ryegrass transcriptome using an RNA-seq strategy[J]. PLoS One,2014,9(8):e103567.
[12]劉 书,刘盼盼,杨伟光,等. 羊草种子萌发相关基因的筛选及表达分析[J]. 草业学报,2018,27(11):58-66.
[13]Hu L X,Li H Y,Chen L A,et al. RNA-seq for gene identification and transcript profiling in relation to root growth of bermudagrass (Cynodon dactylon) under salinity stress[J]. BMC Genomics,2015,16(1):575.
[14]屈兴红,曾洪学. 金冕草(Paspalum notatum)盐胁迫转录组差异性分析[J]. 分子植物育种,2019,17(11):3515-3523.
[15]Cock P J A,Fields C J,Goto N,et al. The Sanger FASTQ file format for sequences with quality scores,and the Solexa/Illumina FASTQ variants[J]. Nucleic Acids Research,2009,38(6):1767-1771.
[16]Grabherr M G,Haas B J,Yassour M,et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology,2011,29(7):644-652.
[17]Li B,Dewey C N.RSEM:accurate transcript quantification from RNA-Seq data with or without a reference genome[J]. BMC Bioinformatics,2011,12:323.
[18]Young M D,Wakefield M J,Smyth G K,et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biology,2010,11(2):14.
[19]李晓院,解莉楠.盐胁迫下植物Na+调节机制的研究进展[J]. 生物技术通报,2019,35(7):148-155.
[20]Zhang M,Smith J A C,Harberd N P,et al. The regulatory roles of ethylene and reactive oxygen species (ROS) in plant salt stress responses[J]. Plant Molecular Biology,2016,91(6):651-659.
[21]鲁 琳,杨尚谕,刘维东,等. 基于转录组测序花烟草响应盐胁迫活性氧清除相关基因的挖掘[J]. 生物技术通报,2020,36(12):42-53.
[22]赵 嫚,陈仕勇,李亚萍,等. 外源GABA对盐胁迫下金花菜种子萌发及幼苗抗氧化能力的影响[J]. 江苏农业学报,2021,37(2):310-316.
[23]赵明德,刘 攀,杨 冲,等. 盐胁迫对青藏高原多年生牧草幼苗生理指标的影响[J]. 生态科学,2018,37(3):123-130.
[24]宋晓敏,吕晓杰,邱智敏,等. 红树植物秋茄类黄酮代谢及其抗氧化活性对高盐胁迫的响应[J]. 西北植物学报,2016,36(12):2461-2468.
[25]李 爽,孫亮亮,白丽丽,等. 类黄酮参与调控中亚滨藜幼苗对盐胁迫的耐受性[J]. 中国生态农业学报,2017,25(9):1345-1350.
[26]Yang L F,Jin Y H,Huang W,et al. Full-length transcriptome sequences of ephemeral plant Arabidopsis pumila provides insight into gene expression dynamics during continuous salt stress[J]. BMC Genomics,2018,19(1):717.
[27]Cao M J,Wang Z,Zhao Q,et al. Sulfate availability affects ABA levels and germination response to ABA and salt stress in Arabidopsis thaliana[J]. The Plant Journal,2014,77(4):604-615.
[28]姚侠妹,纪 敬,岳剑云,等. ABA对盐胁迫下侧柏活性氧代谢及其相关基因表达的研究[J]. 西北植物学报,2017,37(1):105-114.
[29]冷 暖,刘晓巍,张 娜,等. 草地早熟禾干旱胁迫转录组差异性分析[J]. 草业学报,2017,26(12):128-137.
[30]Wang T,Tohge T,Ivakov A,et al. Salt-related MYB1 coordinates abscisic acid biosynthesis and signaling during salt stress in Arabidopsis[J]. Plant Physiology,2015,169(2):1027-1041.
[31]唐立郦. GsZFP1和GsWRKY20基因转化苜蓿及耐盐耐旱性分析[D]. 哈尔滨:东北农业大学,2013.
[32]段俊枝,李 莹,赵明忠,等. NAC转录因子在植物抗非生物胁迫基因工程中的应用进展[J]. 作物杂志,2017(2):14-22.
[33]麻冬梅,秦 楚. AtSOS基因在紫花苜蓿中的表达及其耐盐性研究[J]. 草业学报,2018,27(6):81-91.
[34]谢崇波,金谷雷,徐海明,等. 拟南芥在盐胁迫环境下SOS转录调控网络的构建及分析[J]. 遗传,2010,32(6):639-646.
[35]Zhao X F,Wei P P,Liu Z,et al. Soybean Na+/H+antiporter GmsSOS1 enhances antioxidant enzyme activity and reduces Na+ accumulation in Arabidopsis and yeast cells under salt stress[J]. Acta Physiologiae Plantarum,2016,39(1):19.
[36]Sathee L,Sairam R K,Chinnusamy V,et al. Differential transcript abundance of salt overly sensitive (SOS) pathway genes is a determinant of salinity stress tolerance of wheat[J]. Acta Physiologiae Plantarum,2015,37(8):169.
