贵州地方高粱种质资源遗传多样性分析
2021-09-23胡小兰任明见曾庆鸿邱红波
胡小兰, 任明见,2, 曾庆鸿, 申 涛, 邱红波
(1.贵州大学农学院, 贵阳 550025; 2.国家小麦改良中心贵州分中心, 贵阳 550025;3.贵州省农业科技发展中心, 贵阳 550001)
高粱(Sorghumbicolor(L.) Moench)属于禾本科高粱属,为一年生草本植物。是我国重要的粮食作物,因其水分利用率高、抗旱性强,具有很强的抗逆性和适应性,在我国东北、华北、西北等干旱地区广泛种植[1]。高粱种类丰富,用途广泛,是重要的粮食、饲料和生物能源原料。据统计,中国高粱种植面积有70万~80万hm2[2]。在贵州,高粱具有很高的经济价值,主要用于酿造高档酱香白酒[3]。目前,贵州高粱育种主要面临种质资源贫乏、遗传基础狭窄、品种退化、抗性差、育种技术相对落后等问题[4],因此,对地方种质资源进行遗传多样性分析及聚类十分必要,明确不同种质资源间的亲缘关系可以为优良品种的筛选和针对性引进优良品种提供基础参考。
随着分子生物技术的发展,SSR分子标记技术由于具备多态性高、重复性好、稳定可靠等特点,广泛应用于多种作物的研究中[5]。邵健丰等[6]利用SSR分子标记构建了包含12个连锁群的帚高粱遗传连锁图谱;Atta Ullah等[7]通过SSR分子标记对56个大豆品种间的分子多样性进行分析,结果显示存在显著差异;倪先林等[8]利用26对SSR引物对29份糯高粱种质资源的遗传多样性进行了分析,将29份糯高粱品种聚为两大类。在作物的遗传差异研究中,聚类分析是一种将研究对象分为相对同质的群组的统计分析技术,主要应用于探索性的研究[9],具有简单、直观的优点,在作物研究中常被用来对表型或基因进行分类,获取对种群固有结构的认识。赵庆勇等[10]采用SSR分子标记和表型性状2种聚类方法对30个杂交粳稻亲本的遗传多样性进行比较研究,并分析了这些亲本的遗传差异;田承华等[1]利用SSR分子标记与表型性状进行关联分析找出了高粱中与5个表型性状相关联的4个标记。胡齐赞等[11]基于表型和分子标记对85份芥菜地方种质资源进行聚类分析,丰富了芥菜的遗传基础。
本研究通过对26份贵州地方高粱种质材料进行SSR分子标记和表型性状的遗传多样性分析,为贵州地方高粱杂交亲本选配和分子标记辅助育种提供参考。
1 材料与方法
1.1 试验材料
供试的26份贵州地方高粱种质材料由国家小麦改良中心贵州分中心等单位提供,材料编号、名称及来源详见表1。

表1 26份贵州高粱地方种质资源及来源
1.2 试验方法
1.2.1田间设计
2020年6月将供试材料种植于贵阳市花溪区,国家小麦改良中心贵州分中心试验田,试验田选址环境、水肥管理一致。采用随机区组设计,每区3行,设3次重复,株距18 cm,行距12 cm。
1.2.2田间表型性状调查
成熟后进行表型性状调查,每份材料中间随机取10株,记录样株株高、穗长、穗宽、叶片数、旗叶宽。收获后对调查样株进行试验室考种并记录单穗粒重和千粒重。
1.2.3DNA提取
采用CTAB法[12]提取DNA,经1%琼脂糖凝胶电泳检测其质量和浓度合格后-20 ℃保存备用。从60对SSR引物中筛选出13对多态性引物,标记序列信息来源于文献[1]、文献[12]、文献[13]。PCR反应体系20.0 μL:模板DNA 4 μL、10×PCR buffer(mg2+Plus)2 μL、dNTPs 0.4 μL、5 UTaqDNA聚合酶0.5 μL、上下引物各1 μL, 加ddH2O补足20 μL。引物名称及序列详见表2。PCR扩增产物采用10%非变性聚丙烯酰胺电泳进行检测,银染并拍照[14]。
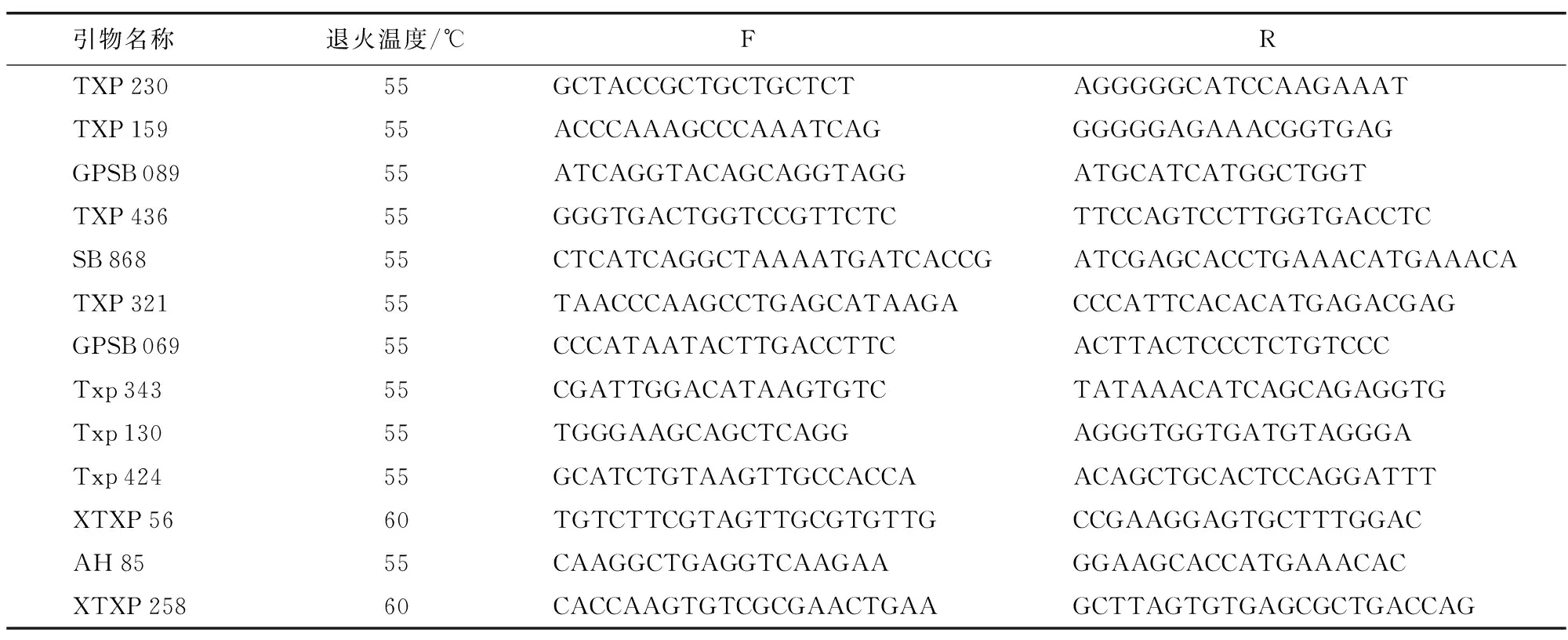
表2 13对引物名称及序列
1.3 统计分析
采用Excel 2003软件、SPSS 21.0软件对各表型性状进行变异性分析和相关性分析,用系统聚类组内联接法进行聚类分析, 聚类结果树状图采用欧式距离绘制。根据PCR扩增产物在10%非变性聚丙烯酰胺凝胶电泳的结果,对条带进行统计,相同迁移位置上,有条带记为1,无条带记为0,建立0/1矩阵,应用NTSYS 2.10 e软件中UPGM法进行聚类分析。根据软件需要将初始数据进行转换,利用POPGENE软件、PIC Cale 0.6软件计算SSR分子标记引物的Shannon信息指数(I)、等位基因数(Na)、有效等位基因数(Ne)、及平均多态性信息量 (PIC)。
2 结果与分析
2.1 SSR分子标记结果
对SSR分子标记结果进行统计分析(表3),数据显示:13对SSR引物共检测出26个等位基因,平均等位基因为2个;有效等位基因数变幅为2.00~1.35,均值为1.68,表明等位基因在群体中分布较均匀;Shannon信息指数变幅为0.43~0.67,均值为0.58;多态性信息含量变幅为0.23~0.37,均值为0.31;观测杂合度变幅为0.49~0.73,均值为0.60,表明杂合位点比例较高;期望杂合度变幅为0.27~0.51,均值为0.40,表明参试材料存在杂合。
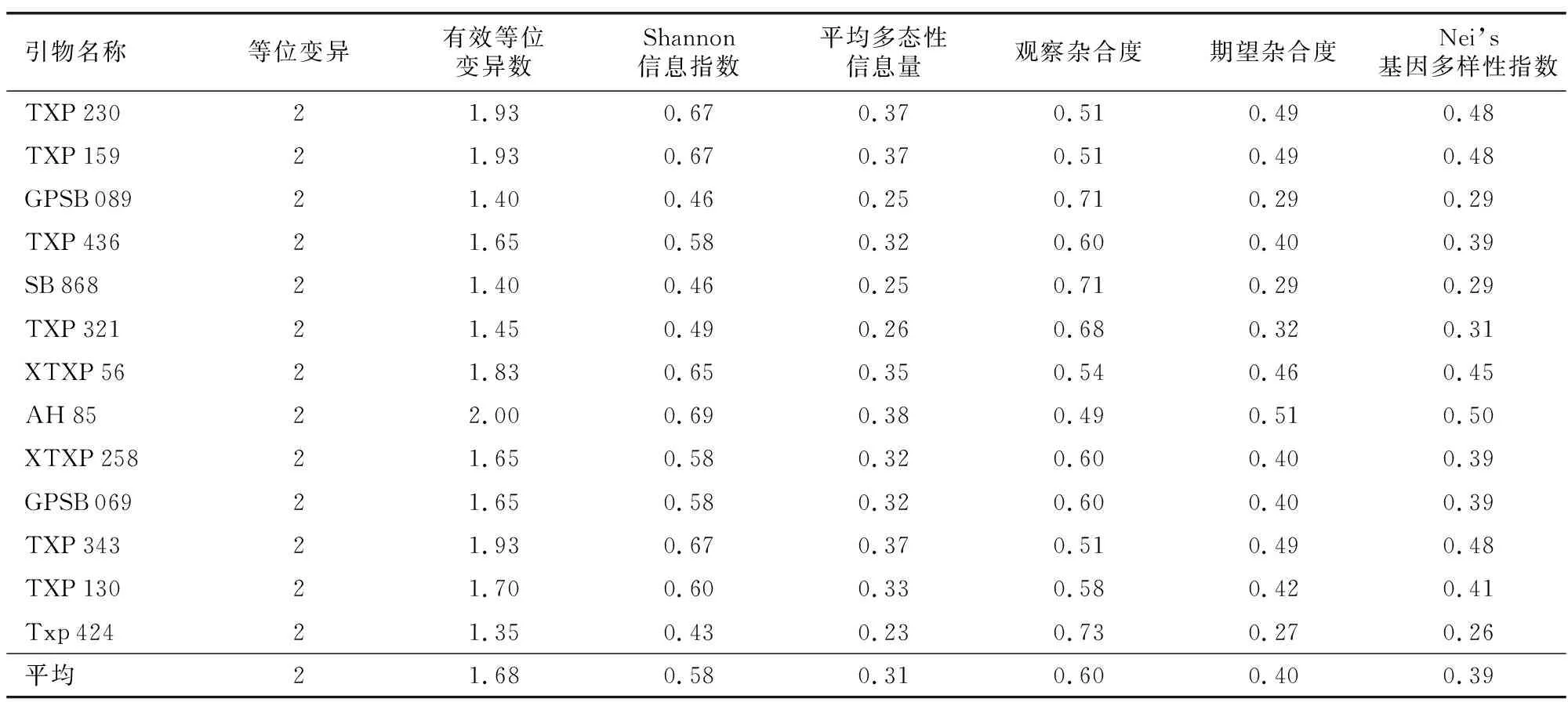
表3 13对引物的多态性
2.2 基于SSR分子标记的高粱种质聚类分析
利用UPGM法对26份供试材料进行聚类分析(图1),结果显示,26份高粱种质的遗传相似系数范围在0.31~0.93之间,在遗传相似系数为0.457时将高粱材料分为四个类群,第Ⅰ类群包含19份材料,遗传相似系数范围在0.53~0.92之间;8、15、16号材料聚为第Ⅱ类群,遗传相似系数范围在0.46~0.54之间;20号材料单独划分为第Ⅲ类群;第Ⅳ类群包含3份材料,遗传相似系数范围在0.54~0.62之间。数据表明,第Ⅳ类群与其他三个类群间的遗传相似性较低,表明类群中种质资源的亲缘关系较远,可以为杂交亲本的选择提供参考;19号茅粱糯2号是通过贵州省农作物品种审定委员会审定的品种(编号:黔审粱2016001),与21号茅台红1号是近似品种(系)材料,遗传相似系数为0.92,二者的聚类结果与其亲缘关系相符。
2.3 表型性状变异性及相关性分析
对供试高粱材料的株高、穗长、穗宽、叶片数、旗叶长、旗叶宽、单穗粒重和千粒重8个表型性状进行变异性分析(表4)。结果显示,株高、穗长和千粒重的偏度小于0,说明这三个表型性状调查结果分布呈负偏态,而其余表型性状偏度均大于0,说明分布呈正偏态。供试材料表型性状变异系数均在10%以上,变异系数范围在13.09%~44.46%之间,均值为22.70%,其中穗粒重变异系数最大,高达44.46%。数据表明供试高粱材料存在遗传变异,且穗粒重存在较大程度的遗传变异,性状选择潜力最大,在选育品种籽粒大小方面有更多的选择,在今后的遗传育种中具有较高的应用价值。
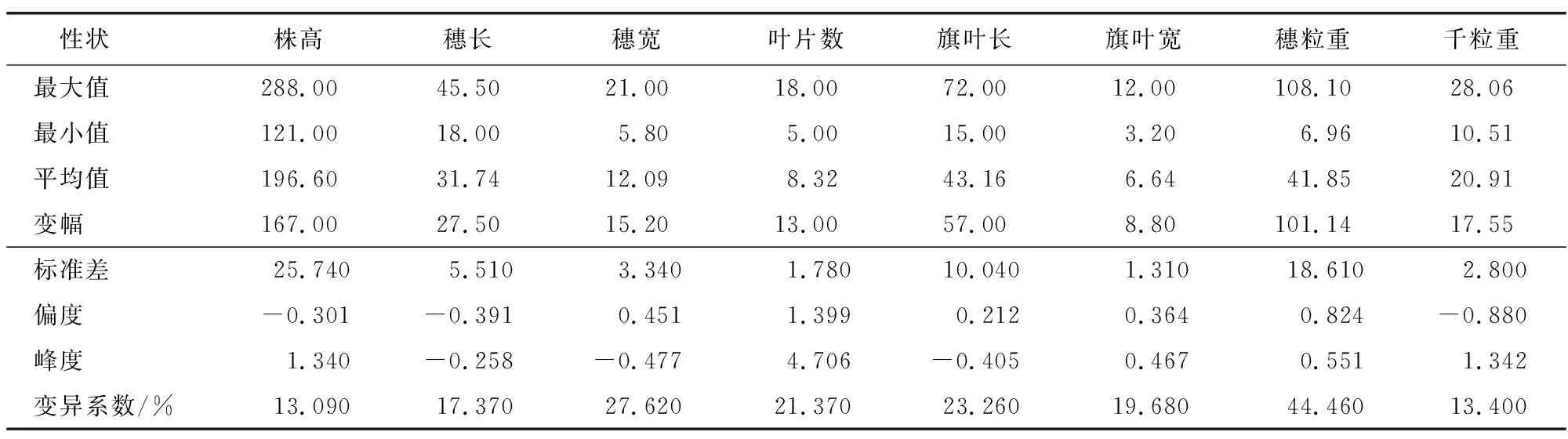
表4 26份高粱种质资源的表型性状表现与分布
对各表型性状进行相关性分析(表5),结果显示:株高、穗长、叶片数、旗叶长和旗叶宽都与穗粒重呈极显著正相关,相关系数分别为0.311、0.324、0.353、0.302、0.277;千粒重与穗宽呈负相关,相关系数为-0.111,与株高呈显著正相关,相关系数为0.128,与穗粒重呈极显著正相关,相关系数为0.291。以上说明供试高粱的株高、穗长、叶片数、旗叶长和旗叶宽对穗粒重的增加有重要影响,穗粒重随着株高、穗长、叶片数、旗叶长和旗叶宽的增加而增加,千粒重随着株高和穗粒重的增加而增加。
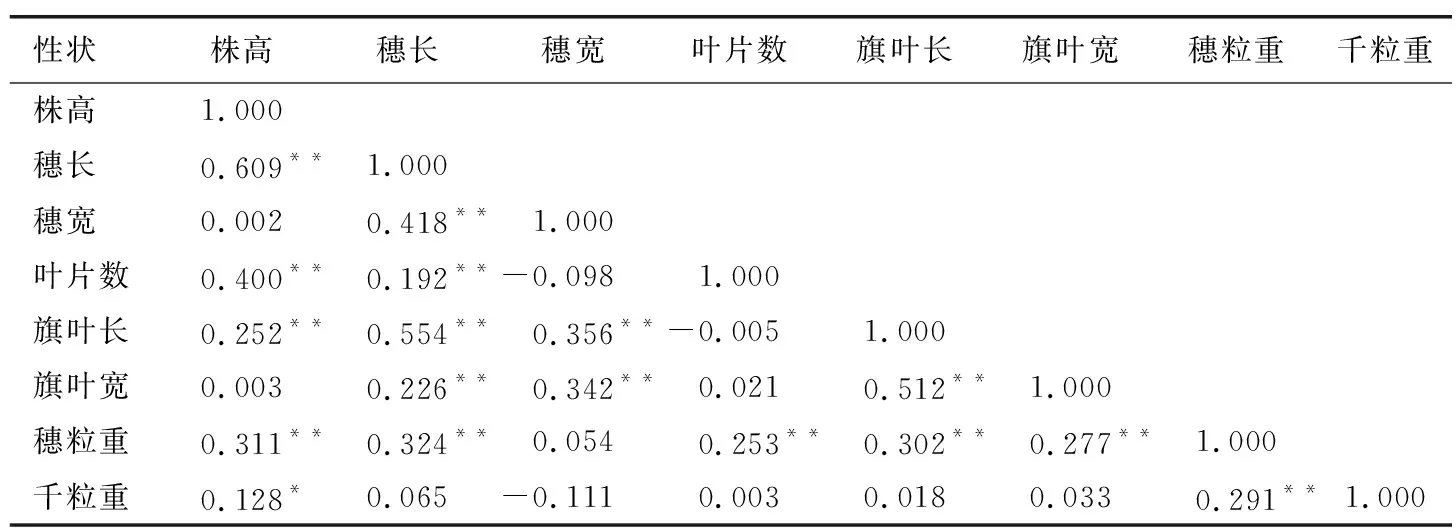
表5 高粱8个表型性状间的 Pearson 相关系数
2.4 基于表型性状的高粱种质聚类分析
基于表型性状进行聚类分析, 用欧式距离绘制聚类结果树状图(图2),结果显示,26份高粱材料在欧式距离为13.5处被分为五大类群,由图2可看出,8、15、16号聚为第Ⅰ类群,这3份材料均属于矮杆、短穗、穗粒重和千粒重中等的种质;第Ⅱ类群有14份材料,均为高杆、长穗、穗粒重和千粒重中等的种质;第Ⅲ类群有7份材料,均为高杆、长穗、穗粒重和千粒重较高的种质;23号材料单独聚为第Ⅳ类群,为高杆、长穗、穗粒重和千粒重高的种质;6号材料也单独聚为第Ⅴ类群,为中高杆、短穗、单粒重和千粒重较低的种质。
3 讨 论
种质资源遗传多样性是进行品种遗传改良的重要前提,目前研究植物遗传多样性多以单独表型性状或分子标记为切入点。前人研究表明,在遗传多样性分析过程中,将表型性状与分子标记相结合进行遗传多样性分析的结果更具有客观性及准确性[15]。本研究通过SSR分子标记和表型性状分析2种方法对贵州地方26份高粱种质资源的遗传多样性进行研究。对两种聚类结果进行比较发现,聚类结果相似,分子标记聚类方法将26份参试高粱材料分为四个类群,第Ⅰ类群的材料包含在表型性状聚类的第Ⅱ类群和第Ⅲ类群中,第Ⅱ类群的材料与表型性状聚类的第Ⅰ类群完全相同,第Ⅳ类群的材料包含了表型性状聚类的第Ⅳ类群和第Ⅴ类群。但两种聚类方法又有所不同,分子标记的第Ⅲ类群单独聚为一类,与表型聚类结果不同,第Ⅳ类群中有一个材料与表型性状聚类结果完全不同,原因在于表型性状是基因型和环境互作的结果,基因在品种引种、选配过程中受环境和人为因素等外部条件影响会产生表达差异,这与赵文杰等[16]研究结果一致。
分子标记结果显示,每对SSR引物平均检测到2个等位基因,其平均H为0.39,平均PIC为0.31,与田承华等[1]、王芳等[13]的研究结果相比,所检测到的平均SSR位点及PIC均偏低,可能与本研究供试的高粱种质资源较少且遗传基础狭窄有关。表型性状分析结果显示,株高对高粱穗粒重和千粒重有重要影响,穗粒重与千粒重呈极显著正相关,表明在适当范围内增加植株高度可以提高高粱产量,这一结果与高杰等[17]研究结果一致。
本研究目的在于通过分子标记和表型性状两方面对贵州地方高粱种质资源进行遗传多样性分析,为后续高粱的杂交亲本选配和分子标记辅助育种提供参考,但由于供试材料较少,只能提供少数种质的参考,若需要了解更多品种间的遗传多样性及亲缘关系,则需要进一步研究。
