野大麦的分子核型分析
2021-09-23李景环
冯 娜, 李景环
(内蒙古师范大学生命科学与技术学院, 呼和浩特 010020)
野大麦(HordeumbrevisubulatumL.)为禾本科大麦属的多年生草本植物[1],在我国,分布于东北、内蒙古、陕西北部、宁夏、甘肃、青海、新疆、西藏等省区[2]。野大麦适口性好、适应性广且具有较强的耐盐性,营养丰富,粗蛋白含量高,是优良饲草。此外,野大麦有较强的耐盐碱、耐寒、抗病虫害等能力,是禾本科小麦族耐盐性最强的盐生植物之一[3-4],可用于改良盐碱地,具有很高的经济价值和饲用价值。一直以来小麦和牧草育种学家选择野大麦作为优良的基因资源,对小麦和饲草进行改良,育成了具有耐盐碱的新品种。杂交育种离不开细胞遗传学的指导,染色体的核型分析就是一种细胞遗传学分析方法。
染色体核型又称染色体组型,核型分析是对细胞有丝分裂中期染色体的各种特征进行定性和定量表述的一种分析方法,包括染色体数目、染色体形态、染色体分子特征等[5]。染色体核型分析广泛应用在物种系统分类、起源与鉴定、亲缘关系分析等方面,而高质量的染色体制片和染色体上清晰可辨的标识是准确进行核型分析的保障[6]。
荧光原位杂交技术可以为核型分析提供清晰的标识,该技术是将荧光素标记的探针代替同位素标记与待测样品中的待测序列以碱基互补配对原则结合在一起,常用的标记物有生物素标记、地高辛标记等。目前,荧光原位杂交技术已经成为一种非常重要的原位杂交技术[7],凭着其特征显著、分辨率高、稳定性强、操作安全便捷等特点,已经应用于产前诊断[8-9]、唐氏综合征、膀胱癌等[10-11]临床医学,以及染色体结构变异的检测和标记基因定位[12]等生物学领域,在细胞遗传学、表观遗传学、分子生物学等方面也有较高的研究价值。在高等真核生物中,45 SrDNA是高度保守的序列,是编码rRNA的基因[13]。45 SrDNA在核仁组织区上[14],一般位于随体染色体的核仁组织区[15],5 SrDNA是编码5 SrRNA的基因。重复序列pAs 1和pSc 119.2在小麦族植物染色体中广泛存在,是微卫星序列。45 SrDNA、5 SrDNA、pAs 1和pSc 119.2序列是植物染色体原位杂交实验中常用的探针序列[16-18]。
以野大麦为试验材料,采用45 SrDNA、5 SrDNA、pAs 1和pSc 119.2这4种序列制备的探针,与野大麦的根尖细胞有丝分裂中期染色体进行荧光原位杂交并分析,构建核型,确立各个标记信号在染色体上的分布特点,以期为野大麦的系统研究和杂交育种提供理论参考,同时为大麦属植物以及小麦族近缘种植物的分类学和细胞学等研究提供基础资料。
1 材料与方法
1.1 材 料
本试验使用的野大麦种子由内蒙古师范大学生命科学与技术学院李景环老师提供。
1.2 方 法
1.2.1野大麦体细胞染色体标本制备
选取野大麦饱满充实的种子放入垫有湿滤纸的培养皿中,放于最适温度为25 ℃的培养箱中培养,待根部长到1.5 cm左右时,于上午剪取根尖作为试验材料。冰水混合物进行预处理,预处理结束后用蒸馏水洗净,置于卡诺固定液固定8 h, 2%的纤维素酶进行解离,改良石炭酸品红染液染色1 h。之后压片和镜检,选取染色体清晰可见的装片,冰冻揭片,梯度乙醇脱水,备用。
1.2.2探针标记
碱裂解法分别提取携带45 SrDNA、5 SrDNA、pAs 1 DNA和 pSc 119.2 DNA的质粒DNA,采用DNA缺口平移法,用地高辛-dUTP标记5 SrDNA探针和pAs 1 DNA探针,生物素标记45 SrDNA和pSc 119.2 DNA探针。4种探针由南京农业大学提供。
1.2.3荧光原位杂交
利用45 SrDNA、5 SrDNA、pAs 1 DNA和pSc 119.2 DNA探针与野大麦的根尖中期染色体进行原位杂交,原位杂交具体步骤参照李景环等[19]的方法,用Olympus BX 51荧光显微镜观察并拍照保存。
1.2.4核型分析
对获得的图片和数据等进行处理,选择30个以上染色体分散良好的中期分裂相细胞进行染色体计数,85%的细胞中染色体数目一致,则该数目确定为该物种的染色体数。选择染色体分散清晰的5个细胞的分裂相进行核型参数的测量和计算,测量染色体长臂值和短臂值,并计算染色体的相对长度和臂比,最后取5个细胞的各项参数平均值作为核型分析的参数。同时结合荧光原位杂交获得的标记信号,对染色体进行同源配对,构建核型分析图和核型公式,核型分析的方法一般参照李懋学和陈瑞阳[20]的标准进行,以Levan等[21]的公式计算相对长度,核型分类采用Stebbins[22]的分类标准进行。
采用染色体分析软件Karyo 3.1进行长度测量,绘制染色体核型图。利用Excel软件计算相对长度等参数,并作出核型参数表。
2 结果与分析
2.1 染色体数目分析
根据染色体核型分析标准,85%以上的细胞具有的染色体数目确定为是该物种的染色体数目[20]。在本实验中,选择30个中期分裂相细胞进行计数得出,野大麦根尖细胞染色体数目为2 n=28,如图1和图2。
2.2 荧光原位杂交标记信号分析
选取5个染色体分散清晰的细胞,用Olympus BX 51进行图片采集,再用Karyo 3.1软件进行长度测量,计算核型参数,同时结合四种探针的荧光原位杂交标记信号的特征,进行同源染色体配对,得到野大麦根尖细胞染色体原位杂交图和染色体核型图,见图1和图2。
由图1和图2可知,野大麦的染色体数目为2 n=28条。45 SrDNA的数目与具有随体的染色体数相同[23],因此野大麦有6对具有随体的染色体,分别位于第6号、第10号、第11号、第12号、第13号和第14号染色体的短臂上。
通过观察图1和图2可以看到,在Olympus BX 51荧光显微镜采集的图片中,用地高辛-dUTP标记的5 SrDNA探针和pAs 1 DNA探针颜色为红色;生物素标记的45 SrDNA和pSc 119.2 DNA探针颜色为绿色。45 SrDNA和5 SrDNA探针同时与野大麦根尖中期染色体进行原位杂交,如图1;pAs 1 DNA和pSc 119.2 DNA探针同时与野大麦染色体进行杂交,如图2;DAPI复染后的染色体呈现为蓝色。由图1可以看到,5 SrDNA位于第10号和第14号染色体的短臂上;45 SrDNA定位在6对染色体上,分别定位于第6号、第10号、第11号、第14号染色体的短臂上,信号较强,第12号、第13号染色体上的45 SrDNA位于近着丝粒的位置上,但是第12号染色体上的45 SrDNA荧光信号强,而第13号染色体上的45 SrDNA荧光信号较弱;第10号和第14号染色体上同时出现了45 SrDNA和5 SrDNA的双色杂交信号。pAs 1 DNA的荧光信号出现在所有的染色体上,大部分位于染色体的端部,第7号和第9号染色体的pAs 1 DNA则位于中部且信号较弱; pSc 119.2 DNA分布于第7号、第10号和第14号共3对染色体上,且都位于染色体短臂的末端,但信号强弱不同,在第7号染色体上pSc 119.2 DNA信号较强,第10号染色体的一条染色体的短臂末端信号较强,但是另一条染色体没有出现荧光信号,第14号染色体的pSc 119.2 DNA信号较弱;pAs 1 DNA和pSc 119.2 DNA双色杂交位点出现在第7号、第10号和第14号染色体上。
2.3 染色体核型参数
核型参数可以反映一个物种的进化程度,核型是由对称向不对称发展[22]。野大麦核型参数的计算,是通过Karyo 3.1软件对染色体的长臂、短臂进行测量,然后用Excel软件计算染色体相对长度和臂比,根据臂比确定染色体的着丝粒类型。野大麦中期染色体的核型参数见表1。
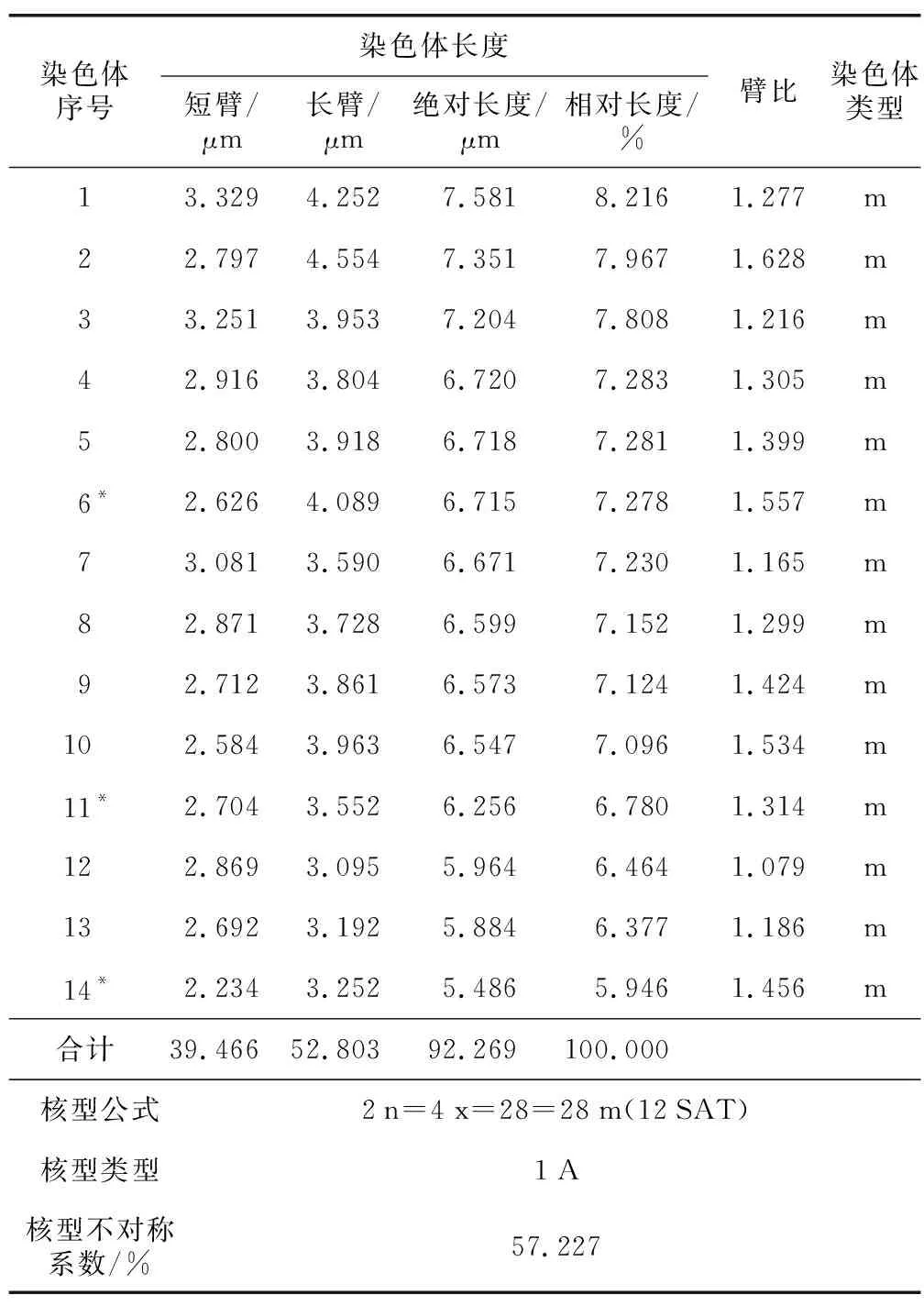
表1 野大麦染色体核型参数
由表1可以看出,野大麦的根尖细胞染色体数目为2 n=28,染色体组总长度为92.269 μm,平均绝对长度为6.591 μm,染色体的绝对长度变化范围为7.581~5.486 μm,染色体的相对长度变化范围为8.216%~5.946%,最长染色体与最短染色体长度比值为1.382;臂比最大值为1.628,最小值为1.079,根据着丝粒位置来分,野大麦的染色体都为m型(中部着丝粒),没有出现臂比值大于2的染色体,核型类型属于1 A型(最对称的核型),核型不对称系数为57.227%。核型公式为2 n=4 x=28=28 m(12 SAT)。
3 讨 论
3.1 荧光原位杂交
荧光原位杂交技术具有灵敏度高、特异性强等优点,可以在同一个染色体片上显示出不同的DNA探针的定位,被广泛应用于各种染色体检测、定位研究中[24]。在试验过程中,最为重要的就是特异性探针序列的选择[24],本研究选择小麦族的45 SrDNA序列、5 SrDNA序列、高度重复序列pAs 1 DNA和pSc 119.2作为探针,这些探针常用来分析小麦族物种的亲缘关系和进化[23-26]。
孔芳等[23]认为,45 SrDNA 的数目与具有随体的染色体数相同,在染色体的次缢痕处,以此确定野大麦有6对染色体有随体。Mantovani等[27]认为,5 SrDNA原位杂交位点数目以一对或两对为主,但在染色体上没有稳定不变的分布位置,这与本研究5 SrDNA的定位结果基本一致。rDNA是进化上高度保守的序列,尤其是在同一物种的同源染色体上的位置和拷贝数基本相同,这为核型分析进行染色体的配对提供了清晰可见的标识,利于准确配对。但是在同属不同物种中rDNA的位置和拷贝数会有所不同,这可以用于物种的分类和演化研究[23]。
pAs 1 DNA序列和的pSc 119.2 DNA在遗传上也是相对保守的,尤其体现在分布位置上。pAs 1 DNA一般集中分布在端部和着丝粒处,对于近缘物种而言都位于大致相同的位置,但是在不同的物种中会表现出不同的拷贝数;本实验显示pSc 119.2 DNA分布于野大麦部分染色体的末端,且信号较强。从本实验结果看出,pAs 1 DNA和pSc 119.2 DNA可以作为一种荧光标记来进行染色体同源配对。
有了明显的荧光标记可以推断染色体的进化,本实验中10号染色体出现了一条没有标记pSc 119.2信号的结果,再结合45 SrDNA和5 SrDNA的标记结果配对,推测10号染色体可能是发生了染色体顶端的缺失。
3.2 野大麦核型
染色体核型分析对植物的系统分类、物种的遗传进化提供了基础[28]。丁鸿等[29]在2012年发表了植物染色体标本的制备和染色体核型分析研究进展,认为核型分析技术是细胞遗传学中最基本最常用的方法,在植物分类、起源、演化等方面都有重大意义,也为以后核型分析的发展做出了展望。本研究根据检测到的45 SrDNA和5 SrDNA荧光原位杂交信号的数量、强弱和位置等,成功将野大麦6对染色体配对,提高了染色体配对的精确性,为野大麦染色体核型模型的建立提供了支持。pAs 1 DNA序列和pSc 119.2 DNA也为核型分析提供了明显的可视标记。由此可见,荧光原位杂交技术使常规的染色体核型分析更为便捷准确。
有关野大麦染色体核型的研究于立华等[30]得到的核型公式为2 n=4 x=28=22 m(4 SAT)+6 sm(2 SAT),本研究结果为2 n=4 x=28=28 m(12 SAT)。两种结果除了染色体数目相同外,其他的核型特征相差很大。表现在具随体的染色体、着丝粒类型、染色体的对称类型。于立华的结果为6条染色体具随体,本研究具随体的染色体为12条,随体的分布除了与物种本身特征有关外,与制片的方法以及染色体识别的方法也有直接关系。常规染色体制图得到的染色体图片,如果随体特别小,就很难识别;孔芳等[23]认为,45 SrDNA的数目与具有随体的染色体数相同,所以带有45 SrDNA探针标记的染色体容易识别,本研究的随体就是结合45 SrDNA探针进行判断的。关于染色体的着丝粒类型,于立华的研究结果是11对中着丝粒染色体,3对近中着丝粒染色体,本研究的结果都是中部着丝粒染色体,对于着丝粒的判断,如果仅靠染色体形状判断一定会有误差,最好的办法是采用着丝粒处序列的荧光标记,本研究和于立华的着丝粒判别都是根据主缢痕的凹陷进行判断的,会存在一定的误差,因此结果不一致。根据Stebbins的核型分类标准将野大麦确定为1 A型,最对称的核型,于立华的结果2 B类型,这个结果主要依赖于染色体长度测量的准确性和配对的准确性,当然造成差异的原因有很多,首先可能是操作手法和处理方法、使用试剂上会有不同导致结果的不同,对数据的处理也有不同。因此要想更准确的进行核型分析,要保证染色体的特征明晰,测量方法误差小,可识别的荧光标记种类适当并且容易辨别,要想更准确地进行核型分析,还应该进行更多标识的探针杂交。
4 结 论
5 SrDNA位于第10号和第14号染色体的短臂上;45 SrDNA位于第6号、第10号、第11号、第12号、第13号和第14号染色体的短臂上;pAs 1 DNA的荧光信号出现在大部分染色体的端部和部分在着丝粒区域;pSc 119.2的荧光信号出现在第7号和第14号染色体短臂的顶端,第10号染色体一条短臂的顶端,另一条染色体可能顶端缺失导致没有出现pSc 119.2荧光信号。
核型公式为2 n=4 x=28=28 m(12 SAT),核型类型属于1 A型,为最对称的核型,核型不对称系数为57.227%,在系统演化上为较原始的种类,进化程度低。
适当全面的荧光标记种类使染色体核型分析更为准确便捷。
