小麦茎基腐病抗性QTL的分析
2021-09-23周淼平杨学明姚金保
周淼平,张 鹏,杨学明,陈 达,姚金保
(江苏省农业科学院粮食作物研究所,江苏南京 210014)
小麦茎基腐病(wheat crown rot,WCR),又称镰刀茎基腐病(Fusarium crown rot),是发生在干旱和半干旱地区的世界性小麦病害,该病害在澳大利亚、新西兰、南美洲、美国西北部太平洋沿岸、加拿大、意大利、北非、中东和中国均有发生[1]。在澳大利亚该病害主要由假禾谷镰刀菌(Fusariumpseudograminearum)引起[2],在中国主要由禾谷镰刀菌(F.graminearum)和亚洲镰刀菌(F.asiaticum)引起,但近几年假禾谷镰刀菌呈上升趋势[3]。病原菌首先侵染小麦的茎基部,在苗期茎基部叶鞘和茎秆出现褐变,严重时引起根部褐变腐烂,造成死苗。后期随着小麦的生长,病原菌沿茎秆向上扩展,使小麦多个节和节间出现褐变,严重时造成枯白穗,从而导致小麦减产。感染茎基腐病的小麦籽粒中还可能残留DON等毒素[4],影响小麦的食用、饲用和种用价值。由于该病害的流行,在澳大利亚,小麦和大麦每年的损失超过9 700万澳元[2];美国西北部太平洋沿岸发病严重田块,产量损失可达35%[5]。近年来,由于常年实施秸秆还田,造成土壤中菌源积累,我国河北中南部、山西南部、陕西中东部、河南和山东大部、江苏和安徽北部的小麦茎基腐病有加重趋势,河南沁阳以及江苏沿海部分田块小麦损失高达30%以上[3,6]。
目前对小麦茎基腐病的防治主要采用农业措施与药剂防治相结合的方法,不仅增加农业成本,而且污染环境,不利于农业生产的可持续发展,因此,种植抗病品种无疑是防治小麦茎基腐病最经济和有效的途径。抗病品种的培育需要抗性稳定的抗源用于杂交配组以及明确的抗性机理指导育种,国内外研究人员对小麦茎基腐病的抗性鉴定方法、抗源的筛选以及抗性机理的解析方面已经取得了一些研究成果。前人采用病麦粒接种、菌液直接浸泡接种等方法先后建立了田间成株期以及室内苗期的抗性鉴定方法;并运用这些鉴定方法对3 400余份常规小麦、硬粒小麦、小黑麦和野生小麦进行了茎基腐病的抗性筛选,筛选到2-49、Sunco、CI12633等一批中抗材料,但没有发现免疫和高抗材料[2,7-10]。遗传分析表明,小麦对茎基腐病的抗性是一个数量性状[11-19]。Wallwork等[11]首先在Kukri的4B染色体上发现抗性主效QTL,随后科研工作者分别对茎基腐病抗源2-49[12-14]、W21MMT70[13,15]、CSCR6[16]、Sunco[13-14]、Ernie[17]、Maco[18]、Otis[18]、IRN497[14]、CPI133814[14]和EGA Wylie[19]的抗性QTL也进行了定位,发现这些QTL分布在小麦的13条染色体上,其中3BL和4B染色体上存在抗性主效QTL,最高可分别解释48.8%[16]和48.0%[11]的表型抗性。Erginbas-Orakci等[20]采用自然群体和DArT标记进行关联分析,在3B和2D染色体上也发现抗茎基腐病QTL。通过分子标记辅助选择,可以将主效QTL和微效QTL进行聚合,从而提高小麦对茎基腐病的抗性,有效降低小麦茎基腐病的危害,如Bovill等[13]将2-49的1D染色体抗性QTL与W21MMT70的3B染色体抗性QTL 聚合后,发现聚合植株茎基腐病严重度比不含抗性QTL的植株降低51.2%。
已经报道的小麦茎基腐病QTL定位研究多采用SSR等分子标记来构建遗传连锁图,这些标记多态性差且数量有限,影响遗传连锁图的基因组覆盖度,从而降低了小麦茎基腐病抗性QTL的检出效率以及QTL定位的精度。近几年,随着二代测序技术的发展以及小麦基因组的拼接完成,开发大量的适合高通量检测的SNP标记成为可能并得到广泛使用,也为小麦茎基腐病抗性QTL的精确定位提供了方便。Yang等[21]利用660K SNP芯片,采用GWAS(genome-wide association studies)方法对234个黄淮麦区小麦品种进行分析,在6A染色体上发现小麦茎基腐病抗性QTL。采用同样方法,Jin等[22]利用55K SNP芯片对358个小麦品种(系)进行关联分析,在5DL 染色体上发现茎基腐病抗性QTL。
本研究采用中抗茎基腐病品种CI12633与感茎基腐病品种扬麦158构建重组自交系群体,用二代测序技术开发SNP标记,结合已有的SSR基因型资料以及群体茎基腐病抗性鉴定结果,以期获得茎基腐病抗性QTL位点,为今后开展抗茎基腐病小麦的分子标记辅助育种奠定基础。
1 材料与方法
1.1 供试材料
CI12633品种是提莫菲维小麦(FriticumtimopheeviiZhuk.)的六倍体衍生系,我国作为白粉病抗源引进,研究发现该品种不仅含有多个抗白粉病基因,而且抗小麦黄花叶病,中抗小麦茎基腐病和小麦纹枯病。扬麦158为江苏里下河地区农业科学研究所培育的品种,曾在长江中下游地区大面积种植,是综合抗性好的广适性品种,中抗小麦白粉病和小麦赤霉病,但感小麦茎基腐病。
以扬麦158为母本,CI12633为父本,配制杂交,采用单粒传的方法培育重组自交系群体,该群体含有198个家系,本研究对其中94个家系进行了基因型分析和茎基腐病抗性鉴定。
1.2 遗传连锁图的构建
通过二代测序方法开发该重组自交系群体的SNP标记,结合已有的群体多态性SSR标记,采用JoinMap 4.0软件构建遗传连锁图。SNP标记的开发以及遗传连锁图的构建见周淼平等[23]的方法,该遗传图谱总遗传距离为2 510.7 cM,包含31个连锁群,含有3 355个分子标记,通过已有的SSR和小麦基因组信息,可以将所有连锁群分配至相应的染色体。
1.3 重组自交系群体的茎基腐病抗性鉴定
群体茎腐病的抗性鉴定在实验室内进行,鉴定方法参照Liu等[24]的方法并略作修改,小麦种子经70%乙醇消毒2 min后,用无菌水冲洗3~4次,置于垫有湿滤纸的培养皿中催芽,待芽长为5 mm左右时,在禾谷镰刀菌孢子悬浮液(含1×106个孢子·mL-1)中接种1~2 min,接种后的种子呈直线均匀排列在灭过菌的湿润纸巾(24×24 cm)上,芽的生长方向一致且均指向纸巾边缘(种子距纸巾边缘2 cm左右),将纸巾沿种子排列方向,从一端向另一端卷起,芽的生长方向向上置于塑料容器中,22 ℃、90%湿度培养14~16 d,调查茎腐病发病情况。
根据病害严重度将茎腐病病级分为0~5级:0级为叶鞘无病症;1级为第一叶鞘病斑长度小于1.0 cm;2级为第一叶鞘病斑长度在1.0~2.0 cm之间;3级为第一叶鞘病斑长度大于2.0 cm,幼苗未萎焉;4级为幼苗出现萎焉病症;5级为幼苗死亡。
群体的每个家系调查10~15株幼苗,计算平均病级,每个试验重复2次,2次重复的平均病级用于数据分析和QTL定位。茎基腐抗性鉴定进行3次技术重复试验,分别称为E1、E2和E3。
1.4 数据分析和QTL定位
采用R软件计算重组自交系群体和亲本茎基腐病抗性鉴定的数据平均值、频率分布、试验间相关性和方差。采用MapQTL 5.0软件进行QTL分析,首先进行QTL区间作图法(interval mapping)进行初步定位,然后采用复合QTL作图法(MQM mapping)进一步详细分析,根据置换检测(permutation test)推荐的LOD值判定是否存在QTL。
2 结果与分析
2.1 群体茎基腐病抗性的鉴定结果
从表1可以看出,3次试验抗病亲本CI12633和感病亲本扬麦158之间的病级差异极显著,群体家系存在超亲分离现象,群体病级分布基本呈正态分布,表明小麦对茎基腐病的抗性是由多基因控制的数量性状(表1,图1)。

图1 群体茎基腐病病级分布
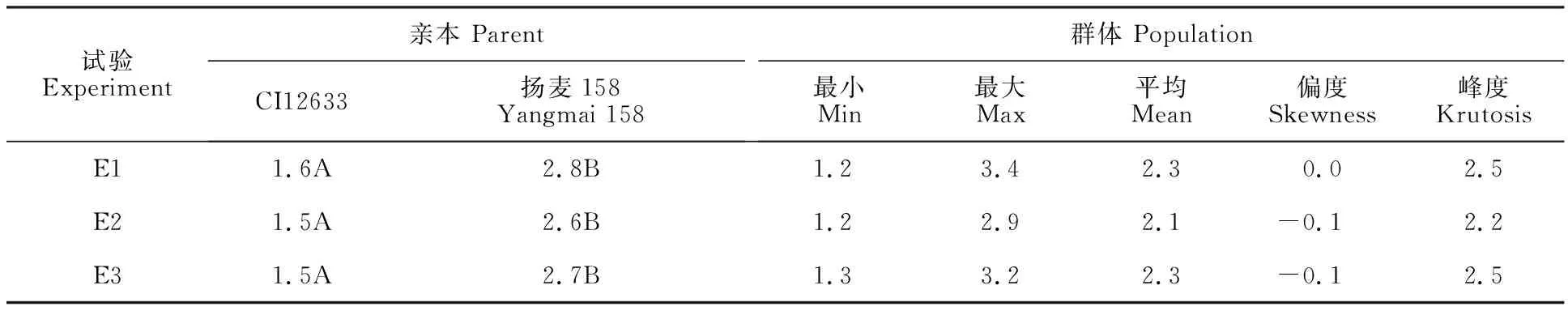
表1 供试小麦群体及其亲本的茎基腐病抗性鉴定结果(病级)Table 1 Identification result(disease grade) of WCR resistance in the population and their parents
群体的3次抗性鉴定试验结果相关性很高,E1与E2、E2与E3、E1与E3的相关系数分别为0.8、0.8和0.6,表明在控温控湿的人工条件下,群体各家系的茎基腐病的抗性在不同试验基本表现一致。方差分析结果(表2)显示,各家系间的茎基腐病抗性差异均达极显著水平,进一步验证群体各家系对茎基腐病的抗性有很大差异。
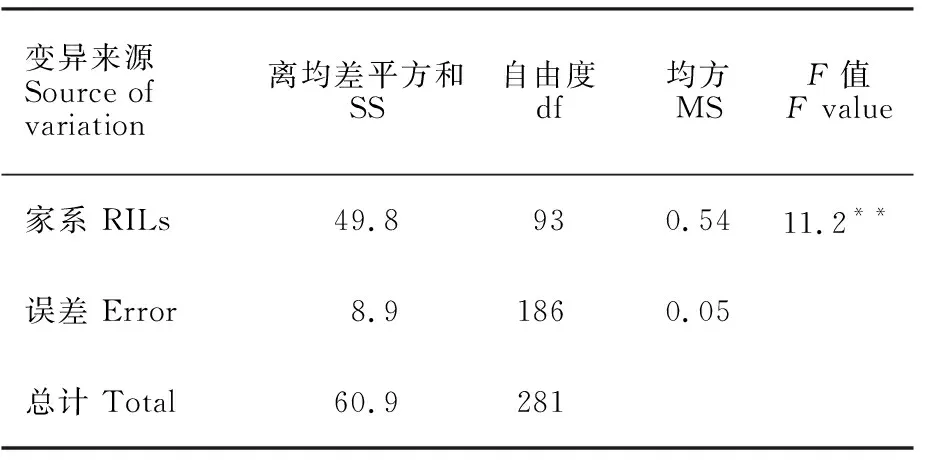
表2 试验的方差分析Table 2 ANOVA of the experiments
2.2 群体茎基腐病抗性QTL的定位结果
结合群体基因型资料和茎基腐病抗性鉴定资料,采用MapQTL 5.0分析软件,分别在1D、2B、3B(2)、7A和7D染色体上定位到6个茎基腐病抗性QTL(图2和表3),其中1D和3B染色体上的抗性QTL来自抗病亲本CI12633,2B、7A和7D染色体上的抗性QTL来自感病亲本扬麦158,单个QTL的表型解释率在9.2%~14.6%之间。1D、2B和3B(与Chr3B_479785994紧密连锁)染色体上的抗性QTL在3次试验中均能检测到,表明这些抗性QTL比较稳定,受环境影响较小,是小麦抗茎基腐病的主效QTL。3B染色体上位于Xgwm181.1-Xgwm340间的QTL和7A染色体上位于Xwmc388-Xgwm635间的QTL只在E1试验中检测到;7D染色体上位于Xgwm37-Xcfa2040间的QTL只在E3试验中检测到,表明这些抗性QTL可能受环境影响较大,只在特定条件下发挥作用。

表3 小麦茎基腐病抗性QTLsTable 3 QTLs for WCR resistance

下划线表示分子标记所在位置。
3 讨 论
3.1 小麦茎基腐病抗性QTL位置比较
澳大利亚茎基腐病的危害较重,因而对小麦茎基腐病的研究较为重视,先后筛选获得了Kukri[11]、2-49[12-14]、Sunco[13-14]、W21MMT70[13,15]、Ernie[17]、CSCR6[16]、EGA Wylie[19]等中抗茎基腐病材料,对这些材料的茎基腐病抗性QTL进行了定位,在3B染色体长臂发现了抗性主效QTL,该QTL最高可解释48.8%的表型变异[16]。本研究在3B染色体上检测到2个茎基腐病抗性QTL,通过小麦物理图谱比对,发现与Chr3B_479785994紧密连锁的QTL与上述文献报道的QTL位置接近,可能为同一QTL;另一QTL位于3B染色体长臂的末端,Poole等[18]也曾在3B染色体长臂Xgwm299-Xgwm181区间检测到抗病QTL,与本研究发现的QTL位置接近,但两者有没有关联,目前尚不清楚。本研究在1D染色体上检测到的茎基腐病抗性QTL与Chr1D_416032418紧密连锁,通过小麦物理图谱比对,发现与2-49[12-14]1D染色体上检测到的QTL位置较为接近,可能为同一QTL。本研究在2B染色体上检测到与Chr2B-518870327紧密连锁的QTL,通过小麦物理图谱比对,发现与Mendos[15]、Sunco[13-14]和Janz[14]中检测到的QTL位置接近,可能为相同抗茎基腐病抗性QTL。本研究在7A和7D染色体上检测到的抗性QTL,与已报道相应染色体的QTL位置不一致,可能为新发现的茎基腐病抗性QTL。
3.2 影响小麦茎基腐病抗性QTL定位的主要因素
小麦茎基腐病抗性的鉴定受环境因素影响较大,在田间,气候条件、小麦生长状况以及小麦纹枯病、小麦根腐病、小麦全蚀病等根部和茎基部病害都会干扰小麦茎基腐病的发生和发展,进而影响抗病QTL的检出效率。从已经报道的茎基腐病抗性QTL表型解释率也进一步验证了这一点。Collard等[12]报道,全部QTL只能解释 40.6%的表型变异;Bovill等[15]报道,检测到的3个QTL也只能解释44.4%的表型变异,这些研究结果说明,由于环境因素和遗传连锁图基因组覆盖度不足的影响,仍有不少QTL未能检出。本研究在室内控温控湿的条件下进行了茎基腐病抗性鉴定,有效减少了由于温度、湿度等环境条件以及其他根部和茎基部病害的影响。另外,采用二代测序方法开发SNP标记,构建高密度群体遗传连锁图,加大对小麦基因组的覆盖度,使得检出的茎基腐病抗性QTL数目较以往研究有所改善,定位精度大幅提高,并且小麦基因组已经测序组装并发布,SNP标记的使用更利于已检出茎基腐病抗性QTL间的比较分析以及候选抗病基因的克隆。
本研究虽然通过控制环境因素以及提高遗传连锁图质量来提高抗病QTL的检出效率,但每次试验检出的QTL只能解释表型变异的40%左右,仍然有不少抗病QTL未能检出,主要原因是进行群体分析的家系数偏少,只有94个,所以,今后的研究需加大群体的家系数目,改良小麦茎基腐病的鉴定方法,进一步提高抗病QTL的检出效率。
4 结 论
采用高密度SNP遗传连锁图结合群体小麦茎基腐病抗性鉴定资料,在1D、2B、3B(2)、7A和7D染色体上检测到6个茎基腐病抗性QTL,单个QTL可解释9.2%~14.6%的表型变异;其中1D和3B染色体的抗性QTL来自CI12633,2B、7A和7D染色体的抗性QTL来自扬麦158;1D、2B和3B(与Chr3B_479785994紧密连锁)染色体上的抗性QTL为稳定的茎基腐病抗性QTL。
