香螺线粒体COXⅠ和CYTB基因遗传多样性研究
2021-09-17张旦旦王煜李卓田莹王荦毛俊霞王许波常亚青郝振林
张旦旦,王煜,李卓,田莹,王荦,毛俊霞,王许波,常亚青,郝振林
(大连海洋大学 农业农村部北方海水增养殖重点实验室,辽宁 大连 116023)
香螺Neptuneacumingii隶属于软体动物门Mollusca腹足纲Gastropoda新腹足目Neogastropoda蛾螺科Buccinida,其壳近菱形,壳质坚硬,颜色为浅褐色,螺层约为7层[1]。香螺为冷水性大型种类,其肉质鲜美肥嫩,营养丰富,富含有人体必需的微量元素[2]。香螺肉可加工成干制品,也可鲜食,深受人们喜爱[3],市场价格近120元/kg,经济价值较高,是非常具有增养殖潜力的螺类。香螺分布范围较小,在中国主要分布于黄、渤海海域[4-5],日本、朝鲜海域也有分布[6]。
目前,关于香螺的研究较少,主要集中于香螺壳质[7]、营养元素[8]、繁殖生物学[9]等方面。近年来,有学者初步开展了香螺遗传多样性的研究,隋娜[10]基于微卫星标记的方法研究了辽宁及山东5个地理群体香螺的遗传多样性,发现地理位置与遗传多样性存在一定的联系。日本学者Azuma等[11]利用分离的多态微卫星标记技术分析了日本北海道海域香螺种群的遗传多样性,发现基因可用于鉴定香螺种群和亲缘关系。然而微卫星技术在PCR扩增时可能会出现无效基因的情况,进而影响群体的鉴定结果[12]。本研究中,通过分析中国黄、渤海海域6个不同地理群体香螺COXⅠ和CYTB基因的遗传多样性水平,试图明确中国香螺遗传多样性水平和种质遗传背景,旨在为香螺健康养殖及其资源保护和种质管理提供科学参考。
1 材料与方法
1.1 材料
试验用香螺于2017年6—10月采自大连市大连湾(DL)、大连市獐子岛县(ZZ)、大连市旅顺盐场(LS)、烟台市八角港(YT)、威海市(WH)、蓬莱市长岛县(PL)6个海域,样本各10尾,用保温箱运回至大连海洋大学农业农村部北方海水增养殖重点实验室。解剖,剪取每尾个体的腹足肌组织,用过滤海水洗净,称重,并于-80 ℃冰箱中保存待用。
1.2 方法
1.2.1 DNA提取、COXⅠ和CYTB基因扩增及测序 剪取香螺腹足肌组织约20 mg,剪碎并装入离心管。采用TIANGEN 海洋动物基因组提取试剂盒提取DNA,用10 g/L琼脂糖凝胶电泳检测PCR产物,并用紫外分光光度计测量DNA的质量和浓度,于冰箱(-20 ℃)中保存备用。
香螺线粒体COXⅠ基因扩增引物为
F:5′ATGCGTTGATTATTTTCKACAAATCA 3′;
R:5′TCTTGAAATCCTARTTGTCCTCATAG 3′。
线粒体CYTB基因扩增引物为
F:5′CTTCCCTTCGTCCTTTCAAT 3′;
R:5′GACAAATATTCCCCAGGAATAAC 3′。
PCR反应体系(50 μL)包括:10×Buffer 5 μL,dNTP(10 mmol/L)5 μL,上、下游引物各2 μL(10 μmol/L),模板DNA(50 μg/μL)5 μL,ddH2O 31 μL。反应条件为:94 ℃下预变性5 min;94 ℃下变性30 s,50 ℃(COXⅠ基因)或55 ℃(CYTB基因)下退火复性30 s,72 ℃下延伸120 s,共进行30个循环;最后在72 ℃下再延伸10 min,在4 ℃下保存。用10 g/L琼脂糖凝胶电泳检测PCR扩增产物并送往生工生物工程(上海)股份有限公司进行双向测序。
1.2.2 序列分析 使用BioEit软件对测序结果进行比对。采用BLAST检索确定目的基因。采用DNASP 5.0(DNA Sequence Polymorphism)软件确认单倍型数,计算6个不同海域群体内核苷酸多样性指数(Pi)、核苷酸差异系数(K)。采用MEGA 5.0软件计算COXⅠ和CYTB基因的碱基组成、变异位点数并构建单倍型分子系统进化树,Bootstrap 重复1 000 次检验置信度。采用TCS 1.21软件制作单倍型网络支系图,以分析6个不同群体的亲缘关系。采用分子方差分析法(AMOVA)分析香螺不同群体间的遗传分化,并计算遗传分化系数(Fst)。
2 结果与分析
2.1 香螺COXⅠ和CYTB基因序列的碱基组成及变异分析
香螺线粒体COXⅠ和CYTB基因长度分别为1 536 bp和1 140 bp。变异位点中,两基因均未发现插入/缺失位点。COXⅠ基因检测到141个多态位点,11种单倍型,CYTB基因检测到34个多态位点,14种单倍型;线粒体COXⅠ基因的T、C、A、G平均碱基含量分别为38.7%、16.2%、27.2%、18.0%,线粒体CYTB基因的T、C、A、G平均碱基含量分别为40.4%、17.0%、28.0%、14.6%(表1)。通过对6个不同群体间COXⅠ和CYTB碱基组成对比发现,两基因的碱基组成较为接近,且A+T碱基含量均大于G+C。

表1 香螺COXⅠ和CYTB 基因序列的碱基组成
2.2 6个不同海域香螺COXⅠ和CYTB基因的单倍型分布
香螺线粒体COXⅠ基因的单倍型分布见表2,其中,WH群体独有的单倍型是COXⅠ-03,YT群体独有的单倍型是COXⅠ-5和COXⅠ-6,ZZ群体独有的单倍型是COXⅠ-8和COXⅠ-9,DL群体独有的单倍型是COXⅠ-10和COXⅠ-11。香螺线粒体CYTB基因单倍型分布见表3,其中,ZZ群体独有的单倍型是CYTB-3、CYTB-5和CYTB-6,LS群体独有的单倍型是CYTB-7、CYTB-8和CYTB-10,YT群体独有的单倍型是CYTB-11和CYTB-14,WH群体独有的单倍型是CYTB-12,PL群体独有的单倍型是CYTB-13。
(2)首先检查SDH电源是否故障,如果有即修复电源,修复后SDH若无告警则流程转入第(4)步。若SDH告警为次要告警,则流程转入第(3)步。若为主要告警,先对两端SDH在ODF处自环,若告警不消除,则需要对ODF和SDH排查和检修,ODF检修为更换尾纤或光接头,SDH检修则为找出故障板卡并更换。若无主要告警,环回方式改成如下,在其中一端ODF处环回给对端,观察对端是否有主要告警,若有告警即可诊断为光链路或中继 SDH故障,需要参考专用保护通道故障定位处理方法解决故障,并消除中继SDH故障。
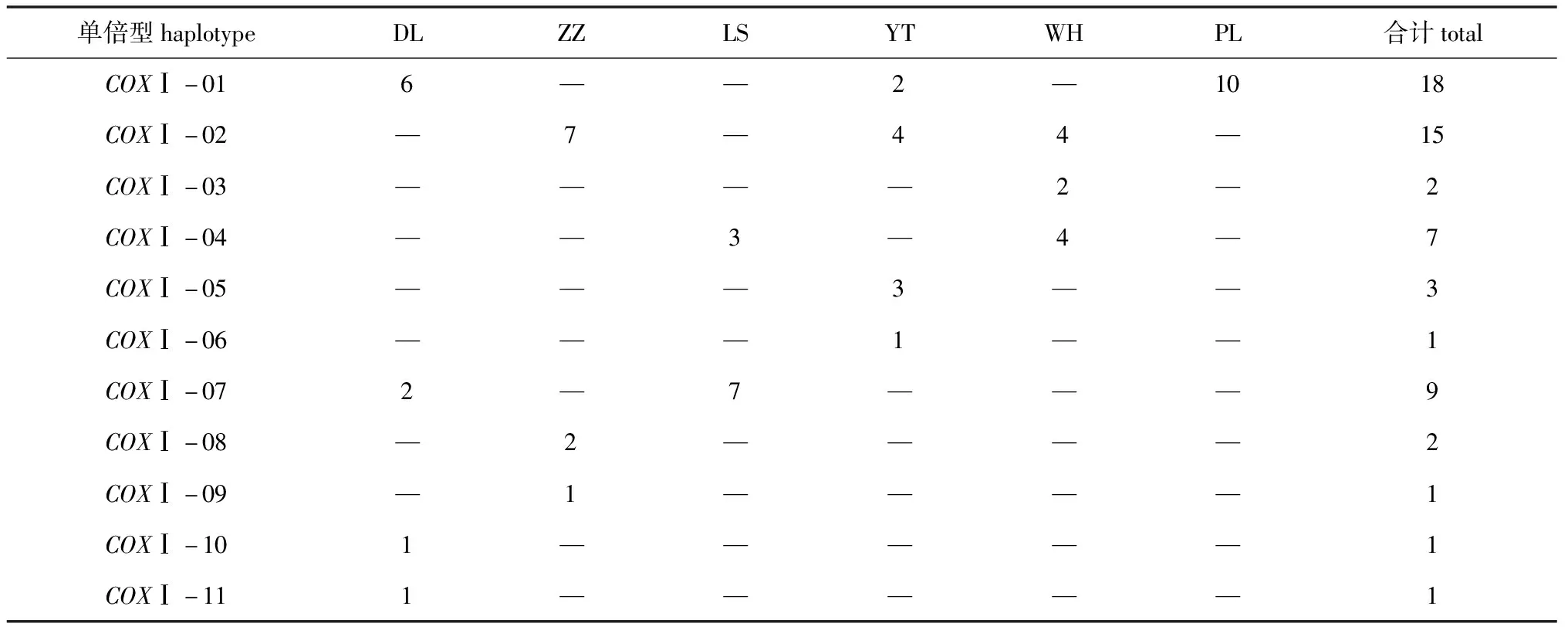
表2 香螺各群体COXⅠ基因单倍型分布情况

表3 香螺各群体CYTB基因单倍型分布情况
2.3 香螺各群体内COXⅠ和CYTB基因的遗传多样性分析
6个香螺群体内COXⅠ和CYTB基因的遗传参数如表4所示,COXⅠ及CYTB基因的平均核苷酸差异数K值变化范围分别为0.000~31.133和1.222~13.444,COXⅠ基因K值变化范围较大,且DL群体最高,为31.133,远大于其他群体;核苷酸多样性指数Pi分析显示,COXⅠ基因中,DL群体的Pi值最高,为0.021 21,PL群体的Pi值最低,为0,而CYTB基因中,LS群体的Pi值最高,为0.012 36,PL群体的Pi值最低,为0.001 14。
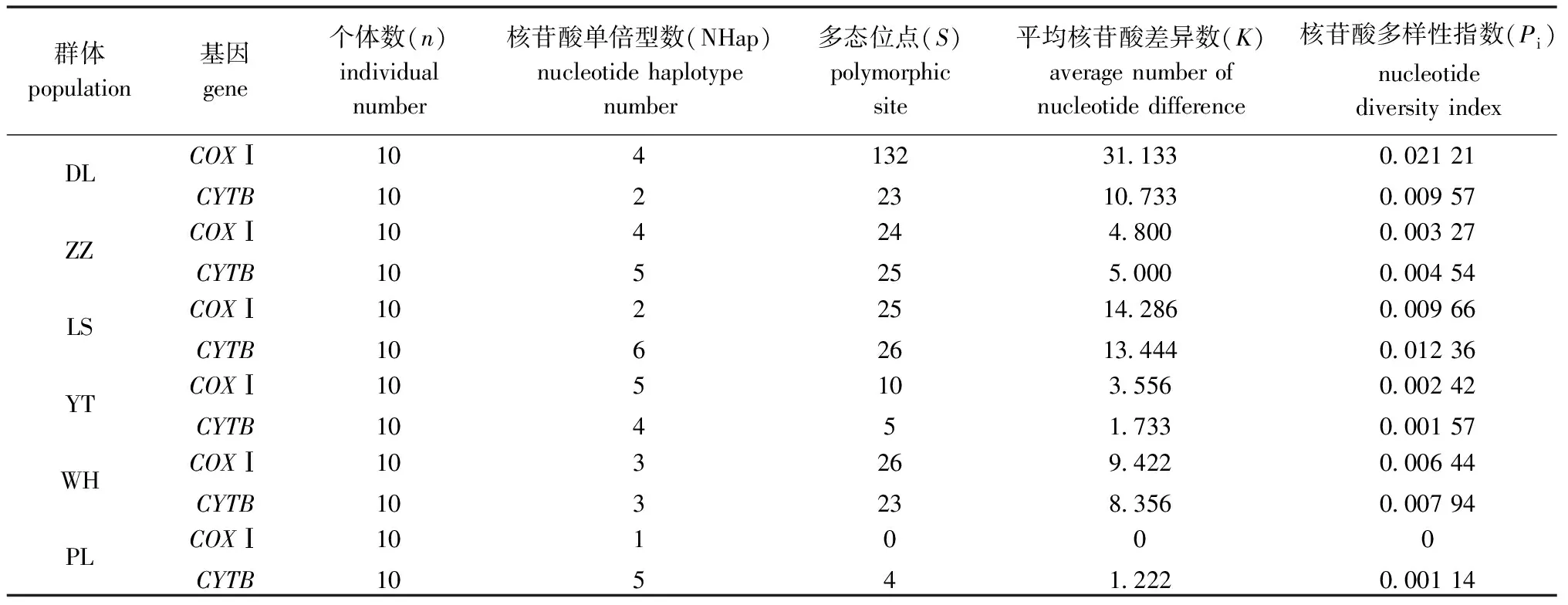
表4 6个不同海域香螺COXⅠ和CYTB基因的群体遗传多样性参数
2.4 香螺各群体间COXⅠ和CYTB基因遗传多样性分析
6个群体间的遗传参数如表5所示,香螺各群体间COXⅠ及CYTB基因K值变化幅度分别为24.971~2.094和11.497~1.395,COXⅠ基因K值变化幅度较大;COXⅠ基因中,YT和PL群体间的K值最低,DL与LS群体间的K值最高;在CYTB基因中,LS和其他群体间的核苷酸差异数与其他群体两两相比差异较大,表明LS群体是6个群体中差异较为明显的群体。另外,在COXⅠ基因中,DL群体与ZZ、YT、WH群体间未发现共享的遗传位点,PL群体与其他5个群体间也未发现共享的遗传位点;但在CYTB基因中,DL群体与ZZ、YT、WH群体间有22个共享的遗传位点,且PL群体与DL、ZZ、LS、YT及WH群体的共享遗传位点数分别是22、44、46、26和44(表5)。

表5 6个不同海域香螺群体间的COXⅠ和CYTB基因平均核苷酸差异数K(对角线下)及共享变异位点(对角线上)
2.5 香螺COXⅠ和CYTB基因单倍型系统进化树及网络图分析
使用MEGA5.0软件制作单倍型系统进化树,结果如图1和图2所示。在COXⅠ基因中,有3个分支的单倍型聚类,分别为具有最大优势分支的单倍型COXⅠ-06、COXⅠ-09、COXⅠ-02、COXⅠ-01、COXⅠ-11、COXⅠ-04、COXⅠ-05,单倍型COXⅠ-03、COXⅠ-07、COXⅠ-08为一个分支,仅单倍型COXⅠ-10单独为一个分支(图1);CYTB基因中,有2个分支的单倍型聚类,一类为最大的优势分支单倍型CYTB-02、CYTB-04、CYTB-08、CYTB-06、CYTB-10、CYTB-09、CYTB-13、CYTB-05、CYTB-11、CYTB-14,另一类为单倍型CYTB-03、CYTB-12、CYTB-01、CYTB-07分支(图2)。
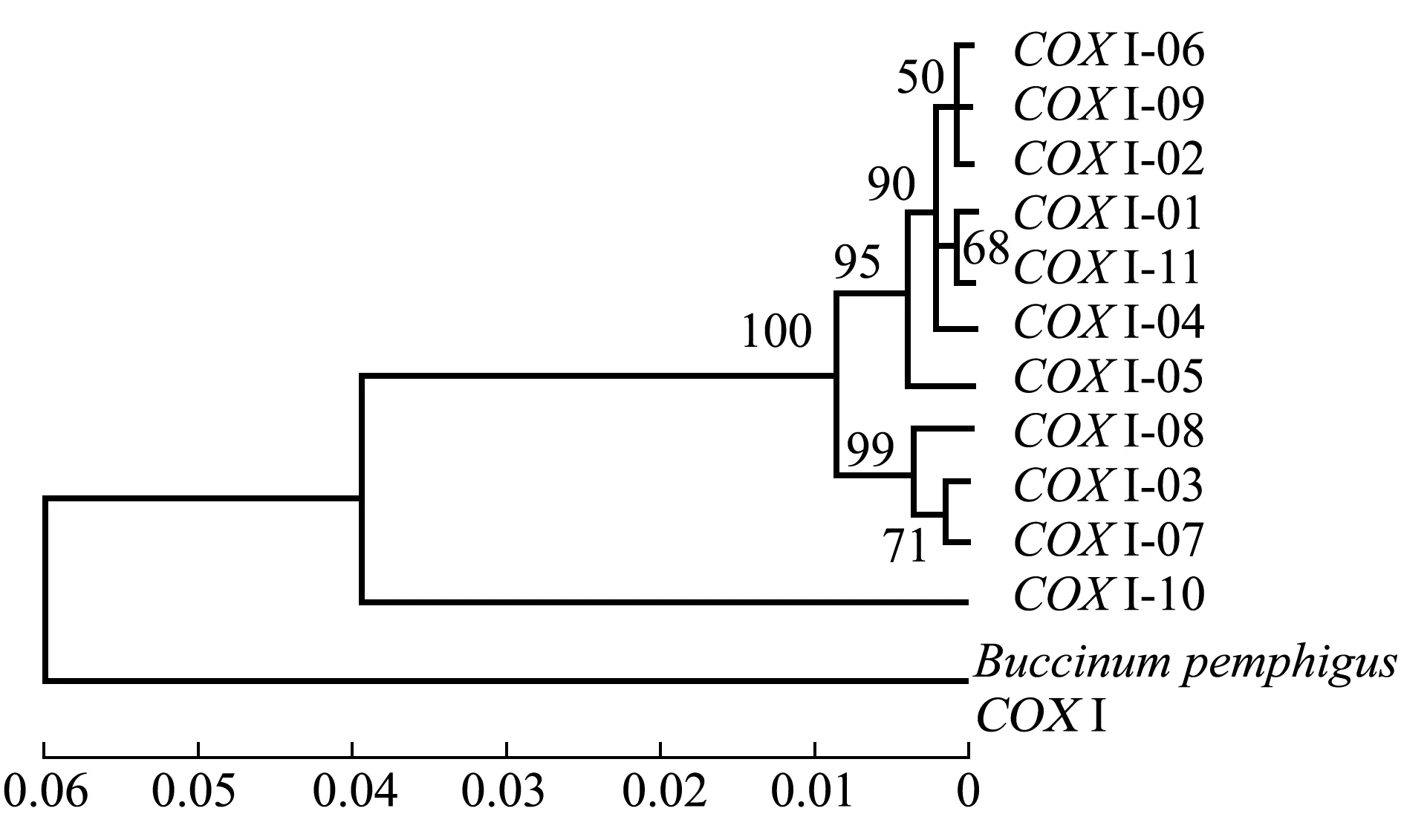
图1 COXⅠ基因单倍型系统进化树
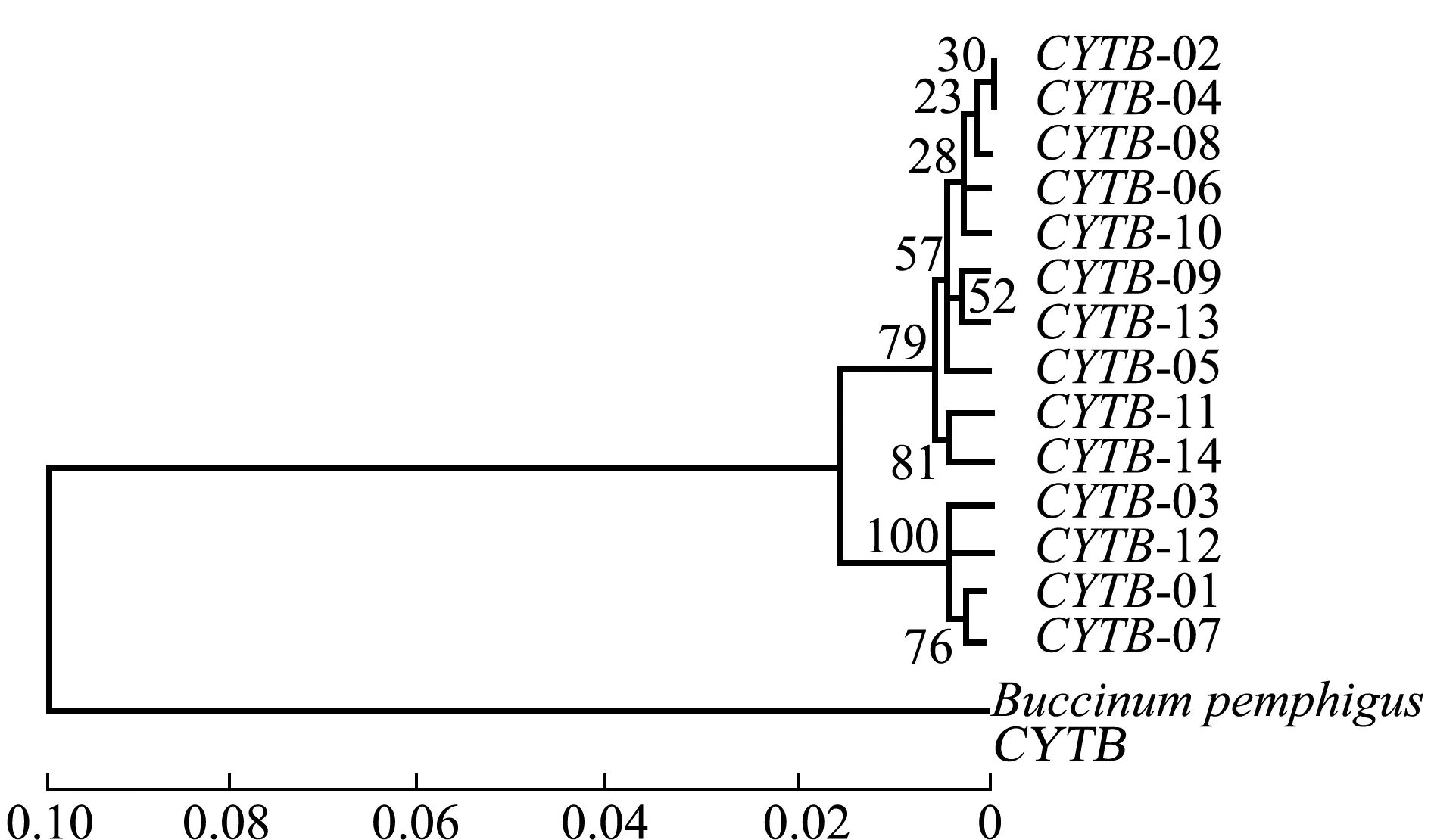
图2 CYTB基因单倍型系统进化树
使用TCS 1.21软件制作单倍型网络图如图3和图4所示,其中,COXⅠ和CYTB基因在不同群体间单倍型交错分布, 且未出现明显地理差异。在COXⅠ基因中,最大的优势分支COXⅠ-02位于网络图的最中心;在CYTB基因中,两分支中均有一个单倍型位于网络图的中心,分别是大分支的单倍型CYTB-02,小分支的单倍型CYTB-01。COXⅠ和CYTB基因单倍型网络图得到的结果与单倍型系统进化树相似。

图中每一个圆圈代表需要进行一次变异,下同。
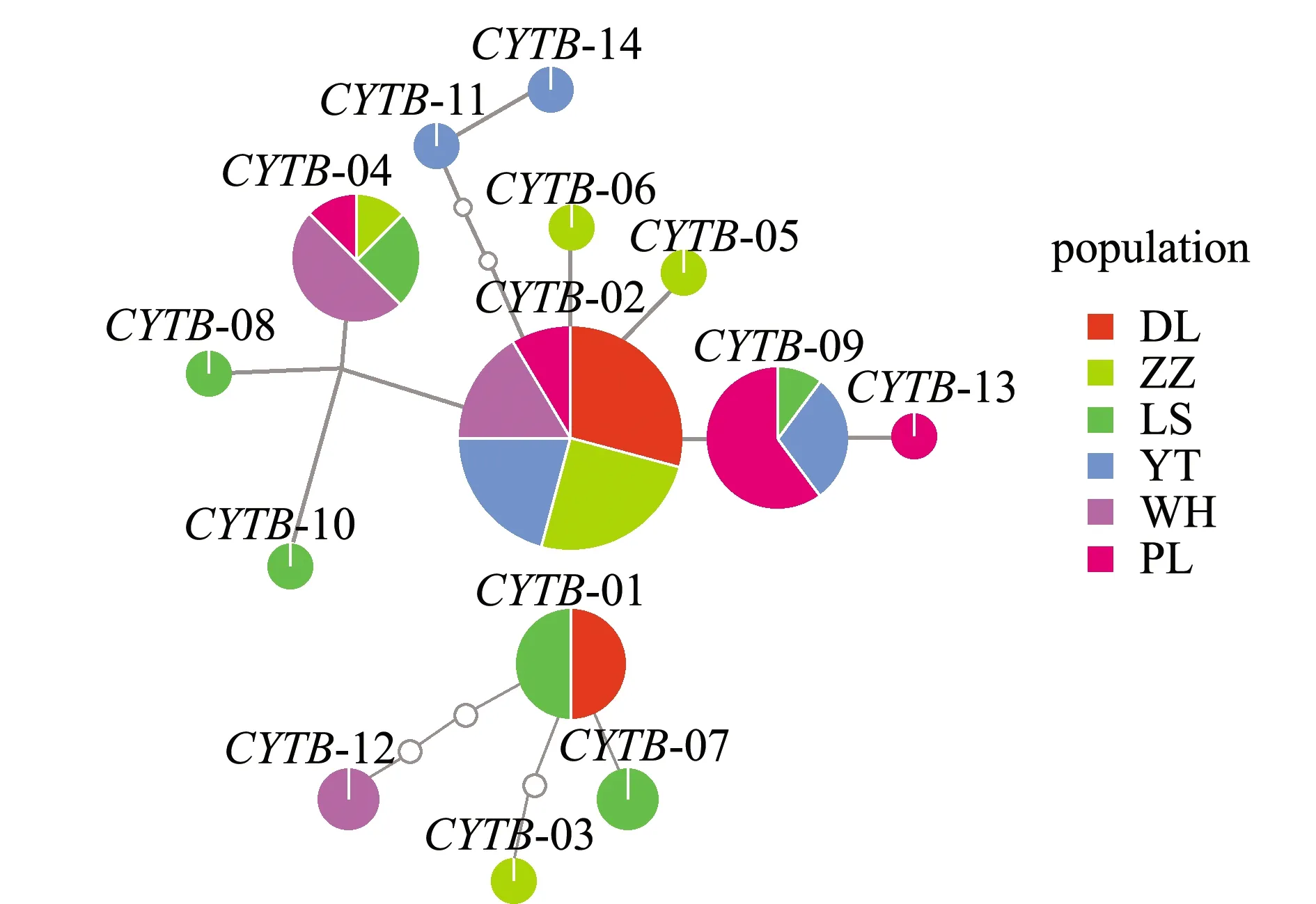
图4 CYTB基因单倍型网络图
2.6 香螺各群体间的遗传变异分析
从表6可见:COXⅠ和CYTB基因各群体间遗传距离分别为0.002~0.018和0.001~0.005;COXⅠ基因中,DL群体与ZZ、LS、YT、WH、PL群体的遗传距离分别为0.014、0.018、0.014、0.015、0.012,其他5个群体间的遗传距离均小于0.012,其中ZZ、YT、PL群体之间的遗传距离最小,分别为0.003、0.002、0.002;CYTB基因中,LS群体与其他5个群体间的遗传距离均为0.005,而其他5个群体间的遗传距离均小于0.005,YT、PL、ZZ群体间的遗传距离最小,均为0.001。
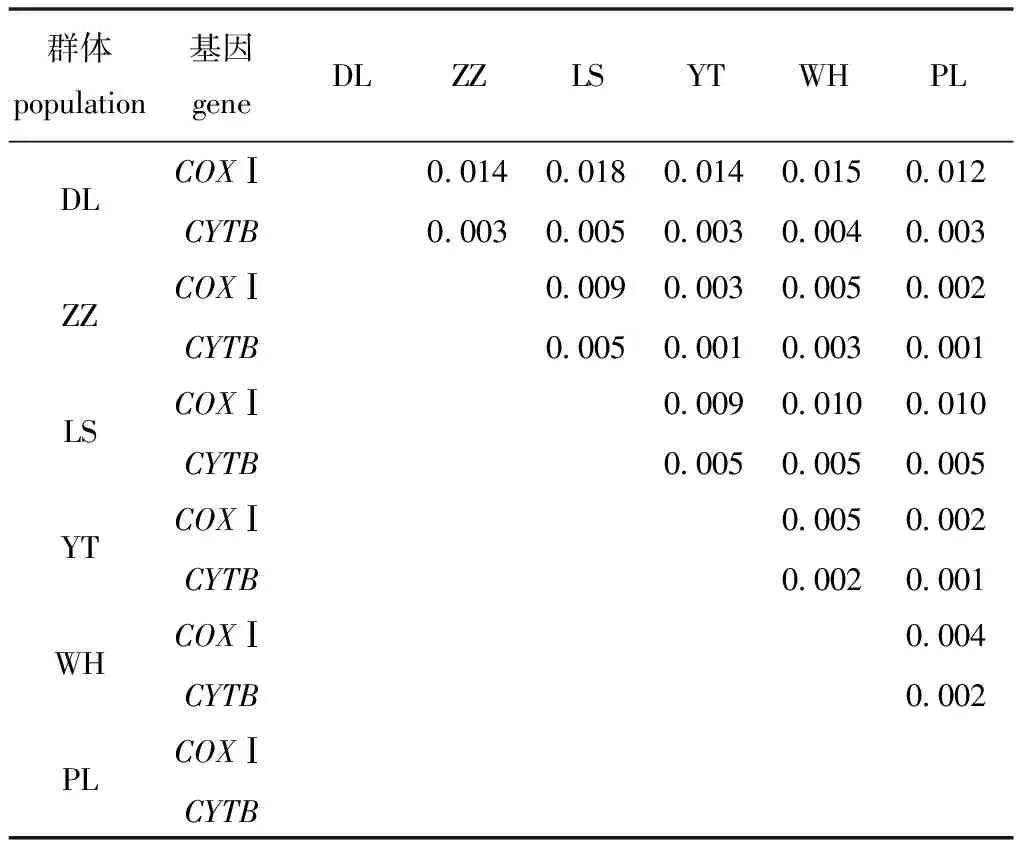
表6 6个不同海域香螺群体间COXⅠ和CYTB的遗传距离
从AMOVA分析结果中(表7)可知:COXⅠ和CYTB基因Fst指数分别为0.099 31和0.180 89,P值均大于0.05,说明群体间的遗传分化并不明显;另外,香螺6个群体内COXⅠ和CYTB基因变异比例分别为90.07%和81.91%,群体间变异比例分别为9.93%和18.09%,说明群体内有较为明显的遗传分化。

表7 基于6个不同海域香螺群体COXⅠ和CYTB基因序列的AMOVA分析
3 讨论
3.1 香螺遗传多样性
遗传多样性是物种生存发展的前提,也是其物种进化的潜力和适应环境能力的基础,更是物种多样性的基础[13]。单倍型多样性和核苷酸多样性是一个物种遗传多样性的衡量指标,因此,单倍型多样性和核苷酸多样性值越大,物种的遗传多样性越丰富,但当研究数量较少时,核苷酸多样性比单倍型多样性更具有说服力[14-15]。根据Grant等[16]提出的标准,核苷酸多样性指数以0.005为临界线,高于0.005说明物种的遗传多样性较丰富。本研究中,COXⅠ和CYTB基因在WH群体中核苷酸多样性指数为0.006 44和0.007 94,均大于0.005,推测可能是因为威海水资源的开发利用较小,遗传多样性较丰富。此外,还可能与其地理位置有关,威海纬度较低,这对气温和水温有一定的影响,使其形成了一定的差异。
3.2 香螺单倍型分布及系统发育
本研究表明,在COXⅠ基因中,单倍型系统进化树和单倍型网络图均有3个大分支,分析结果基本相同,不同群体在单倍型分布上并未出现明显的地理差异,在群体中占比较大的单倍型是该群体中的优势单倍型,有1~2种。在CYTB基因中,单倍型系统进化树和单倍型网络图均出现两个分支,分析结果基本相同,且未发现明显的地域差异,但ZZ、YT、WH、PL群体有独有单倍型,说明交叉现象会在不同群体中出现。其中,最大优势分支单倍型COXⅠ-02和CYTB-02位于网络图最中心且占比最高,依据溯祖理论,祖先类型应是占比最高的单倍型[17],因此,推测中国黄、渤海海域的祖先类型可能为单倍型COXⅠ-02和CYTB-02。
3.3 香螺COXⅠ和CYTB基因的遗传距离和遗传分化
本研究表明,在COXⅠ基因中,从单倍型系统发育树和单倍型网络图可以看出,各群体间产生了穿插渗透的现象,未出现明显的群体差异,且单倍型和变异位点较丰富,说明群体间有较高的遗传渗透率和遗传相似度,这与赵丹等[18]对泥螺BullactaexarataCOXⅠ基因部分片段的研究结果相似。在CYTB基因中,LS群体的单倍型数、多态位点数、平均核苷酸差异数、核苷酸多样性指数、群体间遗传参数和遗传距离等指标均高于其他5个群体,说明LS群体与其他5个群体有较为明显的差异,且遗传背景及遗传多样性方面要优于其他群体,说明物种对环境的适应力与遗传多样性丰富度成正比[19-20]。Shaklee等[21]提出,鱼类在种群的遗传距离标准为0.05~0.30。依据此标准,本研究中6个不同海域群体香螺在COXⅠ基因中,DL群体和LS群体的遗传距离最大,为0.018,小于种群的标准0.05;在CYTB基因中,群体间最大的遗传距离为0.005,小于种群的标准。表明6个不同海域的香螺未达到亚种分化。
本研究中AMOVA分析显示,香螺COXⅠ和CYTB基因群体内变异都远大于群体间差异,COXⅠ基因差异的主要原因是群体内变异为群体,说明COXⅠ基因与其地理位置并未有明显的相关性。王旭[22]通过COXⅠ和16S rRNA分析了黄口荔枝螺Thaisluteostoma和疣荔枝螺Reishiaclavigera的遗传多样性,结果显示,在整个遗传变异中群体内的遗传变异最大,无明显的地理结构和谱系结构,表明群体间不存在显著的遗传分化,本研究结果与之相似。COXⅠ基因中,从单倍型、变异位点数、遗传距离等几个指标中可以发现,DL群体均大于其他5个群体,说明在遗传背景和遗传多样性方面DL群体与ZZ、LS、YT、WH、PL群体相比较为丰富。杨建敏等[23]通过对辽宁至山东的脉红螺Rapanavenosa7个群体16S基因的遗传多样性进行研究,认为大连市大连湾群体是遗传背景最为丰富的群体。本研究中CYTB基因差异的主要原因,推测认为是该基因片段未能出现明显的地域差异。
3.4 香螺资源保护
香螺分布范围和移动范围较小,繁殖力较弱,由于捕捞强度变大,目前出现了显著的资源衰竭[1]。因此,当前要采取有效措施保护其资源。本研究中初步反映了香螺的遗传背景,为制定相关的保护措施提供了依据。笔者认为,可通过多种方式对香螺的资源进行保护:1)保证香螺的饵料资源;2)严格限制捕捞的工具;3)从生产的实际情况考虑,严格控制捕捞期。然而,若从遗传多样性角度分析香螺的资源,还需通过其他技术获取相关的分子数据库。
4 结论
1)中国不同海域间香螺遗传多样性较为丰富,威海群体COXⅠ和CYTB基因的核苷酸多样性指数较高,均大于0.005。
2)不同海域间香螺具有群体独有的单倍型,可作为鉴别不同地理种群的重要依据。
3)不同海域间香螺未出现明显的群体间差异,但群体内变异远大于群体间差异,不同海域间香螺未达到亚种分化水平。
