接种2种不同促生菌对番茄根菌群结构及多样性的影响
2021-09-12冯燕辉熊娟黎烨张婷程林洁金玲月张沁怡蓝灿华田宝玉
冯燕辉 熊娟 黎烨 张婷 程林洁 金玲月 张沁怡 蓝灿华 田宝玉

摘 要:為了解析具有不同IAA分泌特性的促生菌在植物体内的存在模式及其促生机制。以不可产IAA的植物促生菌Lysinibacillus xylanilyticus CZ29和可产IAA的植物促生菌Rhizobium radiobacter GF12为材料,分别接种于盆栽新中蔬四号番茄幼苗根部,以不接种菌株为对照,采用基于细菌16S rRNA基因V5~V7可变区高通量测序的方法,分析2种不同促生菌在植物根内定植情况及其对生长得到促进的植物根内生菌菌群结构和多样性的影响。结果显示:接种2株促生细菌之后,这2株促生菌在生长得到促进的植物根内的定植量无显著变化,说明促生菌接种的促生长作用并非是通过接种促生菌的定植量增加而实现。且经2种不同促生菌分别接种处理后生长得到促进的番茄根内生菌物种丰富度(Chao1指数)以及细菌多样性(Shannon指数)相对于对照以及相互之间均没有显著差异性(P>0.05),表明这2株促生菌的促生长作用与内生菌菌群丰富度和多样性没有直接联系。基于OTU水平的层次聚类和PLS-DA等分析表明,2株促生菌接种均可显著影响番茄根内生菌菌群结构组成,导致原有的内生菌菌群平衡被打破但其具体的影响不同。其中,接种不可产IAA的植物促生菌L.xylanilyticus CZ29的组别中Roseateles、Sphingobium和Rhizobium益生菌受到显著富集;Myroides、Chryseobacterium、Achromobacter及Strenotrophomonas致害菌被显著抑制(P<0.05)。接种可产IAA的植物促生菌R.radiobacter GF12的组别中各菌属菌群平均丰度与对照相比波动都不明显,菌群结构与对照更为相似并表现出内生菌菌群结构组成更加稳定的特点。因此推测,不可产IAA的植物促生菌L.xylanilyticus CZ29是通过富集益生菌抑制有害菌从而促进植物生长。可产IAA的植物促生菌R.radiobacte GF12则是通过同时上调植物内生菌各主要菌群的丰度从而构建更加稳定的内生菌群增大植物的抗刺激能力及分泌IAA而促进植物生长。
关键词:植物促生菌;接种;内生菌;菌群结构;高通量测序
中图分类号:S 641.2 文献标志码:A 文章编号:0253-2301(2021)06-0006-09
DOI: 10.13651/j.cnki.fjnykj.2021.06.002
Abstract: In order to analyze the existence pattern and growth-promoting mechanism of the growth-promoting bacteria with different IAA secretion characteristics in plants, Lysinibacillus xylanilyticus CZ29, the plant growth-promoting bacteria that could not produce IAA, and Rhizobium radiobacter GF12, the plant growth-promoting bacteria that could produce IAA, were used as the materials to be inoculated in the roots of potted tomato seedlings, Xinzhongshu No.4, and the high-throughput sequencing based on the variable region of the bacteria 16S rRNA gene V5-V7 was used to analyze the colonization of the two different growth-promoting bacteria in plant roots whose growth has been promoted and their effect on the structure and diversity of endophytic bacterial flora in plant roots. The results showed that after inoculating the two growth-promoting bacteria, the colonization of the two growth-promoting bacteria in the plant roots whose growth has been promoted did not change significantly, indicating that the growth-promoting effect of the growth-promoting bacteria inoculation was not achieved by increasing the colonization of the inoculated strains. After the inoculation with two different growth-promoting bacteria respectively, there was no significant difference (P>0.05) between the species richness (Chao1 index) and bacterial diversity (Shannon index) of endophytes in tomato roots compared to the control group and between each other, which showed that the growth-promoting effect of these two growth-promoting bacteria was not directly related to the richness and diversity of endophyte flora. The analysis of hierarchical clustering based on OTU level and PLS-DA showed that the inoculation of the two growth-promoting bacteria could significantly affect the composition of the endophyte flora in tomato root, causing the balance of the original endophytic flora to be broken but the specific effects were different. Among them, the probiotics such as Roseateles, Sphingobium and Rhizobium were significantly enriched in the group inoculated with L.xylanilyticus CZ29, the plant growth-promoting bacteria that could not produce IAA; the harmful bacteria such as Myroides, Chryseobacterium, Achromobacter and Strenotrophomonas were significantly inhibited (P<0.05). In the other experimental group inoculated with R.radiobacter GF12, the plant growth-promoting bacteria that could produce IAA, the average abundance of each bacterial genus did not fluctuate significantly compared with the control group, and the microbial community structure was more similar to that of the control group and showed a more stable structure. Therefore, it was hypothesized that L.xylanilyticus CZ29, the plant growth-promoting bacteria that could not produce IAA, promoted the plant growth by inhibiting the harmful bacteria through the enrichment of probiotics, while Rhizobium radiobacter GF12, the plant growth-promoting bacteria that could produce IAA, promoted the plant growth by simultaneously up-regulating the abundance of the main flora of plant endophytes to construct a more stable endophytic flora, thus to increase the anti-irritating ability of plants and secrete IAA.
Key words: Plant growth-promoting bacteria; Inoculation; Endophyte; Microbial community structure; High-throughput sequencing
植物内生菌是指其生活史的某一阶段或全部过程生活于植物各种组织、器官内部或细胞间隙但不导致明显病害的真菌或细菌[1-3],其主要通过从表面严格消毒的植物组织中分离得到[2],既包括中性菌、促生菌,也包括某些潜伏的或条件致病的病原微生物[3-4] ,尽管如此,目前还是普遍认为植物内生菌在植物生长、发育以及健康中发挥着重要作用[3-5]。比如一些促生菌长期稳定定植于植物体内并与宿主形成一种互利共生的关系,使植物具有例如内生固氮、溶磷、抗病害等优势[1,3,5-6],并在全球气候变暖、大量优质农业用地丧失、全球农作物面临巨大环境压力的今天,起到促进植物生长并缓解植物生长压力的作用[7]。但是,在自然界中植物促生菌的来源十分有限。大量的研究数据表明,每克根的根内生环境中的细菌量只有104~108个,植物内生菌无论是从数量上还是物种多样性上都远远少于土壤和植物根际细菌[10-12],且植物宿主根周围的土壤微生物中也仅2%~5%的具有根际定植能力的自由细菌可侵入根部成为促生菌[8-12]。
现有的研究表明,接种特定根促生细菌是改善植物根际和内生細菌促生作用的有效手段。如,Ke等[13]在两种水分条件下,研究了内生假单胞菌A1501接种玉米后对植株生长和植株含氮量的影响;Barriuso等[14]研究10种植物促生长根细菌(PGPR)对松果体生长和菌根生长的促进作用,均证明了促生菌的接种对植物生长有明显促进作用。尽管如此,目前对促生菌引入后其在植物体内的存在模式及其对植物内生菌的影响和真正的促生机制的报道仍旧较少且尚不够深入[15-17]。与此同时,近年来伴随着基于核糖体rRNA基因的宏基因组测序和分析技术的快速发展,对国内占全年蔬菜栽培总面积15%左右[18-19]的番茄植物的根内生菌种群组成及其多样性有了更为深入的了解[9,19],并从中鉴别出了大量丰富且功能多样的促生细菌,包括抑菌、铁载体产生、固氮以及依赖IAA产生的植物生长促进细菌等[20-22]。
因此,本研究在前期解析番茄根微生物组菌群结构组成,以及根内生菌分离、鉴定和活性筛选的基础上,挑选2株具有不同IAA分泌特性的促生菌株研究促生菌接种后生长得到促进的番茄植物根内生菌菌群结构和多样性,旨在解释具有不同IAA分泌特性的接种促生菌在植物体内的存在模式及其促生机制,从而为在实践上构建健康的植物内生微生物组及发展高效的微生物肥料奠定基础。
1 材料与方法
1.1 试验材料与仪器
1.1.1 材料与试剂 实验室前期在番茄根际分离出的2株具有不同IAA分泌特性、对番茄植株具有同等程度促生效果的促生菌,分别为Lysinibacillus xylanilyticus CZ29和Rhizobium radiobacter GF12,其中R.radiobacter GF12具有利用色氨酸分泌IAA的能力,而L.xylanilyticus CZ29则无;FastDNASpin Kit试剂盒。
1.1.2 仪器与设备 IonSSTMXL测序平台;Illumina Miseq高通量测序平台。
1.2 试验方法
1.2.1 接种试验 通过用等浓度的2株菌菌液分别浸泡10粒番茄种子并于盆栽土中播种,同时菌液均匀撒在种子周围土壤中,设置不添加任何微生物对照组,同时试验组、对照组各3个重复。
1.2.2 样品采集及测定 番茄生长1个月后收集每盆长势最好的植株根为一个样,共9个样品。紧接着将番茄根表面消毒后收集到无菌离心管中,按照FastDNASpin Kit试剂盒中要求提取总基因组DNA作为细菌16S rRNA的PCR扩增模板,采用IonSSTMXL测序平台的V5~V7引物799F(5′-AACMGGATTAGATACCCKG-3′)和1193R(5′-ACGTCATCCCCACCTTCC-3′)进行扩增。PCR扩增产物纯化后,扩增子送交上海生物工程科技有限公司进行高通量测序。
1.3 数据处理
扩增子交由上海生物工程科技有限公司进行Illumina Miseq高通量测序。检测数据经CASAVA碱基识别分析转化为原始测序序列。利用cutadapt(version 1.2.1)软件去除引物接头序列,再根据PE reads之间overlap关系,利用pear(version 0.9.6)软件将成对的reads拼接成一条序列,然后按照barcode标签序列识别并区分样品得到各样本数据,最后用Prinseq(version 0.20.4)软件对各样本数据的质量进行质控过滤,得到各样本有效数据。还要使用Usearch(version5.2.236)去除预处理后序列中非扩增区域序列,而后对序列进行测序错误校正,并调用Uchime(version 4.2.40)进行鉴定嵌合体。随后,再将去除嵌合体的序列与数据库代表性序列进行blastn比对,低于阈值的比对结果认为是靶区域外序列,并剔除掉该部分序列,得到最终有效序列。
使用97%相似度的OTU,利用Mothur(version 1.30.1)做rarefaction分析,用R制作曲线图从而分析数据合理性。利用RDP classifier(version 2.12)对OTU进行物种分类,并用blastn将OTU序列与对应数据库进行比对,筛选出OTU序列的最佳比对结果,并对比对结果进行过滤,根据OTU表绘制各个样品不同分类水平物种结构组成柱状图,更加直观地看出样本间的关系及物种构成。采用Alpha多样性指标的Shannon指数、Chao1指数对样品微生物物种的丰富度和多样性进行评估。采用PLS-DA分析来评估样本β多样性及不同样本群落组成相似性和差异等。
2 结果与分析
2.1 2株促生菌在番茄根内的定植情况评估
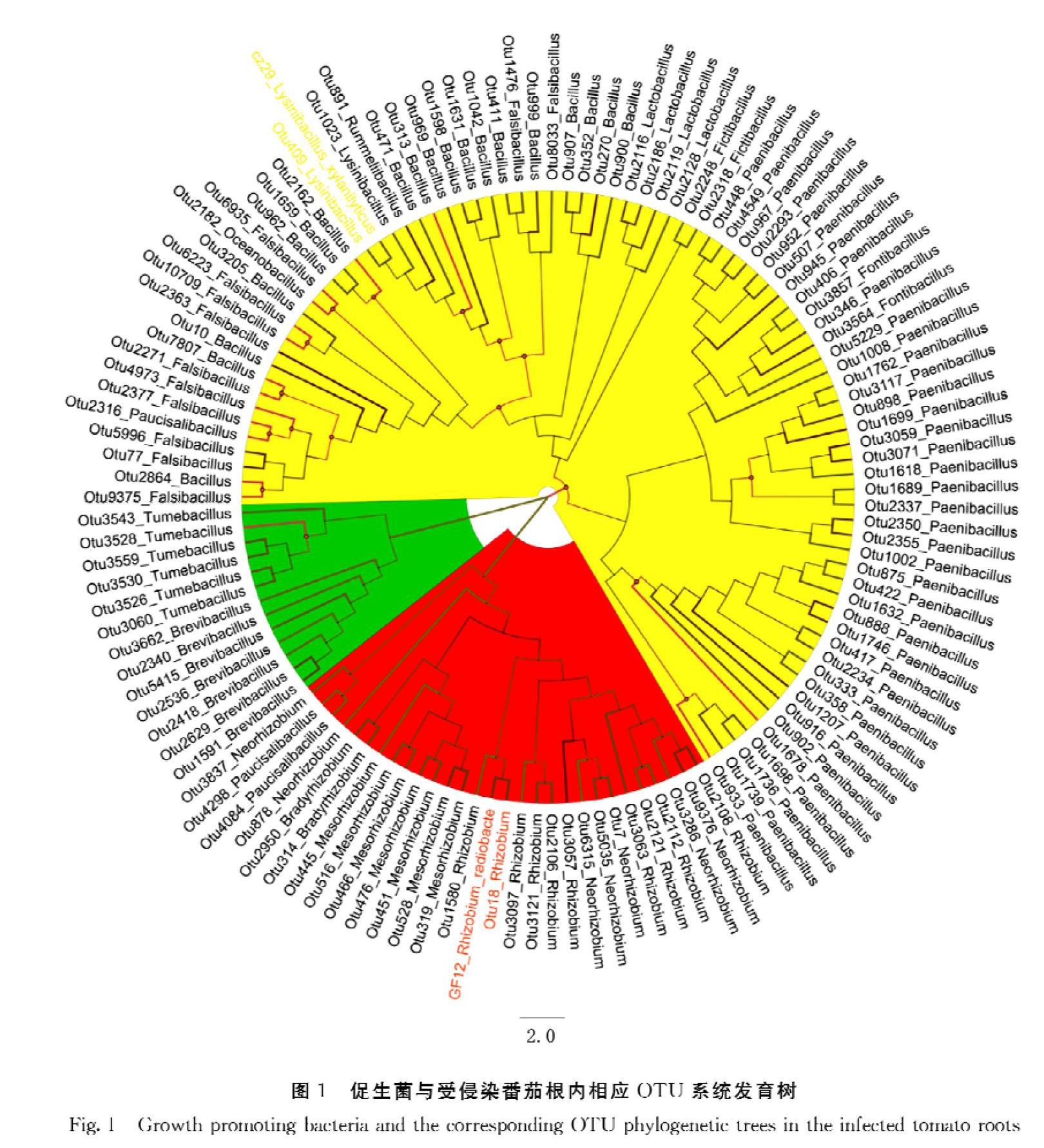
通过受试促生菌的16S rDNA序列与9个样品的Illumina测序聚类的OTU相应序列比对发现,OTU 18与L.xylanilyticus GF12、OTU 409与R.radiobacter CZ29的序列一致性高达99%(图1),说明OTU 18与OTU 409分别为L.xylanilyticus GF12和R.radiobacter CZ29在样品中的OTU。对比其OTU丰度在对照组和试验组中的变化(表1),OTU丰度在相应的处理中没有明显增加(P>0.05),表明2种促生菌接种后它们的定植量无显著变化,因而促生菌接种的促生长作用并非是通过接种菌株的定植量增加而实现。
将这2株促生菌的菌群丰度在组内进行统计学比较,结果显示,在对照组中,Rhizobium与Lysinibacillus无显著区别,于GF12组中也无显著差异,反倒是CZ29组中的Rhizobium丰度(平均3.15%)极其显著地(P<0.01)高于组中的Lysinibacillus(平均0.25%)。除此之外,分别对Rhizobium和Lysinibacillus在3组中的分布进行对比,也仅发现被R.radiobacte CZ29侵染的组别中Rhizobium得到显著富集,充分说明了不管在组间还是组内,Rhizobium侵染、定植能力都更强(图2)。
2.2 促生菌接种对番茄根内生菌菌群结构组成的影响
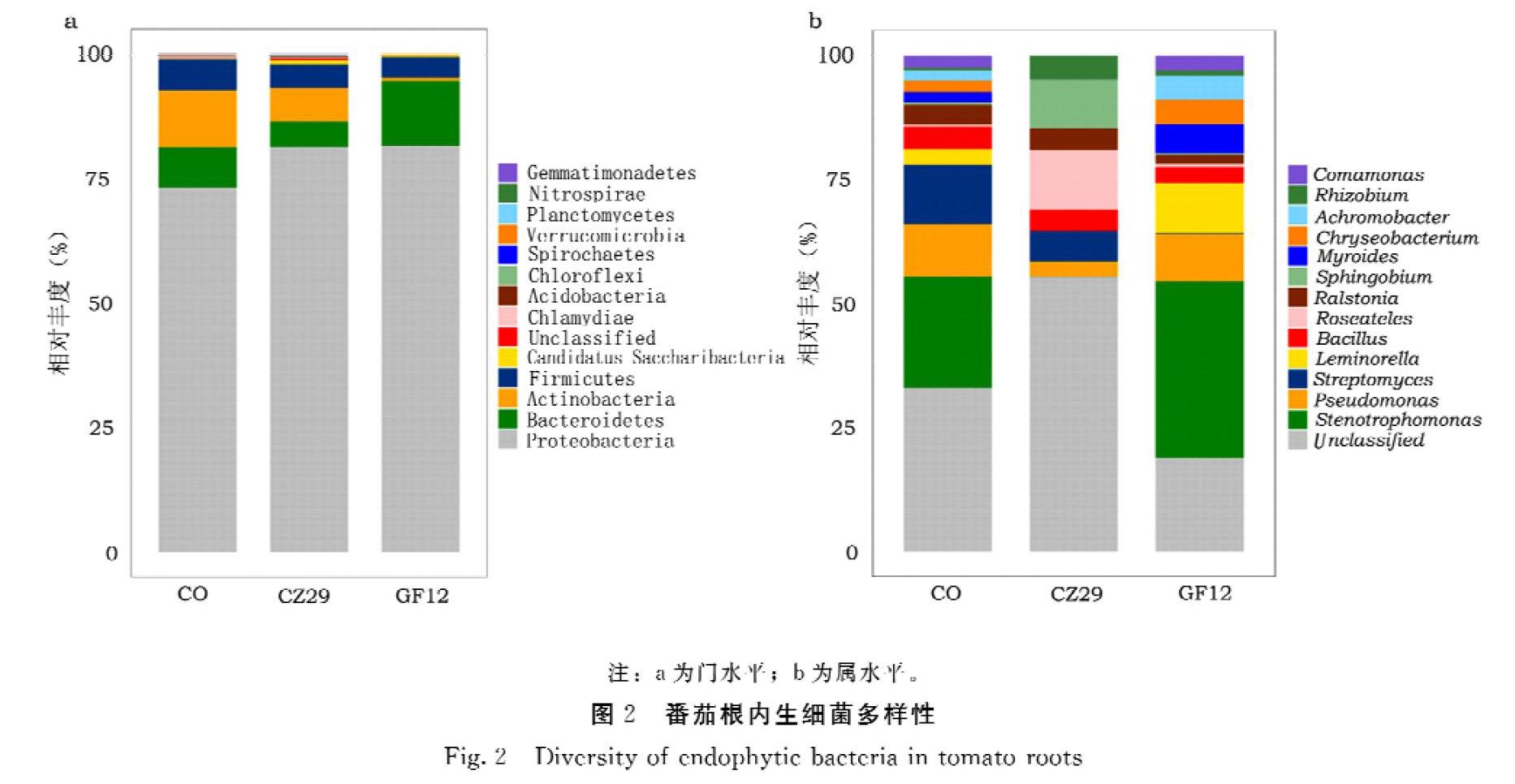
本研究利用blastn将OTU序列与对应数据库进行比对,共注释到20个门分类与373个属分类,其中,各样品相对丰度较高的前14个门与属分别占据了99%和68%以上。由此,对不同样品中细菌群落结构在门和属分类水平上的结果进行对比发现,在门水平下,番茄根内生菌菌群主要由Proteobacteria(73.44%±12.60%)、Bacteroidetes(8.10%±3.71%)、Actinobacteria(11.48%±12.89%)和Firmicutes(6.18%±1.92%)等构成。相对于对照组,经促生菌接种的2个组别中的Proteobacteria菌门平均相对丰度均增长8%左右,Firmicutes菌门则均减少约2%,涨落幅度大体一致。总体上,经促生菌侵染后的处理组在大部分门的丰度上与对照组相比没有显著性差异。但仍可以从图2a看出,2株促生菌接种在门水平下对番茄根内生菌菌群结构组成的影响存在着一定程度的差异,可分泌IAA促生菌接种所带来的影响似乎更小(图2a),这一推测也在属水平下的統计数据中得到了验证。统计结果显示,属水平下的CZ29组中Roseateles(8.24%±1.30%)、Sphingobium(6.72%±1.94%)和Rhizobium(3.15%±1.49%)较于对照(平均丰度分别是0.33%±0.03%、0.44%%±0.62%、0.42%%±0.17%)得到了显著富集(P<0.05);Myroides(0)、Chryseobacterium(0.001%±0.01%)、Achromobacter(0.03%±0.05%)及Strenotrophomonas(0.04%±0.01%)与对照(平均丰度分别为1.72%±0.62%、1.97%±3.18%、1.84%±0.69%、18.66%±5.71%)比则被抑制(P<0.05),Strenotrophomonas仅有对照的0.2%,相反,GF12组各菌属菌群平均丰度波动则都不明显(图2b)。因此,推断L.xylanilyticus GF12与R.radiobacte CZ29接种后对植物生长发育的促生效果虽然基本相同但其促生机制不同。
2.3 促生菌接种对番茄根内生菌菌群丰富度和多样性的影响
9个样品经质量控制后,共获得L.xylanilyticus CZ29处理的167366个、R.radiobacte GF12处理的132009个和对照组的136479个有效序列,共2412种OTU。并由稀释性曲线可知,多数样本序列数在接近10000个时,OTU已经接近饱和,说明再增加测序量对于发现新OTU的边际贡献很小,说明测序数据量合理,所含菌种数较多。现有测序量已经可以反映出样品中细菌丰度信息,满足进一步分析要求。同时,所有样本的测序Coverage指数都大于0.99,说明根际内生菌样品文库种序列基本都被测出,未被测到的概率较低,测序结果可以反映样品真实情况。图3 Alpha多样性分析表明,Shannon指数、Chao1指数在三组处理间差异不显著(P>0.05)。其中,CZ29组Shannon指数(3.94)、Chao1指数(824.65)最高,群落丰富度及多样性最大,GF12组的Shannon指数、Chao1指数(2.93、587.80)最低,因而相对其他两个组别经R.radiobacte GF12侵染后的番茄根内生菌菌群的优势菌突出且丰度更高(图3)。
2.4 促生菌接种对番茄根内生菌菌群结构和β多样性的影响
样本与物种关系图显示了属水平上丰度最高的优势类群与不同组样品的关联关系。图4中显示,部分的优势种群在样本中分布相对较不均匀,Bacteroidetes菌门的Myroides与GF12组相关度更高;Proteobacteria菌门的Stenotrophomonas与CZ29组根样品相关度极低,而在其他两组样品中有明显优势,以上表明促生菌的处理确实对内生菌菌群结构产生了影响(图4)。同时数据也表明L.xylanilyticus CZ29和R.radiobacte GF12促生菌处理对植物根内生细菌菌群结构组成均有明显影响(ANOSIM:R=0.626,P=0.022)。基于不同样品物种组成相似性的PLS-DA分析,CZ29组、GF12组、对照组在第一主成分(21.31%)和第二主成分(11.22%)上均存在一定的相对距离,也补充说明了3组样品间存在差异(图5a)。除此之外,OTU水平的Bray-Curtis分析结果显示(图5b):9个样品聚为2类,且两类差异性明显;其中CZ29_1、CZ29_2和CZ29_3聚为一支,其他样品聚为另一支;另一支下又分为两小支,GF12_1和GF12_3聚为一小支,GF12_2和CO_1、CO_2、CO_3聚为另一小支,表明尽管2株促生菌接种均打破了番茄根内原有内生菌群的平衡,但被R.radiobacte GF12促生菌处理后的番茄根内生菌的群落却与对照组更为相似。
3 结论与讨论
部分内生菌普遍存在于大多数植物中,并提供天然益处,目前大多数的研究报道表明,内生菌的有益作用主要是来自促生菌同时具备固氮能力或分泌IAA等促生特性的影响[23-30],然而促生菌侵染并定植于内生菌群是一个非常复杂的过程,其影响因素众多,如植物、土壤条件、生物地理学因素等,因此直至现在关于促生菌的促生机制研究仍不够明了,同时现阶段促生菌接种相关研究多集中于对根际土壤微生物影响上[31]。故本研究通过高通量分析2株具有相同促生效果、不同产IAA特性的促生菌接种对植物根内生菌群结构和多样性影响,旨在解释接种促生菌导致的菌群变化的促生作用,以期对促生菌促生机制的研究提供参考。
(1)芽孢杆菌和根瘤菌是常见的促生细菌[32],本试验中2株促生菌在番茄根内的定植情况评估结果表明,接种2株促生细菌之后,它们的定植量无显著变化,因而促生菌接种的促生长作用并非是通过接种菌株的定植量增加而实现。且从数据可以看出,Rhizobium较于Lysinibacillus表现出了更强的侵染和定植能力。
(2)Alpha多样性分析中Chao1指数以及Shannon指数相对于对照以及相互之间差异不显著(P>0.05),说明这2株促生菌的促生长作用与内生菌菌群丰富度、多样性不存在明显相关,然而在韦江璐等人的促生菌对土壤细菌群落功能多样性的影响的研究中,各菌株处理后土壤细菌群落物种丰富度指数、优势度指数和均匀度指数却均显著高于对照[33],说明相对于土壤细菌来说,植物内生菌群对外来刺激更能表现出防御能力并具有相对稳定性。比较三组中Chao1指数以及Shannon指数分布可得,CZ29组Shannon指数(3.94)、Chao1指数(824.65)最高,群落丰富度及多样性最大,而GF12组的(2.93、587.80)最低,因而相对其他两个组别经R.radiobacte GF12侵染后的番茄根内生菌菌群的优势菌突出且丰度更高,内生微生物群的结构组成趋于稳定。
(3)基于OTU水平的层次聚类和PLS-DA等分析显示,2株促生菌接种显著影响了番茄根内生菌菌群结构组成,但其影响结果不同。在属水平下,L.xylanilyticus CZ29侵染的组中Roseateles、Sphingobium和Rhizobium益生菌受到显著富集;Myroides、Chryseobacterium、Achromobacter及Strenotrophomonas致害菌则被大量抑制,而GF12组的各菌屬菌群平均丰度相较于对照波动则都不明显。结合以上的结果,经R.radiobacte GF12侵染后的番茄根内生菌菌群的优势菌突出且丰度更高,内生微生物群的组成更加趋于稳定。综上所述,推测L.xylanilyticus CZ29促生菌是通过富集益生菌抑制有害菌从而促进植物生长的。R.radiobacte GF12则是通过同时上调植物内生菌各主要菌群的丰度从而构建更加稳定的内生菌群增大植物的抗刺激能力并分泌IAA而促生。
参考文献:
[1]ROSENBLUETH M,MARTíNEZ-ROMERO E.Bacterial endophytes and their interactions with hosts[J].Mol Plant-Microbe Interact,2006,19:827-837.
[2]SCHULZ BJE,BOYLE CJC,SIEBER TN.Microbial root endophytes[M].Berlin:Springer-Verlag,2006:1-32.
[3]LIU H W,CARVALHAIS LC,CRAWFORD M.Inner plant values:Diversity,colonization and benefits from endophytic bacteria[J].Front Microbiol,2017,8:1-17.
[4]GAIERO J R,MCCALL C A,THOMPSON K A,et al.Inside the root microbiome:Bacterial root endophytes and plant growth promotion[J].American Journal of Botany,2013,100(9):1738-1750.
[5]SANTOYO G,MORENO-HAGELSIEB G,OROZCOMOSQUEDAN MDEL C,et al.Plant growth-promoting bacterial endophytes[J].Microbiological Research,2016,183(5):92-99.
[6]HASSANI MA,ZKURT E,SEYBOLD H.Interactions and coadaptation in plant metaorganisms[J].Annu Rev Phytopathol,2019,57:483-503.
[7]RACHEL B,STEFANR J,GAYATHRI I,et al.Plant Growth-Promoting Rhizobacteria:Context,Mechanisms of Action,and Roadmap to Commercialization of Biostimulants for Sustainable Agriculture[J].Frontiers in plant science,2018,9:1-17.
[8]HASSAN E,DINESH K,MAHESHWARI.Use of plant growth promoting rhizobacteria(PGPRs)with multiple plant growth promoting traits in stress agriculture:Action mechanisms and future prospects[J].Ecotoxicology and Environmental Safety,2018,156:1-22.
[9]TIAN B Y,CAO Y,ZHANG K Q.Metagenomic insights into communities,functions of endophytes,and their associates with infection by root-knot nematode,Meloidogyne incognita,in tomato roots[J].Scientific Reports,2015(5):17087.
[10]BULGARELLI D,SCHLAEPPI K,SPAEPEN S.Structure and Functions of the Microbiota of Plants[J].Annu Rev Plant Biol,2013,64:807-38.
[11]TURNER T R,JAMES E K,POOLE P S.The plant microbiome[J].Genome Biol,2013,14(6):1-10.
[12]CASTRO-SOWINSKI S,HERSCHKOVITZ Y,OKON Y,et al.Effects of inoculation with plant growth-promoting rhizobacteria on resident rhizosphere microorganisms[J].FEMS Microbiology Letters,2007,276(1):1-11.
[13]KE X B,FENG S,WANG J,et al.Effect of inoculation with nitrogen-fixing bacterium Pseudomonas stutzeri A1501 on maize plant growth and the microbiome indigenous to the rhizosphere[J].Systematic and Applied Microbiology,2019,42(2):248-260.
[14]BARRIUSO J,RAMOS SOLANO B,SANTAMARíA C,et al.Effect of inoculation with putative plant growth-promoting rhizobacteria isolated from Pinus spp.on Pinus pinea growth,mycorrhization and rhizosphere microbial communities[J].Journal of Applied Microbiology,2008,105(5):1298-1309.
[15]DARINE T,ALESSIO M,HAROUN BA,et al.Effect of on-field inoculation of Phaseolus vulgaris with rhizobia on soil bacterial communities[J].FEMS microbiology ecology,2011,77(1):221-222.
[16]CAI F,PANG G,MIAO Y Z,et al.The nutrient preference of plants influences their rhizosphere microbiome[J].Applied Soil Ecology,2017,110:146-150.
[17]REINHOLD-HUREK B,BUNGER W,BURBANO C S.Roots Shaping Their Microbiome:Global Hotspots for Microbial Activity[J].Annu Rev Phytopathol,2015,53(1):403-424.
[18]LU J,SHAO G C,CUI J T,et al.Yield,fruit quality and water use efficiency of tomato for processing under regulated deficit irrigation:A meta-analysis[J].Agricultural Water Management,2019,222:301-312.
[19]邵光成,吳世清,房凯,等.生物炭添加提高渍水条件下番茄产量改善品质[J].农业工程学报,2019,35(19):160-167.
[20]CHENG Z Q,LEI S N,LI Y,et al.Revealing the variation and stability of bacterial communities in tomato Rhizosphere microbiota[J].Microorganisms,2020,8(2):170-185.
[21]TIAN B Y,ZHANG C J,YE Y,et al.Beneficial traits of bacterial endophytes belonging to the core communities of the tomato root microbiome[J].Agriculture,Ecosystems and Environment,2017,247:149-156.
[22]MA R Q,CAO Y,CHENG Z Q,et al.Identification and genomic analysis of antifungal property of a tomato root endophyte Pseudomonas sp.p21[J].Antonie van Leeuwenhoek,2017,110(3):11-22.
[23]黎烨,熊娟,张婷,等.番茄根内生细菌的促生及其优势种群的筛选和分析[J].福建农业科技,2019(11):17-24.
[24]KIRAN R G,PAUL F D,ANDREAS E,et al.Soil Inoculation with Bacillus spp.Modifies Root Endophytic Bacterial Diversity,Evenness,and Community Composition in a Context-Specific Manner[J].Microbial ecology,2018,76(3):741-750.
[25]ALMEIDA AR,ALVES M,DOMINGUES I,et al.The impact of antibiotic exposure in water and zebrafish gut microbiomes:A 16S rRNA gene-based metagenomic analysis[J].Ecotoxicology and Environmental Safety,2019,186:1-10.
[26]徐琼,刘洋,曲勤凤,等.高通量测序分析不同地区红腐乳细菌多样性[J].食品科学,2020,41(10):110-116.
[27]雷少楠,程志强,熊娟,等.患根肿病的上海青根内生菌组成和结构的研究[J].中国农学通报,2017,33(33):39-45.
[28]GABRIELA Q,GORKA E,RICARDO A,et al.Radial water transport in arbuscular mycorrhizal maize plants under drought stress conditions is affected by indole-acetic acid(IAA)application[J].Journal of plant physiology,2020,246-247:1-12.
[29]ZHANG Y Z,LI Y P,HASSAN M J,et al.Indole-3-acetic acid improves drought tolerance of white clover via activating auxin,abscisic acid and jasmonic acid related genes and inhibiting senescence genes[J].BMC Plant Biology,2020,20(1):1-12.
[30]GOREN-SAGLAM N,HARRISON E,BREEZE E,et al.Analysis of the impact of indole-3-acetic acid(IAA)on gene expression during leaf senescence in Arabidopsis thaliana[J].News of Science,2020,26:733-745.
[31]韓丽珍,刘畅,周静.接种促生菌对花生根际土壤微生物及营养元素的影响[J].基因组学与应用生物学,2019,38(7):3065-3073.
[32]金月波.水稻根际促生菌筛选及其对水稻促生效果研究[J].辽宁农业职业技术学院学报,2020,22(2):4-7.
[33]韦江璐,覃英,谢显秋,等.促生菌对土壤养分、酶活性及细菌群落功能多样性的影响[J].南方农业学报,2020,51(10):2348-2357.
(责任编辑:柯文辉)
