多形拟杆菌菌株的群体感应信号合成酶的检测及致病机理研究
2021-09-10吴至成王乙妃吴琳
吴至成 王乙妃 吴琳
摘要:解析一株从临床病人血液标本里筛查出来的菌株T6000的基因组序列信息,并对其进行群体感应信号合成酶的基因检测。使用Illumina Miseq测序平台获取该菌的全基因组序列,将其全基因组序列经过注释的基因蛋白质序列提交KEGG数据库进行BLASTp比对分析,确定该菌潜在的重要感应信号合成酶。结果表明:t6000基因组序列全长6074257 bp,其中GC所占的百分比为42.88%。生物信息学分析结果表明t6000具有13种重要感应信号合成酶及致病机理,为药物靶位点的确立及新药的研发奠定理论基础。
关键词:多形拟杆菌;基因组测序;感应信号基因
厌氧菌在某些特定情况下可由正常菌群变为条件致病菌,约占临床内源性细菌感染50-60%,其中类杆菌属细菌分离率最高,常与其他细菌混合感染,造成腹腔内、骨盆及皮肤等感染,有很高的发病率和致死率[1-3]。類杆菌属有超过20种,以脆弱类杆菌、多形类杆菌、普通类杆菌等在临床感染最为常见。
群体感应(quorum sensing, QS)是广泛存在于细菌群体中的依赖细菌密度的信号通讯系统,可通过感知其浓度变化来调节群体行为的现象,如致病菌定植、毒力因子的表达、耐药性和胞外酶的产生,群集运动,生物膜形成和生物发光等[3,4]。因此,研发类杆菌属细菌的的QS系统为新型药物的治疗靶点,可更好地控制细菌群体感染及耐药性的产生,是今后研究的一个重要方向。本研究从临床血液标本中分离出类杆菌t6000,通过Illumina测序平台对其全部基因组进行测序,然后使用相关软件对测序数据进行QS合成酶基因组组装和编码基因预测,以期了解t6000的群体感应信号的类型和作用机制。
1 材料与方法
1.1材料、试剂与仪器
t6000是海南医学院第一附属医院临床病人血液标本里筛查出来的菌株。使用苏州金唯智生物科技有限公司提供的Buffer1、Buffer2、Buffer3、研磨珠等。
1.2 方法
1.2.1基因组DNA的提取
取45ml新鲜培养菌液至50ml离心管中, 4000rpm,5-10min,弃上清;将研磨珠倒入上述收集到的菌体沉淀中,加入1.2ml Buffer1,涡旋打散菌体;加入120ul Buffer2,转速涡旋振荡10min混匀,加入400ul Buffer3,再次涡旋混匀,冰上静置10min;4000rpm 10min将上清移至新的2.0mlEP 管中,上清即为粗提的核酸;电泳并送至金唯智NGS实验室测序。
1.2.2 基因组测、组装与编码基因预测
对以上提取保留的基因组DNA通过Illumina HiSeq 2000测序平台对其全部基因组进行有规律测序,然后使用相关软件(如cutadapt v1.9.1软件)对测序数据进行处理和分析,把基因中的接头序列以及基因的引物去掉,同时也去掉基因中质量在20以内的碱基序列和含量在10%以上的N碱基序列,从而使试验得到更加稳定、有效的数据(clean data)。
对通过以上试验测试得出的数据,利用拼接软件velvet 1. 2. 10 完成序列拼接。采用SSPACE 3.0序列对比软件对所得到的序列与资源库中的序列进行对比分析,并且在序列中通过相互之间的关系对其中插入一些序列片段,从而形成了一条新的序列,即scaffold序列。最后再通过GapFiller1-10软件把gap添加到得到的scaffold序列上,还可以对这条序列进行编辑从而使之得到延伸,就得到了含量在10%以下的N碱基以及质量在20上的碱基序列的scaffold序列,再采用prodigal3.02软件对基因组进行预测即可。
1.2.3基因功能注释
通过上述方法得到基因预测的蛋白序列,使用BLAST软件2.2.31+与基因组百科全书(即KEGG)数据库进行序列比对(E-value<1e-5),完成基因功能注释。
2 结果与分析
2.1 基因组装与编码基因预测
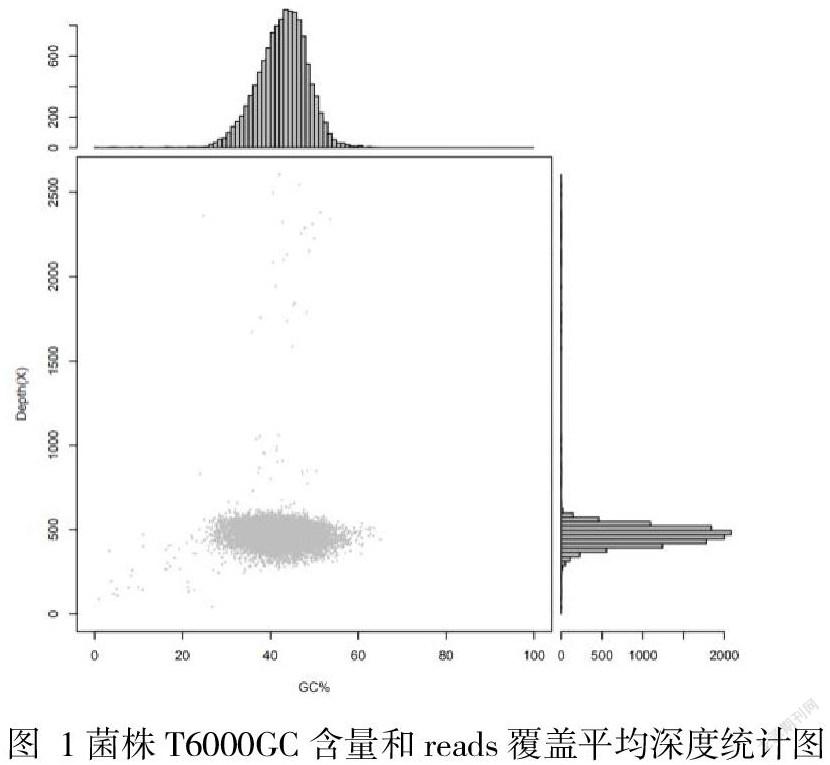
对基因的序列通过Velvet 1.2.20软件进行序列拼接试验(原始的序列19252032个reads,读长2887804800 bp,插入片段大小350bp),拼接获得51个contigs,N50大小为329919bp。经过优化与补洞处理后,获得完整的脆弱拟杆菌株t6000基因组序列,全长6074257 bp,其中GC所占的百分比为42.88%,每一段序列的长度大多数在119103.08bp左右,其中编码区的基因大约在4796左右,在整条序列的基因组所占90.39%左右,平均长度为1144.87bp。根据菌株t6000含量和reads覆盖平均深度统计图可得出(图1),把基因进行拼接后所得到的GC含量的所占比例大约在30~55%,主要分布的序列深度阶段在400~650×的区域之间。
2.2 KEGG数据库基因注释分析
KEGG数据库(即KEGG),基因组信息通过对不同的生物过程进行数据分析和整合可以画出与之相适合的生物代谢通路图 [7]。试验将菌株的所对应的基因序列和KEGG数据库信息对比分析,结果表明,注释基因2544个,占基因总数的53.04%,该菌株的编码基因主要定位在氨基酸生物合成、氨基酸和核苷酸糖代谢和碳代谢等代谢通路。
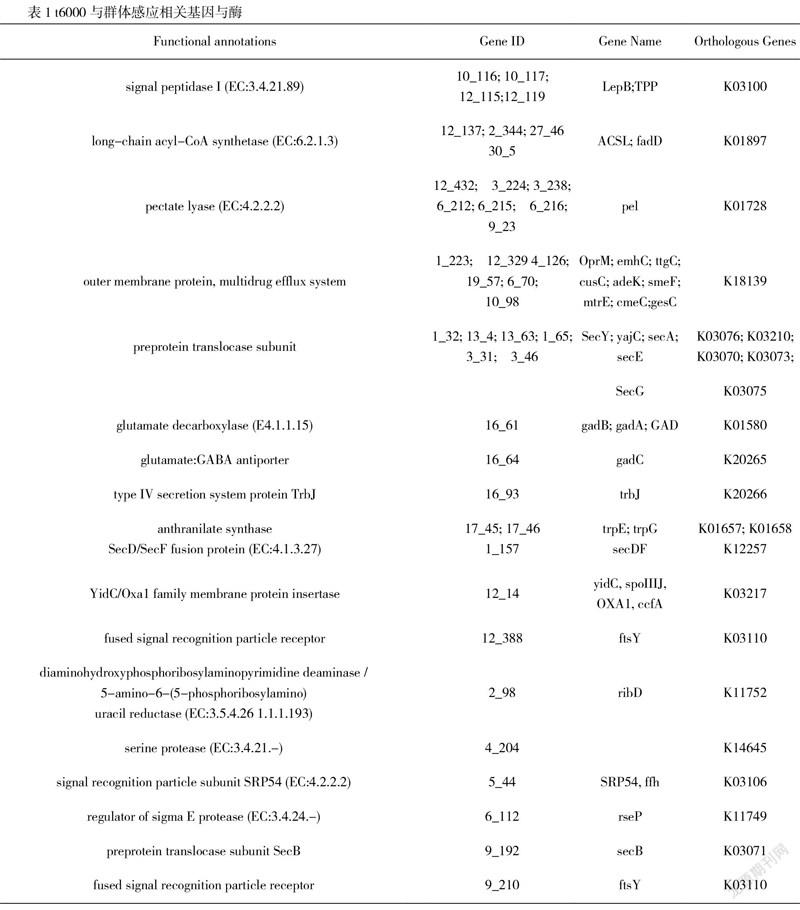
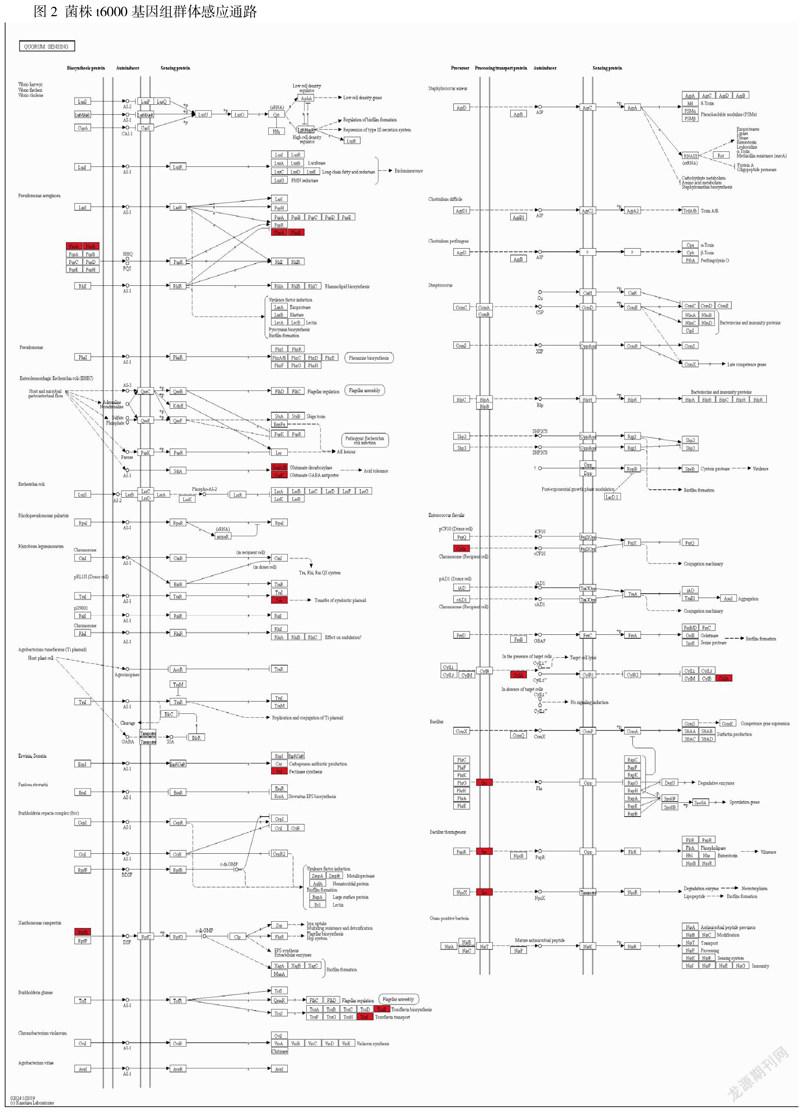
根据KEGG对该菌株的分析结果可知,195个代谢通路中,其中与群体感应相关的基因包括41个,这些基因组成不同的酶类参与调控细菌t6000群体行为,参与的酶及对应的基因信息见表1。将群体感应基因在通路图上展示出来,并与目前已报道的感应信号合成酶的基因进行对比,迅速了解大部分感应信号分子的致病机制和代谢途径,信号通路如下图2所示。通路图中,红色标注的是注释到的基因[6-20]。
多形類杆菌t 6000 可以编码13种群体感应合成酶:PhnA、PhnB调控鼠李糖脂的生物合成;Gad A/B、GadC调控耐酸性质;Trb 调控转运共生质粒;Pel调控角质酶合成;RpfB调控铁摄取、多重耐药性及排出有害物质、鞭毛生物合成、Hrp系统、EPS合成、生物膜的形成、胞外酶等多个过程;ToxE、ToxI分别调控毒黄素的生物合成及转运;CcfA调控接合机制;CylA调控靶细胞裂解;Sec调控降解酶、芽孢形成的基因、毒力、生物膜形成及菌体的衰亡。
参考文献:
[1]Umemura T, Hamada Y, Yamagishi Y, et al. Clinical characteristics associated with mortality of patients with anaerobic bacterema[J]. Anaerobe, 2016,39(6): 4550.
[2]王远贵,张义熙.外科厌氧菌感染与治疗[J].中国现代医生,2008,46(15):122-123.
[3]Goldstein EJC, Citron DM. Resistance trends in antimicrobial susceptibility of anaerobic bacteria, part I[M].Clinical Microbiology Newsletter, 2011,33(1):1-8.
[4]Miller MB, Bassler BL. Quorum sensing in bacteria[J]. Annual Review of Microbiology, 2001,55(1):165-199.
[5]March JC, Bentley WE. Quorum sensing and bacterial cross-talk in biotechnology[J].Current Opinion in Biotechnology, 2004,15(5):495-502.
[6]Case RJ, Labbate M, Kjelleberg S. AHL-driven quorum-sensing circuits: their frequency and function among the Proteobacteria[J]. Isme Journal,2008, 2(4):345-349.
[7]FlavierAB, Clough SJ, Schell MA, et al. Identification of 3-hydroxypalmitic acid methyl ester as a novel autoregulator controlling virulence in Ralstonia solanacearum[J]. Molecular Microbiology,1997, 26(2):251-259.
[8]Kai K, Ohnishi H, Shimatani M, et al. Methyl 3-hydroxymyristate, a diffusible signal mediating phc quorum sensing in Ralstonia solanacearum[J]. Chembiochem,2015,16(16):2309-2318.
[9]Genin S, Denny TP. Pathogenomics of the Ralstoniasolanacearum species complex[J]. Annual Review of Phytopathology,2012,50(1):67-89.
[10]Tao F, Swarup S, Zhang LH. Quorum sensing modulation of a putative glycosyltransferase gene cluster essential for Xanthomonas campestris biofilm formation[J]. Environmental Microbiology,2010,12 (12):3159-3170.
[11]Ryan RP, An SQ, Allan JH, et al. The DSF family of cell–cell signals: an expanding class of bacterial virulence regulators[J]. Plos Pathogens.2015, 11(7):1-14.
[12]Wei Y, Perez LJ, Ng WL, et al. Mechanism of Vibrio choleraautoinducer-1 biosynthesis[J]. Acs Chemical Biology,2011,6(4):356-365.
[13]Spirig T, Tiaden A, Kiefer P, et al. The Legionella autoinducer synthaseLqsA produces an alpha-hydroxyketone signalingmolecule[J]. Journal of Biological Chemistry, 2008,283(26):18113-18123.
[14]Hornung C, Poehlein A, Haack FS, et al. The Janthinobacterium sp. HH01 genome encodes a homologue of the V.cholerae CqsA and L.pneumophila LqsA autoinducer synthases[J]. Plos One, 2013,8(2):142-142.
[15]Pereira CS, Thompson JA, Xavier KB. AI-2-mediated signalling in bacteria[J]. Fems Microbiology Reviews, 2013,37(2):156-181.
[16]Peixoto RJM, Miranda KR, Ferreira EO, et al. Production of AI-2 is mediated by the S-ribosylhomocysteinlyase gene luxS in Bacteroides fragilis and Bacteroides vulgatus[J].Journal of Basic Microbiology, 2014,54(7):644-649.
[17]Lee J, Wu J, Deng Y,et al. A cell–cell communication signal integrates quorum sensing and stress response[J]. Nature Chemical Biology, 2013,9(5):339-343.
[18] Pesci EC, Milbank JB, Pearson JP, et al. Quinolone signaling in the cell-to-cell communication system of Pseudomonas aeruginosa[J]. Proceedings of the National Academy of Scienceof the United States of America, 1999, 96(20): 11229-11234.
[19]Brameyer S, Kresovic D, Bode HB, et al. Dialkylresorcinols as bacterial signaling molecules[J]. Proceedings of the National Academy of Science of the United States of America,2015,112(2):572-577.
[20]Brachmann AO, Brameyer S, Kresovic D, et al. Pyrones as bacterial signaling molecules[J].Nature Chemical Biology, 2013,9(9):573-578.
基金:海南省自然科学基金(417146);
海南省大学生创新创业训练项目(2017089)
[基金项目]海南省自然基金项目(817324)
[作者简介]吴至成(1972-),男,海南海口人,副主任檢验师
[通讯作者]吴琳(1984-),女,海南海口人,副教授
