阿托伐他汀抗人乳腺癌的直接作用靶点的生物信息学分析
2021-09-02戴秋月郎吉萍吕萍郭志刚
戴秋月, 郎吉萍, 吕萍, 郭志刚
(南方医科大学 南方医院 惠侨医疗中心 心血管内科, 广东 广州 510515)
对于全球女性来说,乳腺癌是最常见的癌症,也是癌症死亡的主要原因[1].目前,乳腺癌的治疗方法主要包括手术治疗、放疗、辅助化疗、内分泌治疗和靶向治疗[2].虽然上述方法能降低乳腺癌的死亡率,但考虑到乳腺癌的异质性,仍有一部分患者对这些方法没有反应,因此,研发新的治疗方法对于降低乳腺癌患者死亡率,提高乳腺癌患者生存率尤为重要[3].
他汀类药物是一种3-羟基-3-甲基戊二酰辅酶A(3-hydroxy-3-methyl glutaryl coenzyme A,HMG-CoA)还原酶抑制剂,他汀类药物通过竞争性的抑制HMG-CoA还原酶,抑制肝脏内源性胆固醇的产生,降低异常胆固醇和血脂水平,能够有效地减少冠心病患者或高危人群心血管事件的发生,并可预防中风[4].循证医学以及大量临床试验证实,他汀类药物可以明显降低心血管事件的发生风险,奠定了他汀类药物在调脂方面的基石作用[5-7].他汀类药物常与其他降脂药物联合应用于冠心病的一级和二级预防[8],并且可能会降低乳腺癌复发的风险[9].同时,越来越多的研究证明他汀类药物在肿瘤的治疗中是有效的:阿托伐他汀能通过与有机阴离子转运体(solute carrier organic anion transporter family member 2B1,SLCO2B1)竞争性结合来阻断肾上腺雄激素硫酸脱氢表雄酮(dehydroepiandrosteronesulfate,DHEAS)的摄取,从而减少肿瘤内可用的雄激素库,进而抑制前列腺癌的进展[10];阿托伐他汀与舒林酸联用能够通过增强无瘤小鼠干细胞的多能性,负调控细胞周期进程,从而抗肿瘤活性,抑制肿瘤生长,降低结肠肿瘤的发生率[11];此外,阿托伐他汀还能够显著降低肝癌的发生率,特别是患有糖尿病或肝硬化的患者[12].然而,目前他汀类药物如何抑制乳腺癌的发生和进展的机制尚不明确.本研究利用生物信息学分析,通过搜索公共数据库预测和分析阿托伐他汀抑制乳腺癌细胞的潜在作用靶点,为后续更深入地研究阿托伐他汀提供依据,也为他汀类药物及其类似物的应用奠定基础.
1 材料与方法
1.1 识别阿托伐他汀直接作用蛋白靶点
从DrugBank(https://www.drugbank.ca)中查找阿托伐他汀(atorvastatin)的分子信息及其作用靶点,确定阿托伐他汀的直接作用蛋白靶点(direct protein targets,DPTs).
1.2 构建阿托伐他汀DPTs的PPI网络和分析信号通路
采用 STRING(https://string-db.org/cgi/input.pl)在线数据库构建阿托伐他汀DPTs的蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络,并在操作栏选择分析阿托伐他汀DPTs的KEGG信号通路.设定物种为人种,设置直接及间接关联均不超过50个,筛选出最高置信度(得分0.900)的阿托伐他汀间接作用蛋白靶点(indirect action protein targets,IPTs),同时构建IPTs间的PPI网络,并分析其KEGG信号通路,用Cytoscape软件可视化.
1.3 分析阿托伐他汀DPTs在乳腺癌中的基因组学数据
cBioPortal(http://www.cbioportal.org/)为开放平台,可在其中探索多维度癌症基因组数据.OncoPrint为可视化基因在肿瘤样本中突变、拷贝数、表达情况的工具.将IPTs中与癌症相关的基因导入cBioPortal,搜索选定乳腺癌数据库,分析这些基因在乳腺癌中的表达改变,并通过OncoPrint可视化.
1.4 人乳腺癌芯片数据来源
本研究从美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)公共基因芯片数据平台(gene expression omnibus,GEO)(https://www.ncbi.nlm.nih.gov/geo/)数据库中的GPL570[HG-U133_Plus_2] Affymetrix Human Genome U133 Plus 2.0 Array下载基因芯片数据集GSE5764、GSE21422,获取芯片的标准化数据Series Matrix File(s),并将芯片数据按照组织来源分为乳腺癌组织组和正常乳腺组织组.
1.5 筛选人乳腺癌差异表达基因
用R4.0.3软件读取下载矩阵文件,用Limma函数进行差异表达分析,参数选择|log2FC|>1,错误发现率(false discovery rate,FDR)<0.05,筛选乳腺癌和正常组织间的差异表达基因(differentially expressed genes,DEGs),将2个芯片数据差异分析的结果用韦恩图(http://bioinformatics.psb.ugent.be/webtools/Venn/)进行整合并取交集获得乳腺癌DEGs,利用David在线基因ID转换工具(https://david.ncifcrf.gov/)对DEGs的ID进行转换.
1.6 获取阿托伐他汀对人乳腺癌潜在的治疗靶点基因
将阿托伐他汀的IPTs与乳腺癌的DEGs导入韦恩图(https://online.visual-paradigm.com/cn/)进行整合并取得交集,得到阿托伐他汀在人乳腺癌中的潜在治疗靶点基因.
2 结果
2.1 阿托伐他汀的DPTs
通过DrugBank数据库检索阿托伐他汀,其直接作用蛋白靶点(direct protein targets,DPTs)共有5个:HMGCR(3-hydroxy-3-methylglutaryl-CoA reductase)、DPP4(dipeptidyl peptidase 4)、AHR(aryl hydrocarbon receptor)、HDAC2(histone deacetylase 2)、NR1I3(nuclear receptor subfamily 1 group I member 3)(表1).
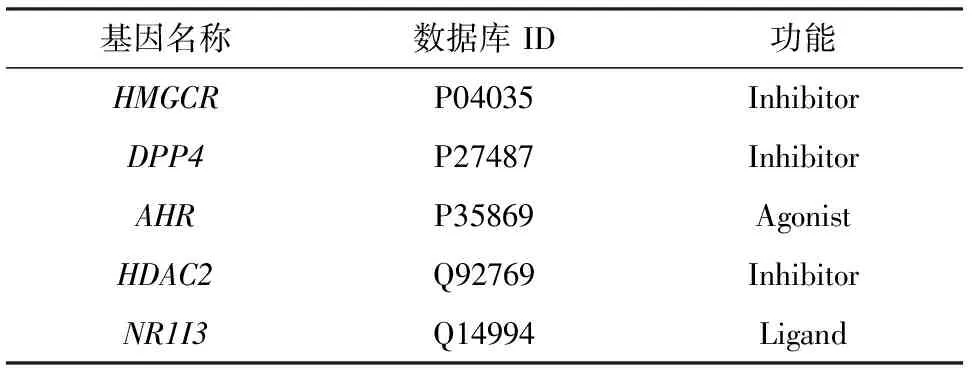
表1 DrugBank中阿托伐他汀的DPTs1)Table 1 Direct protein targets of atorvastatin in DrugBank1)
2.2 阿托伐他汀DPTs间的PPI网络及KEGG信号通路
使用STRING数据库构建阿托伐他汀DPTs间的PPI网络,并分析其KEGG信号通路(图1、表2).结果显示:KEGG通路包括萜类化合物生物合成支柱信号通路、甲状腺激素信号通路、癌症中的转录失调信号通路、亨廷顿氏舞蹈病信号通路.其中,HMGCR和HMGCS1(3-hydroxy-3-methylglutaryl-CoA synthase 1)与萜类化合物生物合成支柱信号通路相关,而HDAC2和SIN3A(SIN3 transcription regulator family member A)与甲状腺激素信号通路、癌症中的转录失调信号通路、亨廷顿氏舞蹈病信号通路相关.
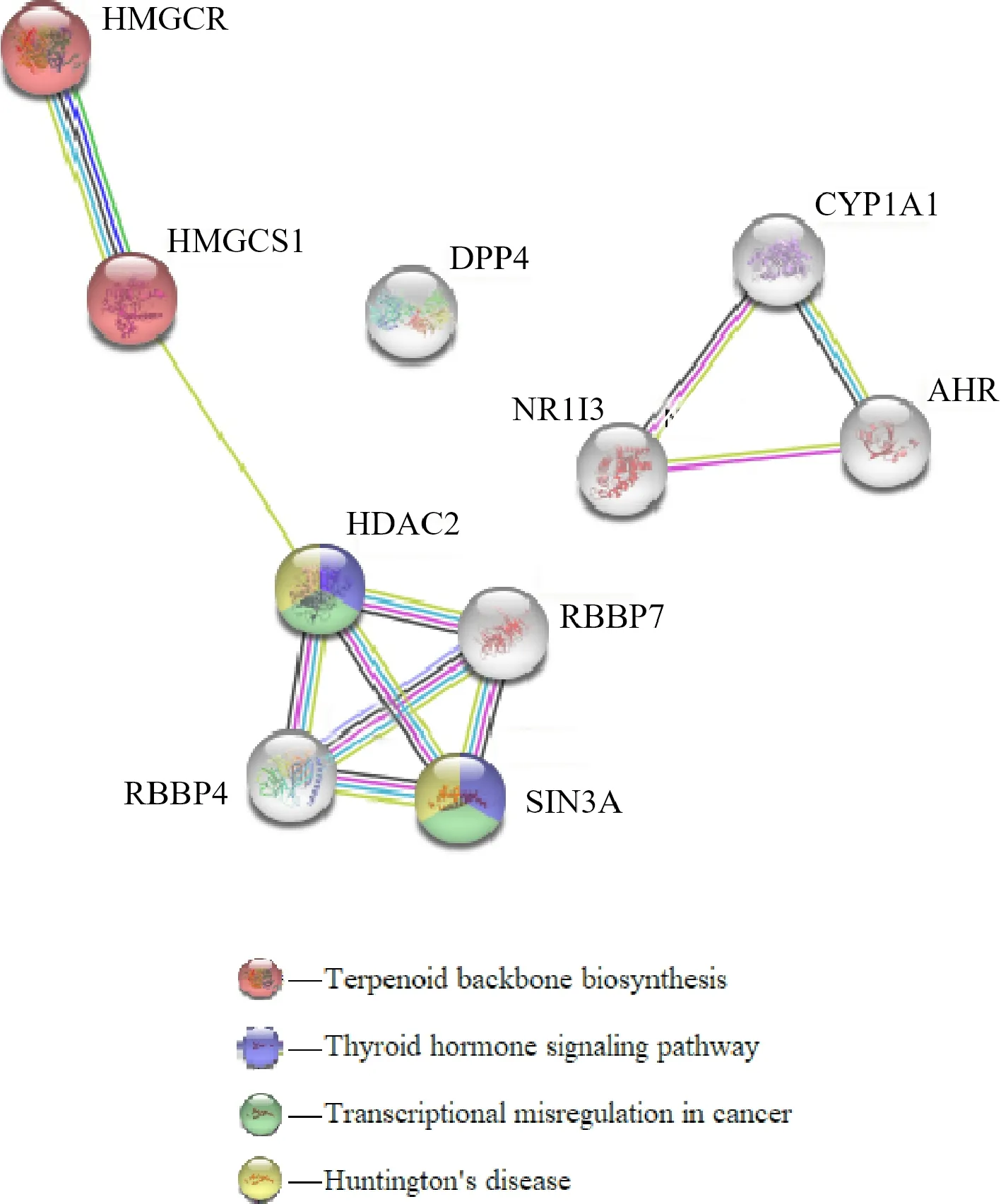
图1 阿托伐他汀DPTs间蛋白-蛋白相互作用(PPI)网络
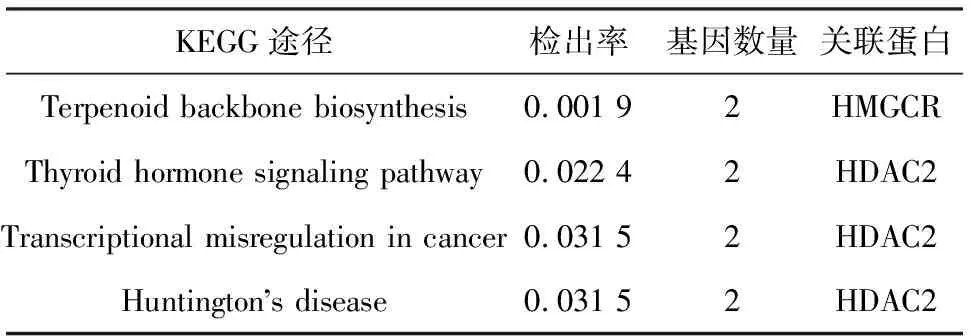
表2 阿托伐他汀DPTs的KEGG通路列表
2.3 阿托伐他汀的IPTs及其KEGG信号通路
利用STRING数据库构建阿托伐他汀的IPTs,分析其KEGG信号通路,并用Cytoscape软件可视化(图2A),其中HDAC2、EP300、HDAC1、RBBP7、RBBP4、CDC5L关联程度最高.KEGG信号通路分析表明,这些基因主要富集于剪接体、病毒性肿瘤、肿瘤、人乳头瘤感染等信号通路(表3),其中14个IPTs富集于肿瘤信号通路,分别为ARNT(aryl hydrocarbon re-eptor nuclear translocator)、ARNT2(aryl-hydrocarbon receptor nuclear translocator 2)、BCL2L1(B-cell lymphoma-2 like 1)、CDKN1A(cyclin dependent kinase inhibitor 1A)、CREBBP(CREB binding protein)、EP300(E1A binding protein p300)、HDAC1(histone deacetylase 1)、HDAC2(histone deacetylase 2)、HSP90AA1(heat shock protein 90 alpha family class A member 1)、MDM2(MDM2 proto-oncogene)、NCOA1(nuclear receptor coactivator 1)、NCOA3(nuclear receptor coactivator 3)、RELA(RELA proto-oncogene, NF-kB subunit)和TP53(tumor protein p53).将这14个IPTs再次导入STRING数据库进行分析,可获得114个阿托伐他汀与肿瘤相关的IPTs(图2B).
2.4 乳腺癌中14个阿托伐他汀抗肿瘤相关靶点的基因特征
为了进一步了解14个与肿瘤相关的阿托伐他汀IPTs的表达变化及功能,本研究利用cBioPortal检测了这14个靶点在乳腺癌患者中的肿瘤基因组改变(图3B).OncoPrint可视化结果表明在3项人乳腺癌研究(MSK研究[13]、METABRIC研究[14]、TCGA研究[15])中,几个基因的变化范围在0.9%到35%,其中TP53发生变化比例最大.2 612例患者(51%)至少有一种ARNT、ARNT2、BCL2L1、CDKN1A、CREBBP、EP300、HDAC1、HDAC2、HSP90AA1、MDM2、NCOA1、NCOA3、RELA和TP53发生改变.其中BCL2L1表现为基因扩增和错义突变,ARNT2表现为基因扩增和截断突变,CDKN1A、HDAC1、HDAC2、HSP90AA1、NCOA1、RELA表现为基因扩增、错义突变和截断突变,CREBBP表现为基因扩增、错义突变、截断突变和基因融合,ARNT、MDM2表现为基因扩增、纯合缺失和错义突变,EP300、NCOA3、TP53变现为基因扩增、错义突变、截断突变和纯合缺失(图3A).
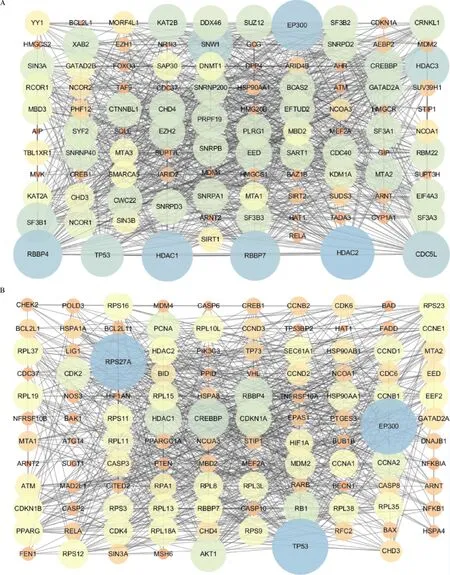
A:阿托伐他汀的IPTs;B:阿托伐他汀与肿瘤相关的IPTs.圆形越大、颜色越蓝表明该基因关联程度越高,圆形越小、颜色越橙表明该基因关联程度越低.两个基因间的连线代表着两个基因存在关联

表3 阿托伐他汀IPTs的KEGG信号通路Table 3 KEGG signaling pathways of the IPTs of atorvastatin
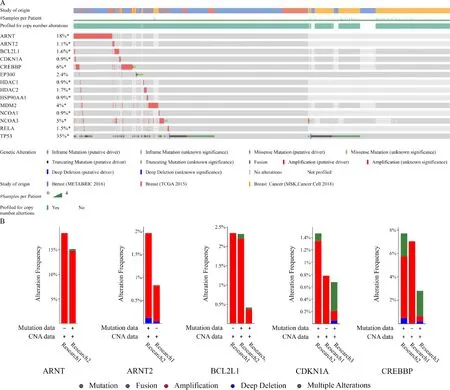
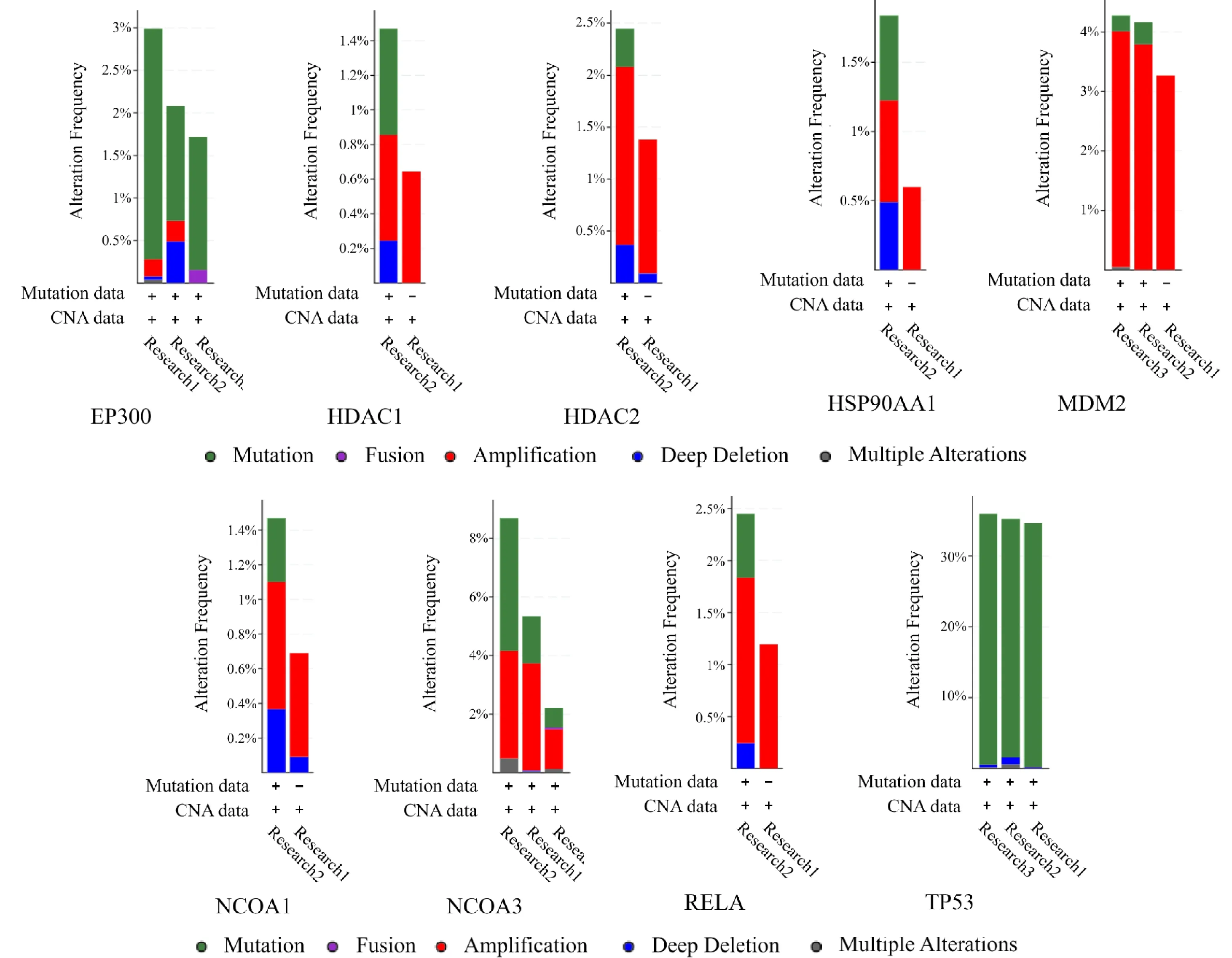
A:OncoPrint可视化14个阿托伐他汀IPTs(ARNT、ARNT2、BCL2L1、CDKN1A、CREBBP、EP300、HDAC1、HDAC2、HSP90AA1、MDM2、NCOA1、NCOA3、RELA和TP53)在3项乳腺癌研究样本中的变化情况;B:14个阿托伐他汀IPTs在3项乳腺癌研究中的变化概览;Research 1:Breast(METABRIC 2016), Research 2:Breast(TCGA 2015),Research 3:Breast Cancer(MSK,Cancer Cell 2018).
2.5 乳腺癌的DEGs
利用R4.0.3软件对GEO中2个乳腺癌表达谱芯片数据进行差异分析并用火山图可视化(图4).使用韦恩图将上述结果进行整合,结果显示在2个芯片中,共有75个DEGs,表达上调的基因21个,表达下调的基因54个(图5).利用David将75个差异基因ID进行转换,获得29个差异表达基因(图6A).
2.6 预测阿托伐他汀抗乳腺癌的潜在靶点
通过比较29个乳腺癌差异表达基因与114个阿托伐他汀间接作用蛋白靶点,本研究发现了1个基因(CCNA2)可能是阿托伐他汀抗乳腺癌的潜在靶基因(图6).
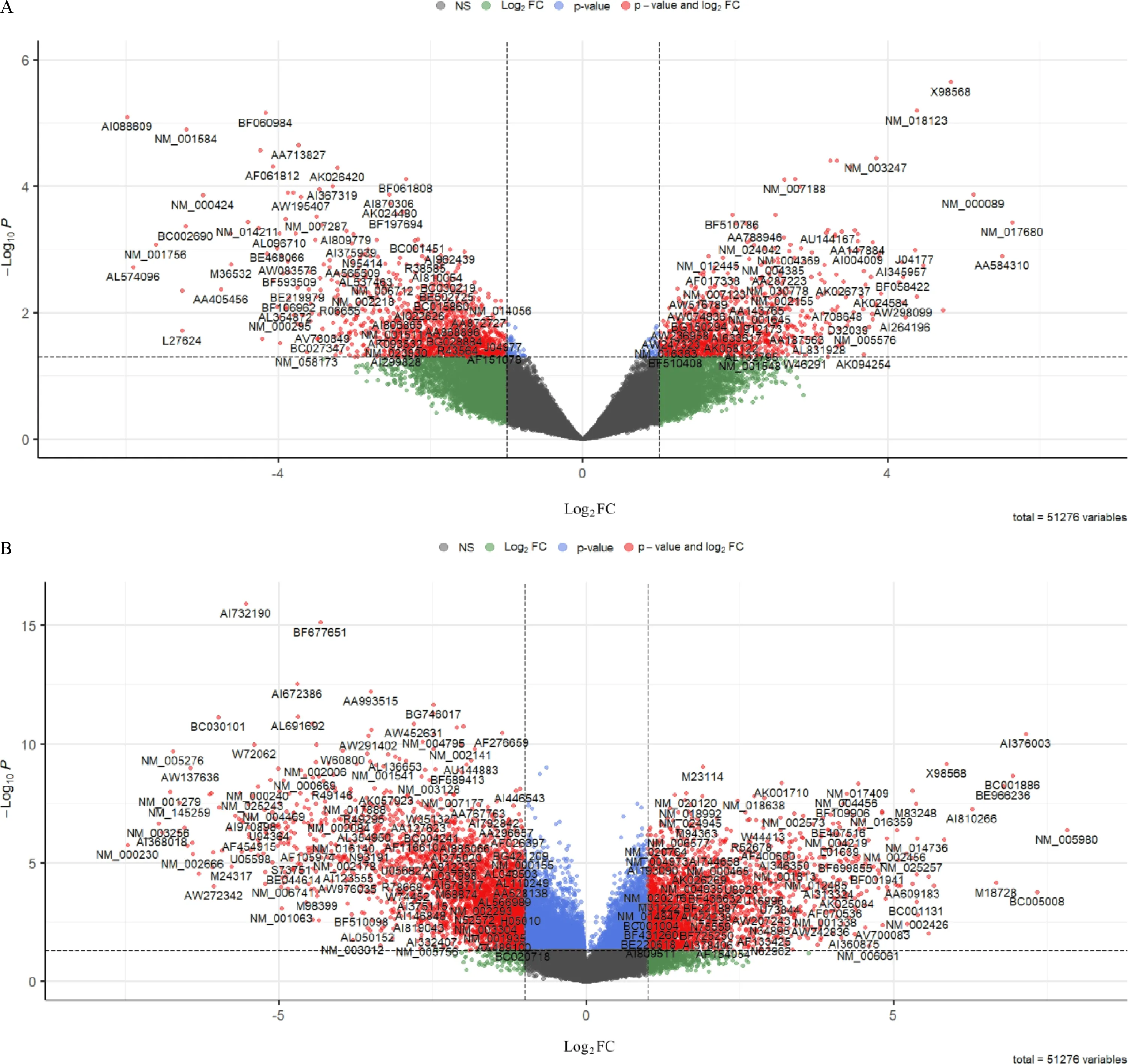
A:GSE5764基因表达谱火山图;B:GSE21422基因表达谱火山图.X轴表示乳腺癌组织与正常组织间基因表达的倍数变化,Y轴表示差异的统计学意义,红色代表显著差异表达的基因.

A:表达上调的基因数量; B:表达下调的基因数量 A:Up-regulated genes; B:Down-regulated genes.
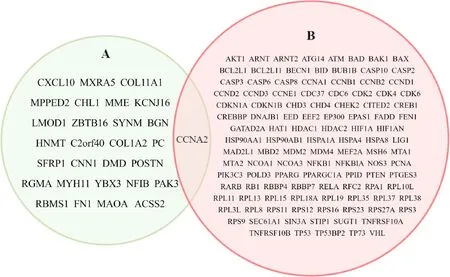
A:乳腺癌的DEGs;B:阿托伐他汀的IPTsA:Differentially expressed genes in breast cancer;B:Indirect action of atorvastatin protein target
3 讨论
乳腺癌是女性最常见的肿瘤,也是35~55岁女性中最常见的死亡原因[16].肿瘤的预防一般分为3级,然而对于乳腺癌,因其病因并未完全明确,一级预防难以实现,目前早期发现乳腺癌的最佳方法是乳腺病的普查,以及加强卫生知识宣传及自我检查[17].同时,乳腺癌的超声表现具有特征性,有利于乳腺癌的诊断[18].虽然乳腺癌的治疗手段丰富且有一定效果,但乳腺癌的发病率逐年上升,发病年龄逐渐年轻化,因此寻找新的治疗方法和手段来降低乳腺癌的复发率,提高患者的生存率尤为重要.
临床上,他汀类药物常用于降低胆固醇水平和冠心病的预防[19],而早在20世纪90年代初,他汀类药物与非心源性死亡,尤其是与肿瘤相关的死亡就引起了广泛关注[20].他汀类药物的抗肿瘤潜力常取决于其亲脂性,亲脂性他汀类药物较亲水性他汀类药物更容易进入包括癌细胞在内的不同组织细胞.一项对乳腺癌细胞系的体外研究表明,相比于亲水性他汀类药物,亲脂性他汀类药物有更强的抗癌功效[21].阿托伐他汀作为亲脂性他汀类药物的一种,近年来有大量的研究表明其能够降低肿瘤发生的风险[22-24],同时也有研究表明,使用阿托伐他汀能够降低患乳腺癌的发病率[25].另一方面,部分乳腺癌的治疗方式,如蒽环类药物、曲妥珠单抗以及放疗,常使患者暴露于潜在的心脏毒性中,导致患者患心血管疾病(cardiovascular disease,CVD)的风险增加,因此在乳腺癌治疗的同时应建立心脏保护策略,所以他汀类药物的使用变得尤为关键[26].阿托伐他汀用于乳腺癌的治疗具有较好的应用前景.
本研究借助网络公共数据库,利用生物信息学方法分析阿托伐他汀抗肿瘤作用的靶点蛋白,通过GEO数据库分析了乳腺癌的过表达基因,发现了阿托伐他汀抗乳腺癌的潜在靶点——细胞周期素A2 (cell cycle cyclin A2,CCNA2). CCNA2是细胞周期蛋白家族中一个重要的成员,是细胞S/G2检查点有关的重要细胞周期调控因子.它的一个重要作用就是激活CDK2调控染色体复制.越来越多的证据表明CCNA2参与了细胞周期检查点的控制,它的功能障碍是肿瘤发生中的早期事件[27].乳腺癌细胞中高表达的CCNA2可促进肿瘤细胞增殖和肿瘤生长,下调CCNA2的表达可抑制乳腺癌细胞增殖、诱导细胞周期S/G2期阻滞和促进肿瘤细胞凋亡,提示CCNA2在乳腺癌发生过程中发挥致癌作用[28].
本研究借助生物信息学的方法确定了阿托伐他汀抗乳腺癌的潜在靶点,为阿托伐他汀的抗癌研究提供了新的途径和新的思路.药物的研究过程中,确定药物靶点蛋白的作用尤为重要.尽管微阵列、蛋白质组学和高通量筛选方法已经被用于寻找药物靶点蛋白,但这些实验方法仍然十分的昂贵和耗时[29].随着大数据信息时代的到来,生物信息学分析逐渐被应用于药物靶点的预测,减少了冗杂的实验过程,降低了实验资源的损耗,是一种更为便捷、灵活的研究方法.不过,由于本研究仅基于公共数据库的生物信息学分析,具有一定的局限性,后续宜通过体内、外实验以及临床样本,进一步分析阿托伐他汀抗乳腺癌的具体通路以及作用机制.
作者贡献声明:
戴秋月:参与研究、设计方案、整理数据、文章撰写;郎吉萍:参与研究,共同探讨提出研究选题制定研究方案;吕萍:参与研究,共同探讨提出研究选题制定研究方案;郭志刚:提供研究经费以及技术、指导等支持,修订、审阅论文.
利益冲突声明:
本研究未受到企业、公司等第三方资助,不存在潜在利益冲突.
