SARS-CoV-2侵袭宿主细胞的关键蛋白酶TMPRSS2的生物信息学分析
2021-08-31张利娜文朝朝杜相欣张雨彤
张利娜,文朝朝,杜相欣,张雨彤,赵 欣,张 策,张 宇
新型冠状病毒肺炎(Corona Virus Disease 2019,COVID-19)是在2019年12月发现的一种由严重急性呼吸综合征冠状病毒2型(SARS-CoV-2)引起的新型呼吸道传染性疾病。当前研究认为,SARS-CoV-2侵袭宿主细胞主要依赖于宿主细胞的某些蛋白酶对病毒刺突蛋白(spikeprotein,S蛋白)的激活以及S蛋白与宿主细胞受体血管紧张素转化酶2(angiotensin converting enzyme-2,ACE2)的结合[1]。跨膜丝氨酸蛋白酶2(transmembrane protease serine-2,TMPRSS2)就是一种介导S蛋白活化以及最初促使病毒进入宿主细胞的关键宿主蛋白酶[2-4]。
1997年,Ariane等[5]首次鉴定出TMPRSS2基因,并发现该基因编码带有丝氨酸蛋白酶结构域的多聚体蛋白。此后,由于TMPRSS2基因在前列腺中高表达,关于TMPRSS2的研究主要集中在了前列腺癌的相关疾病中[6]。由于新冠疫情的影响,加上TMPRSS2与ACE2在呼吸系统上皮细胞中的表达,使得人们对TMPRSS2的研究热情进一步提高。以前的研究已经表明,TMPRSS2在流感病毒和MERS冠状病毒感染宿主过程中起到激活病毒的重要作用[7]。同样,在开展SARS-CoV-2相关研究时,发现TMPRSS2与ACE2在人类支气管、肺泡以及血红细胞中均存在共表达[8]。TMPRSS2蛋白酶通过裂解病毒S蛋白的S1/S2或S2′区域激活S蛋白[1,7,9-10],促进SARS-CoV-2与宿主细胞的ACE2结合,协助病毒进入宿主细胞。不仅如此,TMPRSS2也可能是SARS-CoV-2复制的关键蛋白酶[11],它的表达极大地促进了病毒的复制及合胞体的形成[12]。且另有研究显示,在TMPRSS2存在的状态下,SARS冠状病毒进入细胞的数量增加了2.6倍,而TMPRSS2的靶向消除可明显减少SARS冠状病毒进入细胞的数量[13]。由于以上原因,可以将TMPRSS2形象地看作SARS-CoV-2在人体内的“间谍”。更有趣的是,有研究者提出,尽管患有基础疾病的患者更易受到SARS-CoV-2的感染,但由于患头颈部癌或肺癌的患者体内TMPRSS2表达降低,这些患者反而不易受到SARS-CoV-2的感染[14]。由于TMPRSS2在SARS-CoV-2侵入人体时扮演了如此重要的角色,研究者们已将TMPRSS2抑制剂如甲磺酸卡莫司他等应用于对COVID-19的临床治疗当中[15]。
目前为止,虽然我国在疫情防控工作中取得了阶段性胜利,但新冠肺炎疫情在世界范围内仍然处于紧张形势,并且缺乏高效的特异性抗病毒药物可供临床应用。因此,对SARS-CoV-2侵袭宿主细胞的关键蛋白酶TMPRSS2的作用机制和阻滞途径的进一步研究显得格外紧迫,开发TMPRSS2相关的治疗药物可能是对抗SARS-CoV-2的一项有前途的对策。本文通过生物信息学技术对TMPRSS2基因及蛋白的结构功能做出了预测,结合现有的TMPRSS2相关资料,对TMPRSS2基因及蛋白进行了系统的分析,以期提出对其更全面的了解与认识,为相关抗病毒药物的研究提供借鉴与参考。
1 材料与方法
1.1 材料 TMPRSS2基因与蛋白数据均由美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)获得,2020年11月29日更新的TMPRSS2基因的Gene ID: 7113,TMPRSS2蛋白的GenBank: AAD37117.1。TMPRSS2蛋白的氨基酸序列如图1所示,预测过程中向所有分析软件所提交的蛋白质氨基酸序列均为此序列。

图1 TMPRSS2蛋白的氨基酸序列
1.2 方 法
1.2.1 对TMPRSS2蛋白基本性质的预测及分析基于表1所示软件。

表1 TMPRSS2蛋白基本性质的分析软件及网址
1.2.2 对TMPRSS2基因及蛋白表达情况的预测及分析 基于表2所示的软件或数据库。

表2 TMPRSS2基因和蛋白表达水平的预测软件及网址
1.2.3 对TMPRSS2蛋白各级结构的预测及分析 基于表3所示软件及数据库。

表3 TMPRSS2蛋白结构的预测软件及网址
1.2.4 对TMPRSS2蛋白翻译后修饰位点的预测 基于表4所示软件。

表4 TMPRSS2蛋白翻译后修饰位点的预测软件及网址
1.2.5 TMPRSS2相关的蛋白相互作用关系的预测 以STRING(http://string-db.org/)为预测分析软件,对能够与TMPRSS2蛋白产生相互作用关系的蛋白进行预测,并生成关系网络,同时进行GO及KEGG分析。
1.2.6 TMPRSS2蛋白进化情况分析及进化树的构建 在NCBI的基因库中选取20个不同物种的TMPRSS2蛋白质序列,与智人的TMPRSS2蛋白进行比对,用MEGA-X软件构建系统进化树,并进行序列分析。
2 结 果
2.1 TMPRSS2基本性质的预测结果 TMPRSS2基因位于人类第21号染色体,所处位置为21q22.3,外显子计数为15个。TMPRSS2蛋白共由492个氨基酸组成(图2),其中丝氨酸(Ser)含量最高,为44个,占氨基酸总数的8.90%,组氨酸(His)含量最低,为10个,仅占2.00%。该蛋白的N端氨基酸为甲硫氨酸(Met),其负电荷残基(Asp + Glu)总数为35个,正电荷残基(Arg + Lys)总数为39个。TMPRSS2蛋白的分子式为C2387H3654N650O709S34,总分子量为53 891.24 Da。根据ProtParam对TMPRSS2蛋白的分析,该蛋白的理论等电点为8.12,属于碱性蛋白。假设所有成对的Cys残基均形成胱氨酸,Abs 0.1%(=1 g/L)为2.192时,TMPRSS2蛋白在水中以280 nm测量的理论消光系数为118 145;假设所有Cys残基均消除,Abs 0.1%(=1 g/L)为2.167时,其理论消光系数为116 770。ProtParam对TMPRSS2蛋白在哺乳动物网织红细胞中的预估半衰期约为30 h,计算得出的不稳定性指数(II)为42.73,归类为不稳定蛋白。此外,TMPRSS2蛋白的脂肪指数为72.11,亲水性的平均值(Grand average of hydropathicity,GRAVY)为-0.253。
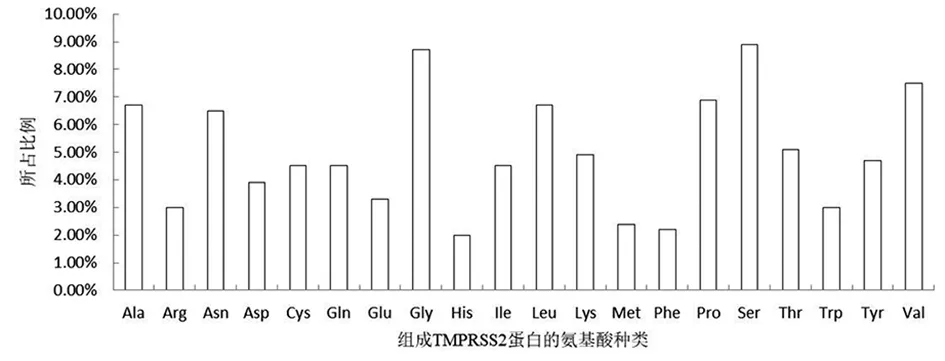
图2 TMPRSS2蛋白的氨基酸构成
为了进一步确定TMPRSS2蛋白的亲疏水性,Protscale对这一项目进行了预测和分析。如图3所示,TMPRSS2蛋白各个氨基酸的亲疏水性得分中,正值得分最高的位点为98位的丙氨酸,得分为2.378;负值得分最高的位点为340位的赖氨酸和341位的苏氨酸,得分均为-2.733。由图中可知,大多数氨基酸的得分分布在负值区域,因此推测TMPRSS2蛋白应为亲水性蛋白,且ProtParam软件对TMPRSS2蛋白的亲水性平均值的预测结果印证了这一推测。

图3 TMPRSS2蛋白的亲疏水性预测
TMPRSS2蛋白信号肽的预测由SignalP 4.0 Server进行。如图4所示,C、S、Y 3个数值分别代表剪切位点预测值、分泌蛋白预测值以及综合预测值,表5给出了3个预测值以及S平均值的具体数值,TMPRSS2蛋白的3个数值均未超过阈值(0.5)。综合分析可以判定该蛋白可能不存在信号肽结构。

表5 TMPRSS2蛋白信号肽的预测值
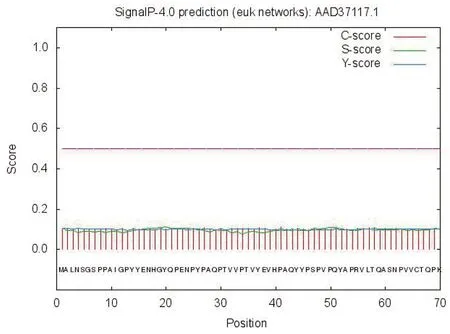
图4 TMPRSS2蛋白信号肽预测
进一步对该蛋白进行非经典分泌蛋白的预测。SecretomeP 2.0 server的预测结果显示,TMPRSS2蛋白的NN-分数为0.816,超过了该软件对哺乳动物非经典分泌蛋白预测的参考阈值0.6,因此可以判定TMPRSS2蛋白是一种非经典分泌蛋白。
蛋白质跨膜结构由TMHMM Server v.2.0进行预测及分析,结果如图5所示。图中蓝线表示蛋白质位于细胞膜内的区域,红线表示蛋白质的跨膜螺旋结构,紫线表示蛋白质位于细胞膜外的区域。经预测,TMPRSS2蛋白共有一个跨膜结构,其氨基酸序列第1-83位位于膜内,第84-106位为跨膜螺旋,第107-492位位于膜外。

图5 TMPRSS2蛋白跨膜结构预测
2.2 TMPRSS2基因及蛋白表达及定位情况的分析结果 TMPRSS2基因在人类正常器官中的表达情况如图6所示。NCBI的Gene数据库显示,对来自代表27种不同组织的95个人的组织样本进行RNA测序后发现,TMPRSS2基因主要表达于人类前列腺组织,其RPKM(reads per kilobase per million reads placed)为(167.21±59.571),其次是结肠(59.926±14.445)、胃(58.94±16.751)、小肠(42.493±12.84)、十二指肠(35.768±4.404)等消化系统器官。表达量最少的器官为骨髓,其RPKM仅为(0.002±0.004),另外在心脏(0.008±0.003)、卵巢(0.039±0.039)等器官中表达量也较少。

图6 TMPRSS2基因的器官分布情况
TMPRSS2蛋白的亚细胞定位由PSORT Ⅱ进行预测,结果如表6所示。从表中结果可知,该蛋白主要分布在细胞核中,该软件是通过Reinhardt法进行细胞质/核识别的,其可靠性为55.5%。此外,该软件通过一种新的信号肽预测方法PSG对TMPRSS2蛋白进行了分析,结果显示其PSG得分为-0.46,因此没有N端信号肽结构,这一结果与SignalP 4.0 Server的预测结果一致。PSORT Ⅱ软件还对TMPRSS2蛋白的膜拓扑类型进行了预测,认为该蛋白属于2型膜拓扑,其胞质尾为1-84号氨基酸,这与TMHMM Server v.2.0预测结果一致。综合上述分析可以证明,我们的预测结果可靠性是较高的。

表6 TMPRSS2蛋白的亚细胞结构定位情况
2.3 TMPRSS2蛋白二、三级结构的预测结果 SOPMA对TMPRSS2蛋白二级结构的预测结果如图7所示。经分析,该蛋白有82个氨基酸组成了α螺旋(蓝色),占比16.67%;96个氨基酸组成了延伸连(红色),占比19.51%;40个氨基酸组成了β转角(绿色),占比8.13%;274个氨基酸为无规卷曲(紫色),占比55.69%。因此,该蛋白主要结构类型为无规卷曲,其占比过半。

图7 TMPRSS2蛋白的二级结构预测
进一步分析了TMPRSS2蛋白的保守结构域,这一预测对该蛋白的生物学功能有着重要意义,通过对保守结构域的分析可以了解或预测蛋白的未知活性及功能。使用NCBI的蛋白质BLAST功能对TMPRSS2蛋白保守结构域进行比对分析,结果如图8所示,该蛋白共拥有3个超家族保守结构域,分别为LDLa超家族结构域、SRCR_2超家族结构域以及Tryp_SPc超家族结构域(表7),而且这些结构域都分布在氨基酸序列第100位以后,因此TMPRSS2蛋白的前100位氨基酸序列在不同物种之间的差异可能是比较大的。

表7 TMPRSS2蛋白的超家族结构域分析

图8 TMPRSS2蛋白的保守结构域
蛋白质三级结构分析由SWISS-MODEL进行预测。该软件针对TMPRSS2蛋白的氨基酸序列使用了50个模板进行分析预测,共得出3个可能的三级结构模型。选取最佳匹配模型(图9a、b、c),该模型GMQE为0.48,QMEAN为-1.88。由于结合位点是不保守的,因此排除TMPRSS2蛋白存在配体的可能性,且该蛋白属于单体蛋白。该模型覆盖了TMPRSS2蛋白第144-491位氨基酸,覆盖率达到71%。该模型的拉氏图如图9d所示,图中绿色、浅绿色、灰色区域代表三级结构不同位点预测结果的匹配程度,它们分别代表“偏好的”、“允许的”和“通常允许的”。拉氏图中的氨基酸与示意图中相同颜色的位点一一对应。综上所述,该三级结构模型作为TMPRSS2蛋白三级结构的预测结果,具有较高的可信度。

(a)TMPRSS2蛋白的三级结构示意图;(b)TMPRSS2蛋白与其同源蛋白相似度的比较;(c)与非冗余蛋白质三维数据文件结构集的比较;(d)TMPRSS2蛋白三级结构预测拉氏图
2.4 TMPRSS2蛋白翻译后修饰位点的预测 蛋白翻译后的修饰对于蛋白质结构与功能而言具有非常重要的意义,通过了解蛋白翻译后的修饰位点,可以为蛋白的深入研究提供更丰富的理论指导。N-糖基化位点的预测由NetNGlyc 1.0 Sever进行,如图10所示,该软件共预测出三个可能的N-糖基化位点,分别为:128 NPSN(得分0.606 9)、213 NTSA(得分0.527 3)、249 NSSR(得分0.633 8)。O-糖基化位点的预测由NetOGlyc 4.0 Server和YinOYang 1.2 Server两个软件进行预测。NetOGlyc 4.0 Server共预测出10个可能的O-糖基化位点,分别为:196 S(得分0.512 398)、204 S(得分0.512 999)、206 S(得分0.554 723)、207 T(得分0.687 043)、214 T(得分0.598 55)、215 S(得分0.601 53)、228 S(得分0.770 603)、232 S(得分0.513 097)、233 S(得分0.848 504)、238 S(得分0.636 903)。YinOYang 1.2 Server共预测出12个可能的O-糖基化位点,结果如图11所示,其中与NetOGlyc 4.0 Server共同预测出的位点共有3个,分别为:204 S、232 S、233 S,综合两个软件的预测结果,这3个位点最有可能成为TMPRSS2蛋白的O-糖基化位点。

图10 TMPRSS2蛋白的N-糖基化位点

图11 TMPRSS2蛋白的O-糖基化位点
蛋白磷酸化位点由NetPhos 3.1 Server进行预测,如图12所示,共得出可能的蛋白磷酸化位点49个(超过阈值0.5),选取其中得分大于0.9的共8个记录如下:15 Y(得分0.948)、71 S(得分0.990)、116 S(得分0.981)、167 S(得分0.992)、254 S(得分0.969)、337 Y(得分0.916)、412 S(得分0.988)、414 Y(得分0.921),它们最有可能成为TMPRSS2蛋白的磷酸化位点。

图12 TMPRSS2蛋白的磷酸化位点
2.5 TMPRSS2蛋白的相互作用预测结果 通过STRING软件对TMPRSS2蛋白的蛋白相互作用关系进行预测。在软件中输入TMPRSS2蛋白名称,选择物种为智人,选择高置信度(0.7),且目标蛋白不超过10个进行分析,结果如图13所示。预测结果共得到9个与TMPRSS2蛋白能够产生相互作用的蛋白,分别为:AR(得分0.940)、NKX3-1(得分0.856)、TMPRSS4(得分0.851)、ETV1(得分0.803)、SLC45A3(得分0.774)、ETV4(得分0.726)、ERG(得分0.726)、FKBP5(得分0.717)、FAM3B(得分0.706)。
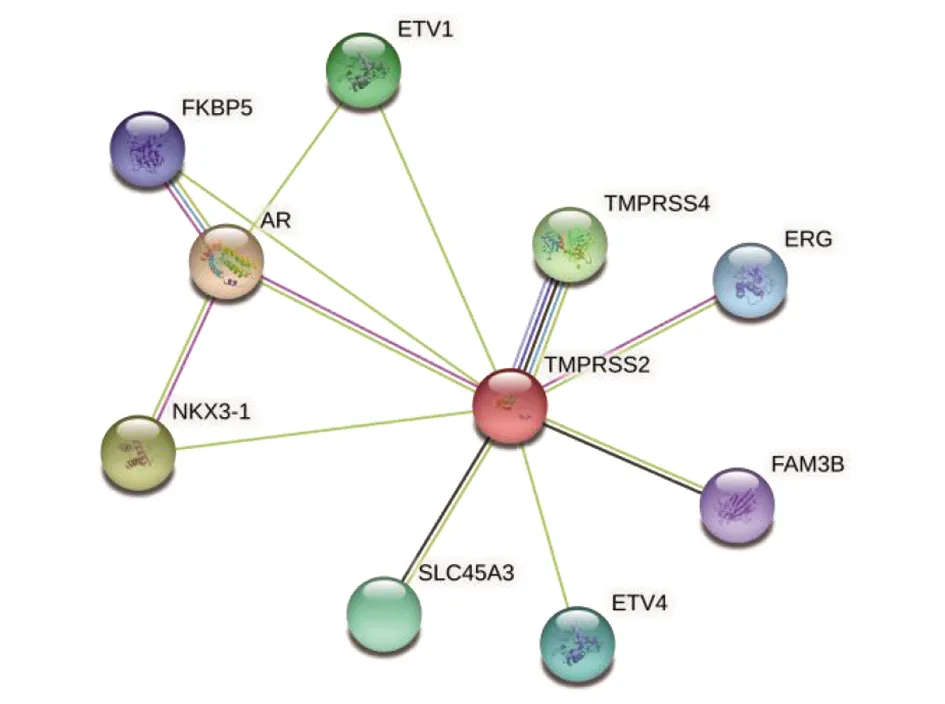
图13 TMPRSS2的蛋白相互作用关系网络
STRING软件对这些蛋白之间发生的生物学关系进行了统计和描述。其中,GO生物过程分析如表8所示,从表中可知,TMPRSS2相关蛋白网络主要参与前列腺细胞增殖、雄激素调控以及RNA转录等生物合成的生物过程中。KEGG通路分析如表9所示,从表中可知,TMPRSS2相关蛋白网络与甲型流感、癌症的途径、前列腺癌、癌症中的转录失调的发生发展有关。

表8 TMPRSS2蛋白互作网络参与的GO生物过程

表9 TMPRSS2蛋白互作网络参与的KEGG通路
2.6 TMPRSS2蛋白的系统进化树构建结果 在NCBI的蛋白库中查找TMPRSS2蛋白,在检索出的所有TMPRSS2蛋白中选择智人、小鼠、黄牛、黑猩猩、琵鹭、家猫、棕熊、鲤鱼、褐头牛鹂、美国鼠兔等20个不同种属的TMPRSS2蛋白氨基酸序列,在MEGA-X软件中进行序列对比后构建系统进化树,结果如图14所示。从序列对比中可以发现,不同物种之间TMPRSS2蛋白氨基酸序列100位之前的序列相似度较差,这与2.3节中做出的分析结果是一致的。

图14 不同物种TMPRSS2蛋白的系统进化树
3 讨 论
本文通过生物信息学手段对SARS-CoV-2侵袭宿主细胞的关键蛋白酶TMPRSS2进行了系统性的预测分析。TMPRSS2蛋白是一种由492个氨基酸组成的亲水性蛋白,它具有一个跨膜螺旋结构,是一种非经典分泌蛋白。TMPRSS2蛋白在前列腺中的表达尤其丰富,其参与的生物过程或通路也多与前列腺癌相关,这些预测结果与之前其在前列腺癌中的研究一致[16]。与此同时,在目前关于SARS-CoV-2的研究与统计中发现,男性患者的患病率和死亡率总体高于女性患者[17],这可能与TMPRSS2在前列腺中的高表达有关。此外,TMPRSS2在人体中的表达可能随着年龄的增长而增加[18],这一结论已在实验鼠身上得到验证[19],这或许可以为老年男性更易患SARS-CoV-2[20]提供一部分解释。翻译后修饰位点的预测中,发现TMPRSS2蛋白共有3个可能的N-糖基化位点,3个置信度较高且均是丝氨酸残基的的O-糖基化位点,以及8个置信度很高的蛋白磷酸化位点(其中5/8是丝氨酸残基),这些预测结果与TMPRSS2的丝氨酸蛋白酶特性完全相符,证明其丝氨酸残基的活跃功能。由于TMPRSS2蛋白在介导S蛋白活化以及促使病毒进入宿主细胞的过程中起到非常重要的作用,因此丰富的翻译后修饰位点将为TMPRSS2蛋白抑制剂的开发提供更多的研究角度。TMPRSS2蛋白共拥有3个超家族保守结构域,分别为LDLa超家族结构域、SRCR_2超家族结构域以及Tryp_SPc超家族结构域。这些保守结构域集中在TMPRSS2蛋白氨基酸序列第100位以后,因此前100位氨基酸可能相对不保守,这一结论在系统进化树的构建过程中被证实,该结论也有助于分析SARS-CoV-2起源于何物种。
在TMPRSS2蛋白互作网络参与的GO生物过程中,RNA聚合酶II转录以及RNA聚合酶II的转录正调控两项生物过程证明,TMPRSS2蛋白及其互作蛋白与RNA的生物合成密切相关。且有研究表明,SARS-CoV-2是一种蛋白包裹的单链正链RNA病毒,因此TMPRSS2蛋白及其互作蛋白对于SARS-CoV-2在宿主细胞内的转录及增殖调控有一定的关系,这也从生物信息学的角度解释了目前研究发现的TMPRSS2蛋白协助SARS-CoV-2复制的可能机制。目前已经有研究者从这一角度着手探索多靶向方案,以阻止与SARS-CoV-2相关的蛋白酶体系的相互作用[21]。另一方面,从TMPRSS2蛋白互作网络参与的KEGG通路中可以看到,与TMPRSS2蛋白网络相关度最高的通路为甲型流感相关通路,研究发现TMPRSS2蛋白参与了甲型流感病毒侵入宿主细胞的过程[22]。新的研究证明,TMPRSS2蛋白互作网络中的TMPRSS4蛋白也参与了流感病毒的激活[23]。在TMPRSS2蛋白互作网络中,TMPRSS4蛋白与TMPRSS2蛋白的作用关系是最为密切的,而且它们共同参与了流感病毒的激活和侵袭,这对跨膜丝氨酸蛋白酶参与其他病毒侵袭宿主的研究具有参考意义。
系统进化树的预测结果显示,人类TMPRSS2蛋白与鼠科动物的TMPRSS2蛋白亲缘关系较近,因此鼠科动物可作为新冠病毒侵入机体时TMPRSS2相关功能研究的模型。此外,鼠科动物属于真兽亚纲啮齿目,这对相关动物为什么容易成为新冠病毒中间宿主的讨论提供了参考。
因为TMPRSS2在全身多组织均有表达[24],且本身不在任何器官中发挥作用[17,25],因此抑制TMPRSS2也不会产生类似于抑制ACE2所导致的副作用[20],所以TMPRSS2成为往后关于冠状病毒靶向治疗的重要候选靶点。目前,尽管多国均研发出了SARS-CoV-2的疫苗,但COVID-19却仍在世界范围内肆虐,并且侵袭性更强的变异类型相继被发现,SARS-CoV-2的超级传播者也不断被报道,世界范围内的疫情形势仍然严峻。本文针对SARS-CoV-2相关蛋白TMPRSS2做出的系统预测及分析,将有助于研究者针对该靶点开发特效药,或为相关通路的抑制研究提供新的思路。
利益冲突:无
