基于多维度特征提取网络的肝脏图像分割
2021-08-19刘蕊续欣莹谢珺
刘蕊,续欣莹,谢珺
(1.太原理工大学 电气与动力工程学院,山西 太原 030024;2.太原理工大学 信息与计算机学院,山西 晋中 030600)
肝癌是世界范围内的主要公共卫生问题,是全球最常见的恶性肿瘤之一[1].肝脏肿瘤最常用的检测手段是计算机断层扫描技术(computed tomography,CT),清晰而准确地描绘肝脏及肿瘤的范围是肝癌诊断过程中至关重要的环节.传统的医学影像处理方法是由放射科医生手动逐张标注病灶区域,工作耗时且人工负担极大[2].随着计算机技术的飞速发展,基于医学图像的计算机辅助诊断技术以其精度高、速度快和成本低的特点在医学图像自动分割的临床应用中得到极大关注.
传统的计算机视觉算法,如阈值分割、区域生长和基于机器学习的方法等,由于受限于先验特征且容易受到噪声影响,并不适合肝脏肿瘤图像分割的临床应用.随着近年来深度学习方法的发展,尤其是卷积神经网络(convolutional neural network,CNN),这种基于数据驱动的生物医学数据检测与分割方法无需手工操作即可实现端到端的病灶结果分析[3],目前已被用于多种医学图像处理,包括肺结节、眼底血管、脑卒中病变和脑肿瘤分割等[4-7].
对于肝脏CT图像,不同病人样本之间肝脏形状和位置略有偏差,同一病例的肝脏形态也会随着切片的不同而发生改变.肝脏与周围组织器官密度相差较小,导致图像的目标对比度较低,边界不清晰.并且CT图像的成像过程引入了大量噪声,这些都是自动分割算法研究中必须解决的关键难点.与此同时,现有条件下难以获取大量的带标注数据,所以从有限的数据中提取充分的特征信息是提升分割准确度的关键,信息提取不充分会造成边缘细节丢失等问题.由于医学CT图像数据的三维特性,数据中包含着单一方向的平面信息和体积方向的空间信息,因此需要采用合适的网络结构,以保证原始数据特征信息的充分获取.
二维网络被广泛应用在医学图像分割中,Ben-Cohen等[8]和Yuan[9]采用了级联网络的思想,将经典的全连接网络进行串联,对肝脏和肿瘤区域进行逐步分割.Tian等[10]在全卷积神经网络(fully convolutional networks,FCN)和CNN的2D肝脏肿瘤分割网络基础上,加入了由长短期记忆模型循环神经网络(long-short term memory,LSTM)和注意力机制组成的语言理解模块,可在分割图像的结果上直接生成诊断报告.Zhou等[11]在UNet结构基础上重新设置了密集跳跃连接方式,可针对不同任务灵活地改变网络深度.Li等[12]将多种经典模块进行组合,同时加强瓶颈处特征的应用,提高了肝脏边界分割效果.但二维网络忽视了体积信息,仅利用单方向上的平面特征不能获得足够高的分割精度.后续研究人员提出了引入体积信息的2.5D网络概念,Han[13]将多张连续切片叠加输入到UNet和Resnet相结合的网络结构中,在避免大幅增加网络复杂度的同时引入了空间信息.Yang等[14]和Yun等[15]采用了多视角分割方法,对3个轴向的2D切片分别进行分割,再使用投票机制将3方向结果进行融合.这种方式虽然引入了一定的空间信息,但是切片的多通道融合会导致特征信息混杂,同时CT数据的各向异性使得不同轴向的切片之间分辨率存在较大差异,为多视角分割带来障碍.三维网络在体积数据利用方面拥有着极大的优势,不仅在单独使用时获得了明显的精度提升,而且能够与二维网络相结合.Li等[16]使用了2D和3D DenseUNet分别提取切片内和切片间特征,之后利用混合特征融合层对2个维度的特征进行融合.Isensee等[17]在肝脏肿瘤分割任务中首先使用3D UNet结构对肿瘤进行粗分割,之后利用2D UNet进行辅助分割,以提升分割精度.三维网络在医学图像的体积信息提取和分割效果上都有明显的优势,可以作为首选方法,但网络复杂度和计算成本之间的平衡依然是制约三维网络灵活应用的问题.
由于医学图像的分割目标往往是尺寸较小的肿瘤或器官,导致医学图像数据只有小部分阳性样本,标签严重不平衡.一些损失函数已经为解决该问题做出一定探索,焦点损失函数[18]通过在交叉熵损失函数的基础上增加调制系数,对分割样本的权重进行了重塑,增加对难分割样本的关注.针对医学图像分割任务,广义Dice损失函数[19]依据标签出现的频率来控制该标签的权重,从而达到平衡各标签训练的效果.指对数Dice损失函数[20]对Dice数值取对数,并加入指数形式,通过标签频率来改善数据的不平衡问题.现有工作的数据不平衡问题多针对二维数据,对三维数据的相关问题研究仍有所欠缺.
为解决上述问题,本文构建了一种多维度特征提取网络RDD-UNet(residual dilated deep supervised-UNet),从体素、轴向和网络层级3个维度上充分提取特征信息,提高了肝脏分割的精确度.
1)本方法采用了改进的三维残差UNet网络结构,加入了长短跳跃连接,融合了上下层网络结构中的多尺度信息,在原始数据体积信息完整保留的情况下充分利用了层内和层间特征.
2)本方法在编码路径中设计了DSC(dilated separable convolution)模块,利用不平衡深度可分离空洞卷积操作,在扩大卷积核感受野的同时,一定程度上缓解了三维网络计算存储空间需求大的问题.
3)本方法设计了混合损失函数,加入改进的Dice损失函数以改善数据不平衡问题,加入SSIM(structural similarity)损失函数以提升肝脏边界处分割效果,同时与深度监督结构相结合,进一步提升肝脏分割精度.
1 RDD-UNet方法
所提网络基于UNet基本结构,加入了残差连接,以长短跳跃连接相结合的方式充分融合层内和层间切片信息;编码路径中设计了不平衡深度可分离空洞卷积模块,在减少网络计算量的同时扩大了感受野,充分提取网络多尺度层间语义信息;同时设计了混合损失函数和深度监督相结合的网络结构,能够在改善小区域数据不平衡问题的同时,提升肝脏边缘分割的精确度.
1.1 RDD-UNet网络结构
本文提出的RDD-UNet网络结构如图1所示,在经典的医学分割UNet[21]基础结构上,将网络维度延伸至三维并增加了网络深度.网络主要包含编码路径、解码路径和深度监督3部分.编码路径由4个编码模块和4个降采样模块组成,编码模块由卷积层、PReLU非线性层和dropout层组成,降采样模块使用步长为2的卷积层代替了一般的池化操作.其中第4个编码模块使用的是不平衡深度可分离空洞卷积模块(DSC模块),在降低三维网络运算量的同时提升了网络特征提取的效率,该模块在1.2节中详细介绍.解码路径和编码路径的结构对称,由4个解码模块和4个上采样模块组成.网络主干通路中加入了UNet长连接和残差短连接[22],将网络层级间不同尺度的语义信息进行融合.输出部分加入了深度监督结构,不同尺度的特征图经过反卷积层、相应倍数的上采样层和Sigmoid层恢复成原始数据尺寸,并结合混合损失函数,得到最终的分割结果,混合损失函数和深度监督的结合使用在1.3节中具体介绍.
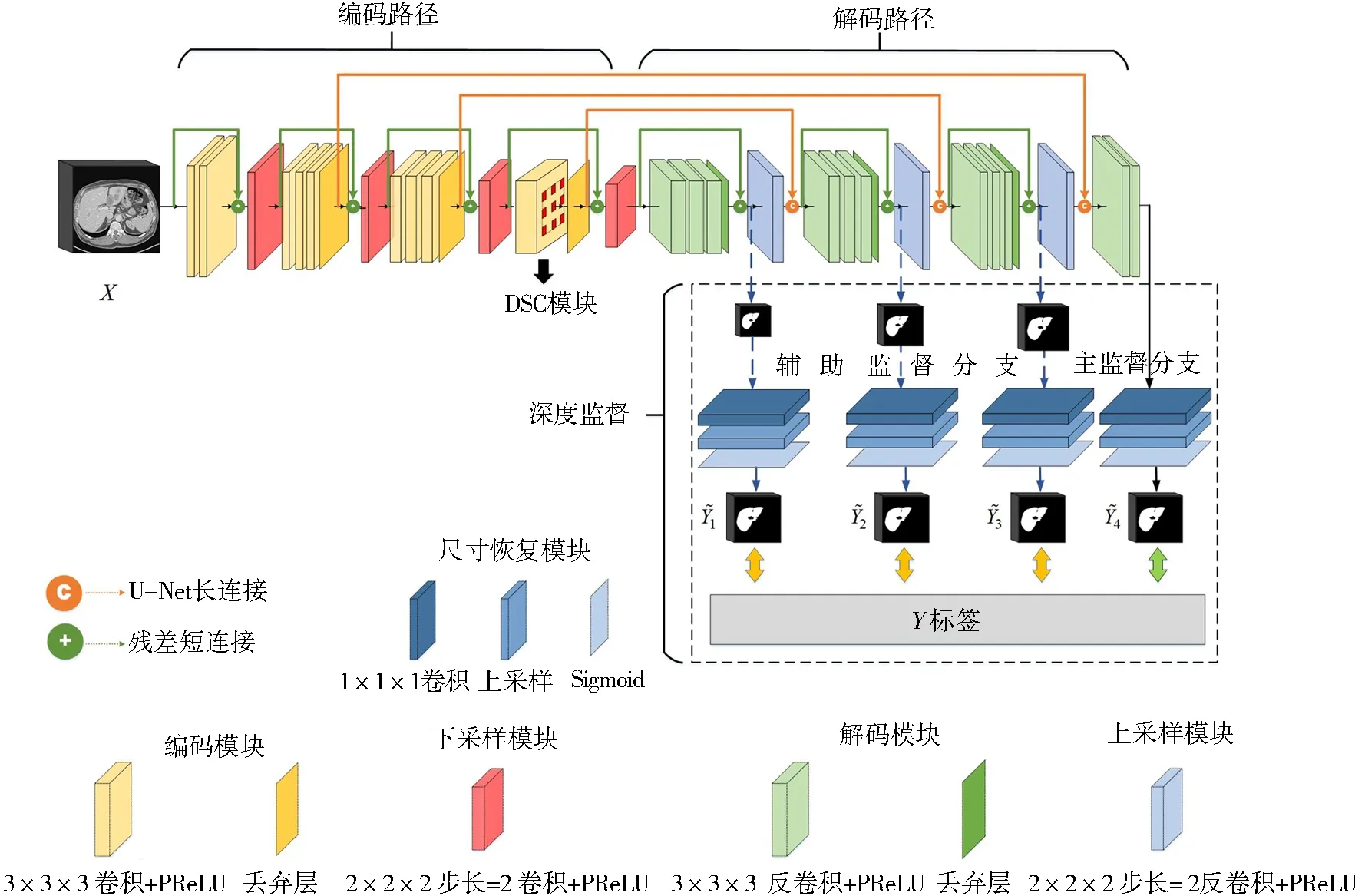
图1 RDD-UNet网络结构Fig.1 Structure of RDD-UNet
该模型的输入X∈h×w×k是长宽为h×w(h=w=512),厚度为k的三维数据,经过预处理和重采样步骤将尺寸缩放到256×256×k(本实验中k=48),经过网络的运算后,深度监督端产生4个输出h×w×k,分别对应不同分辨率的分割结果,和作为网络的辅助监督中间结果,与混合损失函数相结合,共同调节网络的训练过程,得到最终的分割结果该网络可以充分利用CT数据各维度的信息,同时加入的残差连接有助于改善网络深层的梯度指数衰减,促进信息传播,使模型训练更加充分.
1.2 DSC卷积模块
本方法中针对三维网络计算存储空间需求大的问题,在网络中设计了DSC卷积模块,在不平衡多分支结构基础上组合使用深度可分离和空洞卷积,具体描述如图2所示:

图2 DSC模块结构Fig.2 Structure of DSC block
该模块包含3条分支,分别设置了不同的空洞卷积率,大感受野的卷积分支可以更多地提取大目标的抽象特征,而小感受野的卷积分支则更针对小目标的细节信息,各个分支信息最终融合,从体素维度上对输入图像进行多尺度特征信息的提取.图2中,框内包含的2层结构是深度可分离卷积操作,输入特征图Fi∈H×W×D×Ci,其中长宽为H×W,深度为D,输入和输出通道数为Ci和C0.原始卷积操作经过k维卷积核所需参数为k×k×k×Ci×C0,现经过深度分离和逐点卷积分解,参数量为k×k×k×1×Ci+1×1×1×C0×Ci.在本实验中,k=3且C0=128时参数量仅为原始的12%.
按照空洞卷积的原理,如果网络中连续的卷积层使用相同的空洞率,会产生网格效应,即高层特征图中一个像素内容是由底层图像中分散的像素点组成,原理示意如图3所示,这样会造成图像位置信息的丢失.为避免此问题,本方法采用混合空洞卷积的原则[23],DSC模块中2条空洞卷积分支的空洞率分别设置为[2,3]和[2,3,5],保证感受野可以覆盖完整的图像信息,不存在孔洞或者边缘信息丢失.

图3 空洞卷积网格效应Fig.3 Illustration of gridding problem
1.3 混合损失函数和深度监督结构
损失函数是深度学习模型中的重要部分,本方法设计了一种混合损失函数,主要解决肝脏分割的数据不平衡问题,同时提高了肝脏边缘的分割精度.该混合损失函数由以下3部分组成:
1)改进的Dice损失函数(FEL-Dice).Dice系数是医学图像分割中重要的衡量标准,它通过描述预测结果和真实标签之间的重叠区域大小来计算分割精确度.由Dice系数转变成的Dice损失函数[24]已经被广泛应用于医学图像分割任务中,但对于小尺寸目标的分割问题,少量的错误分割点就会导致损失值的大幅上升,影响最终的分割结果.本方法提出了一种改进的Dice损失函数
(1)
其中,N是总体素数量;pi和gi是在i点的预测值和标签值;常量ε用于防止函数分母为0.相比原始Dice函数,对数函数形式增加了损失的非线性程度,参数γ用于改变损失函数的斜率,这样的改进保证了数据中易分割点的损失函数没有大的变化,而难分割点损失值大幅增加,从而增加了网络对难分割点的关注度.在此实验中,由经验取值γ=0.3.
2)二元交叉熵损失函数(binary cross entropy,BCE).这一函数可针对每个体素点计算差值,已被广泛应用在分割网络中.
(2)
3)损失函数借鉴了结构相似度(SSIM),该相似度是图像质量的评判标准之一,通过对比图像之间的亮度、对比度等信息,对图像相似度进行评判,值越大代表两者结构相似性越高.将SSIM作为损失函数引入图像分割网络,可针对肝脏边缘的分割效果进行提升.其损失函数为
(3)
其中,μp和μg表示图像中预测值p和真实值q的平均值;σp和σg表示标准差;σpg表示两者的协方差;常量C1和C2用于防止函数分母为0,根据文献[25]的数据设置为C1=0.012,C2=0.032.
混合损失函数为
Lmul=LFEL-Dice+LBCE+LSSIM.
(4)
为了充分学习各尺度特征信息,本方法进一步将混合损失函数与深度监督结构相结合,利用网络低层和中层特征信息辅助网络训练,加快训练过程的收敛速度,提高最终的分割精度.图1虚线框中圈出的区域为深度监督结构,在解码路径中,网络的最终输出作为主监督分支(黑色箭头表示),在3个解码模块后设置辅助监督分支(蓝色虚线箭头表示).4个侧向输出产生的不同分辨率特征图被送入反卷积层、相应倍数的上采样层和Sigmoid层,恢复成原始数据尺寸,利用混合损失函数与标签进行对比,产生的误差作为深度监督信号,提升网络的训练效果.
网络的主监督分支使用混合损失函数
(5)
3条辅助监督的损失函数均分别使用FEL-Dice损失函数
(6)
网络的总体损失函数由上述两部分共同组成
(7)
其中,超参数α是调节监督信息间比例关系的平衡系数,用于调整辅助监督分支在网络训练过程中的作用强度.α初始取值为0.3,之后随迭代次数的增多而逐渐减小,每50次迭代后进行一次衰减计算,最终趋近于0.具体衰减计算公式为
(8)
其中,M为总迭代次数,t为每50次的迭代次数.
2 实验数据集与评价标准
2.1 实验数据集
本实验使用的数据集是2017 LiTS肝脏肿瘤分割挑战赛的公开数据集,包含131个腹部增强三维CT扫描.由于图像数据通过不同的仪器和来源收集,因此样本间的分辨率和图像质量方面有差异.为了节省时间,在实验中原始CT数据在解剖切面被重采样为256×256尺寸.在预处理过程中,所有扫描的图像经过窗口化操作,CT值控制在[-200,200],以忽略其他器官的干扰,之后进行了直方图均衡化步骤,达到增强图像对比度的效果.
2.2 评价标准
本实验采用的评价标准有Dice系数、体积重叠误差(VOE)、相对体积误差(RVD)、平均对称表面距离(ASD)和最大对称表面距离(MSD)等,计算公式为
(9)
(10)
(11)
(12)
(13)
其中,A和B分别表示预测分割区域和标签区域,S(A)代表A的曲面体素集.
全式(14)中‖·‖代表欧氏距离,d(v,S(A))代表任意体素v到S(A)的最短距离,计算公式为
(14)
3 实验与分析
3.1 实验环境
本实验模型搭建环境为Python3.7,Pytorch框架,ubuntu18.04操作系统,采用NVIDIA TiTanXP 12G内存GPU.网络Dropout率设置为0.3,初始学习率为0.000 1,使用Adam算法进行梯度更新.
3.2 实验结果
3.2.1 混合损失函数与深度监督实验效果
本实验的目的是证明方法所提出的混合损失函数和深度监督网络结构,能够在不平衡数据和目标边界区域表现出极大的效果改善.实验结果如表1和图4所示.从图4a-f中可以看出,混合损失函数结合深度监督结构的效果远高于单一损失函数的结果.从图4c-e中的对比可以看出,混合损失函数能够保留更多的细节信息,对肝脏边缘细节的分割更精确.
此外,深度监督结构是从网络隐含层中集成语义信息的重要组成结构.表1的结果显示,相同损失函数条件下,加入了深度监督的网络效果有明显提升.同时通过对图4c和图4f的比较可以看出,由于深度监督的隐含层特征提取作用,非连通区域的分割效果有了明显改善.

表1 不同损失函数和深度监督结果比较Tab.1 Segmentation results with different loss and deep supervision
3.2.2 空洞卷积与残差连接实验效果
本实验的目的是验证空洞卷积和短残差连接作为重要的多尺度信息采集方法,在RDD-UNet中得到了明显的效果.表2和图5a-e显示了RDD-UNet的消融实验情况.从表2中可以看出RDD-UNet的Dice系数增加约3%,与图5d相比,图5c的结果有明显的优势,这说明空洞卷积对于非连通目标可以准确提取样本点的位置信息.同时,图5e中的大面积肝脏被错误地分割为背景,非连通区域中间边界被错误分割为前景,这些结果说明了加入残差连接可以有效地增加信息融合效果,提升分割精度.

a.原始图像;b.肝脏标签;c-e.分别为RDD-UNet、无空洞卷积、无残差连接模型下的分割结果.图5 不同网络结构的肝脏分割结果示例Fig.5 Examples of liver segmentation result with different network architecture
图6是在测试数据上Dice和VOE指标数值范围的箱型图.从图6结果可以看出,RDD-UNet不仅提高了整体的分割精度,而且对每个样本都有一定程度的提升.
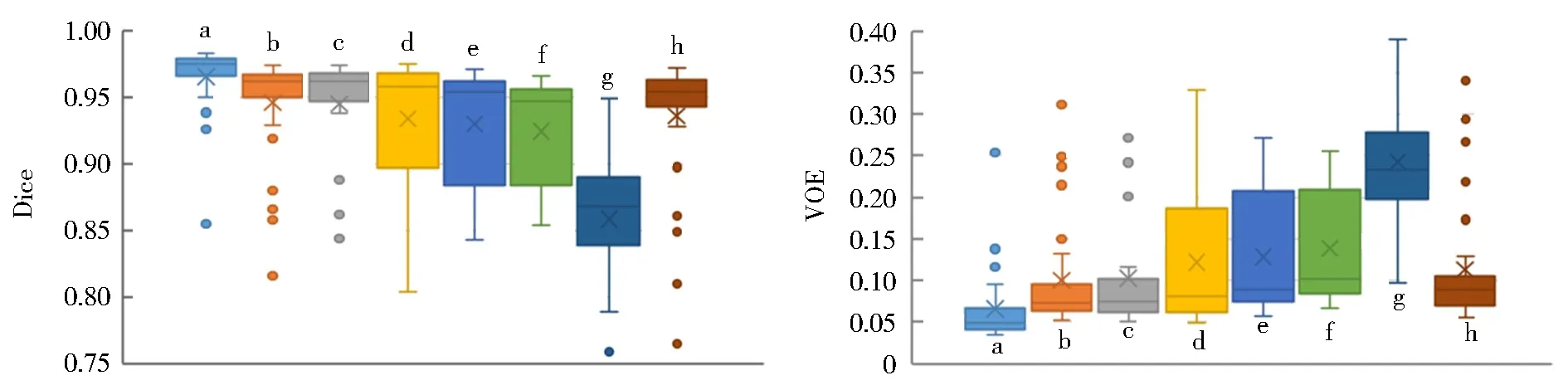
a.RDD-UNet的结果;b-h.分别为FEL-Dice+深度监督、FEL-Dice+无深度监督、BCE+深度监督、BCE+无深度监督、混合损失函数+无深度监督、RDD-UNet+无空洞卷积和RDD-UNet+无残差连接条件下的结果.图6 测试结果箱形图Fig.6 Box plots evaluated on test data
3.2.3 其他方法对比
本方法与Yuan[9]和Tian[10]等学者的方法及H-DenseUNet[16]、UNet++[11]、nnU-Net[17]和Bottleneck[12]等几种方法进行了定量比较,结果如表3所示.其中前2行是2017年MICCIA肝肿瘤分割比赛的冠亚军结果.从表3结果可以看到,RDD-UNet在Dice系数和Dice Global数值均位居榜单高位,VOE得到了当前较好的结果.
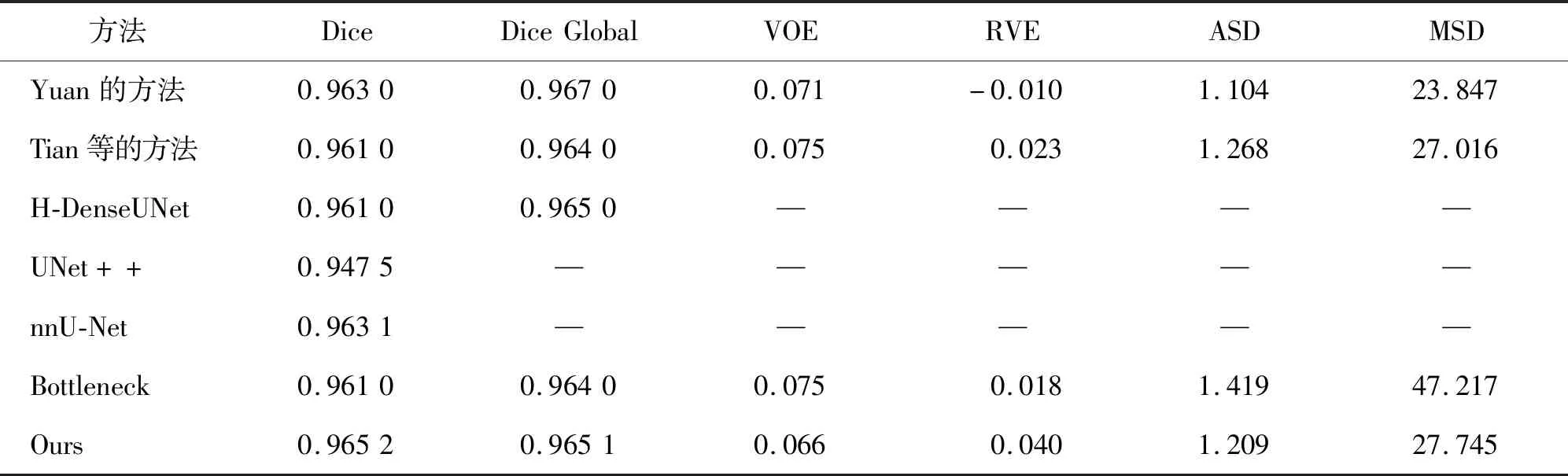
表3 对比实验结果Tab.3 Comparative experimental results
4 结束语
本方法提出了RDD-UNet网络模型,在数据轴向层级采用参数缩减的3D网络提取多方向的有效体积信息,在体素层级运用空洞卷积增大每层的有效特征感知区域,在网络层级运用多种跳跃连接相结合的方式融合多尺度上下文语义信息,与此同时运用混合损失函数和深度监督相配合的结构,提升了肝脏器官边缘的分割精度.本方法在公共数据集LiTS上的Dice分数达到0.965 2,与其他方法相比达到了较高的精度水平.下一步将针对3D网络计算量大的缺点,继续对网络结构进行优化,并加入后处理方法,以实现分割速度和精度的双重提高.
