植物中长链非编码RNA研究进展综述
2021-08-02刘琳营苏晓俊闵玲
刘琳营 苏晓俊 闵玲

摘要:长链非编码RNA(long non-coding RNA,简称lncRNA),通常定义为长度超过200 nt的非编码RNA,是几乎不具有编码能力或具有弱编码能力的RNA转录本。早期的研究质疑lncRNA的重要性,因为与mRNA相比,lncRNA具有低表达和低序列保守性的特点,认为它是RNA聚合酶Ⅱ转录的副产物,故将其存在归因于转录噪声。近年来,随着对非编码RNA的深入研究,lncRNA独特的生物学特性和功能被陆续鉴定,多数研究学者认为lncRNA是一种在生物体中普遍存在且参与基因调控的重要组分。本文主要对植物中发现的lncRNA的种类、发挥功能的分子机制、参与的生物学过程及研究策略等进行综述,为在植物领域进一步研究lncRNA提供依据和参考。
关键词:长链非编码RNA;植物;功能;分子机制;研究策略
中图分类号: S184文献标志码: A
文章编号:1002-1302(2021)12-0012-08
收稿日期:2020-11-07
基金项目:国家重点研发计划(编号:2016YFD0101402)。
作者简介:刘琳营(1994—),女,山东济宁人,硕士研究生,研究方向为lncRNA与棉花花药发育及高温响应。E-mail:linyingliu_257@webmail.hazu.edu.cn。
通信作者:闵 玲,博士,副教授,主要从事棉花生殖发育、环境与生殖发育互作研究。E-mail:lingmin@mail.hazu.cn。
近年来,生物技术的快速发展使研究者证实真核生物的基因组中并非全部序列都可以编码蛋白质,其中大多数是非编码序列 [1]。在以往对于非编码RNA的研究中,备受重视的是一系列短链非编码RNA,如siRNA、miRNA、snRNA等,自1991年科学家证实Xist参与X染色体失活的调控后[2],不断有研究表明,长链非编码RNA(long non-coding RNA,简称lncRNA)在调控真核生物的生命活动中具有重要的作用。
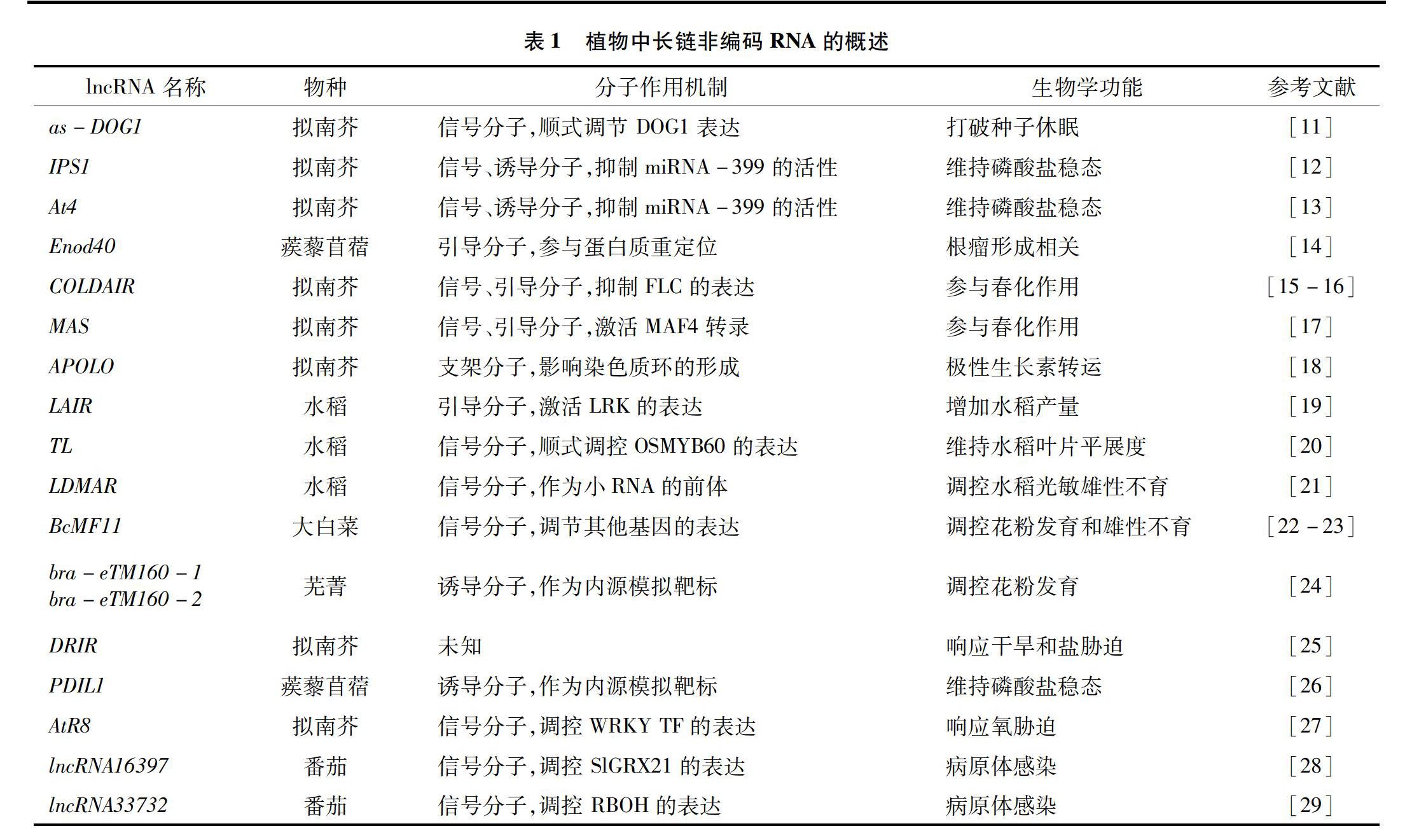
大多数的lncRNA与mRNA一样,能够被RNA Pol Ⅱ转录,具有5′端帽子、3′端poly(A)尾以及选择性剪接位点等结构特点[3-4],一些lncRNA可被RNA Pol Ⅲ转录,植物中也有少数lncRNA是由植物特異性RNA Pol Ⅳ和Ⅴ转录产生的[5-7]。一般来说,真核生物编码蛋白质的序列相对保守,同源性很高,而lncRNA是一类在不同物种中具有低序列保守性的转录本,由此推测,个体和物种间存在差异的原因可能是由于非编码序列差异导致[8-9]。随着高通量转录组测序和表达谱分析技术的快速发展,越来越多的研究表明lncRNA在真核生物的生长发育过程中扮演着十分重要的角色[10]。相比于动物中lncRNA的研究,植物lncRNA的探索仍处于初级阶段,研究主要集中在拟南芥、蒺藜苜蓿、番茄和水稻等植物中(表1)。本文主要对植物lncRNA的分类、分子机制研究进行综述,总结了lncRNA参与植物生长发育和逆境胁迫中的重要调控机制,并对植物中lncRNA的研究策略进行概述,为深入研究植物中lncRNA的功能和分子机制提供科学依据。
1 lncRNA的来源和种类
1.1 lncRNA的来源
在真核生物中,编码蛋白质的基因几乎是通过部分或整体复制以及随后的序列分化而产生的,由于大多数非编码RNA受到较低程度的进化限制,所以只有很少一部分在不同物种之间表现出序列保守性,因此研究者推测lncRNA可能来自以下几种途径:(1)阅读框插入蛋白质编码基因的内含子之间,插入的阅读框与先前的编码序列重新整合形成功能性lncRNA;(2)染色体重排,将2个分开的非转录区连接在一起而产生有多外显子的lncRNA;(3)非编码基因进行逆转座复制形成功能性逆基因或非功能性假基因;(4)局部重复序列串联产生相邻重复的lncRNA;(5)转座因子插入产生功能性lncRNA[11]。
1.2 lncRNA的种类
1.2.1 基于所在基因组位置进行分类
根据lncRNA在基因组中与蛋白编码基因的相对位置,可将其分为正义lncRNA(sense long non-coding RNA)、反义lncRNA(antisense long non-coding RNA)、双向lncRNA(bidirectional long non-coding RNA)、基因内lncRNA(intronic long non-coding RNA)、基因间lncRNA(intergenic long non-coding RNA)5类,其中基因间lncRNA也被称为大型介入性非编码RNA,即lincRNA(large intervening noncoding RNA)[30-31]。
1.2.2 基于分子机制进行分类
大多数lncRNA具有功能,前期研究者发现lncRNA可按分子功能进行以下分类[32-33]。
信号分子:lncRNA可作为信号分子调控邻近基因表达。例如在雌性动物发育过程中,Xist诱导X染色体的失活,该lncRNA在一条X染色体上表达会导致整个染色体沉默[34]。而在植物发育调控中,DOG1(delay of germination 1)基因调控拟南芥种子休眠,as-DOG1(antisense delay of germination 1) lncRNA会抑制DOG1基因的表达,打破种子休眠[11]。
诱饵分子:lncRNA可以作为诱饵,通过招募RNA结合蛋白(RNA binging protein,简称RBP),例如:转录因子、染色质修饰物或者其他调控因子间接调节蛋白质编码基因的表达[32]。此外,lncRNA作为诱饵可以结合miRNAs,阻断miRNAs与其特异性的靶基因互作,这个过程被称为内源模拟靶标(endogenous target mimicry,简称eTM)[33]。在拟南芥中,miR399及其靶基因PHO2(PHOSPHATE 2,编码泛素结合酶相关蛋白的基因)在维持磷酸盐稳态中发挥作用[35]。lncRNA IPS1(INDUCED BY PHOSPHATE STARVATION1)和At4都会竞争性地与miR399结合,导致PHO2上调表达 [12-13,24]。
引导分子:lncRNA可以引导RNA结合蛋白复合体定位到特定位置或招募染色质修饰酶到靶基因,顺式(cis)或反式(trans)作用于靶基因[32]。例如,在蒺藜苜蓿中发现lncRNA Enod40可直接与根瘤中的MtRBP1(medicago truncatula RNA binding protein 1)蛋白结合,将MtRBP1从植物细胞的核斑点重新定位到细胞质颗粒中发挥重要的作用[14]。在拟南芥中发现lncRNA COLDAIR与植物PRC2(polycomb repressive complex 2)蛋白复合体中的亚基CLF(CURLY LEAF)特异结合,指导PRC2复合体到达FLC(FLOWERING LOCUS C)位点,PRC2通过组蛋白H3K27甲基化改变FLC位点的染色质结构,从而抑制FLC的表达,调控开花时间[15-16],这一过程中COLDAIR作为引导分子进行反式调节。最近研究发现lncRNA MAS可以与COMPASS-like复合物中的WDR5a(WD40-REPEAT 5a)结合并将其引导到MAF4(MADS AFFECTING FLOWERING4)位点,从而促进H3K4的甲基转移酶活性并激活MAF4的表达,进而微调开花时间[17]。
支架分子:lncRNA可以作为支架分子行使分子功能。以往有研究认为很多复合物都是用蛋白质作为支架[36]。但近年来研究表明,lncRNA有多种不同的结合功能域,可以与不同的蛋白复合体或效应分子结合,这对复杂的生物信息传递、分子间的相互作用以及对信号本身特异性的精准调控非常重要[37-38]。植物中特有的Pol V可以产生siRNA和相关蛋白,识别其靶基因所需的支架转录本,通过RNA介导的DNA甲基化(RNA directed DNA methylation,简称RdDM)途经进行染色质修饰[39]。RdDM途经主要依赖2种核心蛋白DICER-LIKE 3(DCL3)和ARGONAUTE 4(AGO4),前者切割长双链RNA产生小干扰RNAs(small interfering RNAs,简称siRNAs),这些siRNAs与AGO蛋白结合,形成AGO-siRNAs复合物,Pol V产生的lncRNA作为支架将AGO-siRNAs复合物转运到染色质的目标位点[40-42]。例如,lincRNA APOLO(AUXIN REGULATED PROMOTER LOOP RNA)作为支架RNA参与染色质环的形成[18]。
lncRNA除以上4种分子机制,还是小RNA(siRNA和miRNA)生物合成的前体,如植物中特有的Pol Ⅳ产生的lncRNA,可以由DCL蛋白切割产生siRNA[43];也可能是miRNA的靶标,如miRNA可通过靶向切割lncRNA生成phasiRNA的方式参与植物对长日照胁迫的响应[44]。由于lncRNA在植物中的研究尚处于早期阶段,导致植物中缺乏系统和一致的lncRNA调控机制,随着研究的不断深入,对lncRNA的分子功能划分将更加完善。
2 lncRNA在植物中参与的生物学过程
2.1 lncRNA参与植物生长发育
对lncRNA参与植物生长发育的研究主要集中在生长素运输和信号转导方面。lncRNA ENOD40和lnc351被称为ASCO-RNA(alternative splicing competitor RNA),可以与核斑RNA结合蛋白(nuclear speckle RNA-binding proteins,简称NSRs)相互作用,进而调节基因选择性剪切。在拟南芥中ASCO-RNA能与mRNA竞争性结合NSRs,干扰下游生长素应答基因的选择性剪接,进而影响侧根的生長[45]。此外,ENOD40能参与调节细菌或真菌与豆科植物的共生关系,在共生相互作用中,根瘤菌迅速诱导根瘤菌原基中ENOD40的表达。虽然潜在的分子机制尚不清楚,但ENOD40可以在运输非共生植物细胞生长所必需的代谢物方面发挥作用[46-47]。最近研究表明,生长素调控启动子环APOLO(auxin egulated promoter loop)可以动态调节下游基因PID(PINOID)启动子的环化过程,从而影响PID启动子的活性。PID是植物生长素极性转运的重要调节因子,生长素激活RDD(ROS1、DML2、DML3)介导的DNA去甲基化,促进覆盖PID启动子区域的环打开[18,48]。HID1(HIDDEN TREASURE 1)是长度为236 nt的lncRNA,在拟南芥中,HID1受持续红光调控,抑制了光敏色素互作因子PIF3(phytochrome-interacting factor 3)的转录,促进了幼苗的光形态发生[49]。在水稻中,LAIR(LRK antisense inetrgenic RNA)是由邻近基因LRK(leucine-rich repeat receptor kinase)的反义链转录而来,LAIR可以结合染色质修饰蛋白OsMOF和OsWDR5,并共同定位于LRK1基因组位点,从而激活LRK1转录,提高水稻产量[19]。此外,在水稻中发现的另一个lncRNA TL(TWISTED LEAF)可以通过调控R2R3-MYB基因的表达,维持水稻叶片的平展度[20]。
2.2 lncRNA参与植物的开花调控及生殖发育
有大量研究报道,在植物生殖发育过程中,lncRNA也起到了非常重要的调控作用。在拟南芥中发现lncRNA COLDAIR可以调控FLC(FLOWERING LOCUS)染色质区域的组蛋白甲基化,COLDAIR由FLC的第1个内含子转录而来,春化期间可以与多梳组蛋白复合体PRC2(Polycomb Repressive Complex 2)中的CLF(CURLY LEAF)直接相互作用,指导PRC2复合体到FLC基因位点,PRC2具有H3K27甲基转移酶的作用,通过使H3K27me3富集来抑制FLC基因表达,促进拟南芥正常开花[15,50-51]。近年来,有研究者在拟南芥中发现了一个天然反义转录长链非编码RNA(natural antisense transcript-lncRNAs,简称NAT-lncRNAs),命名为MAS(one NAT-lncRNA,NAT-lncRNA_2962),可结合组蛋白甲基转移酶的关键组分WDR5a(WD40 containing repeat 5a),将其招募到MAF4(MADS AFFECTING FLOWERING4)的基因位点,增强H3K4me3并激活MAF4的表达[17],从而调控拟南芥的开花时间。
除了调节植物开花,lncRNA也会影响花粉发育过程。与水稻光敏雄性不育相关的lncRNA被称为长日照特异性雄性不育相关RNA(long-day-specific male-fertility-associated RNA,简称LDMAR),在长日照环境下,花粉正常发育需要LDMAR的表达,而突变产生的单核苷酸多态性(SNP)改变了LDMAR 的二级结构,导致LDMAR的启动子区域中甲基化作用增强,降低了LDMAR的转录,导致发育中的花粉过早发生细胞程序性死亡,这是造成水稻光敏不育系败育的重要原因[21]。在大白菜中,研究人员基于花粉特异性克隆了一个全长828 bp的cDNA,命名为BcMF11(Brassica campestris male fertility gene 11),是一种全新的非编码RNA,其没有明显的开放阅读框或编码能力,当BcMF11的表达降低时会产生花药绒毡层降解推迟、小孢子分离不同步、花粉粒发育中断等异常现象[22-23],最终导致花粉粒不正常发育,出现败育表型。
随着高通量转录组测序的快速发展,在很多物种中鉴定出与生殖发育有关的lncRNA。有研究表明,在芜菁的花粉发育过程中,lncRNA可以作为内源模拟靶标调节miRNA的功能,其中bra-eTM160-1和 bra-eTM160-2这2个lncRNAs预测是bra-miR160-5p的eTM,研究证实转基因植株的花器官形状虽然正常,但花药中有一半的花粉粒具有小且皱缩、无活力、没有细胞核或花粉内壁缺失的败育表型[24]。在水稻中,同源四倍体水稻在遗传进化方面比二倍体水稻具有更大的优势,但常伴有育性低及种子结实不良等特点,研究人员利用高通量测序发现,与转座因子和减数分裂调控靶点相关的lncRNA可能是花粉和胚囊发育的内源调控因子,其差异表达导致同源四倍体水稻的育性降低[52]。在棉花中,研究人员对棉花的RNA-seq数据分析表明,在花药组织中优势表达的lncRNA较其他组织多,共鉴定到3 925个在花药中高表达的lincRNAs[53]。为了进一步研究lncRNA在棉花花药中的作用,Zhang等对三系杂交棉(胞质不育系、保持系、恢复系)的花药发育过程进行了转录组测序,共鉴定到80 695个参与胞质雄性不育和生育力恢复的候选lncRNAs,随后分别对不育系-保持系和不育系-恢复系差异表达的lncRNA进行GO分析,分析结果显示不育系-保持系在细胞激素代谢过程和氧化还原反应过程中存在明显差异,而不育系-恢复系的差异主要在于棉花育性恢复过程中调控细胞形态形成的功能基因[54]。研究人员对不育系-保持系的花粉母细胞时期、四分体时期和小孢子时期的差异lncRNAs和mRNAs分析时发现,这些差异lncRNAs和mRNAs可能参与了绒毡层细胞降解、小孢子发育、花粉发育以及棉花花药细胞壁的分化、增殖和凋亡等生殖发育过程,进一步的GO和KEGG分析显示它们分别参与植物氧化磷酸化、类黄酮生物合成、戊糖和葡萄糖酸相互转化、脂肪酸生物合成和MAPK信号通路,由于代谢途经异常导致花药败育,最终造成雄性不育[55]。
2.3 lncRNA参与植物的逆境胁迫响应
植物在生长过程中往往会经历高温、干旱、盐碱等非生物胁迫及细菌、真菌、病毒等生物胁迫。由于植物的固着生长,只能通过调节基因表达及代谢物的改变来应对不断变化的环境条件,其中lncRNA在植物响应非生物胁迫中发挥重要的角色。lncRNA DRIR(drought induced RNA)在干旱、盐胁迫和脱落酸处理后被激活,调控植物中ABA信号转导、水运输等响应非生物胁迫相关基因的表达[25]。此外,Li等对水稻进行全转录组链特异性测序,分析发现lncRNA可以参与短期干旱“记忆”反应[56]。近年来,有研究对耐旱甘蓝型油菜及敏旱甘蓝型油菜在干旱和复水条件下进行转录组分析,构建了lncRNA-mRNA共表达网络,进而筛选出了在干旱中参与植物激素信号转导的lncRNA,并且验证了这些lncRNA在油菜中可以正调控靶基因的表达,从而对干旱胁迫作出响应[57]。在低磷胁迫下,拟南芥中的lncRNA IPS1(induced by phosphate starvation1)和玉米中的lncRNA PILNCR1(Pi-deficiency-induced long-noncoding RNA1)都可以模拟miR399的靶标,竞争性地与miR399结合,从而增加miR399靶标基因PHO2的表达,维持植株在低磷胁迫下的正常生长[12,58]。在蒺藜苜蓿中,lncRNA PDIL1(Phosphate Deficiency-Induced lncRNA 1)作为miR399的模拟靶标竞争性地抑制MtPHO2转录本,MtPHO2通过泛素化途经参与Pi转运蛋白的降解[26],表明PDIL1通过间接控制MtPHO2对Pi的转运来保持植物体中相对稳定的磷浓度。
此外,lncRNA在植物响应生物胁迫的过程中也具有關键调控作用。在拟南芥中,RNA Pol Ⅲ转录的lncRNA AtR8在拟南芥根中特异性表达,对野生型和atr8突变体的微阵列分析表明AtR8与防御反应存在较强的关联,在拟南芥处于发育早期、低水杨酸条件或感染紫丁香假单胞菌时,AtR8的积累与2个WRKY基因(WRKY53/WRKY70)的积累呈负相关,由NPR1(nonexpressor of pathogenesis-related gene 1)协同调控植物的防御反应及根系伸长[27,59]。在番茄中,lncRNA33732沉默会导致RBOH基因的表达降低,减少H2O2的含量,导致番茄对番茄疫霉根腐病的敏感性增强,而番茄lncRNA16397可激活SlGRX表达,降低细胞内ROS含量积累,使细胞膜不易损伤,从而增强番茄对致病疫霉的抗性[28-29]。同时,番茄黄化曲叶病毒(tomato yellow leaf curl virus,简称TYLCV)也是番茄作物的毁灭性病害,对番茄的生产具有重要威胁,最新的研究发现易感番茄的lncRNA SILNR1可与TYLCV的小干扰RNA (small-interfering RNAs,简称siRNAs)互作,为TYLCV诱导疾病及宿主抗病毒免疫提供了合理的模型[60]。在棉花中,lncRNAs Ghlnc NAT-ANX 2(L2)和Ghlnc NAT-RLP 7(L3)在棉花对黄萎病菌和灰霉病菌的抗性中发挥重要作用,研究发现对L2和L3进行沉默后会上调其邻近蛋白质编码基因的表达,从而增强棉花的抗病性[61]。
3 植物中lncRNA的研究策略
在真核基因组中非编码序列虽没有蛋白质编码能力,但在调控个体的生命过程中扮演着重要的角色,目前对非编码RNA研究占主导地位的是长度小于200 nt的小RNA(如microRNAs、siRNA、与Piwi蛋白互作的piRNA),其分类和功能机制研究的较为透彻[62]。由于lncRNA在不同物种间具有保守性差、易降解、表达丰度低等特点[5],早期没有引起研究者的重视,随着现代生物测序技术的不断发展,更多的lncRNA被鉴定出来,成为科研领域的热点。
目前对lncRNA的研究一般集中在动物和医学领域,研究的基本策略主要分为:(1)lncRNA的筛选与鉴定。隨着链特异性建库的广泛应用,lncRNA预测算法的不断发展,在拟南芥[17,63]、桑树[64]、甘蓝[65]和棉花[54-55]等植物中筛选和鉴定了大量潜在的lncRNAs。近年来,以单分子测序为特点的第三代测序技术的快速发展,可实现RNA的直接测序以及RNA上修饰的检测[66],从拟南芥[67]、水稻[68]、澳洲棉[69]等物种中鉴定了大量lncRNAs。(2)lncRNA的表达和定位。通过高通量测序筛选的lncRNA需要运用成熟的技术验证其表达水平及定位,包括Northern印记、实时荧光定量PCR(qRT-PCR)、RNA荧光原位杂交(fluorescence in situ hybridization,简称FISH)和瞬时表达等。(3)lncRNA的功能和机制研究。通过高效稳定的植物遗传转化系统可以进一步揭示候选lncRNA的生物学功能。基本策略主要包含功能获得性研究和功能缺失性研究。功能获得性研究主要是通过导入过表达载体实现,功能缺失性研究主要采用CRISPR/Cas9基因敲除和RNAi干涉等方法进行研究。在水稻中,通过对病原体敏感的lncRNA转录组分析发现,过表达ALEX1可以激活茉莉酸途经和对白叶枯病的抗性[70];在番茄中,利用CRISPR/Cas9编辑技术获得lncRNA1459缺失突变体,其果实表现出明显的成熟延迟[71];在番茄中超表达和干涉lncRNA33732可以影响番茄对致病疫霉的抗性[29]。此外,lncRNA与蛋白质的互作研究常用RNA-pull down技术、RNA结合蛋白免疫沉淀(RNA binding protein immunoprecipitation,简称RIP)、RNA纯化的染色质分离(chromatin isolation by RNA purification,简称ChIRP)、目标RNA的捕捉杂交分析(capture hybridization analysis of RNA targets,简称CHART)和交联免疫沉淀(crosslinking-immunopurification,简称CLIP)。因此依据前人研究结果,总结出在植物中可以实现的lncRNA的研究策略(图1)。
4 展望
随着高通量测序技术与生物信息学的飞速发展,越来越多的lncRNA被发现,研究显示,lncRNA并不是之前所认识的“垃圾序列”,而是可以在各种层面上调控基因的表达,如表观遗传[15,17,72]、转录调控[73-74]及转录后调控[12,43,75]。虽然目前对lncRNA的研究取得了一些成果,但研究者所面临的问题仍然很多,如:许多lncRNA的生物学功能未得到阐明;判断非编码转录物是否有功能依然困难;lncRNA作用机制复杂多样,不同的lncRNA研究结果之间的借鉴意义不高。面对这些问题,可以从新的技术策略和思路入手:(1)纳米孔测序技术可以直接对RNA进行测序,并可扩展到lncRNA的全长绘制[67]。(2)单细胞的转录和表观基因组的同时分析可以建立lncRNA与染色质之间的功能联系[76]。(3)对已获得的差异lncRNAs实行靶基因预测、与差异mRNA的共表达分析及转录因子预测。(4)CRISPR/Cas9基因编辑技术可应用在多种植物基因工程中,虽然目前CRISPR基因编辑技术
发展较快,且编辑精度日益提高,为研究重要的植物生长和发育特征提供了有力的支持,但对lncRNA的编辑则需要更特异的sgRNA靶标来确保敲除全长。随着研究技术的逐渐成熟,研究领域的逐渐深入,相信对植物中lncRNA功能的挖掘和调控机制的了解会更加透彻,对探索其在植物生命活动及发育过程中的作用具有十分重要的意义。
参考文献:
[1]张玉婵,陈月琴.长链非编码RNA在植物生殖发育中的调控作用[J]. 生命科学,2016,28(6):640-644.
[2]Borsani G,Tonlorenzi R,Simmler M C,et al. Characterization of a murine gene expressed from the inactive X chromosome[J]. Nature,1991,351(6324):325-329.
[3]Mercer T R,Dinger M E,Mattick J S. Long non-coding RNAs:insights into functions[J]. Nature Reviews Genetics,2009,10(3):155-159.
[4]Zhang J,Mujahid H,Hou Y,et al. Plant long ncRNAs:a new frontier for gene regulatory control[J]. American Journal of Plant Sciences,2013,4(5):8.
[5]Wierzbicki A T,Haag J R,Pikaard C S. Noncoding transcription by RNA polymerase Pol Ⅳb/Pol Ⅴ mediates transcriptional silencing of overlapping and adjacent genes[J]. Cell,2008,135(4):635-648.
[6]Li L,Eichten S R,Shimizu R,et al. Genome-wide discovery and characterization of maize long non-coding RNAs[J]. Genome Biology,2014,15(2):R40.
[7]Matzke M,Mosher R A. RNA-directed DNA methylation:an epigenetic pathway of increasing complexity[J]. Nature Reviews Genetics,2014,15(6):394-408.
[8]Mattick J S. Non-coding RNAs:the architects of eukaryotic complexity[J]. EMBO Reports,2001,2(11):986-991.
[9]Jin J,Liu J,Wang H,et al. PLncDB:plant long non-coding RNA database[J]. Bioinformatics,2013,29(8):1068-1071.
[10]Sun X,Zheng H,Sui N. Regulation mechanism of long non-coding RNA in plant response to stress[J]. Biochemical and Biophysical Research Communications,2018,503(2):402-407.
[11]Fedak H,Palusinska M,Krzyczmonik K,et al. Control of seed dormancy in Arabidopsis by a cis-acting noncoding antisense transcript[J]. Proceedings of the National Academy of Sciences of the United States of America,2016,113(48):E7846-E7855.
[12]Franco-Zorrilla J M,Valli A,Todesco M,et al. Target mimicry provides a new mechanism for regulation of microRNA activity[J]. Nature Genetics,2007,39(8):1033-1037.
[13]Shin H,Shin H S,Chen R,et al. Loss of At4 function impacts phosphate distribution between the roots and the shoots during phosphate starvation[J]. Plant Journal,2006,45(5):712-726.
[14]Campalans A,Kondorosi A,Crespi M. Enod40,a short open reading frame-containing mRNA,induces cytoplasmic localization of a nuclear RNA binding protein in Medicago truncatula[J]. Plant Cell,2004,16(4):1047-1059.
[15]Heo J B,Sung S. Vernalization-mediated epigenetic silencing by a long intronic noncoding RNA[J]. Science,2011,331(613):76-79.
[16]Yamaguchi A,Abe M. Regulation of reproductive development by non-coding RNA in Arabidopsis:to flower or not to flower[J]. Journal of Plant Research,2012,125(6):693-704.
[17]Zhao X,Li J,Lian B,et al. Global identification of Arabidopsis lncRNAs reveals the regulation of MAF4 by a natural antisense RNA[J]. Nature Communications,2018,9(1):5056.
[18]Ariel F,Jegu T,Latrasse D,et al. Noncoding transcription by alternative RNA polymerases dynamically regulates an auxin-driven chromatin loop[J]. Molecular Cell,2014,55(3):383-396.
[19]Wang Y,Luo X,Sun F,et al. Overexpressing lncRNA LAIR increases grain yield and regulates neighbouring gene cluster expression in rice[J]. Nature Communications,2018,9(1):3516.
[20]Liu X,Li D,Zhang D,et al. A novel antisense long noncoding RNA,TWISTED LEAF,maintains leaf blade flattening by regulating its associated sense R2R3-MYB gene in rice[J]. New Phytologist,2018,218(2):774-788.
[21]Ding J,Lu Q,Ouyang Y,et al. A long noncoding RNA regulates photoperiod-sensitive male sterility,an essential component of hybrid rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(7):2654-2659.
[22]Song J H,Cao J S,Yu X L,et al. BcMF11,a putative pollen-specific non-coding RNA from Brassica campestris ssp. chinensis[J]. Journal of Plant Physiology,2007,164(8):1097-1100.
[23]Song J H,Cao J S,Wang C G. BcMF11,a novel non-coding RNA gene from Brassica campestris,is required for pollen development and male fertility[J]. Plant Cell Reports,2013,32(1):21-30.
[24]Huang L,Dong H,Zhou D,et al. Systematic identification of long non-coding RNAs during pollen development and fertilization in Brassica rapa[J]. The Plant Journal:for Cell and Molecular Biology,2018,96(1):203-222.
[25]Qin T,Zhao H,Cui P,et al. A nucleus-localized long non-coding RNA enhances drought and salt stress tolerance[J]. Plant Physiology,2017,175(3):1321-1336.
[26]Wang T,Zhao M,Zhang X,et al. Novel phosphate deficiency-responsive long non-coding RNAs in the legume model plant Medicago truncatula[J]. Journal of Experimental Botany,2017,68(21/22):5937-5948.
[27]Li S,Nayar S,Jia H,et al. The Arabidopsis hypoxia inducible AtR8 long non-coding RNA also contributes to plant defense and root elongation coordinating with WRKY genes under low levels of salicylic acid[J]. Non-coding RNA,2020,6(1):8.
[28]Cui J,Jiang N,Meng J,et al. LncRNA33732-respiratory burst oxidase module associated with WRKY1 in tomato-Phytophthora infestans interactions[J]. Plant Journal,2019,97(5):933-946.
[29]Cui J,Luan Y S,Jiang N,et al. Comparative transcriptome analysis between resistant and susceptible tomato allows the identification of lncRNA16397 conferring resistance to Phytophthora infestans by co-expressing glutaredoxin[J]. Plant Journal,2017,89(3):577-589.
[30]Ponting C P,Oliver P L,Reik W. Evolution and functions of long noncoding RNAs[J]. Cell,2009,136(4):629-641.
[31]Wu L,Liu S,Qi H,et al. Research progress on plant long non-Coding RNA[J]. Plants,2020,9(4):408.
[32]Wang K C,Chang H Y. Molecular mechanisms of long noncoding RNAs[J]. Molecular Cell,2011,43(6):904-914.
[33]Rai M I,Alam M,Lightfoot D,et al. Classification and experimental identification of plant long non-coding RNAs[J]. Genomics,2019,111(5):997-1005.
[34]Pontier D B,Gribnau J. Xist regulation and function explored[J]. Human Genetics,2011,130(2):223-236.
[35]Chiou T J,Aung K,Lin S I,et al. Regulation of phosphate homeostasis by MicroRNA in Arabidopsis[J]. Plant Cell,2006,18(2):412-421.
[36]Good M C,Zalatan J G,Lim W A. Scaffold proteins:hubs for controlling the flow of cellular information[J]. Science,2011,332(630):680-686.
[37]Rinn J L,Kertesz M,Wang J K,et al. Functional demarcation of active and silent chromatin domains in human HOX loci by noncoding RNAs[J]. Cell,2007,129(7):1311-1323.
[38]Spitale R C,Tsai M C,Chang H Y. RNA templating the epigenome:long noncoding RNAs as molecular scaffolds[J]. Epigenetics,2011,6(5):539-543.
[39]Wierzbicki A T. The role of long non-coding RNA in transcriptional gene silencing[J]. Current Opinion in Plant Biology,2012,15(5):517-522.
[40]Daxinger L,Kanno T,Bucher E,et al. A stepwise pathway for biogenesis of 24-nt secondary siRNAs and spreading of DNA methylation[J]. EMBO Journal,2009,28(1):48-57.
[41]Xie Z,Johansen L K,Gustafson A M,et al. Genetic and functional diversification of small RNA pathways in plants[J]. PLoS Biology,2004,2(5):104.
[42]Zheng Q,Rowley M J,Bhmdorfer G,et al. RNA polymerase V targets transcriptional silencing components to promoters of protein-coding genes[J]. Plant Journal,2013,73(2):179-189.
[43]Onodera Y,Haag J R,Ream T,et al. Plant nuclear RNA polymerase IV mediates siRNA and DNA methylation-dependent heterochromatin formation[J]. Cell,2005,120(5):613-622.
[44]Fan Y,Yang J,Mathioni S M,et al. PMS1T,producing phased small-interfering RNAs,regulates photoperiod-sensitive male sterility in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2016,113(52):15144-15149.
[45]Bardou F,Ariel F,Simpson C G,et al. Long noncoding RNA modulates alternative splicing regulators in Arabidopsis[J]. Developmental Cell,2014,30(2):166-176.
[46]Kouchi H,Takane K,So R B,et al. Rice ENOD40:isolation and expression analysis in rice and transgenic soybean root nodules[J]. The Plant Journal:for Cell and Molecular Biology,1999,18(2):121-129.
[47]Compaan B,Yang W C,Bisseling T,et al. ENOD40 expression in the pericycle precedes cortical cell division in Rhizobium-legume interaction and the highly conserved internal region of the gene does not encode a peptide[J]. Plant and Soil,2001,230(1):1-8.
[48]Kakar K,Zhang H,Scheres B,et al. CLASP-mediated cortical microtubule organization guides PIN polarization axis[J]. Nature,2013,495(7442):529-533.
[49]Wang Y,Fan X,Lin F,et al. Arabidopsis noncoding RNA mediates control of photomorphogenesis by red light[J]. Proceedings of the National Academy of Sciences of the United States of America,2014,111(28):10359-10364.
[50]Chekanova J A. Long non-coding RNAs and their functions in plants[J]. Current Opinion in Plant Biology,2015,27:207-216.
[51]Kim D H,Xi Y,Sung S. Modular function of long noncoding RNA,COLDAIR,in the vernalization response[J]. PLoS Genetics,2017,13(7):e1006939.
[52]Li X,Shahid M Q,Wen M,et al. Global identification and analysis revealed differentially expressed lncRNAs associated with meiosis and low fertility in autotetraploid rice[J]. BMC Plant Biology,2020,20(1):82.
[53]Wang M,Yuan D,Tu L,et al. Long non-coding RNAs and their proposed functions in fibre development of cotton (Gossypium spp.)[J]. New Phytologist,2015,207(4):1181-1197.
[54]Zhang B,Zhang X,Zhang M,et al. Transcriptome analysis implicates involvement of long noncoding RNAs in cytoplasmic male sterility and fertility restoration in cotton[J]. International Journal of Molecular Sciences,2019,20(22):5530.
[55]Li Y,Qin T,Dong N,et al. Integrative analysis of the lncRNA and mRNA transcriptome revealed genes and pathways potentially involved in the anther abortion of cotton (Gossypium hirsutum L.)[J]. Genes,2019,10(12):947.
[56]Li P,Yang H,Wang L,et al. Physiological and transcriptome analyses reveal Short-Term responses and formation of memory under drought stress in rice[J]. Frontiers in Genetics,2019,10:55.
[57]Tan X,Li S,Hu L,et al. Genome-wide analysis of long non-coding RNAs (lncRNAs) in two contrasting rapeseed (Brassica napus L.) genotypes subjected to drought stress and re-watering[J]. BMC Plant Biology,2020,20(1):81.
[58]Du Q,Wang K,Zou C,et al. The PILNCR1-miR399 regulatory module is important for low phosphate tolerance in maize[J]. Plant Physiology,2018,177(4):1743-1753.
[59]Wu J,Okada T,Fukushima T,et al. A novel hypoxic stress-responsive long non-coding RNA transcribed by RNA polymerase Ⅲ in Arabidopsis[J]. RNA Biology,2012,9(3):302-313.
[60]Yang Y,Liu T,Shen D,et al. Tomato yellow leaf curl virus intergenic siRNAs target a host long noncoding RNA to modulate disease symptoms[J]. PLoS Pathogens,2019,15(1):e1007534.
[61]Zhang L,Wang M J,Li N N,et al. Long noncoding RNAs involve in resistance to Verticillium dahliae,a fungal disease in cotton[J]. Plant Biotechnology Journal,2018,16(6):1172-1185.
[62]Mattick J S,Makunin I V. Non-coding RNA[J]. Human Molecular Genetics,2006,15(1):R17-R29.
[63]Li S,Liberman L M,Mukherjee N,et al. Integrated detection of natural antisense transcripts using strand-specific RNA sequencing data[J]. Genome Research,2013,23(10):1730-1739.
[64]Song X,Sun L,Luo H,et al. Genome-wide identification and characterization of long non-coding RNAs from mulberry (Morus notabilis) RNA-seq data[J]. Genes,2016,7(3):11.
[65]Shen E,Zhu X,Hua S,et al. Genome-wide identification of oil biosynthesis-related long non-coding RNAs in allopolyploid Brassica napus[J]. BMC Genomics,2018,19(1):745.
[66]Rhoads A,Au K F. PacBio sequencing and its applications[J]. Genomics,Proteomics & Bioinformatics,2015,13(5):278-289.
[67]Cui J Shen N,Lu Z,et al. Analysis and comprehensive comparison of PacBio and nanopore-based RNA sequencing of the Arabidopsis transcriptome [J]. Plant Methods,2020,16:85.
[68]Zhang G,Sun M,Wang J,et al. PacBio full-length cDNA sequencing integrated with RNA-seq reads drastically improves the discovery of splicing transcripts in rice[J]. The Plant Journal:for Cell and Molecular Biology,2019,97(2):296-305.
[69]Feng S,Xu M,Liu F,et al. Reconstruction of the full-length transcriptome Atlas using PacBio Iso-Seq provides insight into the alternative splicing in Gossypium australe[J]. BMC Plant Biology,2019,19(1):365.
[70]Yu Y,Yf Z,Feng Y Z,et al. Transcriptional landscape of pathogen-responsive lncRNAs in rice unveils the role of ALEX1 in jasmonate pathway and disease resistance[J]. Plant Biotechnology Journal,2020,18(3):679-690.
[71]Li R,Fu D,Zhu B,et al. CRISPR/Cas9-mediated mutagenesis of lncRNA1459 alters tomato fruit ripening[J]. The Plant Journal,2018,94(3):513-524.
[72]Bhmdorfer G,Rowley M J,Kuciński J,et al. RNA-directed DNA methylation requires stepwise binding of silencing factors to long non-coding RNA[J]. The Plant Journal,2014,79(2):181-191.
[73]Seo J S,Diloknawarit P,Park B S,et al. ELF18-INDUCED LONG NONCODING RNA 1 evicts fibrillarin from mediator subunit to enhance PATHOGENESIS-RELATED GENE 1 (PR1) expression[J]. New Phytologist,2019,221(4):2067-2079.
[74]Kindgren P,Ard R,Lvanov M,et al. Transcriptional read-through of the long non-coding RNA SVALKA governs plant cold acclimation[J]. Nature Communication,2018,9(1):4561.
[75]Gai Y P,Yuan S S,Zhao Y N,et al. A novel LncRNA,MuLnc1,associated with environmental stress in mulberry (Morus multicaulis)[J]. Frontiers in Plant Science,2018,9:669.
[76]Cao H,Wahlestedt C,Kapranov P. Strategies to annotate and characterize long non-coding RNAs:advantages and pitfalls[J]. Trends in Genetics,2018,34(9):704-721.
