利用小RNA深度测序快速鉴定草莓病毒
2021-07-29何成勇高德航范灵姣李世访王红清
何成勇 高德航 范灵姣 邢 飞 李世访 王红清*
(1.中国农业大学 园艺学院,北京 100193; 2.中国农业科学院 植物保护研究所/植物病虫害生物学国家重点实验室,北京 100193)
草莓(Fragaria×ananassaDuch.)是一种重要的经济作物,在世界各地被广泛种植。由于草莓成熟早和见效快,近几年我国草莓产业发展迅速,栽培面积和产量均居世界首位。然而,由于草莓的多年生长和长期的无性繁殖,导致其生长过程中易受到多种病毒的侵染[1],造成果实品质下降,产量降低[2]。目前,已有20多种病毒[3-6]和1种类病毒[7]被报道能够侵染草莓,当前我国各草莓产区普遍发生的草莓病毒有:草莓斑驳病毒(strawberry mottle virus,SMoV)、草莓镶脉病毒(strawberry vein banding virus,SVBV)、草莓皱缩病毒(strawberry crinkle virus,SCV)和草莓轻型黄边病毒(strawberry mild yellow edge virus,SMYEV);其他病毒如黄瓜花叶病毒[8](cucumber mosaic virus, CMV)、草莓坏死休克病毒[9](strawberry necrotic shock virus, SNSV)、草莓白化病毒[10](strawberry pallidosis associated virus,SPaV)、草莓毛形病毒3(strawberry crinivirus 3,SCrV 3)和草莓毛形病毒4[11](strawberry crinivirus 4,SCrV 4)等在我国部分草莓产区也有报道。草莓病毒大多属于潜隐性病毒,单一侵染时一般不显病症,复合侵染时造成花叶、皱叶、叶柄病变、叶脉褪绿、果实畸形、品质变劣、产量下降和植株矮小等症状,而且一旦植株感染病毒将终生带毒且无法脱除。草莓病毒病严重制约着我国乃至世界草莓产业的可持续发展。因此,快速解析草莓植株中的病毒对于草莓病毒病的防控意义重大。
草莓病毒的传统检测方法有指示植物检测法[12]、电镜检测法[13]、血清学检测法[14]和分子生物学检测法等,但传统的病毒检测方法有其局限性,如指示植物检测法对嫁接技术要求较高,耗时较长,且由于病毒的混合侵染和不同组织病毒含量的差异造成该鉴定方法实际操作难度较大[15];电镜检测法价格昂贵而且对初学者来说掌握的难度比较大,检测结果往往容易受到破碎的植物细胞器的干扰[16];血清学检测法需要根据每种病毒制备特异抗体,过程复杂;较为常用的PCR检测方法需要根据已知病毒序列设计PCR引物,在病毒序列变异度高或检测未知病毒时则很难发挥作用。近年来,sRNA测序技术的发展为植物病毒检测提供了新的思路。sRNA测序的原理是对寄主体内病毒来源的小RNA(virus-derived small interfering RNA, vsiRNA)进行测序和分析,从而鉴定寄主中的病毒种类。sRNA测序因其可同时检测DNA和RNA病毒且不受病毒poly(A)结构的影响等优点已被广泛用于葡萄[17]、西番莲[18]、库尔勒香梨[19]和柑橘[20]等果树病毒的检测中。目前,国内应用sRNA测序鉴定草莓病毒的报道较少。本研究通过sRNA测序技术鉴定了‘丰香’和‘哈尼’2个草莓品种中存在的病毒,并通过RT-PCR对测序结果进行了验证,旨在为草莓病毒的快速鉴定及明确草莓病毒的种类和分布提供研究方法和思路。
1 材料与方法
1.1 试验材料
以‘丰香’草莓和‘哈尼’草莓为试验材料。每个草莓品种各随机采集2份样品,分别于2020年7—8月, 采自山东青岛(‘丰香’草莓)和辽宁东港(‘哈尼’草莓),采集幼嫩的草莓新叶,液氮速冻后于-80 ℃低温冰箱保存备用。
1.2 试验方法
1.2.1总RNA提取与检测
草莓叶片总RNA的提取采用改良的CTAB法[21],利用Nanodrop 2000分光光度计(Thermo Scientific,Wilmington,USA)检测总RNA的浓度与纯度,利用1%琼脂糖凝胶电泳检测RNA的完整性。
1.2.2sRNA文库构建及测序
sRNA测序文库构建及测序由联川生物技术股份有限公司完成。sRNA测序文库制备采用TruSeqTMSmall RNA Sample Prep Kits (Illumina, San Diego, USA)试剂盒。将4份样品分别提取的总RNA等量混合后,首先利用T4 RNA连接酶(Truncated)将一个腺苷化单链DNA3′接头和5′接头相继连接到sRNA上,通过与3′端互补的RT引物进行反转录反应,最后进行PCR扩增得到cDNA序列。用6% polyacrylamide Tris-borate -EDTA胶回收长度大约在140 bp的PCR产物,从而完成整个文库制备工作,构建好的文库用Illumina Hiseq 2500进行测序,测序读取长度为单端1×50 bp。
1.2.3病毒sRNA序列分析
测序获得的原始数据,去除低质量序列后将5′和3′接头序列信息去除,获得clean reads;保留长度在18~25 nt的核苷酸序列,并比对mRNA、RFam和Repbase数据库。随后将序列与宿主基因组比对,过滤比对到基因组的序列。将不能定位到宿主基因组的序列与病毒数据库进行比对并进行热点区分布分析。然后用Velvet软件进行序列拼接,获得较长的contigs。最后,拼接完成后用NT和NR数据库进行病毒注释。
1.2.4RT-PCR验证测序结果
以草莓总RNA为模板合成第一链cDNA,反应体系为:RNA 1 μg、5×M-MLV buffer (Promega)2 μL、dNTP(2.5 mmol/L)、随机引物(6 mer)和Oligo-dT各1 μL,M-MLV和RNase Inhibitor 各0.5 μL,RNase free water补足体系至10 μL,混匀,42 ℃反应60 min,于-20 ℃冰箱保存备用。根据比对得到的病毒核苷酸序列[22-24],设计特异引物(表1),用于RT-PCR检测,引物由北京六合华大科技有限公司合成。PCR扩增体系为:cDNA 2 μL、2×pfu PCR Mastermix(天根生化科技有限公司)10 μL、上下游引物(10 μmol/L)各1 μL,ddH2O补足体积至20 μL,充分混匀。PCR反应程序:94 ℃预变性5 min,94 ℃变性30 s,Tm退火30 s,72 ℃延伸30 s,30次循环,72 ℃终延伸10 min。PCR产物回收纯化后连接到pTOPO载体(艾德莱)上,连接产物转化大肠杆菌DH5α,挑取单克隆测序验证。

表1 用于检测5种草莓病毒的引物序列Table 1 Primers used for five strawberry viruses detection
2 结果与分析
2.1 sRNA测序结果与分析
对‘丰香’和‘哈尼’2个草莓品种混样后进行sRNA测序,对sRNA测序数据进行分析,结果在混合样品中获得的sRNA总数为15 403 693,去除低质量序列,保留去除接头序列后长度在18~25 nt 的序列,筛选获得的clean reads数为11 685 131,占sRNA总数的75.86%。研究表明感染病毒的植物体内富集的病毒sRNA主要分为3个类型:21、22和24 nt,本研究从感染病毒的草莓样品中富集到的sRNA长度主要为21、24和22 nt(图1),reads数分别为4 080 830,3 585 518和1 996 530。获得的clean reads数的占比与sRNA的长度分布均表明从样本中获得的sRNA质量可靠。

图1 sRNA长度分布统计Fig.1 Statistics of sRNA length distribution
2.2 sRNA序列的拼接与注释
应用Velvet软件对sRNA进行序列拼接,获得较长的contigs。拼接后的序列用NT数据库进行病毒注释(表2)。结果显示共有242个contigs比对到5种草莓病毒,其中98个contigs比对到草莓白化病毒(SPaV),长度在34~191 nt,相似性为77%~100%,在基因组上的覆盖度为31%;61个contigs比对到草莓镶脉病毒(SVBV),长度在34~116 nt,相似性为88%~100%,在基因组上的覆盖度为50%;51个contigs比对到草莓斑驳病毒(SMoV),长度在34~123 nt,相似性为82%~100%,在基因组上的覆盖度为23%;16个contigs比对到草莓毛形病毒3(SCrV 3),长度在51~131 nt,相似性为92%~100%;16个contigs比对到草莓毛形病毒4(SCrV 4),长度在41~154 nt,相似性为77%~94%。综合比对到草莓病毒的contigs数、覆盖度和相似性,推测混合样本中含有这5种病毒的可能性大小依次为:SVBV、SPaV、SMoV、SCrV 3和SCrV 4。
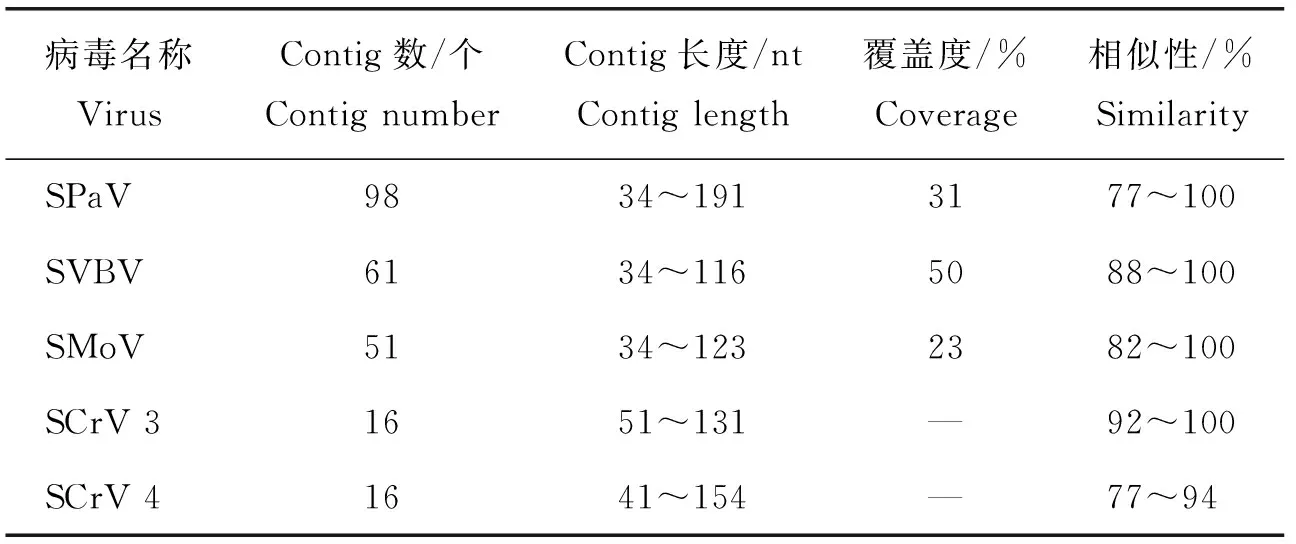
表2 sRNA测序检出草莓病毒数据统计Table 2 Statistics of detected strawberry viruses by sRNA sequencing
2.3 RT-PCR验证
为验证sRNA测序结果的可靠性,根据比对到的草莓病毒序列设计5种病毒的特异引物(表1),分别对4份样品进行RT-PCR扩增。结果显示,部分样品中分别扩增得到大小为462、278、517、517和549 bp的特异性条带(图2)。PCR产物测序结果经与GenBank中登录序列比对后发现,SMoV、SVBV和SCrV 3在4份样品中均检测到,而SPaV和SCrV 4只在2份‘哈尼’草莓样品中检测到。RT-PCR结果表明,本研究中经sRNA测序比对到的草莓病毒均存在于草莓样本中。RT-PCR与sRNA测序结果一致,说明利用sRNA测序鉴定草莓病毒的结果可靠。

M,DNA标准DL2 000;PC,阳性对照; CK,健康植株;1~2,‘丰香’草莓;3~4,‘哈尼’草莓。M, DL2 000 DNA Marker; PC, Positive control; CK, healthy plant;1-2, ‘Toyonoka’; 3-4, ‘Honeoye’.图2 5种草莓病毒的RT-PCR扩增Fig.2 RT-PCR amplification results of five kinds of strawberry viruses
3 讨论与结论
RNA沉默(RNA silencing)是植物抗病毒的主要机制,植物体内的DCL蛋白可将病毒来源的双链RNA(dsRNA)或中间复合体加工形成大量长度在21~24 nt病毒来源的小干扰RNA(vsiRNA)[25-26]。sRNA深度测序技术检测病毒是基于RNA沉默原理的一种病毒检测技术,对感染病毒的植株进行sRNA深度测序,经过生物信息学分析,获得的序列信息中包含与病毒序列高度一致的siRNA序列,再通过重新组装获得相关的目标病毒基因组的信息,可为发现新的病毒和鉴定病毒提供较充足的证据。本研究利用sRNA深度测序技术,从2份‘丰香’草莓和2份‘哈尼’草莓的混合样品中检测到SMoV、SVBV、SPaV、SCrV 3和SCrV 4共5种草莓病毒,后续利用RT-PCR验证测序结果时在2份‘丰香’草莓样品种检测到SMoV、SVBV和SCrV 3等3种病毒,而在2份‘哈尼’草莓样品中同时检测到这5种病毒,这与sRNA测序结果一致,证实了该方法鉴定草莓病毒的准确性和可靠性。
本研究检测出的5种病毒中,SMoV和SVBV是国内较为常见的草莓病毒,另外3种病毒在国内报道较少。Ding等[27]2017年首次在我国福建报道了SPaV能够侵染草莓;韩晓玉等[28]在河南郑州和新乡的草莓上也检测到SPaV;本研究中检测到SPaV的草莓品种‘哈尼’采自辽宁东港,说明该病毒分布的地理跨度较大。目前国内仅在这3个省市的草莓上检测到该病毒,至于其他草莓产区是否存在SPaV,将是今后草莓病毒检测需要关注的地方。SCrV 3和SCrV 4同属毛形病毒属,Chen等[11]2018年在福建福州地区首次报道了SCrV 3和SCrV 4,但在后续的调查中并没有检测到这2种病毒,说明这2种病毒的分布并不广泛。本研究中,SCrV 4仅在‘哈尼’草莓样品中检测到,而SCrV 3在‘哈尼’草莓和采自山东青岛的‘丰香’草莓品种中均检测到。本研究是第2次报道这2种病毒在国内其他草莓产区的存在,因此,其发生和分布值得进一步关注。
草莓病毒的复合侵染是造成草莓衰退病的主要诱因,严重降低草莓的品质和产量。谢雪花[29]利用sRNA测序在长沙地区的草莓上同时检测到CMV和SVBV;李伟佳等[9]利用RNA-Seq在同一草莓植株上同时检测到2~3种病毒;本研究在同一植株上最少检测到3种病毒,最多检测到5种病毒,说明草莓病毒的复合侵染较为普遍。需要注意的是,本研究中被病毒复合侵染的草莓植株并未表现出明显的病毒病症状,这对于草莓病毒病的发现和预防带来一定难度。至于复合侵染是否对草莓产量有影响将是下一步研究的重点。
本研究利用sRNA测序技术从2个草莓品种中共检测到5种病毒,并利用RT-PCR技术进行了验证,首次在我国生产的同一个草莓品种中检测到5种病毒。本研究可为草莓病毒的鉴定和监测工作提供借鉴。
