球状轮藻叶绿体全基因组的组装与特征分析
2021-07-15胡莎莎张冬群卿人韦兰利琼
方 琰, 胡莎莎, 张冬群, 吕 婕, 李 锐, 卿人韦, 兰利琼
(四川大学生命科学学院生物资源与生态环境教育部重点实验室, 成都 610065)
1 引 言
轮藻(Charophytes)是一种广布全球的大型水生藻类,形态高度分化,拥有与陆地植物最近的亲缘关系,两者共同构成链型植物门[1](Streptophytes).由于轮藻在植物进化史中的特殊分类地位,其可将其作为植物从水生到陆生环境的一种过渡适应机制模型[2],轮藻对生境水质要求较高,可作为一种生物指示剂[3].近年来,由于环境污染问题日益加剧,轮藻被许多国家纳入濒危物种名录[4-5],然而我国对轮藻的研究和保护还未得到充分重视.
现生轮藻科(Characeae)分为轮藻族(Chareae)和丽藻族(Nitelleae).如今基于形态和系统发育分析的分类矛盾争议点大多集中在种属间,如通过传统的形态学观察只能根据雌雄同株或异株来区分球状轮藻(Charaglobularis)和弧枝轮藻(Characonnivens)两个种.Schneider等人[6]在系统分类研究中均发现利用单一叶绿体基因matK对二者无法进行有效区分;Nowak等人[7]通过2个核基因18S rDNA, ITS1和3个叶绿体基因rbcL,atpB,matK的联合数据对瑞典14种轮藻属植物进行系统发育关系研究,并结合形态学分类对比分析表明,种间遗传分化程度较低,物种的界定非常不明确,形态分类和分子分类结果存在冲突;Wood的传统分类学认为灯枝藻属是轮藻属和丽枝藻属的姐妹群,拟丽藻属与前三者相比关系较远,但任玲萱等人通过对轮藻科植物18S rDNA、rbcL、atpB联合聚类分析表明,灯枝藻属与拟丽藻属具有很强的亲缘关系,与传统分类学观点存在分歧[8].现今基于几个常用分子片段联合分析的轮藻科部分系统发育结果较为混乱,无法较好的解决轮藻科植物内部分类结构不清晰等问题,所以扩大数据量并利用基因组进行系统发育研究已成为大势所趋.
叶绿体是一种重要的植物细胞器,其DNA一般为双链环状结构,长约120~180 kb,在光合作用等多种代谢途径中起到关键作用[9].叶绿体起源于蓝藻的内共生学说,因其具有母系遗传的特性使其较核基因组和线粒体基因组更为保守[10].叶绿体基因组含有大量变异速率适中的功能基因,可用作DNA条形码广泛应用于系统发育、种群分析等方面的研究[11-12].大多数叶绿体基因组具有典型的四分体结构,包括一个大的单拷贝(LSC)区,一个小的单拷贝(SSC)区和两个反向重复序列(IRa和IRb).自从烟草(Nicotianatabacum)[13]和地钱(Marchantiapolymorpha)[14]叶绿体基因组序列公布以来,科研工作者们对叶绿体基因组的关注度不断增加.Rochaix[15]于1978年得到了第一个衣藻叶绿体全基因组图谱.此后藻类叶绿体基因组数据不断增长.藻类叶绿体基因组大小差别较大,最小的刺松藻(Codiumfragile)仅为86 kb,最大的伞藻(Acetabularia)可达到2 000 kb[16].
Monique等人[17]对普生轮藻(Charavulgaris)进行了测序并得到了完整的叶绿体全基因组序列,研究分析表明,在迄今为止测定的所有链型植物门叶绿体基因组数据中,轮藻具有最大的叶绿体基因组,最长的单拷贝区和最高的A+T含量,造成这种情况的主要原因是叶绿体基因组内基因间隔区的扩张和内含子长度的增加.普生轮藻叶绿体基因组结构也与陆生植物有较大不同.Orton等人[18]利用三种轮藻植物并结合其他绿藻植物和有胚植物的叶绿体全基因组探究了叶绿体基因的保留和/或丢失模式,以了解陆地植物的祖先是如何进化出适应陆地的生存机制.
随着高通量测序技术的不断发展,植物叶绿体全基因组的测序变得越来越容易[12].在众多轮藻科植物研究中,基于叶绿体全基因组的研究十分匮乏.在这里,我们对球状轮藻叶绿体全基因组进行了测序、组装和拼接,旨在揭示球状轮藻叶绿体全基因组结构特征,丰富其叶绿体基因组信息,并为后续探究轮藻科植物物种鉴定和系统进化关系提供理论依据.
2 材料与方法
2.1 实验材料
本文所用的球状轮藻藻体采集于四川省成都市五凤溪.将长势较好的新鲜藻体用流水洗净,清理表面杂质杂藻,去除假根及较老的结,再用双蒸水反复冲洗后液氮速冻并于-80 ℃冷藏保存待用.
2.2 方 法
2.2.1 DNA提取及测序 采用植物基因组提取试剂盒(Tiangen Biotech Co.,Ltd,Beijing,PA,China)提取上述处理好的球状轮藻样品DNA,送至北京诺和致源生物信息科技有限公司,检测合格后,利用Covaris超声波破碎仪随机打断叶绿体DNA生成300~500 bp的DNA小片段,在序列末端修复、加A尾、加测序接头,再经过纯化、PCR扩增等一系列操作构建好整个球状轮藻文库,之后用Qubit 2.0进行初步定量,并稀释文库,再用Agilent 2100对文库的插入片段进行检测,当大小符合预期后,采用Q-PCR法对文库有效浓度进行准确定量分析以保证文库质量.文库经检测合格后,根据目标下机数据量的需求和有效浓度把不同文库pooling至flowcell后使用Ⅱllumina novaseq 6000进行双末端测序,得到测序的原始读序(Raw Reads),最后使用FastQC v0.11.7软件评估并过滤掉低质量reads得到纯净读序(Clean Reads),从NCBI(National Center for Biotechnology Information)上下载普生轮藻(Charavulgaris)叶绿体全基因组作为参考序列,并将所有reads映射其上进行最后质量分析.
2.2.2 叶绿体全基因组的组装、注释及物理图谱构建 我们采用GetOrganelle软件[19]对Clean Reads进行组装,经过尝试,最终设定word size值为101,其他参数值默认,利用软件Geneious11.0.4将结果与Charavulgaris进行比较,验证其组装效果并进行下一步注释[20],之后对输出文件进行手动修正后将最终注释结果上传至在线网站OGDRAW(http://ogdraw.mpimp-golm.mpg.de)得到球状轮藻叶绿体全基因组完整图谱[21].
2.2.3 简单重复序列分析 使用MISA (MIcroSAtellite identification tool)软件(http://pgrc.ipk-gatersleben.de/misa/)对球状轮藻叶绿体全基因组进行简单重复序列分析,重复单元参数设置为单碱基重复、二碱基重复、三碱基重复、四碱基重复、五碱基重复、六碱基重复分别为10、6、4、3、3、3.设置2个SSR之间最小距离值为100 bp[22].
2.2.4 密码子偏好性分析 根据注释结果提取球状轮藻叶绿体全基因组所有的CDS序列,并依次写入fasta文件,使用EMBOSS 6.4.0 (http://emboss.open-bio.org/)在线软件分析密码子使用率,使用软件CodonW 1.4.2计算同义密码子使用度[23],所有参数均设定为默认值.
2.2.5 系统发育分析 选取从NCBI数据库中下载的轮藻族普生轮藻(Charavulgaris)和丽藻族无色丽藻(Nitellahyalina)叶绿体全基因组数据,并以鞘毛藻科的Chaetosphaeridiumglobosum和双星藻科的Zygnemacircumcarinatum作为外类群,与本研究所得到的球状轮藻叶绿体全基因组共5个一起提取共有CDS基因,并使用MEGA 7.0软件采用邻接法(Neighbor-joining, NJ)构建系统发育进化树,自展值(Bootstrap, BS)重复抽样1 000次.
3 结果与分析
3.1 叶绿体基因组结构特征
本实验测序结果中,Clean Reads占Raw Reads的99.55%,该值大于90%表明数据质量较为理想,可用于后续实验分析.组装拼接得到的球状轮藻叶绿体基因组全长180 652 bp,GC含量26.6%,其具有高度保守的典型四分体结构,其中LSC(large single copy)区长131 709 bp,GC含量为25.14%、SSC(small single copy)区长27123 bp,GC含量为23.6%,IR(inverted repeat)区长10 910 bp,GC含量为39.0% (表1).
球状轮藻叶绿体基因组序列共注释出包括9对重复基因在内的137个基因,其中包括94个蛋白质编码基因、37个tRNA基因和6个rRNA基因(表2).LSC区包括78个CDS和26个tRNA;SSC区包括14个CDs和1个tRNA;而IR区则只有2个CDS,5个tRNA和3个rRNA.并且ndhF基因横跨IRB区和SSC区(图1).
球状轮藻叶绿体基因组共有17个基因含有内含子,包括10个CDS,6个tRNA和1个rRNA.其中ycf3基因有两个内含子,ndhB、atpF、rps16、trnK(uuu)、trnV(uac)、trnG(ucc)、trnL(uaa)、clpP、petB、rpl16、rpl2、trnI(gau)、trnA(ugc)、rrl、ndhA基因均只具有一个内含子,基因matK位于基因trnK(uuu)的内含子区域,rps12基因有一个反式剪接内含子.

图1 球状轮藻叶绿体基因组图谱Fig.1 The gene map of C. globularis

表2 球状轮藻叶绿体全基因组基因信息
3.2 球状轮藻简单重复序列分析
简单重复序列(SSR)又称微卫星序列(Microsatellite DNA),是基因组中由一到六个碱基重复组成的基本单位重复多次所构成的一段DNA,普遍存在于真核生物的核、线粒体及叶绿体基因组中,因其具有良好的通用性而被广泛应用于物种鉴定及遗传差异性分析.本研究根据所选参数,从球状轮藻叶绿体基因组中共检测出87个SSR位点,其中单碱基重复最多,有24个,占比27.59%且均为A/T型;二碱基重复有21个,占比24.14%;三碱基重复最少,只有2个,占比13.5%;四碱基重复有20个,占比22.99%;五碱基重复有6个,占比6.90 %;六碱基重复有3个,占比均为3.45%;球状轮藻SSR类型中有11个复合型SSR,占比12.64%(表3).分析可知球状轮藻基因组中的SSR绝大部分由A和T构成.
在球状轮藻所有的SSR序列中,最长可达117 bp,最短只有10 bp,平均长度为19.56 bp,其中单碱基重复的平均长度为9.23 bp,二碱基重复的平均长度为15.33 bp,三碱基重复的平均长度为13.5 bp,四碱基重复的平均长度为14 bp,五碱基重复的平均长度为15 bp,六碱基重复的平均长度为18 bp,复合型SSR的平均长度为23.44 bp,重复序列长度为10~20 bp的最多,有72个(占82.75%),重复序列长度为21~40 bp的有5个(占5.75%),重复序列长度大于40 bp的有9个(占10.34%).SSR重复单元的重复次数为3~15次(不计算复合型SSR),其中重复次数为3~10次的有68个(占比78.16%),重复次数为11~15次的有19个(占比21.84%)(表4).
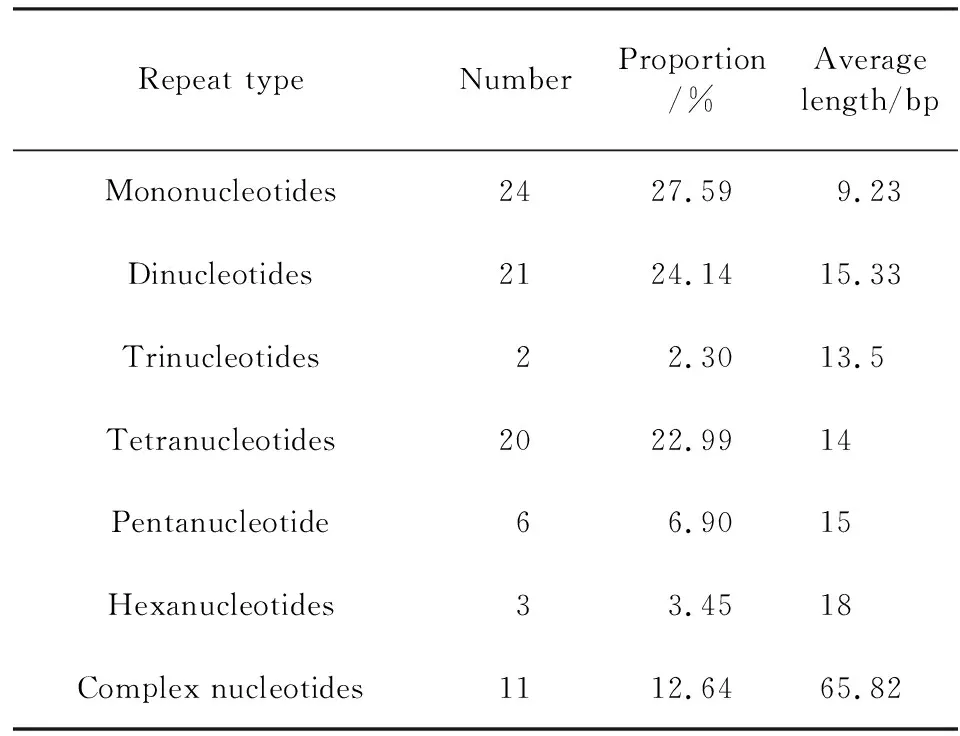
表3 球状轮藻叶绿体基因组SSR数据统计

表4 球状轮藻叶绿体基因组SSR预测
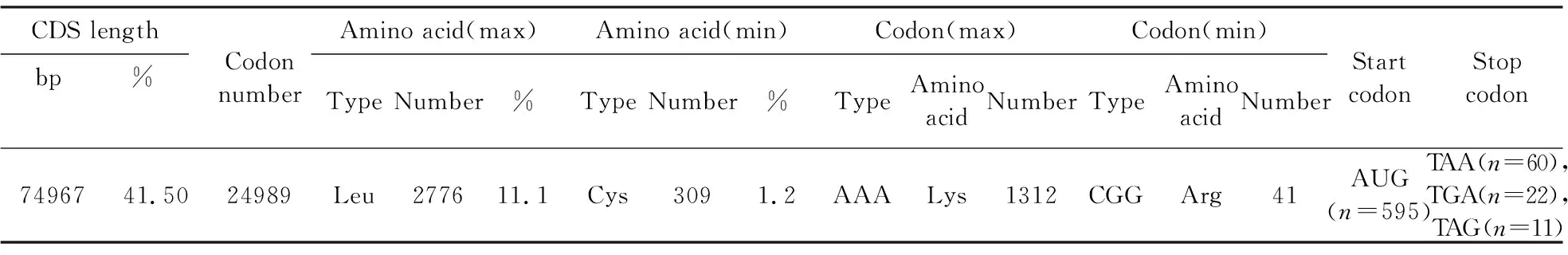
表5 球状轮藻叶绿体基因组密码子使用数据统计
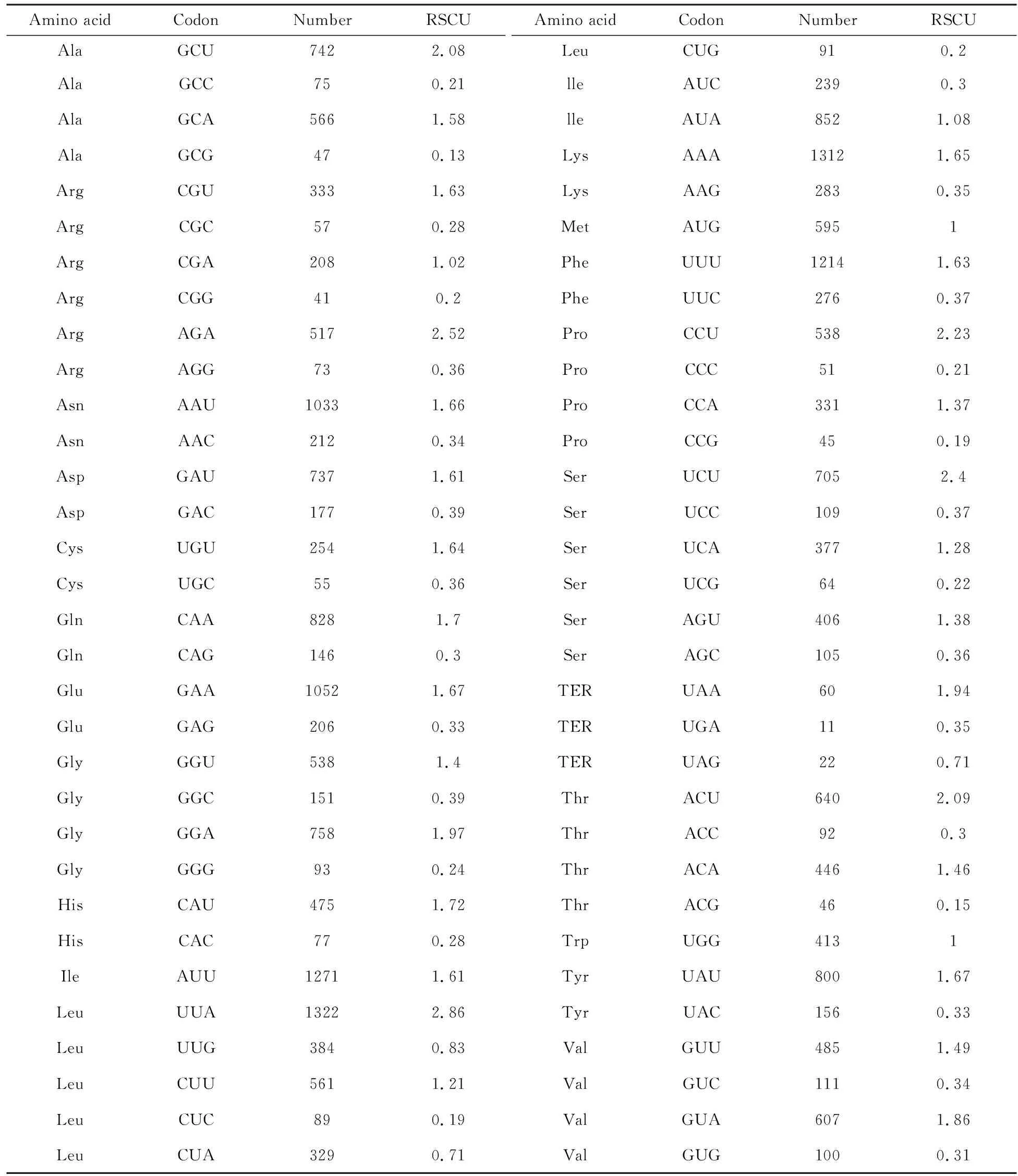
表6 球状轮藻叶绿体基因组密码子使用率
3.3 球状轮藻密码子使用偏好性分析
密码子由mRNA中3个相邻的核苷酸组成,在翻译时代表一种氨基酸.由于密码子存在简并性,即不同的密码子可以编码相同的氨基酸,同义密码子出现频率的高低反应了密码子使用的偏好情况.同义密码子使用度(RSCU)是衡量密码子偏好性的重要指标,如果密码子使用没有偏好,则该密码子的RSCU值等于1.当某一密码子的RSCU值大于1,则表明其的使用频率相对较高,具有偏好性.
在组装得到的球状轮藻叶绿体全基因组编码序列中,去除掉ycf20基因的一个重复序列后利用CodonW1.4.2对剩下的93条CDS序列进行密码子组成和偏好性分析,球状轮藻叶绿体基因组的蛋白质编码基因全长741 967 bp,共包含24 989个密码子.密码子编码氨基酸最多的是亮氨酸(Leu),有2 776个(占11.1%),密码子编码氨基酸最少的是半胱氨酸(Cys),有309个(占1.2%).这些密码子中使用使用最多的是AAA,编码赖氨酸(Lys)并出现了1 312次,使用最少的是CGG,编码精氨酸(Arg)并出现了41次(表5).除色氨酸(Trp)只有一个密码子外,其余的氨基酸均有2~6个密码子.此外,起始密码子AUG没有偏性(RSCU=1);在终止密码子中,TAA的使用最为频繁(RSCU>1),占终止密码子总数的64.52%.通过结果分析发现,球状轮藻叶绿体基因组密码子偏好A和T,编码同义密码子使用度(RSCU)大于1的密码子均以A/U结尾.
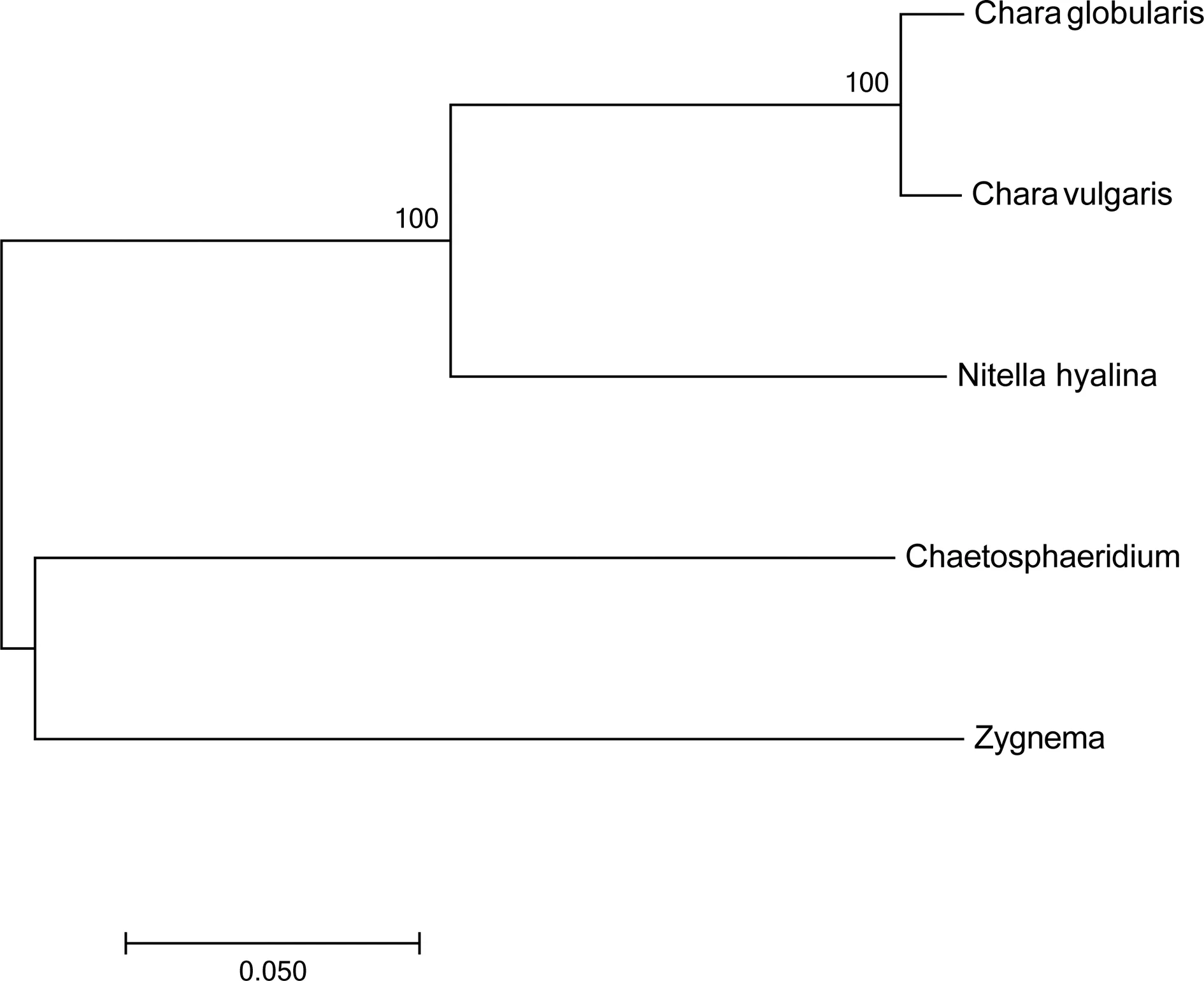
图2 基于叶绿体全基因组82个共有CDS构建的NJ系统发育树(“100”表示支持率为100%)Fig.2 Neighbor Joining phylogenetic tree constructed by 82 common CDS of complete chloroplast genomes("100" means the bootstrap values is 100%)
3.4 球状轮藻系统发育分析
选取包括球状轮藻在内的轮藻科三个种,并以鞘毛藻科的Chaetosphaeridiumglobosum和双星藻科的Zygnemacircumcarinatum作为外类群,提取出五个叶绿体全基因组共有CDS序列82个,采用邻接(NJ)法进行系统发育树的构建(图2).结果显示进化树的置信度均为100%,置信度大于90%则表明聚类结果可信度较高.在系统发育树中,球状轮藻与普生轮藻聚在一起成为轮藻族,表明二者的亲缘关系更近,与同源性可达100%;丽藻族的无色丽藻单独成为一个分支,轮藻族与丽藻族聚在一起成为姐妹群.
4 讨 论
轮藻植物因其独特的进化地位一直备受学者关注,但仅仅基于形态学和部分分子片段的分类学研究仍不能很好的解决部分种属间分类混乱的现象,叶绿体基因组具有母系遗传、序列保守和结构简单等特点,相比于几个分子片段,叶绿体全基因组可以反应更多的遗传信息.
本文首次揭示了球状轮藻叶绿体全基因组的结构特征,结果表明球状轮藻与大部分植物的叶绿体基因组结构相似,具有高度保守的四个区域结构,与已经报道的普生轮叶绿体藻基因组的结构、大小十分相似.球状轮藻共注释出了137个基因,包括94个蛋白质编码基因,37个tRNA和6个rRNA,有17个基因具有内含子,其中matK基因位于trnK(uuu)的内含子区域,rps12基因有一个反式剪接内含子,球状轮藻与其他链型植物叶绿体基因组在总体结构、基因数量和内含子含量上存在差别,例如Staurastrum和Zygnema的叶绿体基因组仅仅具有8和13个内含子,Spirogyramaxima叶绿体基因组则丢失了一个IR区域[18].与已报道的无色丽藻叶绿体全基因组相比,球状轮藻具有2个特殊蛋白质编码基因psaM、matK,3个tRNA基因包括trnK(uuu)、trnG(ucc)和trnL(uaa),但全长却比无色丽藻小21 148 bp,主要是由于叶绿体基因组内基因间隔区的扩张造成的.球状轮藻具有其他链型植物门植物所不具有的rpl12、trnL(gag)、rpl19、ycf20四个基因,rpl12和rpl19分别编码50S核糖蛋白体CL12和50S核糖蛋白体CL19;轮藻植物也是第一个报道rpl12基因的链型植物[19].该基因是一个特殊的反式剪切基因,3’端和5’端均位于LSC区域内,未知功能基因ycf20在IR区有2个拷贝,ndhF基因横跨IRB和SSC区.因轮藻具有特殊的进化地位和基因特点,后续可以加大样本量,从特殊基因入手进行系统发育研究,探究其对轮藻植物的影响.
叶绿体的简单重复序列既具有核基因组SSR的高多态性、多等位性、共显性等特点[24],又具有单亲遗传模式的结构简单、相对保守等特点[25-26],所以有较好的种间、种内遗传变异区分能力,已成为区分物种的重要分子标记而被广泛应用[27].球状轮藻叶绿体基因组中共检测出87个SSR位点,且大部分SSR都位于非编码区,只有少数位于编码区,其中单碱基重复最多,有27.59%均以A/T单碱基重复,碱基偏好性明显;其余所有的二碱基重复至六碱基重复中均含有AT,较多的SSR位点存在表明球状轮藻叶绿体基因组可能更易发生重排.
对球状轮藻的密码子偏好性进行分析得知,球状轮藻包含24 989个密码子.密码子编码氨基酸最多的是亮氨酸(Leu),密码子编码氨基酸最少的是半胱氨酸(Cys).这些密码子中使用使用最多的是AAA,编码赖氨酸(Lys),使用最少的是CGG,编码精氨酸(Arg).此外,起始密码子AUG没有偏性,同义密码子使用度(RSCU)为1,终止密码子中只有UAA的同义密码子使用度(RSCU)大于1,编码同义密码子使用度(RSCU)大于1的密码子均以A/U结尾.
在轮藻科内部研究物种间系统发育关系往往利用DNA条码进行单基因建树,但由于其序列较短,信息位点较少,不同的基因建树的结果往往不同,存在一定的局限性.叶绿体是高等植物共有的细胞器,含有足够信息位点的叶绿体基因组已被证明可有效判断系统发育关系,甚至是在较低的分类学水平下植物之间也有较强的分类学意义,为物种间系统发育的研究提供了新的思路[28].本实验通过对球状轮藻叶绿体全基因组的系统发育研究表明,球状轮藻与轮藻族的普生轮藻聚在一起成为一个姐妹群,表明二者的亲缘关系更近,这与任玲萱等人基于18S rDNA、rbcL、atpB联合聚类分析的结果一致[8],也符合球状轮藻的传统形态分类地位[29],但由于轮藻科植物的叶绿体全基因组信息较少,现今具有详细基因信息的叶绿体全基因组只有C.vulgaris和N.hyalina,后续应扩大轮藻科植物叶绿体全基因组数据库,以便更好的对其进行深入研究.
球状轮藻叶绿体全基因组结构与序列信息的揭示,为其遗传背景的研究和系统进化关系的探索奠定了基础.利用叶绿体结构基因组特征探究和验证轮藻系统发育与进化关系,为解决轮藻科部分物种分类混乱的问题提供了新的解决方案.后续作者将通过增加样本容量、加入更多有效分子片段并结合形态学分类特征等对轮藻科植物的分类学地位进行进一步探究,以期解决轮藻植物部分分类混乱的问题.
