向日葵全基因组NBS抗病基因密码子使用偏好性分析
2021-07-01刘洋路妍景岚
刘洋 路妍 景岚


摘要:为分析了解向日葵(Helianthus annuus L.)核苷酸结合位点(nucleotide binding site,简称NBS)型抗病基因编码时各密码子的使用情况,以向日葵全基因组NBS型抗病基因的255条基因序列为来源,利用CodonW软件对每个基因序列进行密码子统计分析。结果表明,向日葵NBS型抗病基因使用A/T(U)结尾的密码子占比大于G/C结尾的密码子,且有效密码子数(effective number of codon,简称ENC)平均值为51.40,整体偏好性较弱,但第3位碱基偏好使用以A/T(U)结尾的密码子。从同义密码子相对使用频率(relative synonymous codonw usage,简称RSCU)分析得到第3位为A、T结尾的密码子占密码子总数(RSCU>1)的比例最高。ENC-GC3s分布图表明,碱基突变是影响密码子偏好性的关键因素。同时相关性分析结果还表明,ENC与A3s、T3s呈极显著负相关关系,多种不同分析都印证了向日葵NBS型抗病基因序列偏好于以A/T(U)结尾的密码子。分析向日葵NBS型抗病基因密码子偏好性,通过优化密码子来提高NBS抗病基因在向日葵中的表达,可为向日葵分子改造和抗病育种提供理论依据。
关键词:向日葵;NBS抗病基因;密码子偏好性;密码子相关参数;相关性分析
中图分类号: S565.501;Q943.2 文献标志码: A 文章编号:1002-1302(2021)10-0043-05
向日葵(Helianthus annuus L.)别称太阳花、朝阳花属菊科(Asteraceae)向日葵属(Helainthus L.),是世界各地均有栽培的重要的油料作物。原产地位于南美洲,起初用于观赏。现我国新疆、内蒙古、黑龙江等北方地区均有栽培。据张一宾等在2015年的统计,全球向日葵种植面积约为2 540万hm2,同比上年增长1.0%[1]。向日葵生长发育的过程中受到许多病原物侵染是向日葵产量下降的主要原因,而培育和科学利用抗病品种是控制植物病害最经济有效的方式。抗病品种防御抵抗力强,能大大减少田间防治的成本,还能减少因使用农药造成的环境污染。
当植物受到病原菌刺激后会发出一系列的信号,并分泌抗病基因编码的抗病蛋白(R蛋白),通过识别病原菌分泌出的效应因子(Avr)后,再将信号进行传导继而引发一系列防御反应,最终就可以迅速地抑制病原菌对植物的侵染[2]。核苷酸结合位点作为R基因所编码的蛋白中高度保守的区域之一,在抗病过程中起重要作用[3]。核苷酸结合位点(nucleotide binding site,简称NBS)型蛋白有3个保守结构域[4],分别是磷酸结合环(P-loop,别称Kinase-1a)、激酶2a(Kinase-2a)、激酶3a(Kinase-3a),它们一起构成了抗病蛋白的(ATP/GTP)水解和抗病信号的传递[5]。除了这3个保守结构域外,NBS还含有其他几个保守区域,像GLPL和MHD等[6]。有研究表明,植物NBS型抗病基因是植物抗病基因中最大的一类,在植物自然免疫中起着重要的作用。
密码子(codon)指mRNA或DNA中每相邻的3个核苷酸编为一组序列,编码某一种特定氨基酸,是生物体内信息传递的基本环节。密码子偏性是生物体中编码同一种氨基酸的同义密码子的非均衡使用现象,由于这一现象与DNA和蛋白质相关联,所以具有重要的生物学意义。在生物体的遗传密码中,除了仅含有1个密码子的色氨酸和甲硫氨酸以外,所有其他氨基酸都具有1个以上的简并密码子。编码相同氨基酸的密码子是同义密码子(synonymous codon),在不同物种或基因中具有选择上的偏好,即为密码子选择偏好性(codon usage bias,简称CUB)。对于相同的物种,不同的蛋白编码密码子在基因中以不同的频率出现。对于相同氨基酸,编码氨基酸的不同密码子的概率在不同的蛋白之间也会有所不同。因此,生物体基因会对其他氨基酸上的简并密码子的选择具有一定的偏好性,结合密码子偏好性改造相关基因的密码子从而选择合适的表达系统,从而提高抗病基因在向日葵体内的表达,对于优化改进向日葵抗病体系具有重要意义。
近年来,随着科学技术的不断发展,越来越多物种的全基因组序列已经公布出来。对基因片段或全基因组序列密码子偏好性方面的研究也成为重点关注对象,像兰科植物(Orchidaceae Juss.)[7]、拟南芥[Arabidopsis thaliana (L.) Heynh.][8]、水稻[9]、柑橘(Citrus reticulata Blanco)[10]、葡萄(Vitis vinifera L.)[11]、香蕉(Musa nana Lour.)[12]、菠萝[Ananas comosus (Linn.) Merr.][13]的密码子偏好性均有报道。
2017年英国《Nature》杂志报道了法国科研人员通过测序获得了高质量的向日葵参考基因组,加速了向日葵育种计划[14]。随着向日葵全基因组序列测序的完成,笔者分析向日葵NBS型抗病基因编码时各密码子的使用情况,今后可以通过优化密码子来提高NBS抗病基因在向日葵中的表达,为向日葵抗病育种提供理论依据。
1 材料与方法
在https://www.ncbi.nlm.nih.gov/genome/?term=txid4232[orgn]上下载向日葵全基因组蛋白质序列,根据生物信息学分析P-loop、Kinase-2a、Kinase-3a、GLPL等4个保守结构域在基因中的峰度值,以NBS的保守结构域(P-loop、Kinase-2a、Kinase-3a、GLPL)为关键信息在Excel中进行检索及筛选,共得到255个符合条件的基因序列。再通过NCBI下载相应NBS FASTA格式的碱基序列。利用CodonW软件计算并统计分析向日葵NBS抗病基因组密码子使用情况。CodonW软件分析的数据主要包括同义密码子第3位上4种碱基的出现频率(A3s、G3s、C3s、T3s)、全基因組的GC含量、最优密码子使用频率(frequency of optimal codons,简称FOP)、密码子偏爱指数(codon bias index,简称CBI)等;采用同义密码子的相对使用度、密码子适应指数(codon adaption index,简称CAI)和有效密码子数(effective number of codon,简称ENC)3个指标对密码子偏好性进行评价。RSCU表示编码氨基酸同义密码子间的相对概率[15];RSCU的计算为某一密码子使用的频率和使用时预期频率的比值。若某一密码子的RSCU>1,则表示该密码子是相对使用频率较高的密码子,反之亦然;RSCU=1则代表该密码子无明显偏好性[7],其计算公式如下:
RSCUij=Xij1ni∑nij=1Xij。
式中:RSCUij表示编码第i个氨基酸第j个同义密码子间的相对概率;Xij是编码第i个氨基酸的第j个密码子的出现次数;ni是编码第i个氨基酸的同义密码子的数量(值为1~6)。
CAI的范围为0~1,越靠近1即表示这个基因的表达水平越高。ENC的范围为20~61,ENC越小,相应的内源基因的表达量就越高。
2 结果与分析
2.1 全基因组NBS抗病基因序列的密码子组成分析
分析结果(表1)表明,全基因组GC含量为36.50%~44.90%,平均GC含量为40.16%。对密码子第3个位置上的碱基类型进行统计学分析,C、G的出现频率平均值分别是22.05%、25.64%,而A和T(U)的出现频率分别为39.03%、40.90%。这些结果表明,向日葵NBS型基因使用A/T(U)结尾的密码子占比为79.93%,比G/C结尾的密码子占比高,向日葵基因组ENC平均值为51.40,说明向日葵密码子整体偏好性较弱。
2.2 同义密码子使用偏好分析
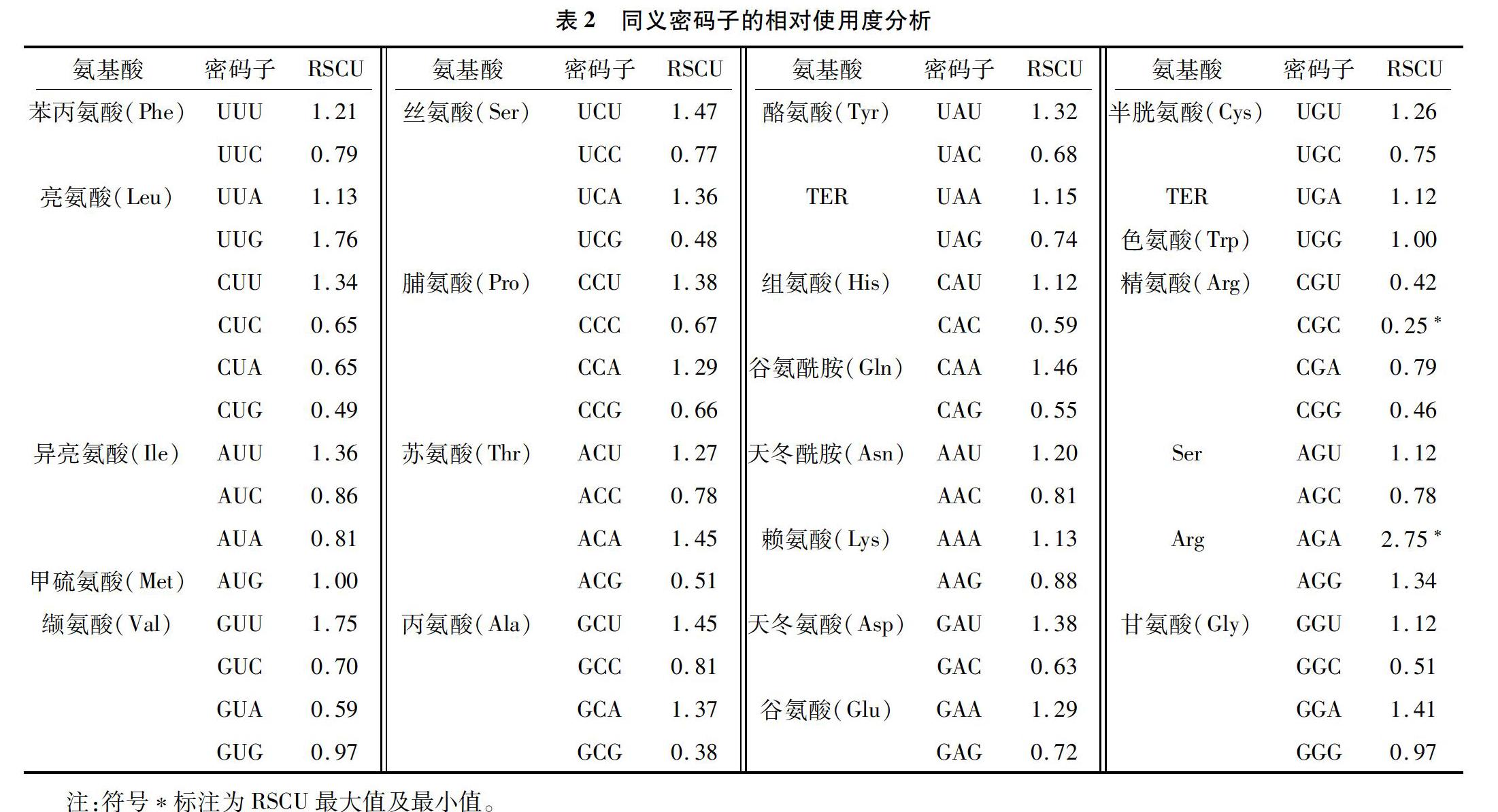
用CodonW软件计算并统计分析向日葵全基因组NBS抗病基因中所有氨基酸对同义密码子的偏好性、使用频率等数据。由表2可知,RSCU>1的密码子共29个,为高频密码子,且第3位为A、T(U)、C和G的密码子数量分别占RSCU>1密码子总数的41.38%、51.72%、0、6.90%。其中第3位为T(U)的密码子数量最多,第3位为C的密码子数量最少,表明向日葵NBS抗病基因编码时偏好使用以A/T(U)结尾的密码子,较少使用以C结尾的密码子。密码子AGA的RSCU最大,为2.75,该密码子的偏好性最强;编码氨基酸Trp、Met中的UGG、AUG的RSCU均等于1,表示向日葵NBS型基因中密码子UGG、AUG使用没有偏好性;氨基酸Arg中的密码子CGC的RSCU最小,为0.25,密码子偏好性最弱。
2.3 最优密码子的鉴定
将向日葵全基因组NBS基因序列按照ENC的大小进行排序,选取上下限区域各5%的序列数据形成高表达库和低表达库,再计算2个表达库中各个密码子的ΔRSCU,选取高表达库中RSCU大于1而在低表达库中小于1,共鉴定出 5个最优密码子(表3),即AUA、GUG、ACC、UAA、GGG。其中UAA密码子的ΔRSCU 为1.15,其偏好性最强。
2.4 ENC绘图分析
通过绘制ENC与GC3s 的相关性散点图,直观地解释密码子使用偏好性,可以反映无选择压力条件下二者之间的关系。通过对向日葵全基因组NBS型抗病基因GC3s与ENC的关联性进行分析(图1)可知,大部分基因点都落在期望曲线附近,ENC与GC3s 呈极显著相关关系(P<0.001,r2=0.465 8),这说明碱基突变对密码子偏好性的影响较强[16]。与期望曲线相距较远的受自然选择压力较大。处于曲线以下的基因点,其ENC相对较低,对应的内源基因表达量也相对越高,对密码子的使用具有更显著的偏好性;反之,处于曲线以上的基因点对密码子使用的选择性较低。
2.5 密码子评价参数的相关性分析
密码子各参数相关性分析结果(表4)表明,T3s与A3s、CAI呈极显著正相关关系(P<0.01,下同),而与C3s、G3s、CBI、ENC、GC3s和GC呈极显著负相关关系;C3s与G3s、CBI、FOP、ENC、GC3s和GC呈极显著正相关关系,而与A3s、CAI呈极显著负相关关系;A3s与G3s、CBI、FOP、ENC、GC3s和GC呈极显著负相关关系,与CAI呈显著负相关关系(P<0.05);G3s与CAI、FOP呈极显著负相关关系,与ENC、GC3s呈极显著正相关关系;CAI与CBI、FOP呈极显著正相关关系,与GC3s呈极显著负相关关系;CBI与FOP、ENC、GC3s和GC呈极显著正相关关系;FOP与GC呈极显著正相关关系;ENC与GC3s和GC呈极显著正相关关系;GC3s和GC呈极显著正相关关系;上述相关性分析结果表明,密码子上第3个碱基的不同类型会显著影响基因的表达水平。总之,基因在表达时对密码子第3位上的碱基类型具有不同选择性,且偏好选用GC含量较大且A/T为第3位结尾的同义密码子,从而影响基因表达水平。
3 讨论与结论
密码子偏好性是是翻译过程中的重要调控参数,不同生物的密码子使用偏性的差异非常不同。其中高表达密码子使用偏性较大,所对应的生物体中有非常丰富的tRNA;密码子偏好性还与mRNA的二级结构等因素相互影响[17-18]。另外,大多数密码子的3个碱基在进化过程中都会发生突变,不同碱基的突变所表现的意义也不同。前2个位置碱基的突变一般为非同义突变会改变基因的功能,第3个碱基的突变多为同义突变对基因功能无影响。因此,分析第3位碱基的影响因素可能会揭示出密码子偏好性的原因所在。本研究做了多种不同的统计分析,结果均表明向日葵NBS型抗病基因序列偏好于以A/T(U)结尾的密码子。向日葵全基因组平均GC含量为40.16%,大量研究表明双子叶植物的GC含量通常小于50%,而单子叶植物往往具有較高的GC含量,二者的密码子偏好性具明显差异[19]。不同植物的ENC均有所差异,萝卜ICE1基因密码子的ENC为51.35[20],菠萝基因组密码子的ENC为58.41[13],蓼科大黄属植物CHS基因密码子的ENC为34.35[21],红藻rbcL基因密码子的ENC为41.89[22]。分析所得,向日葵NBS型抗病基因密码子的ENC为51.40,香蕉NBS型抗病基因的ENC为56.40[23],水稻NBS-LRR基因ENC变化范围为31.22~59.47,相较于拟南芥(45.41~59.1)更大[24],更加表明不同物种不同基因所对应的密码子偏好性均有所差异(表5)。
同时相关性分析结果还表明,ENC与A3s和T3s大小呈极显著负相关关系,再次证明向日葵NBS型抗病基因序列偏好于以A/T(U)结尾的密码子。这也与水稻[24]、甘薯与三浅裂野牵牛[25]的NBS抗病基因偏好于以A/T(U)结尾相一致。从ENC-GC3s分布图中可以直观看出密码子的偏好性主要来自于碱基的突变,部分来自于自然選择压力。向日葵NBS型抗病基因使用A/T(U)结尾的密码子占比为79.93%,比G/C结尾的密码子高,且ENC平均值为51.40,说明向日葵NBS型抗病基因密码子整体偏好性较弱,但第3位碱基偏好使用以A/T(U)结尾的密码子。 从同义密码子相对使用频率分析得到,第3位为A、T结尾的密码子占密码子总数(RSCU>1)的比例最高,密码子AGA的RSCU最大,为2.75,该密码子的偏好性最强;通过计算RSCU高低表达库的平均值,鉴定出AUA、GUG、ACC、UAA、GGG等5个最优密码子。多种不同分析都印证了向日葵NBS型抗病基因序列偏好于以A/T(U)结尾的密码子。研究分析密码子偏好规律并优化密码子来提高NBS抗病基因在向日葵中的表达,未来可以对向日葵进行分子改造和抗病育种提供理论依据和指导作用。
参考文献:
[1]张一宾,顾林玲. 近年来全球向日葵种植面积及农药使用市场与品种[J]. 现代农药,2018,17(1):16-18.
[2]郑永杰,伍艳芳,李 江,等. 樟树NBS-LRR类抗病基因家族分析与CcRNL基因克隆[J]. 生物技术通报,2018,34(2):142-149.
[3]Liu J L,Liu X L,Dai L Y,et al. Recent progress in elucidating the structure,function and evolution of disease resistance genes in plants[J]. Journal of Genetics and Genomics,2007,34(9):765-776.
[4]Bent A F. Plant disease resistance genes:function meets structure[J]. The Plant Cell,1996,8(10):1757-1771.
[5]Lukasik E,Takken F L. STANDing strong,resistance proteins instigators of plant defence[J]. Current Opinion in Plant Biology,2009,12(4):427-436.
[6]森晓红. 向日葵NBS型抗锈病相关基因的克隆与分析[D]. 呼和浩特:内蒙古农业大学,2014.
[7]李 蓉,谢析颖,王雪晶,等. 兰科植物FNR基因的密码子偏好性分析[J]. 热带作物学报,2018,39(6):1137-1145.
[8]Mukhopadhyay P,Basak S,Ghosh T C. Differential selective constraints shaping codon usage pattern of housekeeping and tissue-specific homologous genes of rice and arabidopsis[J]. DNA Research,2008,15(6):347-356.
[9]Liu Q P. Mutational bias and translational selection shaping the codon usage pattern of tissue-specific genes in rice[J]. PLoS One,2012,7(10):e48295.
[10]Ahmad T,Sablok G,Tatarinova T V,et al. Evaluation of codon biology in Citrus and Poncirus trifoliata based on genomic features and frame corrected expressed sequence tags[J]. DNA Research,2013,20(2):135-150.
[11]续 晨,蔡小宁,钱保俐,等. 葡萄基因组密码子使用偏好模式研究[J]. 西北植物学报,2012,32(2):409-415.
[12]曲俊杰,黄 羽,卢 江,等. 香蕉基因组密码子使用偏好性分析[J]. 南方农业学报,2017,48(1):14-19.
[13]陈 哲,胡福初,王祥和,等. 菠萝密码子使用偏好性分析[J]. 果树学报,2017,34(8):946-955.
[14]Badouin H,Gouzy J,Grassa C J,et al. The sunflower genome provides insights into oil metabolism,flowering and Asterid evolution[J]. Nature,2017,546(7656):148-152.
[15]Sharp P M,Li W H. The codon adaptation index:a measure of directional synonymous codon usage bias,and its potential applications[J]. Nucleic Acids Research,1987,15(3):1281-1295.
[16]Ma Q P,Li C,Wang J,et al. Analysis of synonymous codon usage in FAD7 genes from different plant species[J]. Genetics and Molecular Research,2015,14(1):1414-1422.
[17]Novoa E M. Lluís ribas de pouplana.speeding with control:codon usage,tRNAs,and ribosomes[J]. Trends in Genetics,2012,28(11):574-581.
[18]胡振民,万 青,李 欢,等. 茶树CsNRT1.1基因密码子使用特性分析[J]. 江苏农业学报,2019,35(4):896-903.
[19]冯瑞云,田 翔,程 宏,等. 蒙古黄芪异黄酮合成酶基因密码子偏好性分析[J]. 山西农业科学,2019,47(3):297-303.
[20]孙继峰,韩太利,徐立功,等. 萝卜ICE1密码子使用偏性分析[J]. 核农学报,2018,32(3):477-485.
[21]马盛超,余鑫梅,姚慧鹏. 蓼科大黄属植物CHS基因密码子偏爱性分析[J]. 分子植物育种,2017,15(6):2171-2178.
[22]李国灵,陶 文,高诗晨,等. 红藻rbcL基因密码子偏爱性分析[J]. 分子植物育种,2020,18(1):109-117.
[23]方 辉,曲俊杰,孙嘉曼,等. 香蕉全基因组NBS抗病基因密码子使用偏好性分析[J]. 分子植物育种,2017,15(3):883-889.
[24]张晓峰,薛庆中. 水稻和拟南芥NBS-LRR基因家族同义密码子使用偏好的比较[J]. 作物学报,2005,31(5):596-602.
[25]陈观水.甘薯抗病相关基因的克隆与分析[D]. 福州:福建农林大学,2007.
