基于DCE-MRI影像的深度多任务学习联合预测乳腺癌病理信息研究∗
2021-06-16袁成成范明许茂盛王世威厉力华
袁成成范 明许茂盛王世威厉力华∗
(1.杭州电子科技大学生物医学工程与仪器研究所,浙江 杭州310018;2.浙江省中医院放射科,浙江 杭州310006)
乳腺癌居于全球女性因癌死因首位[1],在乳腺癌的诊断和治疗过程中,乳腺癌病理报告是其主要依据[2]。常见的病理信息包括乳腺癌的组织学分级,分子分型及Ki-67的表达等。诺丁汉组织学分级系统将其分成低、中、高3个组织学级别[3],组织学等级越高代表乳腺癌的恶性程度越高,乳腺癌的组织学等级已被证明能够产生与乳腺癌临床行为有关的重要信息[4]。肿瘤细胞增殖指数(Ki-67)是与细胞增殖相关的核抗原[5],Ki-67的增殖活性与乳腺癌肿瘤的分化、侵袭和转移程度以及预后相关[6]。乳腺癌分子分型主要可分为4种[7],包括管腔上皮A型(Luminal A型)、管腔上皮B型(Luminal B型)、人类表皮生长因子受体2过表达型(HER-2型)和基底细胞样型(Basal-like型)。乳腺癌分子分型对于选择合适的个性化治疗方法具有重要意义,有助于乳腺癌的精准诊疗[8]。
影像检查是乳腺癌早期诊断的常用方法。磁共振成像(MRI)技术被认为是重要的乳腺检查方式,对乳腺癌的诊断具有较高的灵敏度和特异性,并且没有辐射[9]。磁共振成像具有多种参数成像方式,动态对比增强磁共振成像(DCE-MRI)就是其中的一种,DCE-MRI具有良好的特异性,可以提供肿瘤的形态、血流特性和血管分布等信息[10]。现有的基于机器学习的传统特征提取方法配合分类器的算法是目前DCE-MRI影像中乳腺癌病理信息预测的重要方法之一[2],其人工提取特征的方法具有一定的局限性和主观性。而深度学习可以直接从影像中提取特征,很好的弥补了这一不足。深度学习(deep learning)可以直接提取影像特征[11],但深度学习通常需要重新设计并训练一个新的深度卷积网络,这需要耗费大量的时间资源和计算资源[12]。迁移学习(transfer learning)可以认为是在最小人工监督代价下进行机器学习的一种崭新策略[13]。迁移学习是运用大规模数据集中预训练好的网络模型,将该模型嵌入在其他任务模型中作为特征提取器,可以重用复杂的结构和优化的权重,大大节省了时间资源及计算资源。而且大规模图像数据集上的实验结果证明了预训练网络提取到的特征具有良好的判别性[14]。鉴于此,可以使用迁移学习去预测DCEMRI影像中乳腺癌病理信息。
值得说明的是,在乳腺癌病理信息预测研究中,以往通常采用单任务学习模型,将乳腺癌病理信息分析看成是单一分类任务。这种方法的缺陷在于没有考虑到利用不同任务间的相似性来提高模型的预测性能,而多任务学习方法正好弥补了这一缺陷。多任务学习是指在一个神经网络中同时完成2个相互关联的任务,利用不同任务之间的相似性来辅助决策,在图像处理或者语音处理任务中,多任务学习能够显著改进实验结果[15]。本文尝试使用多任务学习去联合预测乳腺癌病理信息。尽管多任务学习可以提高模型的预测性能,但是并非所有的任务都可以用多任务学习进行训练。如果任务不相关,采用多任务学习不能取得好效果,有时还会使模型准确度降低[16]。因此,使用多任务学习的前提是必须保证任务之间具有相关性。
本文提出了一种基于DCE-MRI影像的深度多任务学习联合预测乳腺癌病理信息算法。多任务学习是和单任务学习相对的一种方法。一般来说,标准的算法理论是一次学习一个任务,而多任务学习是一种联合学习,多个任务之间部分参数共享以达到相互学习的目的。在实验过程中,我们分别用单任务学习与多任务学习对乳腺癌病理信息进行预测,实验结果表明,利用深度多任务学习方法对于预测乳腺癌的病理信息,有较好的预测效果。
1 方法
1.1 患者病理信息数据
本研究采集的数据来自于浙江省中医院,经过数据筛选,最终采用202例病例作为本次研究的数据集。从浙江省中医院采集的病理报告中,可以得到每个病例的病理信息情况。数据集中的患者均为女性,年龄分布在29~83岁之间,平均年龄约为53岁,其中绝经前102例,绝经后100例。分子分型Luminal A型的病例共计38例,Luminal B型的病例共计100例,HER-2型的病例共计28例,Basal-like型的病例共计36例,组织学分级为Ⅰ级和Ⅱ级的病例共计86例,Ⅲ级的病例共计116例。Ki-67低表达的病例共计50例,Ki-67高表达的病例共计152例。
对组织学分级、Ki-67表达分别进行统计,卡方检验(Chi-Square Test)和方差分析(ANOVA)用于确定统计学显著性,计算得到的P值低于0.05时认为具有统计学意义。Ki-67对绝经情况、年龄和肿瘤最大径做卡方检验和方差分析,P值分别为0.1212,0.1043,0.4236,组织学分级对绝经情况、年龄和肿瘤最大径做卡方检验和方差分析,P值分别为0.9036,0.1396,0.263,说明Ki-67与组织学分级对绝经情况、年龄、肿瘤最大径不显著相关,Ki-67对Luminal A及组织学分级做卡方检验,P值均小于0.001,组织学分级对Ki-67及Luminal A做卡方检验,P值也均小于0.001,说明Ki-67、组织学分级及Luminal A型任意两个任务之间显著相关。
1.2 患者影像数据采集
本次研究所采用的医学影像来自浙江省中医院放射科,使用德国西门子3.0T的MRI扫描设备,采用专用的8通道双乳房线圈。所有乳腺MRI检查均采用俯卧位,DCE-MRI包括1例对比增强前序列,即蒙片系列(S0序列)和5例对比增强后序列(S1~S5)的双侧矢状面图像,采用脂肪抑制T1加权三维序列。DCE-MRI参数设置如下:重复时间(TR)为4.51 ms,回声时间(TE)为1.61 ms,矩阵为448×448,翻转角度(FA)为10°,视野(FOV)为340 mm×340 mm,层厚为1.0 mm,像素分辨率为0.759 mm×0.759 mm,以4 mL/s的速度静脉注射Gd-DTPA,剂量为注射造影剂60 s后采集第一个序列,随后按60 s的时间间隔获得5个连续的对比增强影像序列。
1.3 患者腺体影像分割
采用乳腺癌DCE-MRI进行影像研究,从采集的乳腺癌原始影像中分割出含肿瘤的腺体区域。对于DCE-MRI原始影像一般有144张切片,选取最大肿瘤切片对其进行研究分析。根据病理报告和影像,标注出含肿瘤的腺体区域。采用人工方法在原始影像中分割出标注区域,原始影像图片矩阵为448×448,去除大量黑色区域部分,在保证其像素分辨率不变的情况下将其变成224×224矩阵。最后用S1,S2,S3,S4,S5分别减去S0序列(蒙片序列),得到其减影序列分别用S0,S1,S2,S3,S4,S5表示。其具体实验步骤如图1所示。
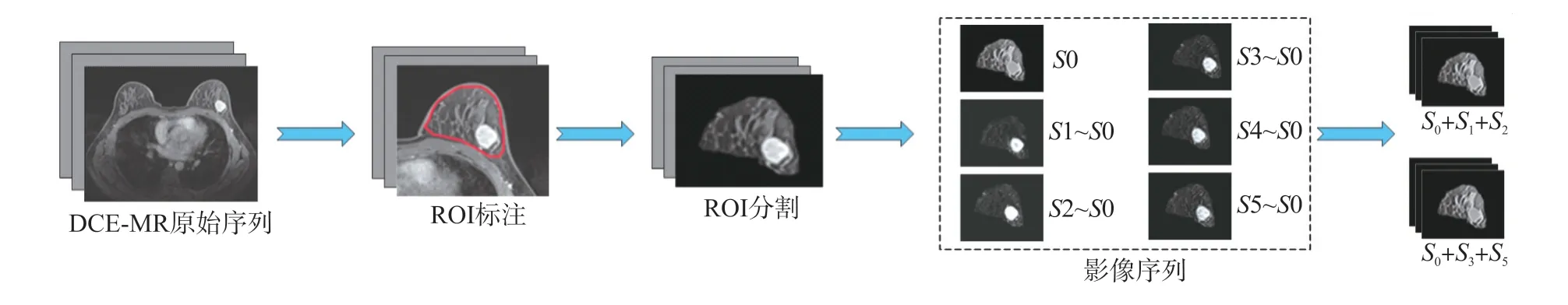
图1 腺体影像分割过程
1.4 腺体影像数据扩充
影像数据扩充的目的是将影像特征尽可能的分布在扩充的影像中,以增加深度学习的运算样本,以便获得更多影像的内在特征。首先将数据集随机划分为训练集与测试集,划分比例约为3∶2。故训练集为122例病例,测试集为80例。在对数据集进行扩充时,仅对训练集数据进行扩充,测试集数据保持不变。因为在训练集中样本分布不均匀,所以在对数据集进行扩充时对样本多的进行欠采样,对样本少的进行重采样。扩充的影像主要是通过对原始影像进行旋转和镜像得到的。在对原始影像预测其Ki-67的高低表达中,训练集中Ki-67高表达91例,低表达31例,对Ki-67高表达扩充得到的影像图片为776张,Ki-67低表达扩充得到的影像图片为752张;在对原始影像预测其分子分型是否为Luminal A型中,训练集中Luminal A型阳性23例,阴性99例,对阳性扩充得到的影像图片为736张,阴性扩充得到的影像图片为792张;在对原始影像预测其组织学分级是否为Ⅲ级中,训练集中组织学分级Ⅲ级69例,非Ⅲ级53例,对组织学分级Ⅲ级扩充得到的影像图片为720张,组织学分级非Ⅲ级扩充得到的影像图片为808张。
1.5 基于迁移学习的特征提取
对上文进行预处理后的影像进行特征提取。在乳腺癌病理信息预测过程中,采用深度卷积神经网络直接从DCE-MRI中提取特征,由于训练一个完整的深度卷积神经网络模型需要消耗大量的时间资源与计算资源。本文引入了迁移学习的方法,通过迁移在大数据集ImageNet上预训练好的模型权重参数[17],并重新设计其分类层,从而减少模型收敛拟合时间,生成性能优异的特征提取模型。在进行迁移学习过程中,保存其分类层前特征提取的网络结构与模型参数迁移到新的神经网络。假设迁移结构层数为n层,冻结前m层即前m层的模型参数永远保持不变,而后n-m层需要针对新的数据集进行权重训练。
首先利用VGG16卷积神经网络进行迁移学习提取影像特征。VGG卷积神经网络模型在图像分类和目标检测任务中都表现出非常好的结果[18]。VGG16是VGG卷积神经网络的一种,我们选择其全连接前特征提取网络结构作为迁移层,其网络结构如图2所示。VGG16输入的影像尺寸为224×224×3,前两个224表示图片的长和宽,3表示通道数,输出的特征尺寸为7×7×512。
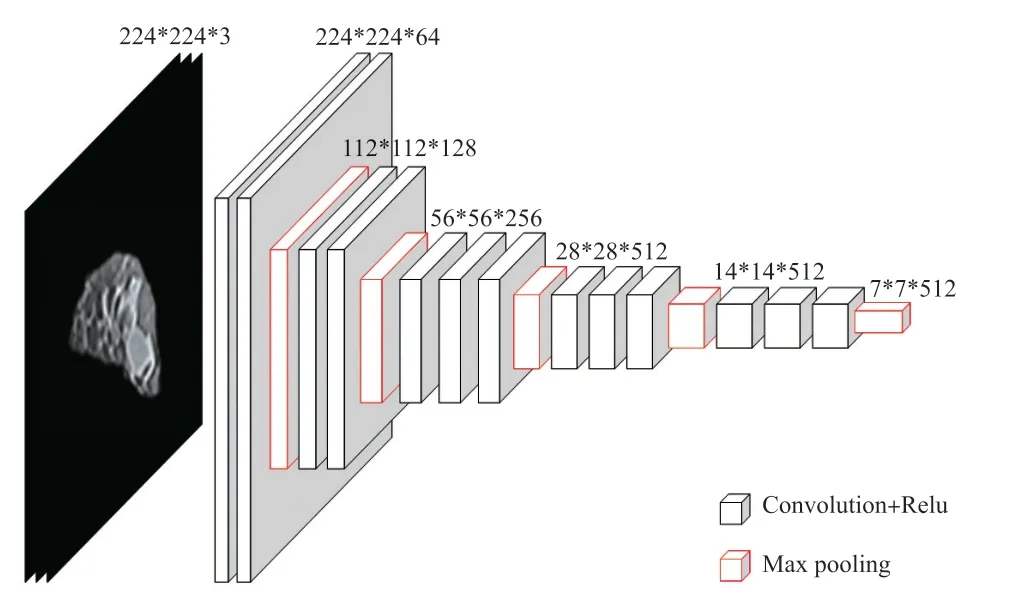
图2 VGG16网络框架图
残差网络与DenseNet因其特有的“短路连接”在图像分类上被普遍使用,本文用到的迁移网络有ResNet50和DenseNet121,ResNet50输入的图片尺寸为224×224×3,输出特征尺寸为7×7×2048。DenseNet121输入的图片尺寸为224×224×3,输出特征尺寸为7×7×1024。
1.6 基于深度学习的病理信息预测模型
对上文提取的特征进行病理信息预测。通过迁移学习提取出的特征维度较高,首先我们使用全局平均池化将特征降为一维,然后我们运用全连接层逐级降维,为了解决深度神经网络容易过拟合的问题,我们引入了dropout[19]层。针对于单任务学习及多任务学习我们设计了不同的病理信息预测模型。单任务学习病理预测模型如图3所示,多任务学习病理信息模型如图4所示。多任务学习方法除了共享特征提取层,还在分类层中引入了共享层,即在分类时有一部分特征是两个任务都拥有的。其中任务A与任务B的维数始终相同,且任务A、共享层及任务B的总维数始终为400,共享层的维数为超参数可调。单任务学习和多任务学习都在FC1后面及FC2后面引入dropout层。
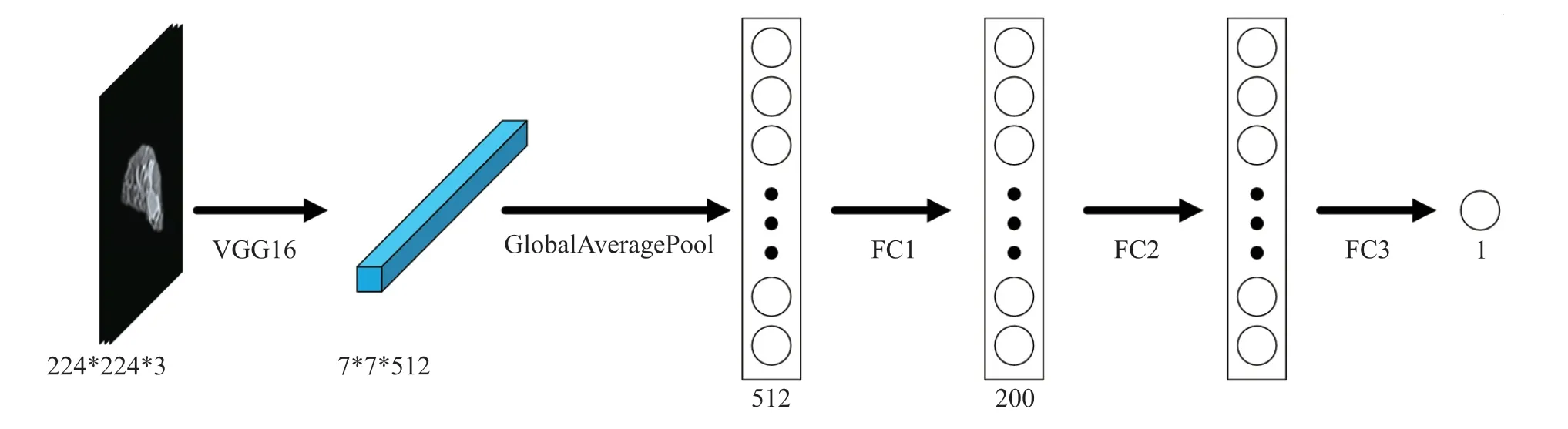
图3 单任务学习病理信息预测模型
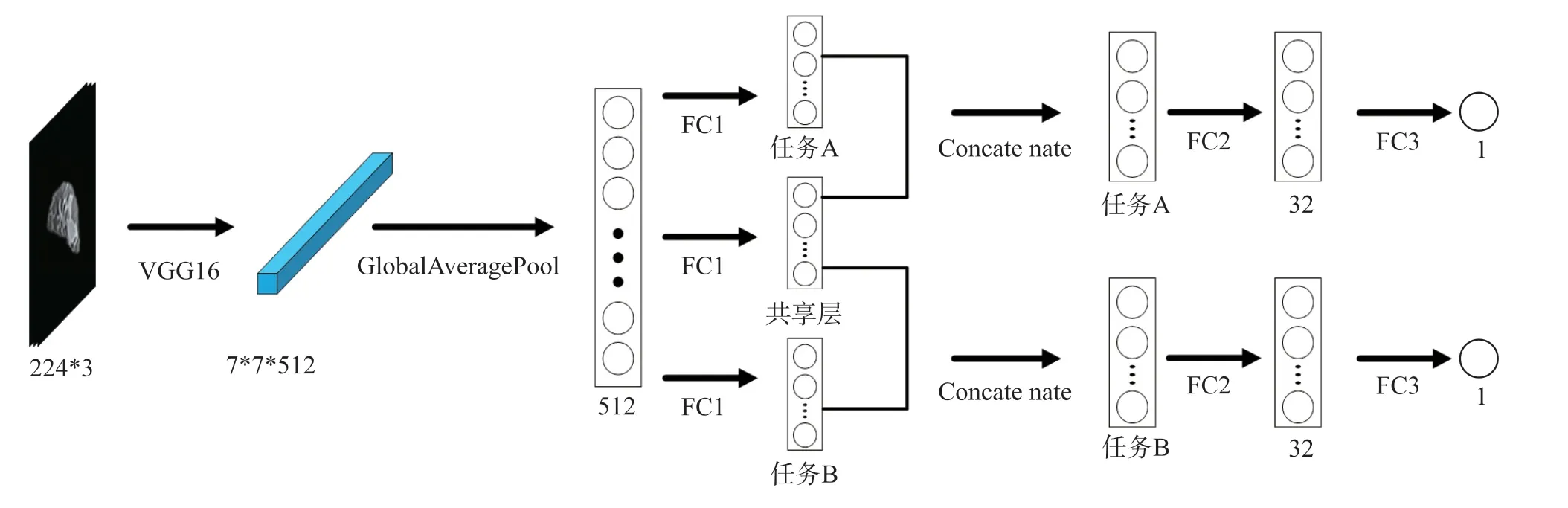
图4 多任务学习病理信息预测模型
2 结果
2.1 基于单任务学习的单序列影像病理信息预测
DCE-MRI具有6个序列,对6个单序列影像分别进行单任务学习,并建立病理信息预测模型。对各个单序列影像进行预测分析,通过基于VGG16的迁移学习预测乳腺癌的病理信息,实验结果如表1所示。通过对表1分析可知,对于预测Ki-67表达任务,S0序列表现最优,预测Ki-67的AUC值为0.752。对于预测Luminal A型任务,S0序列表现最优,预测Luminal A型的AUC值为0.722。对于预测组织学分级任务,S3序列表现最优,预测组织学分级的AUC值为0.695。
2.2 基于单任务学习的多序列影像病理信息预测
从DCE-MRI影像中选取3个减影序列组成多序列影像,对多序列影像进行单任务学习,并建立病理信息预测模型。首先我们选择连续的3个序列S0,S1,S2作为输入的多序列影像。为防止偶然性,我们把6个序列分为高中低3份,分别在3份中随机选择1个序列,最终选择了S0,S3,S5作为输入的多序列影像作为补充。对多序列影像进行预测分析,通过基于VGG16的迁移学习预测乳腺癌的病理信息,实验结果如表2所示。
通过对表2分析可知,对于预测Ki-67,Luminal A型及组织学分级任务,S0+S1+S2多序列影像分别对应的AUC值为0.793,0.730,0.715。S0+S3+S5多序列影像分别对应的AUC值为0.772,0.729,0.712。均优于最优单序列影像对应的AUC值0.752,0.722,0.695。
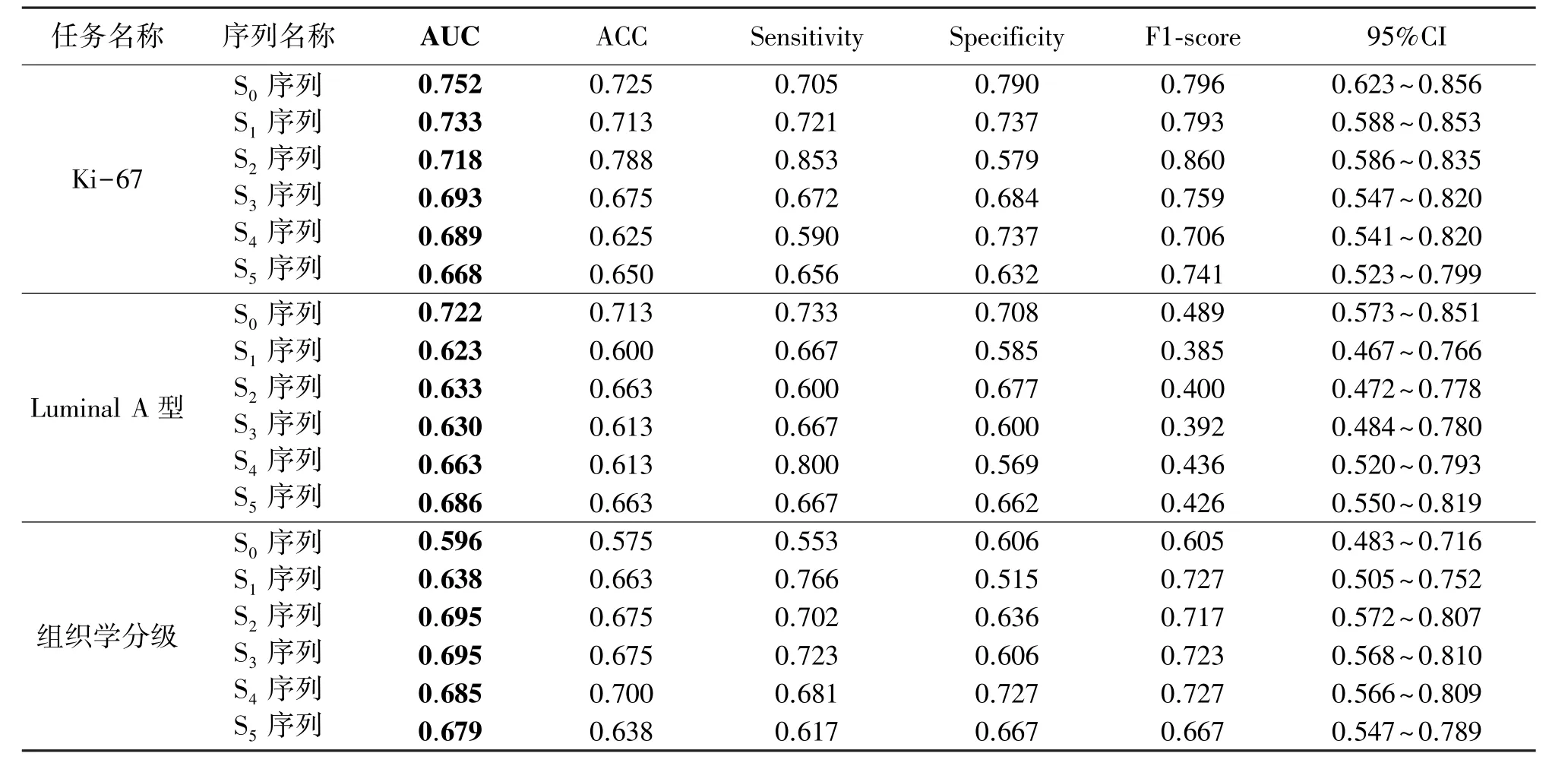
表1 基于单任务学习单序列影像病理信息预测
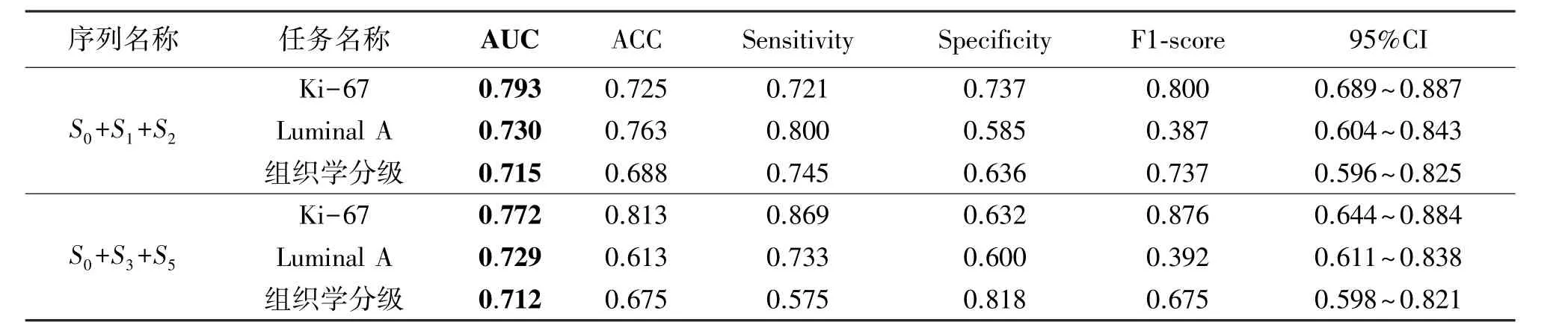
表2 基于单任务学习多序列影像病理信息预测
2.3 基于CNN的多序列影像病理信息预测性能比较
对多序列影像采用不同的卷积神经网络对其进行特征提取,对提取的特征进行单任务学习,分别建立基于不同卷积神经网络的病理信息预测模型。残差网络与DenseNet因其特有的“短路连接”在图像分类上被普遍使用,本文主要选择了VGG16,Res-Net50和DenseNet121进行迁移,因多序列影像效果优于单序列影像,故选择多序列影像作为迁移学习的输入图像。实验结果如表3所示。
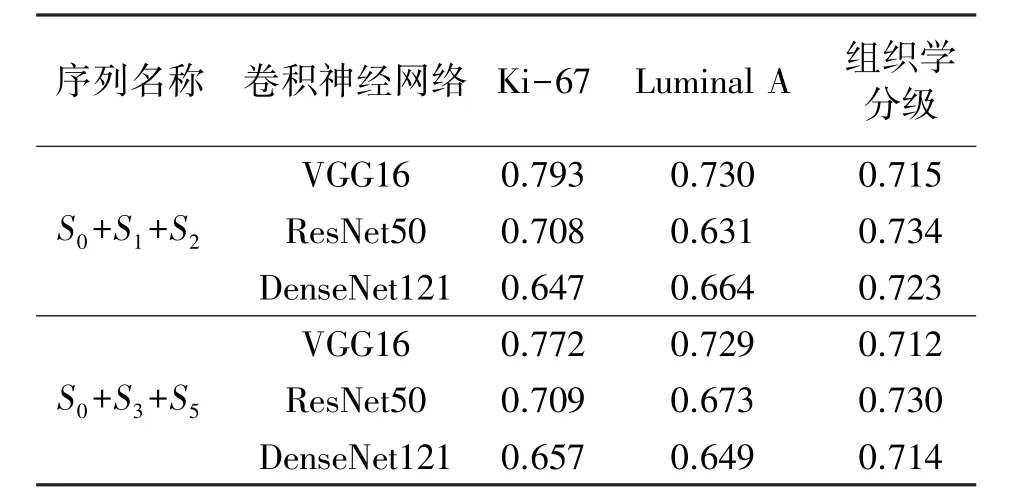
表3 基于CNN多序列影像预测比较(AUC)
通过对表3分析可知,对于预测组织学分级任务,ResNet50与DenseNet121稍高于VGG16,对于预测Luminal A 与 Ki-67任务,ResNet50与DenseNet121低于VGG16,综合考虑三个任务,因此本文最终选择VGG16作为最终的迁移网络。
2.4 基于多任务学习的多序列影像病理信息预测
对多序列影像进行多任务学习,并建立病理信息预测模型。通过上文的数据分析可知,Ki-67表达、Luminal A型与组织学分级之间任意两个任务显著相关,分别对其进行深度多任务学习联合预测,S0+S1+S2多序列影像作为输入影像,其实验结果如表4所示。通过对表4分析可知,多任务学习的病理信息预测结果优于单任务学习。
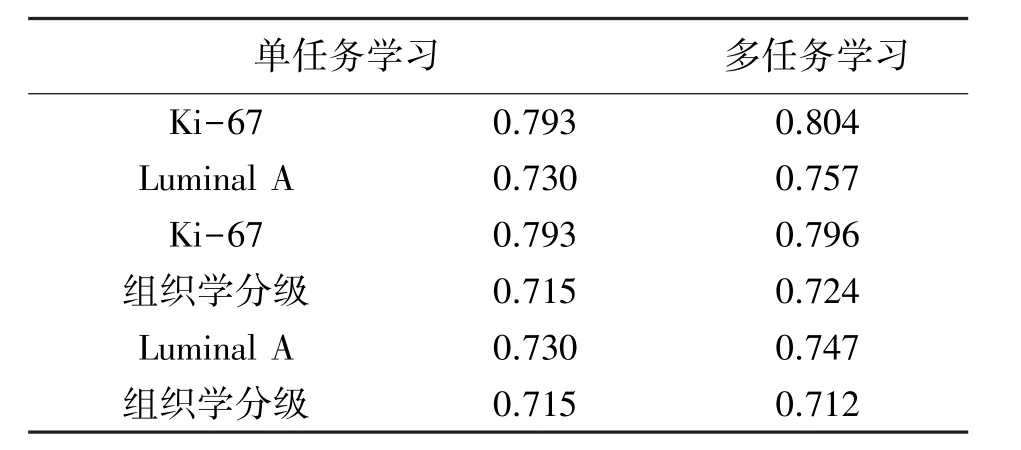
表4 多序列影像单任务学习与多任务学习比较(AUC)
2.5 单任务学习与多任务学习预测模型比较
对上文所示单任务单序列、单任务多序列以及多任务多序列的病理信息预测模型进行比较,其预测Ki-67表达,分子分型和组织学分级结果的ROC曲线,如图5所示。由图5可知,在单任务学习中,多序列影像相比于单序列影像具有更好的性能。而对比单任务学习,多任务学习的ROC曲线相比而言更偏向于左上方,说明多任务学习方法具有更好的分类性能。综上所述,多任务学习方法相比于单任务学习方法可以提高预测乳腺癌病理信息的性能。
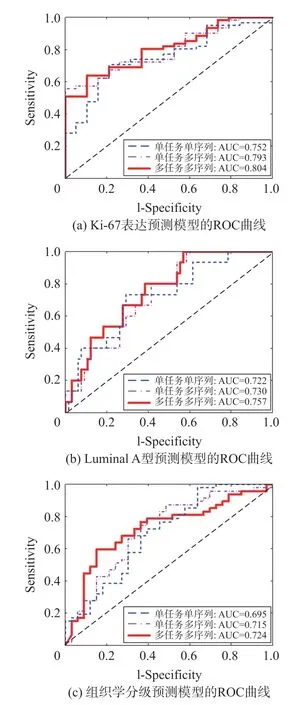
图5 病理信息模型预测比较
3 讨论
在本研究中,采用深度多任务学习的方法来预测DCE-MRI影像中乳腺癌常见的病理信息指标,包括Ki-67表达,Luminal A型及组织学分级。对采集的DCE-MRI影像分割出含肿瘤一侧的腺体区域,得到其6个减影序列,并对分割后的减影序列进行裁剪。首先将数据集随机分为训练集与测试集,并将训练集的数据进行数据扩充,测试集数据保持不变。其次构建基于迁移学习的预测模型分别提取单序列影像以及由随机3个序列组成的多序列影像的高维特征,随后,采用单任务学习方法以及特征共享的多任务学习方法分别对基于Ki-67表达与Luminal A型、基于Ki-67表达与组织学分级、基于Luminal A型与组织学分级进行预测,并计算AUC评估单序列影像预测模型、多序列影像预测模型以及多任务影像预测模型的性能。结果表明,采用多序列影像,可以提高单序列影像模型预测乳腺癌病理信息的性能,采用多任务学习,可以提高单任务学习影像模型预测乳腺癌病理信息的性能。
基于深度多任务学习预测DCE-MRI影像乳腺癌病理信息的研究是一个较新的探索领域,Tian等[20]设计了交通场景中完成主体识别任务算法,通过多任务学习提高了主体识别的准确率和召回率。范正光等[21]使用深度多任务学习方法,建立声学模型,能够高效的完成语音识别任务。但深度多任务学习很少应用在DCE-MRI影像中。本课题组的前期研究显示,通过深度卷积神经网络对乳腺癌分子分型预测的受试者的DCE-MRI影像进行分析,有一定的预测效果[22]。采用机器学习,DCE-MRI和DWI通过多任务学习可以提高Ki-67表达和分级的预测能力[23-24]。笔者首次尝试使用深度多任务学习的方法应用于DCE-MRI影像乳腺癌Ki-67表达,Luminal A型与组织学分级预测的研究中,预测性能基本达到预期效果。
本研究存在一定的局限性:一是数据样本规模较小,在未来的研究中可以采用大型独立的多参数影像数据集,对预测模型的鲁棒性进行进一步的验证;二是在对Ki-67表达、Luminal A型以及组织学分级3个任务进行预测时,只进行基于2个任务的多任务学习研究,后续会进一步进行基于3个任务的多任务学习研究。
4 结论
本文提出了基于DCE-MRI影像的深度多任务学习的乳腺癌病理信息预测算法。实验结果表明,使用深度多任务学习,可以对乳腺癌的前期诊断和个性化临床治疗方案的选择提供有价值的参考。在后续研究中,会进一步研究基于Ki-67表达,Luminal A型,组织学分级的多任务学习的研究,并且引入更多显著性相关的任务,增加该技术在临床上的实用性。
