革兰氏阳性与阴性细菌基因组DNA提取方法的优化
2021-06-15云飞梁林鲍彦彬石雅丽孙亚超李永丽兰辉
云飞 梁林 鲍彦彬 石雅丽 孙亚超 李永丽 兰辉
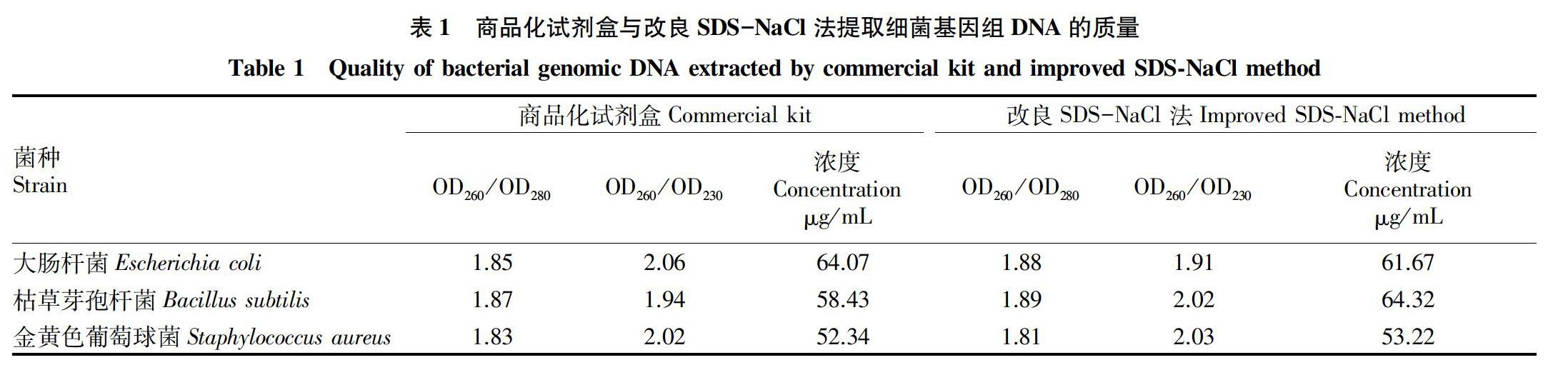
摘要 在分析现有提取微生物基因组DNA的原理和方法基础上,对SDS-NaCl法进行了改良,以商品化微生物基因组提取试剂盒提取的基因组结果为对照,利用改良的SDS-NaCl法,分别提取大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)的基因组DNA。结果表明,改良的SDS-NaCl法提取的大肠杆菌、枯草芽孢杆菌和金黄色葡萄球菌基因组DNA的 OD260/OD280分别为1.88、1.89、1.81,浓度分别为61.67、64.32、53.22 μg/mL;而商品化试剂盒提取大肠杆菌、枯草芽孢杆菌和金黄色葡萄球菌基因组DNA OD260/OD280分别为1.85、1.87、1.83,浓度分别为64.07、58.43 、52.34 μg/mL。结果证明改良SDS-NaCl法提取的革兰氏阳性和阴性细菌的基因组DNA可用于后续试验如全基因组测序和PCR等,同时可快速获得大量的高质量微生物基因组。
关键词 细菌基因组DNA;革兰氏阳性细菌;革兰氏阴性细菌;SDS-NaCl法
中图分类号 Q933文献标识码 A文章编号 0517-6611(2021)10-0098-03
doi:10.3969/j.issn.0517-6611.2021.10.026
开放科学(资源服务)标识码(OSID):
Optimization of Methods for Genomic DNA Extraction from Bacteria
YUNFei1,LIANG Lin2,BAO Yan-bin2 et al
(1. Department of Mining Technology, Inner Mongolia University of Technology, Hohhot, Inner Mongolia 010051;2. School of Chemical Engineering and Technology, Inner Mongolia University of Technology, Hohhot, Inner Mongolia010051)
Abstract Based on the analysis of the principles and methods of genomic DNA extraction, the SDS-NaCl method was modified. The results of genomic DNA extraction by commercial genomic extraction kit were compared with those by SDS-NaCl method modified by our laboratory. The results showed that the OD260/OD280 of the genomic DNA of Escherichia coli, Bacillus subtilis and Staphylococcus aureus were 1.88, 1.89 and 1.81, and the concentration were 61.67,64.32and 53.22 μg/mL respectively.The OD260/OD280 by commercial genomic extraction kit were 1.85, 1.87 and 1.83, and the concentration were 64.07,58.43 and 52.34 μg/mL respectively. The results showed that the genomic DNA extracted by the modified nacl method could be used in subsequent experiments such as whole genome sequencing and PCR, and a large number of high quality microbial genomes could be obtained rapidly.
Key words Genomic DNA;Gram positive bacterium;Gram negative bacterium;SDS-NaCl method
目前,随着高通量测序技术日益成熟,微生物全基因组DNA的完成图测序和重测序已成为微生物分子生物学与代谢组学研究中的重要手段,其中微生物基因组DNA的质量对测序结果影响很大。
目前,提取微生物基因组DNA的方法主要有液氮研磨法[1-4]、冻融法[5]、加热煮沸法[6]、碱裂解法、溶菌酶法[7-9]、SDS(十二烷基磺酸钠)法[6,10]、CTAB(十六烷基三甲基溴化铵)法[10]、Chelex-100煮沸法[11]、吸附柱法[12]、磁珠法[13-14]。
SDS-NaCl法具有裂解細菌彻底和提取基因组DNA纯度较好的特点。笔者利用改良的SDS-NaCl法,分别提取大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)的基因组DNA(其中大肠杆菌是革兰氏阴性菌,枯草芽孢杆菌和金黄色葡萄球菌是革兰氏阳性菌),同时以商品化细菌基因组提取试剂盒提取结果为对照,通过与商品化试剂盒提取效果进行比较,获得一种操作简单、成本低廉,且可快速获得较高质量的革兰氏阳性和阴性细菌的基因组DNA。
1 材料与方法
1.1 菌种与试剂
菌种来自内蒙古工业大学基因工程实验室保存的大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)。
细菌基因组DNA提取试剂盒购买于北京天根生化科技有限公司。蛋白酶K、溶菌酶、SDS、苯酚、氯仿和无水乙醇均为国产试剂。
1.2 试验方法
1.2.1 商品化试剂盒的提取。
提取步骤参见北京天根生化科技有限公司细菌基因组DNA试剂盒说明书。
1.2.2 改良的SDS-NaCl法。
将细菌在LB培养基中(酵母提取物5 g,胰蛋白胨10 g,NaCl 10 g,1 000 mL去离子水,用5 mol/L的NaOH调节其pH至7.0,121 ℃蒸汽灭菌20 min)培养8~10 h后,取1 mL新鲜菌液放入离心管,10 000 r/min离心1 min后尽可能除净上清;加入200 μL溶菌酶,悬浮菌体;加入10 μL RNaseA轻轻混匀;加入20 μL蛋白酶K(20 mg/mL),室温静置1 min;然后加入500μL的裂解液(100 mmol/L pH 7.0 Tris-HCl,500 mmol/L pH 8.0 EDTA,20 mmol/L NaCl,10%SDS),50 ℃温浴20 min;加入300 μL的苯酚,上下混匀;再加入氯仿300 μL,混匀;10 000 r/min,5 min;转移上清液至新离心管;加入300 μL的氯仿,混匀,离心10 000 r/min,5 min,转移上清液到新离心管;加入1 mL无水乙醇,在-20 ℃中沉淀10 min;离心10 000 r/min,15 min,弃上清液;加入1 mL 70%的乙醇洗涤,离心10 000 r/min,5 min,弃上清液;在室温下干燥15 min;然后加入30 μL ddH2O。取1 μL DNA溶液进行琼脂糖凝胶电泳,同时通过紫外分光光度计检测浓度。
2 结果与分析
2.1 商品化试剂盒提取的3种细菌基因组DNA
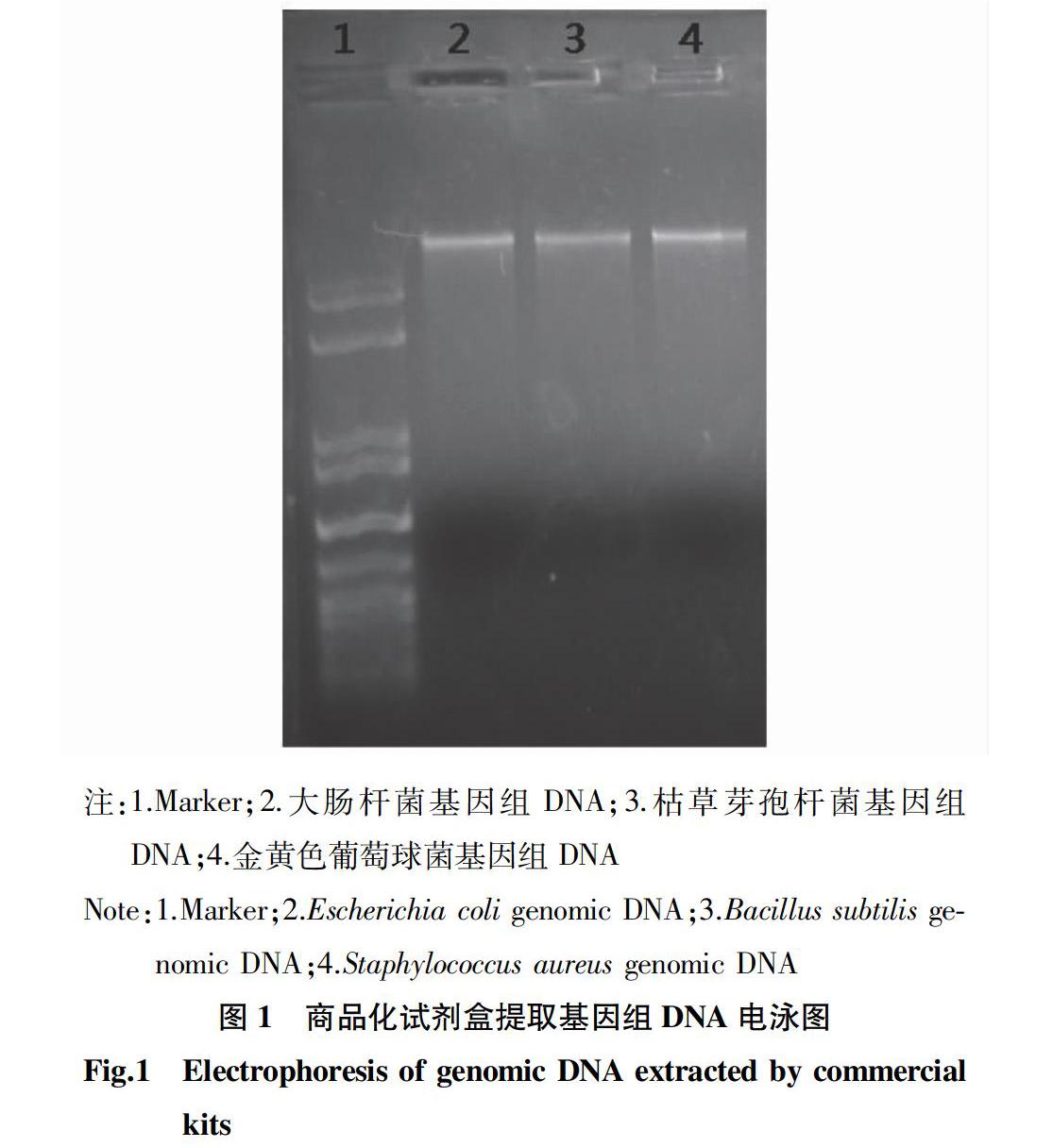
利用商品化试剂盒提取大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)的基因组DNA,经凝胶电泳后检测DNA的质量,结果见图1。从图1可以看出,3种细菌的基因组DNA经电泳后条带整齐清晰,无蛋白和RNA残留,DNA质量较好。
2.2 改良SDS-NaCl法提取的3种细菌基因组DNA
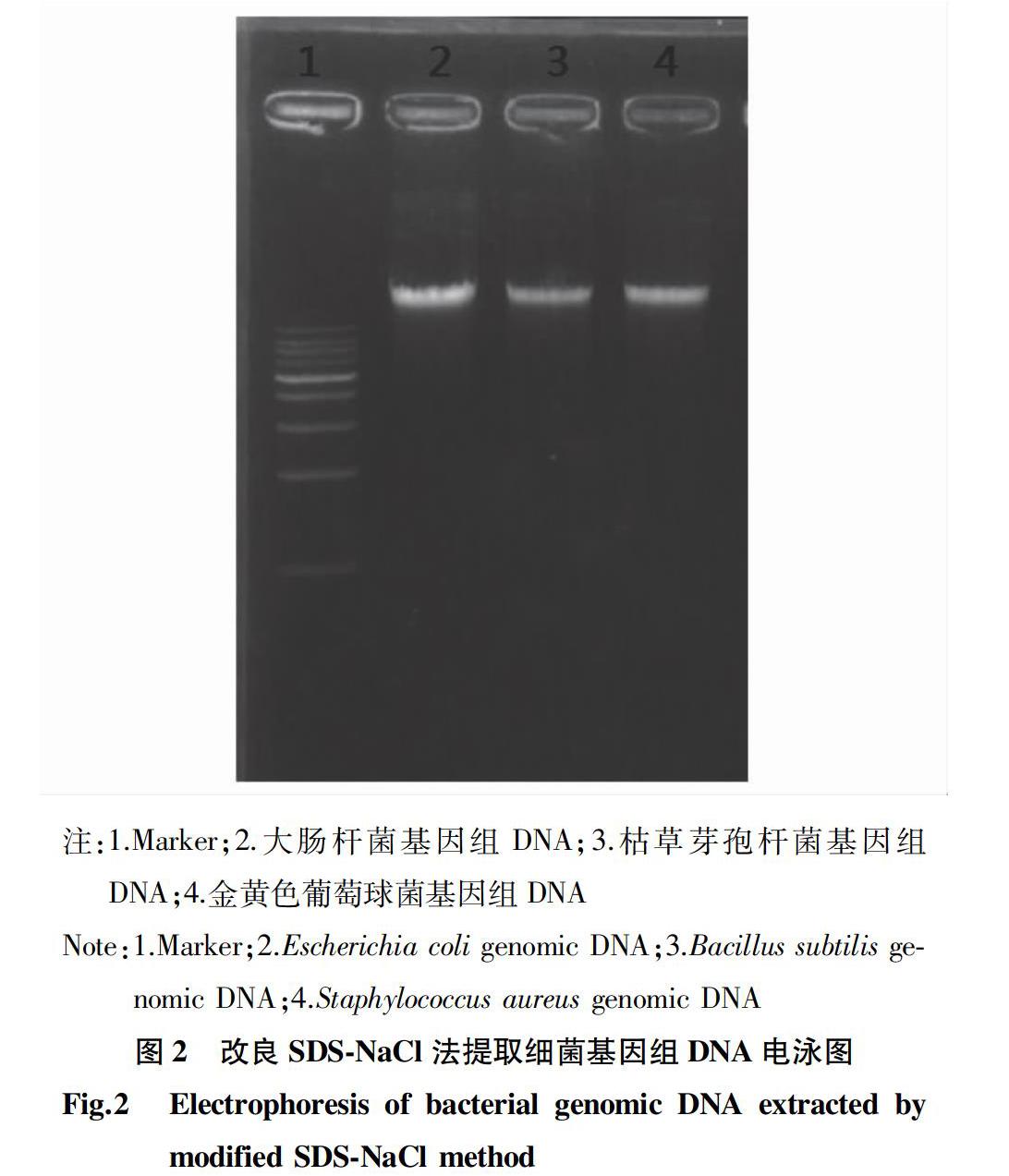
利用改良SDS-NaCl法提取大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcusaureus)的基因组DNA,经凝胶电泳后检测DNA的质量,结果见图2。从图2可以看出,3种细菌的基因组DNA经电泳后条带整齐清晰,与商品化试剂盒提取结果相比,DNA条带较
粗,说明改良SDS-NaCl法提取DNA的量更多,DNA的质量更好。
2.3 基因组DNA 的浓度及纯度
根据紫外分光光度法结果(表1)分析这2种方法提取细菌基因组DNA的质量,商品化试剂盒法提取大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)基因组DNA的OD260/OD280分别是1.85、1.87和1.83,OD260/OD230分别是2.06、1.94和2.02,浓度分别是64.07、58.43和52.34 ng/mL;改良SDS法提取的大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)和金黄色葡萄球菌(Staphylococcus aureus)基因组DNA的OD260/OD280分别是1.88、1.89和1.81,浓度分别是61.67、64.32和53.22 ng/mL。结果表明,经改良的SDS 法提取的3种细菌基因组 DNA 纯度和浓度均较好(图3),且操作步骤简单,成本低,可用于大量提取细菌基因组DNA。
3 结论
微生物基因组提取是进行微生物分子生物学与代谢分析的基本操作,基因组的质量与数量直接影响下游试验的结果,因此根据试验样品的特点对微生物基因组提取方法进行不断改良是必需的,这样可以有效提高试验结果的准确性与可信度。如对于特殊环境微生物基因组的提取,如瘤胃微生物等,就必须对试验方法进行改良。该研究将目前常用的微生物基因组提取方法进行比较,同时将革兰氏阳性细菌与阴性细菌的基因组提取特点进行比较,以便今后对于特殊环境微生物基因组进行提取方法的摸索与改良。
参考文献
[1] ZHAO X M,DUSZYNSKI D W,LOKER E S.A simple method of DNA extraction for Eimeria species [J].Journal of microbiological methods,2001,44(2):131-137.
[2] 刘国兴,刘源涌,张辉,等.东北梅花鹿瘤胃微生物基因组的DNA提取[J].黑龙江畜牧兽医,2015(11):227-229.
[3] 刘水平,罗志勇.分子生物学实验中使用液氮研磨组织的方法[J].实验教学与仪器,2005,22(3):29.
[4] 刘薇,方国庆,刘立成,等.奶牛瘤胃微生物DNA提取法的研究与优化 [J].中国畜牧杂志,2011,47(13):71-75.
[5] 馮唐锴,赵文亮.细菌基因组DNA提取方法简化的研究[J].景德镇学院学报,2014,29(6):42,47.
[6] 高秋月,景奉香,李海燕,等.基于磁珠的细菌基因组DNA快速提取方法[J].安徽农业科学,2010,38(21):11071-11074.
[7] 常健,王琪.晋西北酸粥发酵过程中微生物基因组DNA提取方法的比较[J].食品工业科技,2016,37(2):209-212,233.
[8] 戴方伟,宋晓明,周莎桑,等.大鼠肠内容物细菌基因组DNA提取方法的比较[J].中国实验动物学报,2011,19(3):246-249.
[9] 夏涵,府伟灵,陈鸣,等.快速提取细菌DNA方法的研究[J].现代预防医学,2005,32(5):571-573.
[10] 刘晓侠,林建平,岑沛霖.微生物基因组DNA提取方法的比较与改进[J].嘉兴学院学报,2007,19(3):48-50.
[11] 陶兴玲,雷琼,马立安.一种快速提取土壤微生物DNA的方法[J].长江大学学报(自科版),2018,15(2):54-58.
[12] PANOVA M,ARONSSON H,CAMERON R A,et al.DNA extraction protocols for whole-genome sequencing in marine organisms[J].Methods in molecular biology,2016,1452:13-44.
[13] OLSON N D,MORROW J B.DNA extract characterization process for microbial detection methods development and validation[J].BMC Research Notes,2012,5(1):668.
[14] ZHOU J Z,BRUNS M A,TIEDJE J M.DNA recovery from soils of diverse composition [J].Applied & environmental microbiology,1996,62(2):316-322.
