混合Filter与改进自适应GA的特征选择方法
2021-06-11邱云飞高华聪
邱云飞,高华聪
辽宁工程技术大学 软件学院,辽宁 葫芦岛125100
特征选择是指为了降低数据维度,在保证特征集合分类性能的前提下,从原始特征集合中选出具有代表性的特征子集。对于特征选择方法,按照分类器在算法选择特征过程中的参与方式进行分类,可将其分为三类:过滤式(Filter)、包装式(Wrapper)和嵌入式(Embedded)。过滤式的特征选择方法先对初始特征进行过滤,再用过滤后的特征训练模型,所以过滤式方法有计算量小、易实现的优点,但分类精度较低;包装式的特征选择方法由于在其特征选择过程中需要多次训练分类器,计算开销通常比过滤式特征选择要大得多,但分类效果要好于过滤式;嵌入式特征选择在分类器训练过程中将自动地进行特征选择,利用嵌入式特征选择,分类效果明显但参数设置复杂且时间复杂度较高。
遗传算法(GA)是一种基于种群的迭代的元启发式优化算法,对初始化个体通过算法的编码技术和一些基本的遗传算子选择、交叉、变异等操作,依据个体的适应度值进行选择遗传,经过迭代,得到适应度最高的个体[1]。遗传算法以生物进化为原型,具有良好的全局搜索能力,可以快速地将解空间中的全体解搜索出,并且利用它的内在并行性加快求解速度。但在实际应用中,遗传算法容易产生早熟收敛的问题,采用何种选择方法既要使优良个体得以保留,又要维持群体的多样性,一直是遗传算法中较难解决的问题。目前,基于遗传算法的特征选择方法受到了广泛关注:Ferriyan等人[2]改进两点交叉,将一点交叉用于遗传算法参数,使遗传算法运行速度更快;An[3]提出了改进的遗传算法应用于特征选择,将多种群并行和竞争策略引入遗传算法;Vaishali等人[4]提出在预处理阶段利用遗传算法从数据集中选取基本特征,并在数据集上应用多目标进化模糊分类器以获得较高分类精度。以上针对遗传算法的研究中没有兼顾特征子集和分类精度,所以本文提出了一种改进的自适应遗传算法,在选择操作中通过融合选择方式保证了种群的多样性和准确性,同时把特征数加入到评价函数中,设置参数平衡特征子集和分类精度,并采用自适应的形式动态调整交叉和变异概率,解决遗传算法陷入局部最优的问题,使算法准确快速收敛到最优解。
Relief[5]算法是一种简单高效的过滤式特征选择方法,但局限于解决二分类问题。随后Robnik-Šikonja等人对其进行扩展,得到了ReliefF算法[6]。ReliefF算法主要用于解决多分类和数据缺失、冗余等问题。ReliefF系列算法运行效率高、稳定性好,对数据类型没有限制,属于特征权重算法,算法会赋予所有和类别相关性高的特征较高的权重,但算法的局限性在于不能有效地去除冗余特征。
互信息指测量两个随机数据样本之间的相关性。通常,互信息描述数据集之间的依赖程度。如今,互信息已经广泛应用于特征提取的研究中,Sharmin等人[7]将MI作为一种度量标准,用于创建一个基于x检验的同时选择特征和离散化的框架;王金杰等人[8]提出一种基于混合互信息和粒子群算法的过滤式-封装式的多目标特征选择方法,能够更好地降低特征个数和分类错误率。
混合方法结合了两种或多种经过充分研究的算法,形成了解决特定问题的新策略。混合方法通常利用子算法的优势,因此与传统方法相比更加稳健。Zhang等人[9]提出了一种混合的过滤器和包装器方法,使用自举策略创建了特征的子集,计算每个特征子集的分类精度以找到优化的子集;Thejas等人[10]提出一种过滤器和包装器相结合的方法,将Kmeans聚类与标准化互信息混合并利用随机森林的贪婪搜索方法得到最优特征集;马超[11]提出了结合ReliefF和改进乌鸦搜索算法的特征选择算法,从而获得特征子集。
上述特征选择算法虽然都取得了一定的成果,但都没有快速地消除冗余特征,在一定程度上降低了分类算法的性能。因此,考虑到ReliefF算法高效的计算效率、互信息的去冗余和遗传算法较优的全局搜索能力,本文提出了混合Filter模式与改进自适应GA的特征选择方法(ReFS-AGA)。该方法采用过滤式与封装式混合的特征选择模型,先用ReliefF算法设置阈值去除权重较低的特征;然后应用归一化互信息,修正传统互信息多特征值敏感问题并消除冗余特征;最后利用改进的自适应遗传算法迭代进化,融合选择算子,并设计新的评价函数,引入特征数和分类精度从而得到最优特征子集。
1 基于Filter模式的特征选择
1.1 ReliefF算法
Relief算法是一种著名的过滤式特征选择方法,会根据各个特征和类别的相关性赋予特征不同的权重,权重小于某个阈值的特征将被移除。算法从训练集D中随机选择一个样本R,然后从和R同类的样本中寻找最近邻样本H(Near Hit),从和R不同类的样本中寻找最近邻样本M(Near Miss),并根据以下规则更新每个特征的权重:如果R和Near Hit在某个特征上的距离小于R和Near Miss上的距离,则说明该特征对区分同类和不同类的最近邻是有益的,则增加该特征的权重;反之,如果R和Near Hit在某个特征的距离大于R和Near Miss上的距离,说明该特征对区分同类和不同类的最近邻起负面作用,则降低该特征的权重。以上过程重复m次,最后得到各特征的平均权重。特征的权重越大,表示该特征的分类能力越强。Relief算法的运行时间随着样本的抽样次数m和原始特征个数N的增加而线性增加,因而运行效率非常高。
假设一个样本X有p个特征,S为样本量n的训练样本集,F即{f1,f2,…,fp}为特征集,一个样本X由p维向量(x1,x2,…,xp)构成,其中xj为X的第j个特征值。
Relief算法可以解决名义变量和数值变量,两个样本X和Y的特征值的差可由下面的函数来定义。
当xk和yk为名义变量时,
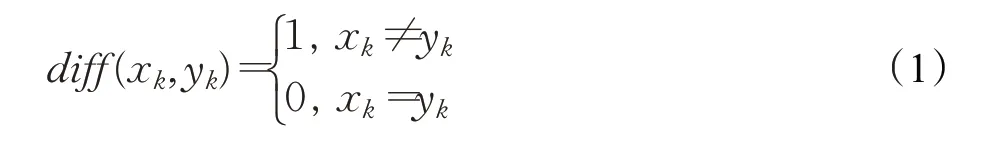
当xk和yk为数值变量时,

νk为归一化单位,把diff值归一化到[0,1]区间权重更新公式为:
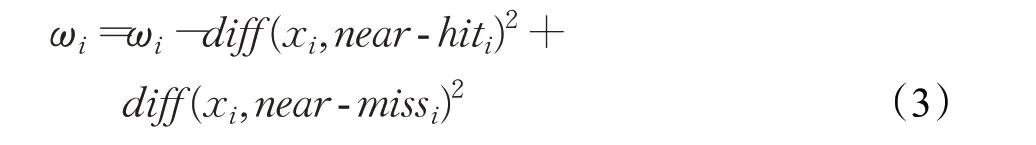
由于Relief算法比较简单,但运行效率高,并且结果也比较令人满意,因此得到广泛应用,但是其局限性在于只能处理两类别数据,因此后人对其扩展得到了可以处理多类别问题的ReliefF算法。该算法在处理多类问题时,每次从训练样本集中随机取出一个样本R,然后从和R同类的样本集中找出R的k个近邻样本(Near Hit),从每个R的不同类的样本集中均找出k个近邻样本(Near Miss),然后更新每个特征的权重,如下式所示:
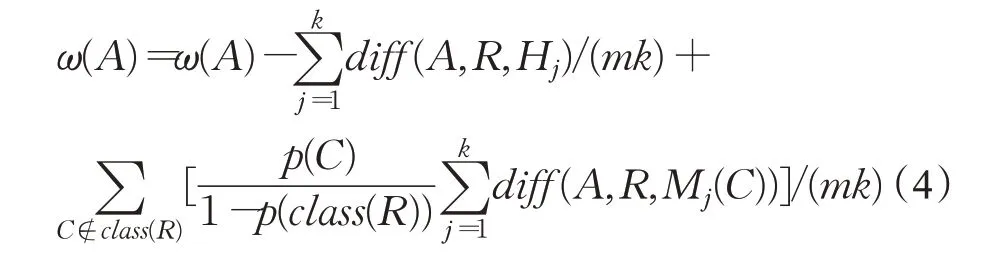
式中,diff(A,R,Hj)表示样本R1、R2在特征A上的差,Mj(C)表示类C∉class(R)中第j个最近邻样本,如公式(5)所示:

算法1reliefF算法流程
输入:训练集D,抽样次数m,特征权重阈值δ,最近邻样本个数k。
输出:各个特征的特征权重T。

1.2 互信息
在信息论中,熵用来衡量一个随机事件的不确定性。假设对于一个离散随机变量X(取值集合为S,X的概率密度分布函数为p(x),x∈S)进行编码,自信息I(x)是变量X=x时的信息量,定义为:

那么随机变量X的熵定义为:

熵越高,则说明随机变量的信息越多;反之,熵越低,则说明随机变量的信息越少。
在得知某一确定信息的基础上获取另外一个信息时所获得的信息量称为条件熵。对于两个离散随机变量X和Y,假设X取值集合为S,Y取值集合为T,其联合概率分布满足p(x,y),则在Y条件下X的条件熵为:
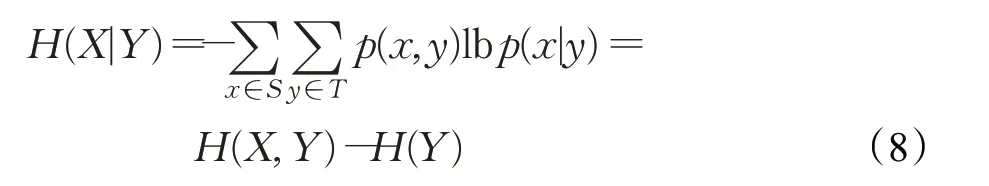
互信息(Mutual Information)是衡量已知一个变量时,另一个变量不确定性的减少程度,可以描述两个随机变量之间的共有信息量。互信息如公式(9)所示:
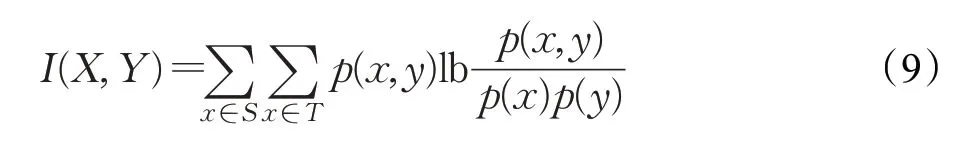
其中,p(x)是变量x的概率密度,p(y)是变量y的概率密度,p(x,y)是联合概率密度。
互信息越大说明两个随机变量之间的相关程度越高。公式(9)在对变量的计算时倾向于选择多值特征。因此本文用对应的熵做分母对其进行归一化,将值限制在[0,1]之间,SU(X,Y)值越接近1,表示两个变量之间相关性越高,当其值为0时,表示这两个变量是不相关的,由此得到归一化互信息(Symmetrical Uncertainty),其公式为:
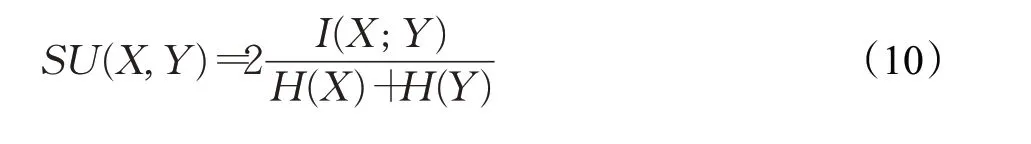
2 基于自适应遗传算法的特征选择
2.1 传统遗传算法(GA)
遗传算法是受到生物进化过程启发的搜索和优化技术,基于适者生存的规则,通过各种遗传操作搜索最优解。遗传算法中的种群(population)代表着问题可能存在的解集,而一个种群则由经过基因(gene)编码的一定数目的个体组成,每个个体都对应于一个可能的问题解决方案。遗传算法将根据不同编码方式随机产生初始种群,按照适者生存优胜劣汰的原理迭代进化,直至找到合适的解集。在每一次迭代过程中,都会通过适应度(fitness)大小选择合适的个体,并进行组合交叉(crossover)和变异(mutation),产生出新的种群。由于每次迭代时都完成一次进化操作,所以迭代后产生的新种群将会优于初始种群。当最优个体的适应度达到给定的阈值,或者最优个体的适应度和群体适应度不再上升时,或者迭代次数达到预设的代数时算法终止,末代种群中的最优个体可以作为问题近似最优解。遗传算法的算法描述如算法2所示。
算法2遗传算法流程
输入:种群中的个体数n,交叉概率pc,变异概率pm。
输出:具有高适应度的个体。


遗传算法的主要特点是直接对结构对象操作,具有更好的全局寻优能力;采用概率化寻优方法,能自动获取和指导优化搜索空间,自适应地调整搜索方向。在解决大规模非线性、非连续和复杂的综合性大型问题有着巨大的优势,易于计算机实现、操作简单、对问题域有超强描述能力。但由于其交叉概率和变异概率的值固定,可能会陷入局部最优解。
2.2 改进的自适应遗传算法
2.2.1 染色体编码
对搜索空间中的问题解进行编码使其映射为遗传种群中的个体是自适应遗传算法的首要步骤。根据微阵列数据维度高规模大的特点,本文将采用二进制编码方式对个体进行编码,将被选中的特征置1,其余置0。
2.2.2 遗传算子
在选择操作中,轮盘赌方法对个体采用回放式的随机采样方法,使种群中的每个个体都有机会被选择,从而保证了种群的多样性。然而,由于采样方式的随机性,使得它有时不选择适应度高的个体,导致收敛速度变慢。针对轮盘选择算子的不足,本文提出采用最佳保留和轮盘赌相结合的方式,直接保留适应度值较高的5%个体,其余95%个体采用轮盘赌的方式进行选择,这样能将较优个体和平庸个体都得到保留。
交叉操作和变异操作是遗传算法中的两个关键操作,交叉操作会在全局生成新的个体,保证算法的全局搜索能力,变异操作保证算法的局部搜索能力。传统遗传算法中交叉概率(pc)和变异概率(pm)是固定的预定义变量,pc值过大会使具有高适应度值的个体丢失,过小会使陷入局部最小值使搜索停滞。pm值过大会使遗传算法变成随机搜索算法,过小会限制算法的搜索能力。自适应参数调节通过动态调整交叉概率和变异概率的值,既使得个体多样性得到提高,又保证了算法的收敛速度。在改进的自适应遗传算法中,pc和pm的值可以按照公式(11)和(12)进行调整:
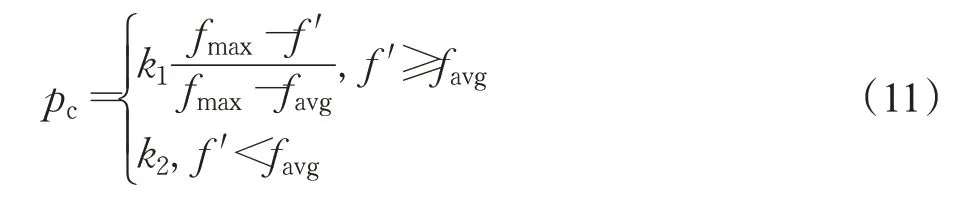
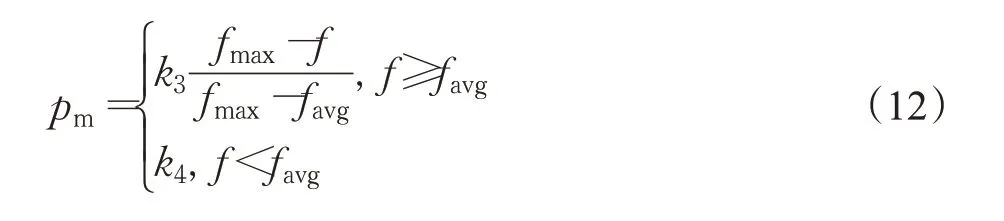
在公式(11)、(12)中,fmax表示算法进行搜索操作时所有个体适应度的最大值,favg表示平均适应度,f′表示即将进行交叉操作中的父母的较大适应度,f表示个体的适应度值,k1、k2、k3、k4表示四个控制变量。本文中设置k1=0.9,k2=0.6,k3=0.2,k4=0.01。
2.2.3 改进的适应度函数
适应度值体现了种群中的个体对环境的适应能力,算法应用选择操作来从父代种群中选择适应度值高的个体遗传到下一代,经过多次迭代逐渐获得问题的最优解。
特征选择的目的是保留较少的有效特征以提高算法的分类性能。在分类问题中,不同数量特征特征子集极有可能具有相同的分类精度。但是,遗传算法如果较早找到特征数量最多的子集,则将忽略特征较少的子集。为了解决类似问题,本文提出了一种新的适应度函数(评价函数),用来评估特征选择算法的性能,公式(13)把搜索到的特征子集数与分类精度融合到适应度函数中。

其中,acc为个体的分类精度,n为通过算法选择后的特征数目,N为特征总数,α和β是用户自定义参数,用来平衡分类准确率和所选特征数对适应度函数的贡献。改进的自适应遗传算法的优化过程如图1所示。
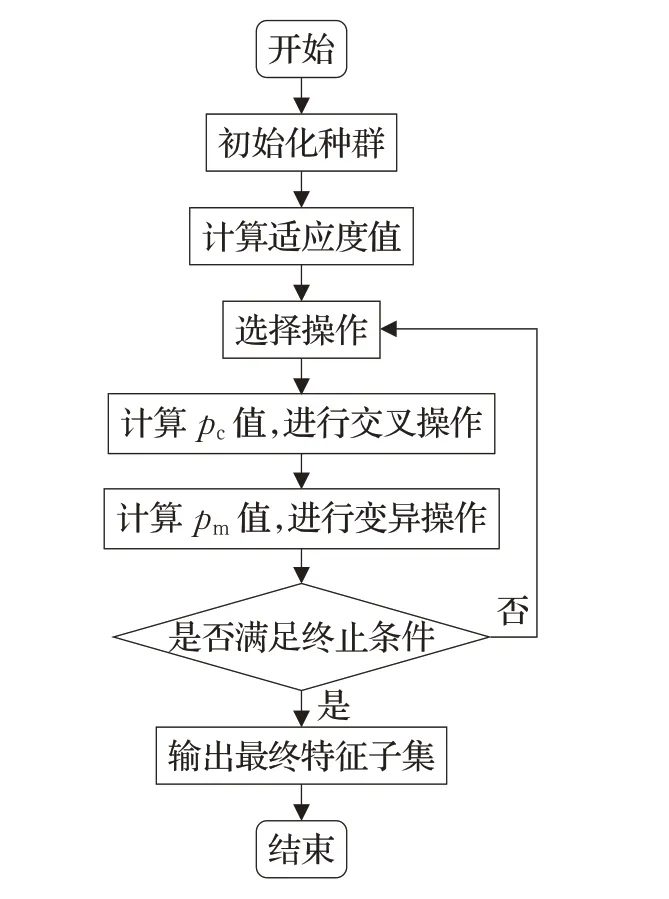
图1 改进的自适应遗传算法流程图
3 混合Filter与改进自适应GA的特征选择方法(ReFS-AGA)
目前,数据仍存在高维度、大规模、高冗余等特性,这使得人们在处理和分析数据时存在巨大挑战。因此本文提出了一种混合Filter模式与改进自适应算法的特征选择方法(ReFS-AGA),首先使用ReliefF算法去除不相关特征,进行特征降维,再结合互信息去除冗余较高特征,经过滤后的特征子集构成自适应遗传算法的初始种群,最后引入改进的自适应遗传算法,优化选择算子,并改进评价函数平衡特征子集和分类精度,快速找出最佳特征子集。算法流程图如图2所示。
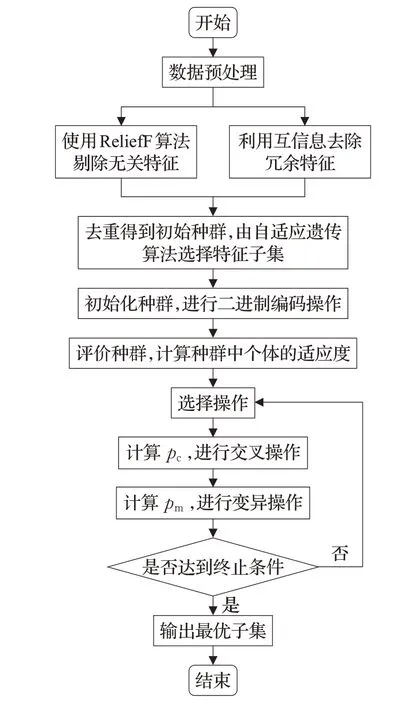
图2 ReFS-AGA算法流程图
ReFS-AGA算法的详细步骤可描述如下:
(1)采用ReliefF算法计算原始数据集的特征权重值,去除无关特征得到特征子集A。设置特征子集A中特征数为原始特征数的10%。
(2)采用标准互信息公式(10)计算原数据集中特征与类的相关性,并按其值降序排列对应特征,得到特征子集B。
(3)将特征子集A与特征子集B合并,并去除重叠特征,组成自适应GA算法的初始种群。
(4)初始化种群,采用二进制编码对种群中的个体进行编码。人口规模根据问题空间确定;尺寸越大,自适应GA越容易搜索最佳解决方案,并且时间越长。在这项工作中,种群大小M设定为50,采用二进制编码对种群中个体进行编码。
(5)根据公式(13)计算适应度值,并通过阈值选择出适应度高的个体。
(6)根据公式(11),计算出pc的值,使用交叉操作生成新的个体。
(7)根据公式(12),计算出pm的值,使用变异操作生成新的种群。
(8)是否满足终止条件,如果是则转到(9),如果否,转到(5)。
(9)根据解码规则输出最佳特征子集。
ReliefF算法核心是根据被选择的样本和不同类别的最近邻样本的距离来评价特征,能有效剔除无关特征,运行效率高但去冗余度较差;归一化互信息通过找到对同一类别中具有强依赖性的特征达到去冗余的目的;自适应遗传算法作为一种不确定搜索算法,初始种群会影响其性能,混合Filter模式与自适应GA的特征选择方法利用ReliefF算法和互信息融合,去除高维数据中的不相关特征和冗余特征,使得自适应遗传算法具有较好的搜索起点,同时根据特征数和分类精确度改进适应度函数并对参数进行自适应调整,使得算法具有更快的收敛速度和更好的寻优能力,从而高效地获得特征子集。
4 实验与分析
4.1 实验设置
为了评估本文所提出的算法对高维度微阵列数据的处理性能,将分别在Breast Cancer Wisconsin、SPECTF Heart、Colon Cancer、SRBCT、Leukemia、Lung Cancer、Breast和Lymphoma基因表达数据集中使用该算法。以上八个数据集都是公共高维数据集且被广泛使用。数据集的简要描述如表1所示。
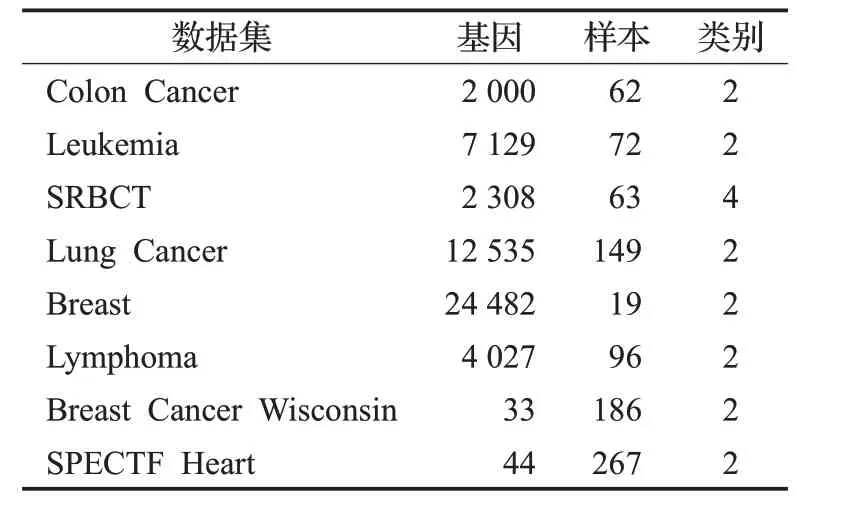
表1 基因表达数据集
实验平台为PC机(Windows7,Intel Core i5-2410 M CPU@2.30 GHz),使用的软件为MATLAB R2017a和Pycharm。本文所使用的经典分类器是SVM和Naive Bayes。
4.2 实验结果分析
为了较好地验证本文所提出的特征选择算法的有效性,分别从改进的自适应遗传算法和混合filter与改进的自适应遗传算法(ReFS-AGA)两个方面分析。在进行实验之前,采用Min-Max标准化方法对所有的数据集进行预处理,将其归一化。在所提出的ReFS-AGA算法中,需对参数进行设置,其中ReliefF算法中近邻值k设置为10,在归一化互信息的计算中阈值设置为0.15在自适应遗传算法中种群大小P设置为30,适应度函数中的参数α和β分别设置为0.8和0.2。最大迭代循环数为500,对于小于1 000个案例的数据集,采用五折交叉验证,对于大于1 000个案例的数据集,采用十折交叉验证,分类精确度取算法每次叠后的平均值。
4.2.1 改进的自适应遗传算法
为了验证所提出的改进自适应遗传算法的有效性,使用基于轮盘赌的遗传算法(GA)、自适应遗传算法(AGA)[12]和本文改进的自适应遗传算法(改进AGA)在表2中两个数据集上进行测试,每个算法分别运行30次,分别对比特征数和分类精度的平均值。
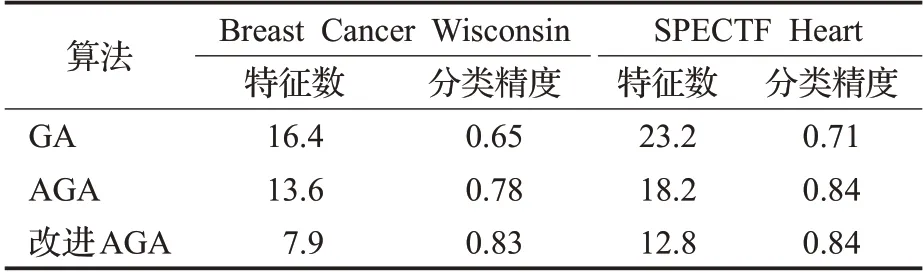
表2 不同遗传算法实验结果
根据表2实验结果可以看出,在Breast Cancer Wisconsin数据集上,改进AGA选择的特征数明显少于GA和AGA,同时分类精度也得到提高;在SPECTF Heart数据集上,虽然改进AGA在分类精度与AGA相差不大,但改进后的算法选择了更少的特征。由此可以证明本文提出的改进AGA不仅保证了分类精度,而且取得了最小特征子集。
4.2.2 ReFS-AGA算法
为了探究ReFS-AGA算法的优化性能,将从运行时间、作用在相同数据集上的不同特征方法对分类精度的影响和特征数对所选特征分类精度的影响三个方面设置对比实验。
(1)运行时间
为了评价ReFS-AGA算法的效率,在表1前六个数据集上分别应用ReFS-AGA算法和自适应遗传算法,比较其运行时间,其中自适应遗传算法的参数与上述相同,时间取算法独立运行30次的均值,结果如表3所示。
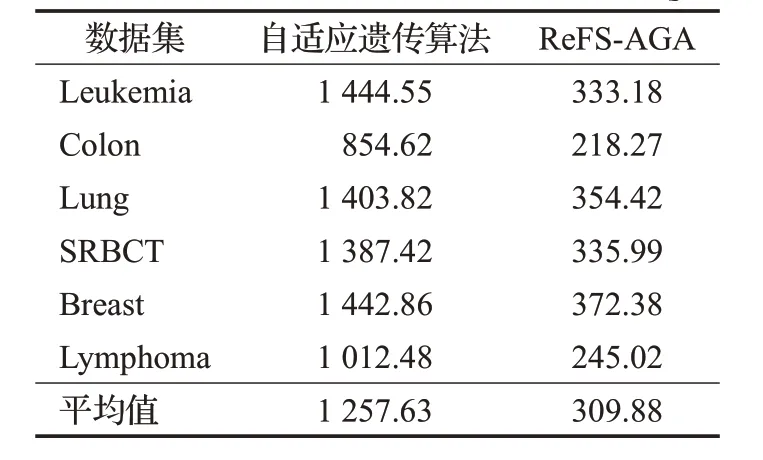
表3 算法的运行时间s
通过表3可以看出,在六个数据集上应用ReFS-AGA算法进行特征选择所需要的运行时间远远小于仅应用自适应遗传算法的所需时间。这说明ReFS-AGA混合算法充分发挥了Filter模式快速高效的优点,快速去除冗余特征的同时将特征降维,为自适应遗传算法提供较优的搜索起点,很大程度地提高了算法的运行效率。
(2)不同分类方法对分类精度的影响
为了充分证明所提出算法的准确性,本组实验选择四种经常使用的特征选择方法与所提出方法进行比较。其中,CFS(Correlation-based Feature Selection)[13]采用基于特征与标签相关性的启发式方法来评价特征的重要性。mRMR-GA[14]是一种二阶段选择算法,该算法先应用MRMR过滤高维微阵列数据中的嘈杂基因,然后通过遗传算法筛选特征子集。mRMR-PSO[15]同样作为二阶段选择算法,采用MRMR去除冗余基因后应用PSO算法选择特征子集。
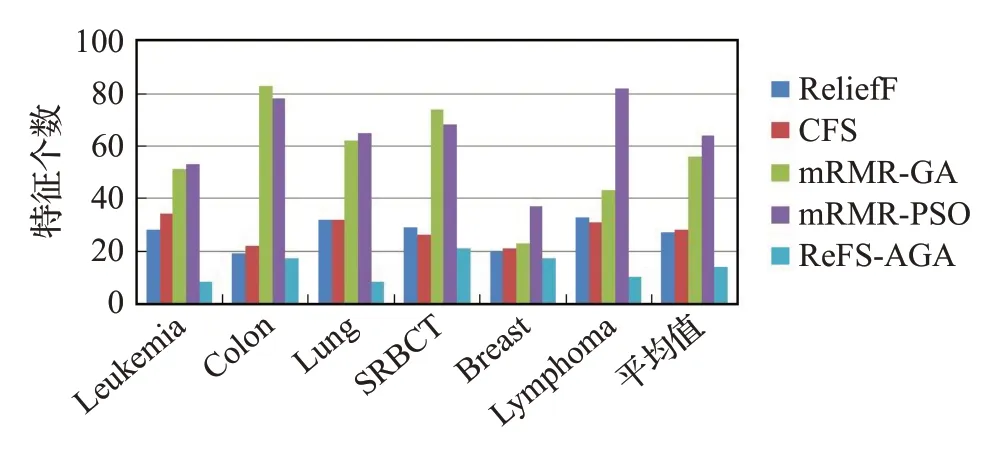
图3 不同特征选择算法所选的特征数目
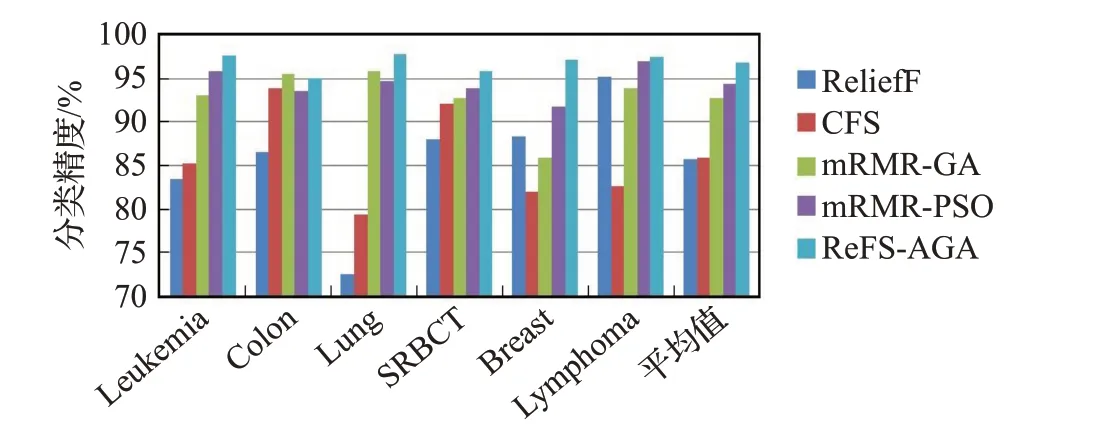
图4 不同特征选择算法基于SVM的分类精度比较
图3和图4分别给出了五种分类方法作用在六个数据集上的所选择的特征个数和其基于SVM分类器的分类精度。根据图3可以看出ReFS-AGA算法选择的特征个数最少,而mRMR-PSO算法选择的特征个数最多,当算法ReFS-AGA使用在SRBCT数据集上选择特征时,所选的特征个数稍高于其使用在另三个数据集,而在Leukemia和Lung数据集上所选的特征数较少。根据图4的实验结果,可以发现本文提出的ReFS-AGA算法在六个数据集上的分类精度最高,其次是mRMR-PSO和mRMR-GA算法,而ReliefF算法的分类精度最低。在对Colon Cancer数据集进行分类时,ReFS-AGA算法的分类准确率稍低于mRMR-GA,在对其余三个数据集进行分类时,ReFS-AGA算法的分类精度都高于其他四种算法。将图4中的分类精度结合图3中的特征子集个数分析,ReFS-AGA算法在Colon Cancer数据集上所选择的基因数远小于其他四种算法但其分类精度却没有明显下降,说明了特征选择的有效性。在Lymphoma数据集上虽然ReFS-AGA算法的分类精度与mRMR-PSO算法的分类精度相差不大,但mRMR-PSO算法所选择的特征数却远远大于ReFS-AGA算法,这说明了ReFSAGA算法具有较好的降维效果。通过平均值可以看出,在不同特征选择算法取得最小特征子集数时,ReFSAGA算法的平均分类精度比ReliefF算法提高了11.18个百分点,比mRMR-GA算法提高了4.04个百分点,这说明相比于Filter特征选择算法中的经典ReliefF算法和基于GA的二阶段特征选择算法mRMR-GA,ReFSAGA算法在保证了特征子集维度的情况下,有效地提升了分类精度。结合表1发现,ReFS-AGA算法不仅能解决多分类问题,还能在处理二分类问题时筛选出较少的特征并获得最佳的分类精度。
(3)特征数对分类精度的影响
为了探究ReFS-AGA算法所选特征数对分类精度的影响,使用SVM和Naive Bayes两种不同的分类器评估六个数据集的分类结果,每个分类精度是重复30次分类过程的平均结果。使用SVM分类器对数据集分类时,设置惩罚系数和伽马值分别为0.12和0.13;并且内核函数是Sigmoid。
从图5、6的分类结果中可以看出,对于经ReFSAGA算法选择后特征子集进行分类时,分类精度不会随着基因数的增加单调增加,在SRBCT数据集上随着基因数的增加分类精度稍有下降,在其他五个数据集上分类精度则呈现平稳趋势。这说明对于相对少量基因的数据集分类时,在较少的基因之间存在的相对简单的映射会使得分类更容易。随着基因数量增加,基因之间复杂的隐含联系和噪声可能影响分类精度。结合表1分析,Leukemia、Colon Cancer、Lung Cancer、Breast和Lymphoma属于二分类数据集,而SRBCT属于多分类数据集,在处理多分类数据时,基因数的增加会使得基因之间相关性变强,从而影响分类精度。另外,对比图5、6发现,采用SVM分类时,在Leukemia、Colon Cancer、Lung Cancer、SRBCT、Breast和Lymphoma数据集上分别在特征数为8、17、8、21、17和10时取得最大分类精度。采用Naive Bayes分类时,在Leukemia、Colon Cancer、Lung Cancer、SRBCT、Breast和Lymphoma数据集上分别在特征数为18、19、9、30、21和20时取得最大分类精度,在Lymphoma数据集上使用Naive Bayes分类所得到的分类精度明显低于使用SVM的分类精度,而其余五个数据集上两个分类器所得到的分类精度差距不大,这说明虽然Naive Bayes算法与SVM算法总体相差不大,但SVM算法在处理高维数据具有更好的性能和较高的准确性。图5和图6中对特征子集使用不同分类器时,所有数据集的分类精度高于85%,这证明了ReFS-AGA方法的稳健性。
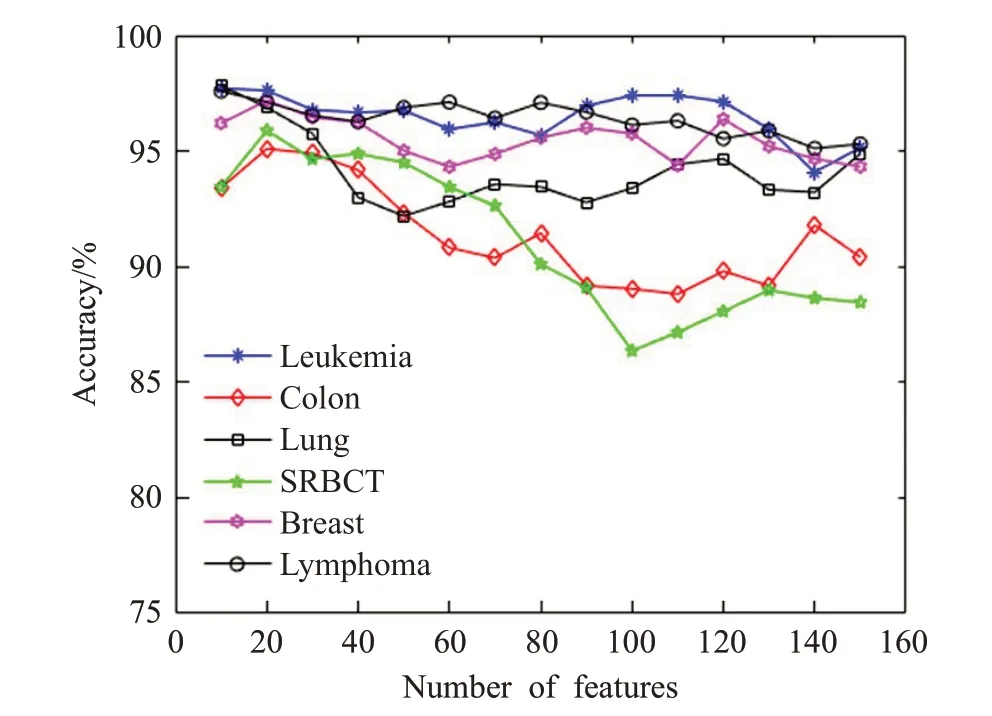
图5 不同数目特征基于SVM的分类精度
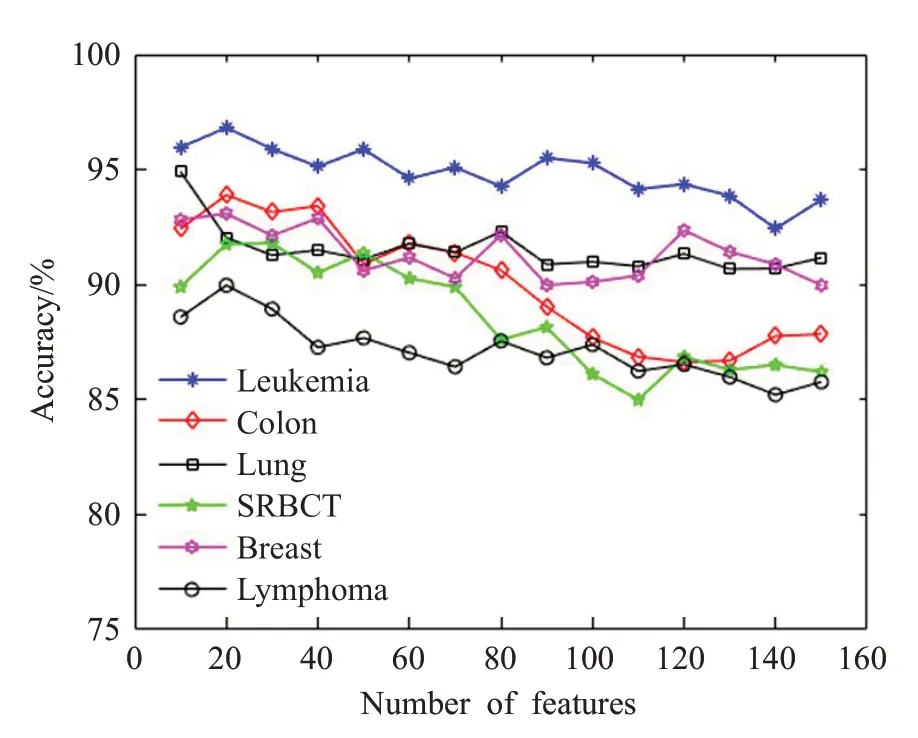
图6 不同数目特征基于Naive Bayes的分类精度
5 结束语
针对高维小样本数据中大量无关特征导致的分类任务繁重、分类精度低等问题,本文提出了一种混合Filter模式与改进自适应遗传算法的特征选择方法,充分结合了过滤式算法的高效快速和自适应启发式算法的准确率高的优点,先使用过滤式特征选择,迅速去除了无关数据,弥补了自适应遗传算法速度慢的不足,为下一阶段提供良好的初始种群;再应用改进的自适应遗传算法随机探索搜索空间,用特征数优化评价函数,最小化保留特征以使子集中仅保留最佳特征,自适应地调整参数,避免算法过拟合,在迭代进化的过程中选择特征子集,最终使算法不仅适合多分类问题,而且相比其他算法有更高的准确率和更小的特征子集。本文将ReFS-AGA算法应用在不同数据集上,通过对比ReFSAGA与ReliefF、CFS、mRMR-AGA和mRMR-PSO算法可以看出,ReFS-AGA算法在降维和提高分类精度方面均有优秀表现,并且通过对比不同分类器对所选特征子集的分类结果可以看出分类精度都保持在85%以上,说明了ReFS-AGA算法的稳健性。基于以上叙述,证明了ReFS-AGA算法不但快速可以筛选出含有重要信息的特征子集,有效地降低数据维度,而且可以在不同类型的数据集上获得优秀的分类精度。未来的工作需要进一步优化算法,使其更加适应样本分布不平均的多分类数据集。
