基于EST-SSR标记的油松种子园遗传多样性分析
2021-06-11代剑峰金雨晴周先清贾海宽张鸿景
代剑峰,党 磊,金雨晴,周先清,贾海宽,张鸿景
(1.河北省林业科学研究院,河北 石家庄 050061;2.河北省林木良种技术创新中心,河北 石家庄 050061;3.北京林业大学,北京 100083;4.平泉市国有七沟林场,河北 承德 067500;5.呼伦贝尔市红花尔基林业局国家樟子松良种基地, 内蒙古 呼伦贝尔 021112)
油松(Pinus tabulaeformis)是我国特有的针叶树种,其天然林和人工林广泛分布于国内的 14个省(区)[1],在木材生产和生态保护上占据重要地位。自20世纪70年代起,我国开始建立油松良种初级种子园,目前包括河北在内的很多地区都已开展有关高世代油松种子园的研究工作[2-5]。
种子园的遗传多样性在一定程度上反映了育种群体配置的合理性以及遗传改良的能力,随着种子园向更高世代发展,如何建立有效的育种群体逐渐成为种子园建设的首要问题,对种子园遗传多样性的研究也越来越迫切[6-12]。利用分子标记对种子园的遗传多样性进行研究是目前较为常用的方法,其中简单序列重复(Simple sequence repeat, SSR)标记具有可靠性高、操作简单且在同属间具备较高通用性等优点,广泛应用于各种林木遗传分析中[13-15]。由于针叶树基因组非常庞大且复杂,导致 SSR引物的开发成本较高[16-17],利用SSR引物对油松种子园遗传多样进行分析的研究较为少见,目前只在内蒙和山西两处种子园有过相关的报道[18-19]。近年来,随着高通量测序技术的发展,利用植物物种的转录组序列来设计的微卫星分子标记引物,即EST-SSR引物,使引物开发的成本大幅降低[20-22],能够批量设计出大量有效引物用于遗传多样性研究,极大地提高了针叶树遗传分析的效率[23-26]。
河北省平泉七沟油松良种基地油松初级种子园始建于1984年,面积56 hm2,共使用217个优树无性系。优树接穗主要来源于辽河源大窝铺、唐山遵化市清东陵、承德平泉市宽城县和七沟镇。本试验以基地内来源清晰的153个无性系为研究对象,利用EST-SSR分子标记分析它们的遗传多样性,探讨无性系之间的亲缘关系和分化程度,对油松高世代种子园建设、遗传改良策略的制定以及优良家系的筛选提供参考。
1 材料方法
1.1 植物材料和基因组DNA提取
油松材料由河北省平泉国有林场良种基地提供,选取基地内油松初级种子园中种源较为清晰的153个无性系作为试验验样本,分别来自于4处天然油松群落:大窝铺(Dawopu)(N41°19′,E118°32′)6个、东陵(Dongling)(N40°11′,E117°37′)97个、宽 城(Kuancheng)(N40°36′,E118°29′)11个 和七 沟(Qigou)(N41°02′,E118°32′)39个。采 集各无性系幼嫩针叶,装于自封袋中冷藏带回,样品储存于-20 ℃直至提取基因组DNA。
利用CTAB法提取各样本针叶基因组DNA,将DNA溶于ddH2O后,利用Nano Drop 2000(Nano Drop 2000/2000c, Thermo Scientific, USA)检测DNA浓度,最终将DNA样本稀释至20 ng/μL备用。
1.2 引物筛选及SSR-PCR反应
本文利用油松及其近缘物种针叶组织转录组信息[21,27],设计了70对EST-SSR引物用于多态性引物的筛选,对所有正向引物的5’端进行M13修饰,以便与荧光标签(FAM,HEX,TAMRA,ROX)结合,利用琼脂糖电泳法对引物进行筛选。
PCR采 用20 μL体 系,包 括10 μL 2×TaqPCR master mix (Biomed Technologies, Beijing, China),4 μL(4 pmol/L)正向/反向混合引物,4 μL (4 pmol/L) 荧光 标 签(Fluorescent-dye-labeled M13 primer),2 μL(20 μg)基因组DNA。PCR扩增条件如下: 94 ℃10 min,94 ℃ 30 s,退火60 ℃ 30 s,每个循环减0.5 ℃,延伸72 ℃ 45 s,进行20个循环。95 ℃ 30 s,退火50 ℃ 30 s,延伸72 ℃ 45 s,进行20个循环。最终延伸72 ℃ 5 min。
1.3 数据分析
试验采用毛细管电泳法,将扩增产物用ABI3730测序仪(3730XL, Applied Biosystems,USA)进行检测,检测结果利用GeneMarker version 2.20(SoftGenetics, State College, Pennsylvania,USA)读取,并记录SSR位点信息(引物gp01对应位点记为GP01,以此类推)。每个位点的等位基因数(Number of observed alleles,N)、近交系数(Coefficient of inbreeding,Fis) 等利用GenALEx version 6.5[28]计算获得;每个位点无效等位基因频率(Invalid allele frequency,Pn)通过FreeNA[29]计算得出;利用SPAGeDi[30]计算出等位基因总数(Total number of alleles,Nt)、每个位点的平均等位基因数(Average number of alleles,Na)、等位基因丰富度(Allelic richness,AR)、有效等位基因数(Effective number of alleles,Nea)、群体独有等位基因数(Np)、观测(Observed heterozygosity,Ho)和期望(Expected heterozygosity,He)杂 合 度;哈 迪—温 伯 格 平衡(Hardy-Weinberg equilibrium, HWE)通过ARLEQUIN[31]进行检测;群体差异系数(Population differentiation coefficient,Fst)由GenALEx软 件 利用多位点比较法(999 permutations)计算得出,群体间差异度(PairwiseFst)也同样由该软件进行计算;包含无效等位基因以及剔除无效等位基因的群体差异系数Fst(ENA)通过FreeNA进行计算,该参数用于评估群体间的遗传分歧以及无效等位基因对遗传差异的潜在影响;NJ Tree图由SplitsTree软件构建并绘制。
2 结果与分析
2.1 引物筛选
利用随机选取8个试验样本的混合DNA对70对引物进行PCR初筛,共有32对引物扩增出了预期大小的片段,其余扩增没有PCR产物。再用这32对引物分别对8个试验样本逐一进行PCR扩增,其中20对引物扩增出了单态位点(或多态性低),另外12对为多态位点,图1为引物gp08的扩增结果。由此,选用这12对引物进行后续试验,引物信息如表1。

图1 引物gp08扩增产物检测Fig.1 Amplified product detection of primer gp08

表1 引物信息Table 1 Information of primers
2.2 无性系及种源群体的遗传多样性
利用GeneMarker version 2.20读取毛细管电泳结果(图2),整理后发现153个无性系在12个SSR位点上,一共检测到了62个等位基因,每个位点的等位基因数为2~10个,平均5.17个,其中在位点GP04检测到了10个等位基因,位点GP02只检测到了2个等位基因。各位点无效等位基因频率(Pn)的检测结果显示,有5个位点(GP2、GP6、GP10、GP11、GP12)几乎没有无效等位基因,GP13的Pn值最高达到了0.162 4,其它位点的Pn值均未超过0.08。期望杂合度(He)的最大值出现在了位点GP01(0.757),所有位点的期望杂合度(He)和观测杂合度(Ho)的平均值分别为0.414和0.403。有多个位点(GP02、GP04、GP06、GP10、GP11和GP12)出现了Ho大于He的情况,表明在这些位点上存在杂合子过量的情况。各位点的近交系数(Fis)较小,平均Fis为0.018 6(表2)。
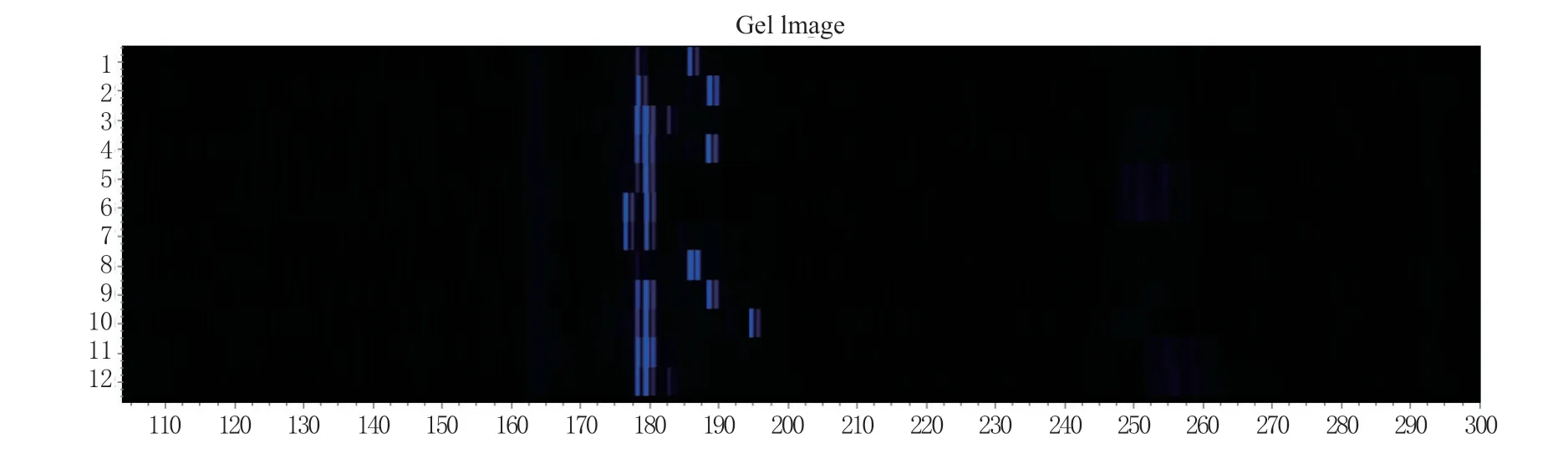
图2 引物gp01在部分样本中的扩增条带Fig. 2 Amplification bands of primer gp01 in partial samples
如表3所示,根据样本来源,153个无性系被分为4个种源群体:大窝铺、东陵、宽城和七沟。在3个群体的9个位点上发现了17个特殊等位基因(Np),出现频率从0.005到0.045。4个群体检测到的等位基因数(Nt)分别为28(大窝铺)、55(东陵)、35(宽城)和44(七沟),平均等位基因数依次为2.33、4.58、2.92和3.67。大窝铺、东陵和七沟3个群体的平均有效等位基因数(Nea)、等位基因丰富度(AR)、观测杂合度(Ho)和期望杂合度(He)基本一致,都略高于宽城群体,这可能与群体样本数量以及样本选择有关。
2.3 无性系种源差异及群体分化
153个无性系在12个位点上的群体差异系数(Fst)都很小,均值仅为0.015 6。去除无效等位基因后,Fst)(ENA)也没有发生显著的变化,均值为0.017 2。
同时,对4处种源群体进行两两比较,PairwiseFst最大值出现在大窝铺群体和宽城群体之间(0.071 7),最小值在东陵群体和七沟群体之间(0.009 9),均未达到显著水平,表明各群体间没有明显的遗传分化(表4)。
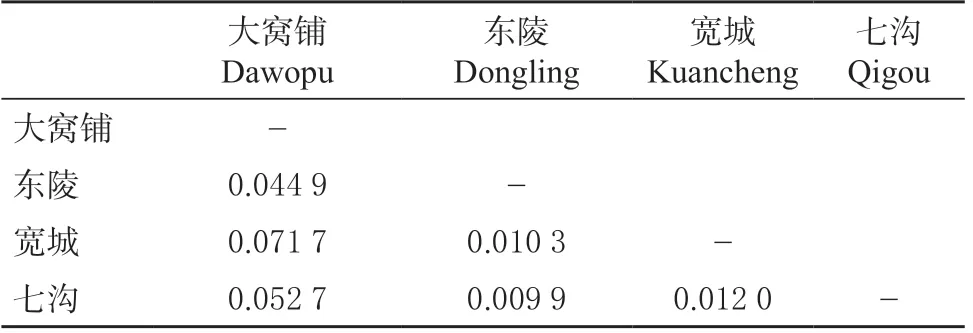
表4 4处种源群体的遗传分化Table 4 Genetic differentiation of groups between 4 provenances
图3为依据153个样本的遗传距离绘制的NJ 树图,从图中可以看到聚类的结果并没有表现出明显的种源相关性,大部分情况下都是多个种源的无性系相互混杂。表明虽然种源地不同,但这些无性系之间共享了大部分的遗传信息。
依据个体基因型进行主成分分析(PCA),前3个成分贡献了79.54%的变异(分别为73.54%,3.09%和2.91%)。图4为4个群体的无性系分别在PC1与PC2的二维坐标图(4A)和PC1与PC3的二维坐标图(4B)上的投影,从图中可以看出4个群体是相互重叠的,很难进行区分。这进一步证明所有无性系的亲缘关系较近,没有明显的群体界限。

3 讨论与结论
3.1 EST-SSR引物筛选
随着各物种cDNA文库的不断丰富,与传统SSR引物相比利用转录组序列设计EST-SSR引物速度更快、成本更低且具备良好的属间通用性,因此近年来被广泛的应用于种质资源的评价和利用、遗传多样性检测与分析以及种子园交配系统评价等领域[18,25,32]。然而,由于EST-SSR的有效扩增率低、多态性差,在使用前首先要进行大量的引物筛选工作[33]。本试验从油松及其近缘物种的70对ESTSSR引物中筛选出32对能够有效扩增的引物,其中12对为多态性引物,引物多态率为17.14%。造成EST-SSR引物多态率较低的原因可能由于松科植物是比较原始的树种,序列相对保守、变异程度相对较低,此外植物样本之间的基因差异较小也可能造成EST-SSR引物多态率较低。本研究用这12对引物对油松种子园的育种群体进行标记,共检测出62对等位基因,多样性信息较为丰富,从基因水平上为油松种子园的多样性分析提供了参考信息。此次筛选出的引物也可以为其他同类种子园多样性的研究提供可靠的引物资源。

图4 主成分分析图Fig.4 Principal component analysis(PCA)
3.2 油松种子园的遗传多样性
种子园的遗传多样性在一定程度上反映了育种群体配置的合理性以及遗传改良的能力,丰富的遗传多样性是种子园实现育种目的的基础。本研究对油松种子园的153个无性系单株遗传多样性的分析结果表明,由这153个无性系单株构成的群体在12个多态性位点上平均等位基因数为5.17,期望杂合度(He)和观测杂合度(Ho)分别为0.414和0.403。与程翔等[18]利用EST-SSR对黑里河油松种子园遗传多样性进行分析时得到的结果(He=0.366,Ho=0.446)基本一致,高于李悦等[34]利用同工酶标记得到结果(He=0.329,Ho=0.285),又低于袁虎威等[5]利用SSR标记得到的结果(He=0.570 3,Ho=0.595 5)。造成这种差异的原因可能与EST-SSR标记的位点多态性高于同工酶标记而低于SSR标记有关。本文结果表明该处种子园有着较为丰富的遗传多样性,具备一定的育种潜力。
3.3 油松种子园无性系的遗传分化
本试验选用的153个无性系按来源可以分为4个种源群体:大窝铺、东陵、宽城和七沟。通过分析发现它们的群体间平均差异系数(Fst)为0.015 6,该结果与马尾松(0.018 1)[35]和侧柏(0.011)[36]类似,远低于樟子松(0.219 4)和挪威云杉(0.749)等[37-38]针叶树种,说明该种子园的油松群体遗传分化水平较低,主要差异来源于群体内部。同时,NJ树图聚类和主成分分析的结果进一步印证了以上观点。导致这种分化不明显的原因可能是:该处种子园的种源地都分布于河北省东北部和北部山区,气候条件类似,加之人类的频繁引种活动,最终使得该区域内的油松基因交流频繁。为了避免近交的发生,油松种子园在繁育高世代良种时,一方面应当结合分子标记对亲本群体进行筛选,另一方面应当从更广泛的区域引进油松良种扩充亲本群体。
由于病虫害问题导致种子园连年欠收,一直没能获得足够的子代群体,因此结果尚需要子代数据的进一步佐证,后续的研究中对种子园的交配系统进行全面的分析,完善结论。另外,由于种子园的优树绝大部分都源于遵化市的清东陵,而源于其他种源地的资源较少,导致本文中各种源群体大小出现不平衡,这是否会影响结果产生偏差有待进一步验证。
