抗生素抗性基因在大气环境中的研究进展
2021-05-29贺红梅张文斌张小荣贺凯张丽
贺红梅,张文斌,张小荣,贺凯,张丽
抗生素抗性基因在大气环境中的研究进展
贺红梅1,张文斌2,张小荣2,贺凯3,张丽1
(1. 萍乡学院 材料与化学工程学院,江西 萍乡 337000;2. 隆回六中,湖南 邵阳 422208;3. 隆回县烟草专卖局,湖南 邵阳 422210)
抗生素抗性基因(ARGs)作为一种新的环境污染物,受到了广泛的关注,目前关于大气中ARGs的研究很少。大气环境是ARGs的潜在储存库,空气传播是抗生素抗性细菌引起重要疾病的主要途径。文章在查阅大量国内外文献的基础上,系统综述了大气环境中ARGs的污染现状、潜在传播途径及其机制和相关的检测方法,并指出了当前形势下我国开展对大气中ARGs研究的必要性和重要性,为相关机构开展气溶胶中ARGs研究提出几点建议。
抗生素抗性基因;耐药菌;大气环境
抗生素是由微生物、化学合成和半合成的类似化合物产生的次级代谢产物,能抑制微生物生长[1]。作为治疗人类感染性疾病的治疗剂,如今它也被广泛用于畜牧业和水产养殖业[2]。兽用抗生素最初用于动物疾病预防和治疗、饲料添加剂、生长促进剂,目前远远超过了其作为动物治疗剂的用途[3]。大量使用抗生素可能会对人类和微生物系统产生强大的选择压力[4–6]。抗生素耐药性对人类健康威胁主要有:(1)残留的抗生素可能会改变微生物组,并促进耐药菌(ARB)的出现和选择[7];(2)对环境微生物产生选择压力并导致抗生素抗性基因(ARGs)和ARB蓄积的潜在危害[5]。由于治疗性抗生素使用效率的降低,导致临床上抗生素抗性病原体引起患者感染住院增加,甚至出现治疗失败和死亡现象[8]。目前关于ARGs研究主要集中在其来源、环境行为与传播机制、细菌耐药对人体健康的影响以及ARGs污染控制等方面[9–12],且大多数研究围绕水和土壤中的ARGs。相比其他环境介质,国内外对大气中ARGs的研究相对较少。根据近几年国内外文献资料,本文对大气中ARGs的污染现状、潜在传播途径及其机制和相关的检测方法进行了系统综述,并指出了当前形势下我国开展大气中ARGs研究的必要性和重要性,为相关机构开展气溶胶中ARGs研究提出几点建议。
1 大气中ARGs的污染现状
与其他相似污染物不同,ARB和ARGs可以通过空气颗粒物传播,该空气颗粒物具有许多附着点并使其ARB和ARGs在高污染的大气中更稳定地悬浮。研究表明空气传播被认为是ARGs传播的重要途径。与其他介质相比,大气中的ARB和ARGs黏附在空气颗粒后易于被人体吸入,更深地沉积在肺部,对人体健康构成直接的危害。有学者研究香港17个幼儿园室内空调过滤器灰尘和小孩尿液样本中的ARGs和ARB。结果发现幼儿园室内存在16种ARG和、的多重耐药细菌[13]。在污水处理厂(细格栅和污泥浓缩池)、浴室、实验室、医院和室外等6类大气样品中检测到19种ARGs,其中在雾霾天室外检测到的ARGs总浓度最高(9×105copies/m3),而最低的是在浴室环境中(4.2×104copies/m3),医院大气环境中的ARGs种类最多。此外,在这6个大气环境中发现的ARGs中主要是β-内酰胺类ARGs[14]。房文艳等学者研究了深圳市某典型社区农贸市场内空气微生物及ARGs,发现活禽交易区可培养细菌浓度远高于一般室内区域,其中PM2.5中所含菌量占总菌量42%以上;活禽交易区空气介质中、、和检出率高达70%,周边环境空气样品中,随着与活禽交易区距离的增加,空气微生物含量和ARGs丰度呈显著下降趋势[15]。有学者收集我国北部的三个特大城市(北京、天津和石家庄)的灰尘,发现灰尘中含有大量的ARGs,并且夏季的ARGs含量要比冬季高,在夏季磺胺类、四环素类和β-内酰胺类TEM-1的含量分别可达到~107.1copies/g、106.4copies/g、107.3copies/g,这可能是夏季人类活动频率比冬季高造成的[16]。Ling等对猪场和乳制品厂内部环境、诊所和避难所室外环境的空气样品进行定量检测,结果表明在所有空气样品中,、均有检出,并且养殖场和室内环境均高于室外,而仅在养殖场中检出[17]。McEachran等研究了养牛场空气PM10颗粒物,检出、、和[18]。Chandan等研究雾霾时期大气样品,被检出64种耐药基因,大气可能是重要的耐药基因储存库[19]。Gilbert等发现医院空气中微生物气溶胶含有、和基因[20]。
2 大气中ARGs的危害
大多数依附空气传播的病原体具有多重耐药性[21–24],人类呼吸道疾病的增加也可能与水平方向上的ARB和ARGs的传播增加有关,不同体系的微生物中携带的ARB和ARGs通过雾化作用释放到大气环境中,依附具有许多附着点的空气颗粒物传播,其中促进空气传播的生物颗粒物为生物气溶胶,主要包括细菌、真菌孢子、植物花粉、藻类以及细胞排泄物或碎片[21],占颗粒物(PM)的30%∽80%[25]。这意味着生物气溶胶中可能存在大量的致病性和非致病性微生物,并对人类健康和气候产生重大影响。生物气溶胶具有许多附着点并使其ARB和ARGs在高污染的大气中更稳定地悬浮,通过云滴,冰晶以及降水等作用循环到环境中的其他介质,影响水文循环和气候[21]。此外,ARB和ARGs粘附在空气颗粒后易于通过呼吸作用吸入,更深地沉积在人体肺部,导致呼吸系统症状、传染病、急性毒性以及癌症,从而也成为人类健康的主要问题[26]。除病原体外,空气中ARGs还可通过不同的人类活动或风向使其重新雾化而直接排放[27](图1),这对人类的健康构成了严重威胁。然而关于大气中ARGs污染概况及其吸入健康影响的研究十分缺乏,可能是由于难以捕获足够数量的生物气溶胶以进行ARGs识别和进一步量化。
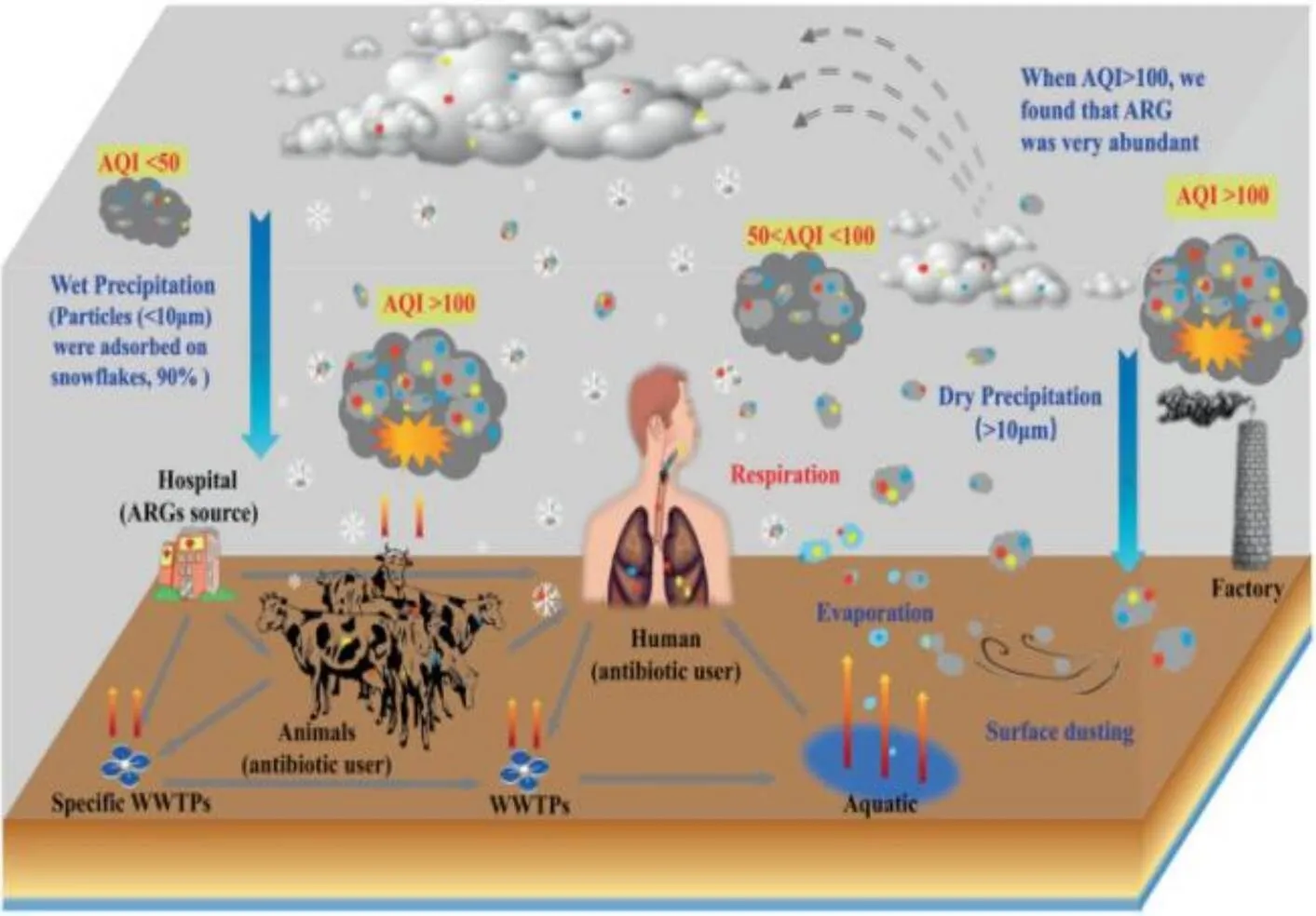
图1 ARGs环境回路示意图[28]
3 大气中ARGs的传播机制及其途径
微生物系统中抗生素的出现可能导致正常敏感细菌的遗传或突变,使细菌能够存活并进一步繁殖,成为带ARGs的ARB。ARGs扩散传播的主要机制是垂直基因转移(VGT)和水平基因转移(HGT)。其中VGT是通过微生物自身繁殖使得ARGs在水体、土壤、大气不同环境介质中扩散传播(图2)。HGT是以包括质粒、整合子和噬菌体在内的移动遗传元件(MGEs)作为载体,通过接合、转导和转化等作用传播ARGs[28,29](图3)。大气中细菌群落主要由四个门组成:Actinobacteria、Bacteroidetes、Firmicutes和Proteobacteria[30]。但是在同一水平上空气传播细菌的可变性,主要取决于它们对环境条件的适应性。此外气象参数(大气温度、风速、太阳辐射、相对湿度、大气压强、蒸汽压力、降雨、降雪)和理化因素(氮氧化物、悬浮物[PM2.5、PM10]、CO、SO2、O3等)以及采样位置可以直接影响空中细菌群落的组成[31–34]。
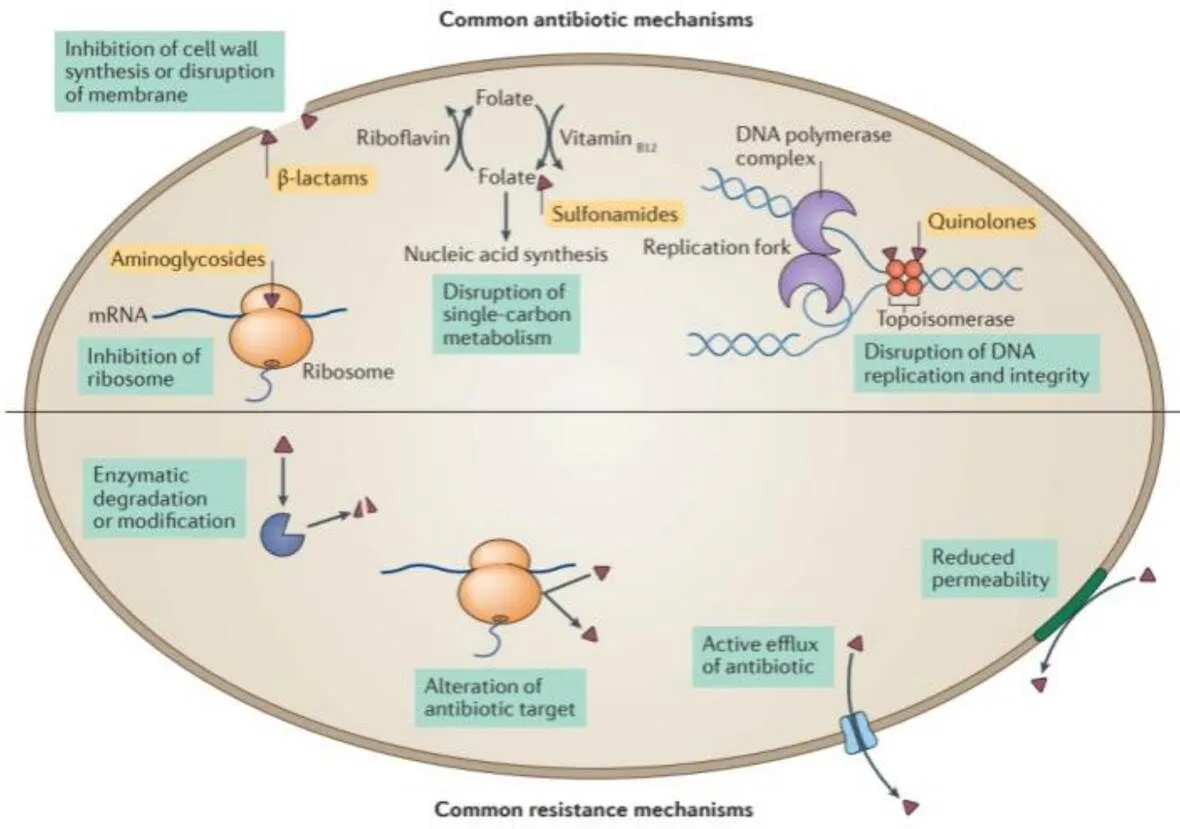
图2 ARGs扩散传播主要机制
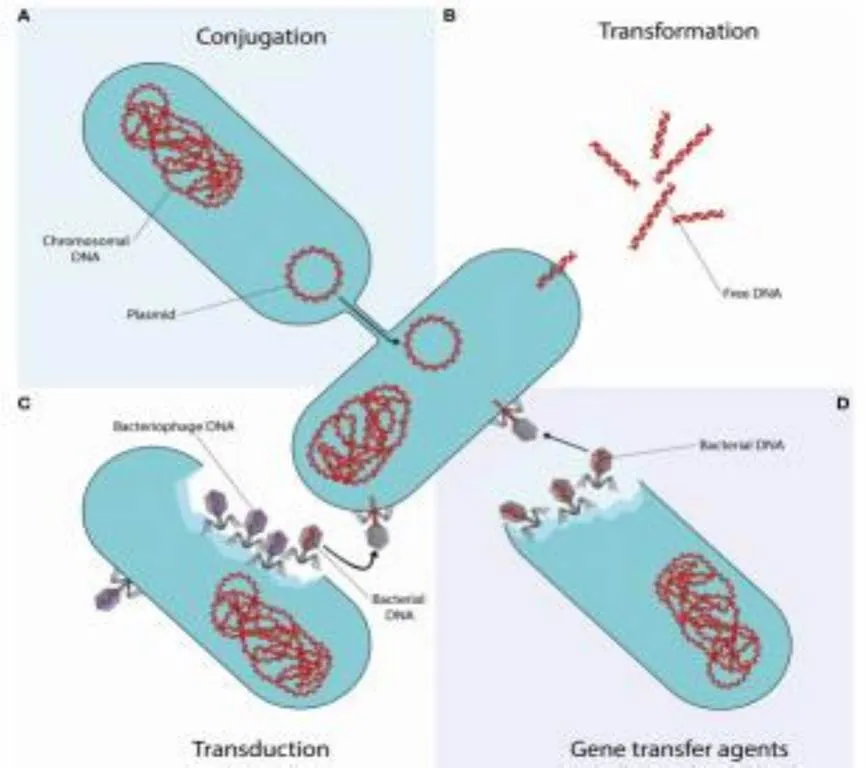
图3 基因水平转移途径
4 大气中ARGs的采样和检测方法
目前已有多种分子生物学法被用于检测ARGs。这些方法主要有普通聚合酶链式反应(Polymerase chain reaction,PCR)、实时荧光定量PCR、宏基因组学方法等。PCR基本原理是:对特定核苷酸片段进行指数级扩增,在扩增反应结束后,再通过凝胶电泳对产物定性分析。一般步骤为:样品DNA提取→目标基因引物设计→PCR扩增反应→凝胶电泳验证扩增产物。该技术可直接在空气样品中提取总DNA,再利用PCR方法分析样品中的ARGs。该方法不需要对微生物进行培养,可将环境中低浓度目标DNA或RNA 进行扩增和检测,结果更加准确,快速灵敏[35,36]。
普通PCR只能对ARGs定性检测,而实时荧光定量PCR (Real-time quantitative PCR,RT-QPCR)技术,从数量上可以更直观地表征环境中ARGs的水平,有助于摸索其在环境介质中的迁移和传播等行为。RT-QPCR 技术原理是:通过荧光标记探针,把核酸扩增、杂交、光谱分析和实时检测计算技术结合起来,然后借助荧光信号检测PCR产物[37]。荧光标记物与扩增产物结合后,被激发的荧光强度就与扩增产物量成正比,从而可实现精确定量,准确表征环境的抗性基因水平。RT-QPCR 技术可进行实时监测和连续分析,自动化程度高,它不需要通过电泳确认扩增产物的长度,具有特异性更强、能有效解决PCR污染问题等优点,被广泛应用于环境微生物生态系统中特定基因定量检测。
宏基因组学方法(Metagenomics)是将样品中的DNA直接克隆到载体并导入宿主细菌中,进而筛选目的基因进行测序分析等。宏基因组方法用于检测ARGs的流程:宏基因组DNA提取与纯化→宏基因组文库的构建→宏基因组文库的筛选和分析[38,39]。大气中ARGs 采集借鉴微生物气溶胶的采集方法,目前已用于大气中ARGs的采集方法和检测技术主要如下表1。

表1 大气中ARGs的采样技术和检测方法
5 研究展望
我国对大气中ARGs 的研究尚处于起步阶段。大气环境作为 ARGs 的潜在储存库在一定程度上缺乏详细的研究数据,本文简要综述了大气中 ARGs 的研究现状,对未来相关研究开展提出几点建议:
(1)由于空气具有流动性,不同季节不同大气环境ARGs污染的种类、浓度和其影响因素应是未来研究的重点。
(2)空气样品通常因为不稳定性因素太多,样品收集方法和提取技术对结果影响较大,我们应该在不同领域(例如,纺织工业、食品工业、医药和生物修复)中研究空气传播细菌活性和生物勘探。在今后的研究中致力于优化大气中ARGs 的样品采集和定性定量检测方法,可以考虑新一代测序方法以测定更多的ARGs,为大气中ARGs的削减提供新思路。
[1] DEMAIN A L, SANCHEZ S. Microbial drug discovery: 80 years of progress[J]. The Journal of antibiotics, 2009, 62(1): 5–16.
[2] NISHA A R. Antibiotic Residues-A Global Health Hazard[J]. Veterinary world, 2008, 1(12): 375–377.
[3] GELBAND H, MILLER-PETRIE M, SURAJ P, et al. The state of the world's antibiotics 2015[J]. Wound Healing Southern Africa, 2015, 8(2): 30–34.
[4] National Academies of Sciences, Engineering, and Medicine. Environmental Chemicals, the Human Microbiome, and Health Risk: A Research Strategy[M]. The National Academies Press, 2018.
[5] QIAO M, YING G G, SINGER A C, et al. Review of antibiotic resistance in China and its environment[J]. Environment international, 2018, 110: 160–172.
[6] PRUDEN A, PEI R, STORTEBOOM H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado[J]. Environmental science & technology, 2006, 40(23): 7445–7450.
[7] CHO I, BLASER M J. The human microbiome: at the interface of health and disease[J]. Nature Reviews Genetics, 2012, 13(4): 260–270.
[8] HIDRON A I, EDWARDS J R, PATEL J, et al. Antimicrobial-resistant pathogens associated with healthcare-associated infections: annual summary of data reported to the National Healthcare Safety Network at the Centers for Disease Control and Prevention, 2006–2007[J]. Infection Control & Hospital Epidemiology, 2008, 29(11): 996–1011.
[9] LUO Y I, MAO D, RYSZ M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental science & technology, 2010, 44(19): 7220–7225.
[10] ZHAI W, YANG F, MAO D, et al. Fate and removal of various antibiotic resistance genes in typical pharmaceutical wastewater treatment systems[J]. Environmental Science and Pollution Research, 2016, 23(12): 12030–12038.
[11] DANG B, MAO D, XU Y, et al. Conjugative multi-resistant plasmids in Haihe River and their impacts on the abundance and spatial distribution of antibiotic resistance genes[J]. Water research, 2017, 111: 81–91.
[12] WANG J, MAO D, MU Q, et al. Fate and proliferation of typical antibiotic resistance genes in five full-scale pharmaceutical wastewater treatment plants[J]. Science of the Total Environment, 2015, 526: 366–373.
[13] LI N, CHAI Y, YING G G, et al. Airborne antibiotic resistance genes in Hong Kong kindergartens[J]. Environmental Pollution, 2020, 260: 114009–114017.
[14] WANG Y, WANG C, SONG L. Distribution of antibiotic resistance genes and bacteria from six atmospheric environments: Exposure risk to human[J]. Science of the Total Environment, 2019, 694: 133750–133758.
[15] 房文艳, 高新磊, 李继, 等. 城市社区农贸市场空气微生物及抗生素抗性基因研究[J]. 生态毒理学报, 2015, 10(5): 95–99.
[16] ZHOU H, WANG X, LI Z, et al. Occurrence and distribution of urban dust-associated bacterial antibiotic resistance in Northern China[J]. Environmental Science & Technology Letters, 2018, 5(2): 50–55.
[17] LING A L, PACE N R, HERNANDEZ M T, et al. Tetracycline resistance and class 1 integron genes associated with indoor and outdoor aerosols[J]. Environmental science & technology, 2013, 47(9): 4046–4052.
[18] MCEACHRAN A D, BLACKWELL B R, HANSON J D, et al. Antibiotics, bacteria, and antibiotic resistance genes: aerial transport from cattle feed yards via particulate matter[J]. Environmental health perspectives, 2015, 123(4): 337–343.
[19] PAL C, BENGTSSON-PALME J, KRISTIANSSON E, et al. The structure and diversity of human, animal and environmental resistomes[J]. Microbiome, 2016, 4(1): 1-15.
[20] GILBERT Y, VEILLETTE M, DUCHAINE C. Airborne bacteria and antibiotic resistance genes in hospital rooms[J]. Aerobiologia, 2010, 26(3): 185–194.
[21] FRÖHLICH-NOWOISKY J, KAMPF C J, WEBER B, et al. Bioaerosols in the Earth system: Climate, health, and ecosystem interactions[J]. Atmospheric Research, 2016, 182: 346–376.
[22] KIM K H, KABIR E, JAHAN S A. Airborne bioaerosols and their impact on human health[J]. Journal of Environmental sciences, 2018, 67: 23–35.
[23] DELORT A-M, AMATO P. Microbiology of Aerosols[M]. John Wiley &Sons, 2017.
[24] POLYMENAKOU P N. Atmosphere: a source of pathogenic or beneficial microbes?[J]. Atmosphere, 2012, 3(1): 87–102.
[25] XIE Z, FAN C, LU R, et al. Characteristics of ambient bioaerosols during haze episodes in China: A review[J]. Environmental Pollution, 2018, 243: 1930–1942.
[26] DOUWES J, THORNE P, PEARCE N, et al. Bioaerosol health effects and exposure assessment: progress and prospects[J]. The Annals of occupational hygiene, 2003, 47(3): 187–200.
[27] ZHU G, WANG X, YANG T, et al. Air pollution could drive global dissemination of antibiotic resistance genes[J]. The ISME Journal, 2021, 15(1): 270–281.
[28] MARTÍNEZ J L, COQUE T M, BAQUERO F. What is a resistance gene? Ranking risk in resistomes[J]. Nature Reviews Microbiology, 2015, 13(2): 116–123.
[29] VIKESLAND P J, PRUDEN A, ALVAREZ P J J, et al. Toward a Comprehensive Strategy to Mitigate Dissemination of Environmental Sources of Antibiotic Resistance[J]. Environmental science & Technology, 2017, 51(22): 13061–13069.
[30] CUTHBERTSON L, AMORES-ARROCHA H, MALARD L A, et al. Characterisation of Arctic bacterial communities in the air above Svalbard[J]. Biology, 2017, 6(2): 29–50.
[31] FUJIYOSHI S, TANAKA D, MARUYAMA F. Transmission of airborne bacteria across built environments and its measurement standards: a review[J]. Frontiers in M23icrobiology, 2017, 8: 2336–2352.
[32] BOWERS R M, CLEMENTS N, EMERSON J B, et al. Seasonal variability in bacterial and fungal diversity of the near-surface atmosphere[J]. Environmental science & technology, 2013, 47(21): 12097–12106.
[33] GANDOLFI I, BERTOLINI V, AMBROSINI R, et al. Unravelling the bacterial diversity in the atmosphere[J]. Applied microbiology and biotechnology, 2013, 97(11): 4727–4736.
[34] ŠANTL-TEMKIV T, GOSEWINKEL U, STARNAWSKI P, et al. Aeolian dispersal of bacteria in southwest Greenland: their sources, abundance, diversity and physiological states[J]. FEMS microbiology ecology, 2018, 94(4): 31–40.
[35] 高盼盼, 罗义, 周启星, 等. 水产养殖环境中抗生素抗性基因 (ARGs) 的研究及进展[J]. 生态毒理学报, 2009, 4(6): 770–779.
[36] 孙平勇, 刘雄伦, 刘金灵, 等. 空气微生物的研究进展[J]. 中国农学通报, 2010, 26(11): 336–340.
[37] 王丽梅, 罗义, 毛大庆, 等. 抗生素抗性基因在环境中的传播扩散及抗性研究方法[J]. 应用生态学报, 2010 (4): 1063–1069.
[38] MONIER J M, DEMANÈCHE S, DELMONT T O, et al. Metagenomic exploration of antibiotic resistance in soil[J]. Current opinion in microbiology, 2011, 14(3): 229–235.
[39] WRIGHT G D. The antibiotic resistome: the nexus of chemical and genetic diversity[J]. Nature Reviews Microbiology, 2007, 5(3): 175–186.
[40] 高敏, 仇天雷, 秦玉成, 等. 养鸡场空气中抗性基因和条件致病菌污染特征[J]. 环境科学, 2017, 38(2): 510–516.
[41] 曾庆涛. 农田土壤典型抗生素抗性基因污染及其土——气迁移研究[D]. 浙江大学, 2019.
[42] 高新磊, 邵明非, 贺小萌, 等. 污水处理厂空气介质抗生素抗性基因的分布[J]. 生态毒理学报, 2015 (5): 89-94.
[43] 董玉芳. 城市农贸市场空气微生物群落特征及抗生素抗性基因污染[D]. 黑龙江: 哈尔滨工业大学, 2014.
[44] 刘长利, 郑国砥, 王磊, 等. 养猪场空气中抗性基因和条件致病菌污染特征[J]. 应用生态学报, 2018, 29(8): 2730–2738.
[45] MCEACHRAN A D, BLACKWELL B R, HANSON J D, et al. Antibiotics, bacteria, and antibiotic resistance genes: aerial transport from cattle feed yards via particulate matter[J]. Environmental health perspectives, 2015, 123(4): 337–343.
[46] XIE J, JIN L, LUO X, et al. Seasonal disparities in airborne bacteria and associated antibiotic resistance genes in PM2. 5 between urban and rural sites[J]. Environmental Science & Technology Letters, 2018, 5(2): 74–79.
[47] ECHEVERRIA-PALENCIA C M, THULSIRAJ V, TRAN N, et al. Disparate antibiotic resistance gene quantities revealed across 4 major cities in California: a survey in drinking water, air, and soil at 24 public parks[J]. ACS omega, 2017, 2(5): 2255–2263.
[48] BENGTSSON-PALME J, LARSSON D G J, KRISTIANSSON E. Using metagenomics to investigate human and environmental resistomes[J]. Journal of Antimicrobial Chemotherapy, 2017, 72(10): 2690–2703.
[49] LIS D O, PACHA J Z, IDZIK D. Methicillin resistance of airborne coagulase-negative staphylococci in homes of persons having contact with a hospital environment[J]. American journal of infection control, 2009, 37(3): 177–182.
[50] HU J, ZHAO F, ZHANG X X, et al. Metagenomic profiling of ARGs in airborne particulate matters during a severe smog event[J]. Science of the Total Environment, 2018, 615: 1332–1340.
[51] LI B, YANG Y, MA L, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes[J]. The ISME journal, 2015, 9(11): 2490–2502.
Research Progress of Antibiotic Resistance Genes in Atmospheric Environment
HE Hong-mei1, ZHANG Wen-bin1,2, ZHANG Xiao-rong2, HE Kai3, ZHANG Li1
(1. School of Materials and Chemical Engineering, Pingxiang University, Pingxiang, Jiangxi 337000, China; 2. Longhui NO.6 Middle School, Shaoyang, Hunan 422208, China; 3. Long Hui County Tobacco Monopoly Bureau, Shaoyang, Hunan 422210, China)
Antibiotic resistance genes (ARGs) have
widespread attention as an emerging environmental pollutant, and there are few studies on ARGs in the atmosphere. The atmospheric environment is a potential reservoir of ARGs, and diseases by antibiotic-resistant bacteria are mainly from airborne transmission. Based on a review of a large amount of domestic and international literature, the paper systematically elaborates the current status of contamination of ARGs in the atmospheric environment, potential route of transmission and their mechanisms and relevant detection methods, and points out the necessity and importance of conducting research on ARGs in the atmosphere in China under the current situation, and puts forward several suggestions for relevant institutions to carry out research on ARGs in aerosols.
antibiotic resistance genes; drug-resistant bacteria; atmospheric environment
X831
A
2095-9249(2021)06-0051-05
2021-11-16
江西省教育厅科学研究项目(GJJ161263)
贺红梅(1984—),江西萍乡人,实验师,硕士,研究方向:环境微生物。
〔责任编校:吴侃民〕
