植物CRISPR/Cas技术研究进展及其在林业科学研究中的应用
2021-05-26胡凯强廖家凯席飞虎高鹏飞魏文桃丁家治顾连峰
胡凯强, 廖家凯, 席飞虎, 高鹏飞, 陈 凯, 魏文桃, 丁家治, 苗 苗, 顾连峰
(1.福建农林大学林学院;2.福建农林大学生命科学学院,福建 福州 350002)
林木物种世代周期长,缺乏便捷有效的遗传转化体系,这对优良种质资源的培育极其不利.传统的林木基因工程受到基因组杂合度高、倍性复杂[1]等条件的约束,无法对基因组进行精准修饰,导致林木物种的遗传多样性难以得到有效利用.随着生物大数据的快速发展,继早期的归巢核酸内切酶(meganucleases)、锌指核酸内切酶(zinc finger endonuclease, ZFN)、类转录激活因子效应物(transcription activator-like effector nucleases, TALENs)以及由铈—乙二胺四乙酸复合物执行切割功能的化学方法[2]之后产生的CRISPR/Cas系统可在基因组中引入定点突变,其组装复杂性、编辑效率等问题得到明显改善,为林木基因功能验证与解析、突变体创制、快速育种等提供参考.
1 CRISPR/Cas系统在植物中的研究现状
1.1 CRISPR/Cas系统的研究历程
科学家最早在负责大肠杆菌(Escherichiacoli)碱性磷酸酶的同工酶转换的iap基因区附近发现了一种“重复—间隔—重复”的基因序列[3].2000年Mojica et al[4]发现多种原核生物均具有这类序列,其间隔序列唯一且长度固定,重复序列通常为包含24~40 bp的短部分回文序列.随后这种特异性片段被命名为成簇规律性间隔短回文重复(clustered regularly interspaced palindromic repeats, CRISPR),两侧的同源基因被命名为CRISPR相关基因Cas[5].Horvath et al[6]发现当病毒入侵时,细菌会整合新间隔序列并对噬菌体产生抗性.研究[7]发现“重复—间隔—重复”中的重复序列来源于细菌和古生菌自身,间隔序列来自入侵的噬菌体病毒等.CRISPR/Cas系统的发展历程如图1所示.2013年Yang et al[8]首先在人类细胞中建立基因编辑工具,实现靶基因的多重编辑,CRISPR/Cas系统自此诞生.同年,在拟南芥(Arabidopsisthaliana)、水稻(OryzasativaL.)等植物中利用CRISPR/Cas9系统成功进行基因靶向实验[9-11],这是CRISPR/Cas系统在植物研究中的首次报道.2014年由CaMV 35S启动子驱动的Cas9系统靶向甜橙(CitrussinensisL.)CsPDS基因,叶片通过柑橘黄单胞柑橘亚种[12](Xanthomonascitrisubsp.citri, Xcc)促使农杆菌渗透,该系统在林木物种中首次得到应用[13].虽然叶片出现白化斑点,但由于它是瞬时遗传转化,并没有获得稳定遗传的编辑后代.随着研究的深入,该技术逐渐在调控植物内源基因表达[14]、性状改良[15]以及抗病毒研究[16]等领域得到广泛应用.
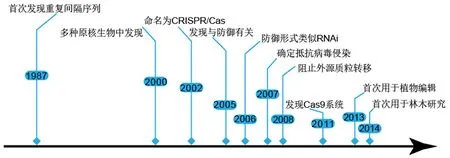
图1 CRISPR/Cas技术研究的关键事件Fig.1 Key events for CRISPR/Cas technology
1.2 CRISPR/Cas系统的缺点及对策
1.2.1 序列限制 Cas蛋白会识别不同的原间隔序列临近基序(protospacer adjacent motif, PAM),但是PAM序列在基因组上的分布频率有限,在一些目的基因上无法寻找到合适的位点,这严重制约了全基因组范围内的编辑.开发识别不同PAM序列的新Cas蛋白则可以有效降低PAM序列的依赖性.通过特异性识别NAG、NAGG和NAAG等PAM系统有效扩展了CRISPR/Cas靶向基因组的范围[17].2019年CRISPR/Cpf1(CRISPR/Cas12a)系统首次在木本植物葡萄柚(C.paradisi)中成功编辑CsLOB1等位基因,解决了Cas9系统因受到单核苷酸序列多态性限制,其单个sgRNA难以修饰两个等位基因的难题[18].
1.2.2 脱靶、编辑效率低 脱靶是指Cas蛋白对目标靶基因以外的其他序列进行切割,从而产生不必要的突变.Yang et al发现SpCas9-NG系统不仅会靶向水稻基因组,而且会靶向sgRNA本身,这极大地增加了脱靶风险[19].科研人员针对这一问题已搭建多种生物信息学平台,以设计高特异性gRNA元件.Xie et al开发的CRISPR-PLANT可为8种植物基因组预测低脱靶位点的sgRNA[20].基因编辑工具包CRISPR-GE不仅能用于设计目标sgRNA,还可以用于预测脱靶位点以及后续的突变分析[21].通过开发突变体Cas9-HF1产生较弱的Cas9-DNA关联度来确保核酸酶仅在完全匹配的DNA位点进行切割[22].
该技术还存在编辑效率低等问题,这与Cas9的密码子优化以及靶点处的GC含量有很大关系[23,24].同种或相近物种密码子优化和70%左右GC含量的靶位点有助于提高打靶效率.除此之外,温度、光照等环境因素也会影响编辑效率,因此该技术目前基本上局限在实验室内而无法得到大规模田间应用.科学家们如今已经通过开发温度敏感型CRISPR/Cas12a系统[25]等来提高植物基因编辑效率.
1.3 CRISPR/Cas技术在植物研究中的新方向
1.3.1 单碱基编辑技术 单碱基编辑技术能够实现对单一碱基的编辑,与传统的CRISPR/Cas技术相比具有精准且不会产生随机突变等优点.最初的单碱基编辑系统是可以实现C·G到T·A转换的胞嘧啶编辑器(CBEs).随后Liu et al[26]开发了腺嘌呤碱基编辑器(ABEs),介导基因组DNA中A到G的转化.Qin et al[27]在棉花(GossypiumhirsutumL.)中实现了由A·T到G·C的精确编辑,由GhBE3产生的碱基编辑可从T0代遗传到T1代.Wen et al[28]在玉米(ZeamaysLinn.)中建立优化的基于靶向差异sgRNAs的代理报告系统(discriminated sgRNAs based surrogate system, DisSUGs),以实现单碱基编辑细胞的高效富集,提高了突变细胞的筛选效率.单碱基编辑技术的应用使植物的遗传改良更加精确与安全.
1.3.2 RNA碱基编辑 RNA编辑是一种转录后水平的基因修饰,依赖PPR、MORF等蛋白家族发挥作用,可以有效提高编辑的精确性,增加基因产物的多样性.Zhang et al[29]首先证明RNA在哺乳动物中可以被编辑,并使用失活的Cas13蛋白(dCas13)建立了一种没有严格序列限制、可以编辑包含致病性突变的全长转录本的系统.Jesser et al[30]利用CRISPR/Cas13a能够靶向噬菌体基因组的特性在烟草中实现对芜菁花叶病毒(turnip mosaic virus,TuMV)和GFP融合转录本的干扰,为植物的抗病毒研究提供了一种新的技术手段.植物中的RNA编辑多存在于自身的细胞器内[31],目前人工开发的编辑系统非常少,应用前景广阔.
1.3.3 系统优化 传统的CRISPR/Cas系统使用固定的几种启动子驱动Cas蛋白和sgRNA.常用的玉米泛素Ubi等启动子太长会导致单一载体只能连接较少的sgRNA,否则会导致载体过大,难以转化和降低编辑效率.基于此,科研人员在OsU3/U6的基础上筛选优化出4个更加精简的启动子(mOsU3、mOsU6a、mOsU6b和mOsU6c).使用这些启动子能够提高单一载体sgRNA表达盒的组装数量,进而提高多靶位点编辑的能力[32].此外,Zhang et al[33]在柑橘(CitrusreticulataBlanco.)中使用YAO基因的启动子,借助CRISPR/Cas9系统进行了快速高效的基因编辑,这类启动子在植物花粉中高表达,有助于将编辑性状遗传给后代.这些新元件为CRISPR/Cas技术的深入研究以及植物的遗传改良等提供了便利.
2 CRISPR/Cas系统的递送与编辑鉴定方式
2.1 CRISPR/Cas系统在植物中的递送方式
将CRISPR/Cas系统递送进植物细胞内是该系统发挥作用的前提.目前植物领域应用最多的主要有传统的转基因与非转基因递送方法,纳米材料的应用则提供了更加便捷高效的递送方法.
2.1.1 转基因递送 转基因递送方法主要是农杆菌介导的质粒转化,这是最为常见且可以获得稳定表达后代的一种方式.Zhu et al[34]通过农杆菌介导转化山橘(Fortunellahindsii)愈伤组织,靶向编辑其F.hindsii基因.Lin et al[35]通过PEG介导转化绿竹(Bambusaoldhamii)、粟(Setariaitalica)、玉米等材料的原生质体进行基因编辑.Andersson et al[36]通过对分离的马铃薯原生质体进行瞬时转化和再生,对淀粉合酶相关基因进行功能完全性敲除.此外,还可通过基因枪等粒子轰击的方法将该系统导入到植物体内,但是昂贵的费用限制了其应用.
2.1.2 非转基因递送 转基因法导致外源基因的随机插入,因此具有潜在的生态安全性隐患.而体外组装的核糖核蛋白复合体(RNPs)可以在递送进细胞后立即切割靶标,无需转录和翻译,迅速降解,从而避免转基因植株的产生,提高生物安全性.Woo et al[37]将体外预组装的RNPs成功递送到拟南芥、莴苣(Lactucasaliva)等植物中,并在再生植物中达到46%的突变效率.CRISPR/Cas9相关的RNP也已经成功递送到葡萄(Vitisvinifera)、苹果(Maluspumila)等林木物种原生质体当中,实现了无转基因编辑[38].此外,农杆菌被用来介导质粒转化,在烟草(Nicotianatabacum)中进行瞬时表达[39],在再生白化植株中用特异性PCR检测到5株植株不含T-DNA.这为CRISPR/Cas系统的非转基因递送提供了一种新方法,尤其适合童期较长、多靠营养繁殖的林木物种.
2.1.3 新颖的递送方式 纳米材料为CRISPR/Cas的递送开辟了另一条路径.Doyle et al[40]通过叶面喷施含有CRISPR/Cas9质粒包被的纳米颗粒,实现了SPO11基因的编辑.该技术简单、高效、成本低廉,使非模式植物和转化困难植物的基因组编辑成为可能.发育调控因子(DRs)可促进植物在叶腋处的再生,利用该原理将分别含有DRs和sgRNA的表达载体共转化稳定表达Cas9的烟草,获得了白化芽并且成功遗传给后代[41].Daniel et al[42]提前生成过表达Cas9的本氏烟,然后将能够表达sgRNA的RNA病毒转化到提前生成的植株上,利用病毒可以迁移到生殖器官的特点在后代中创造有效突变.这两种方法省时高效,可以消除组织培养的冗长过程和遗传转化的壁垒.
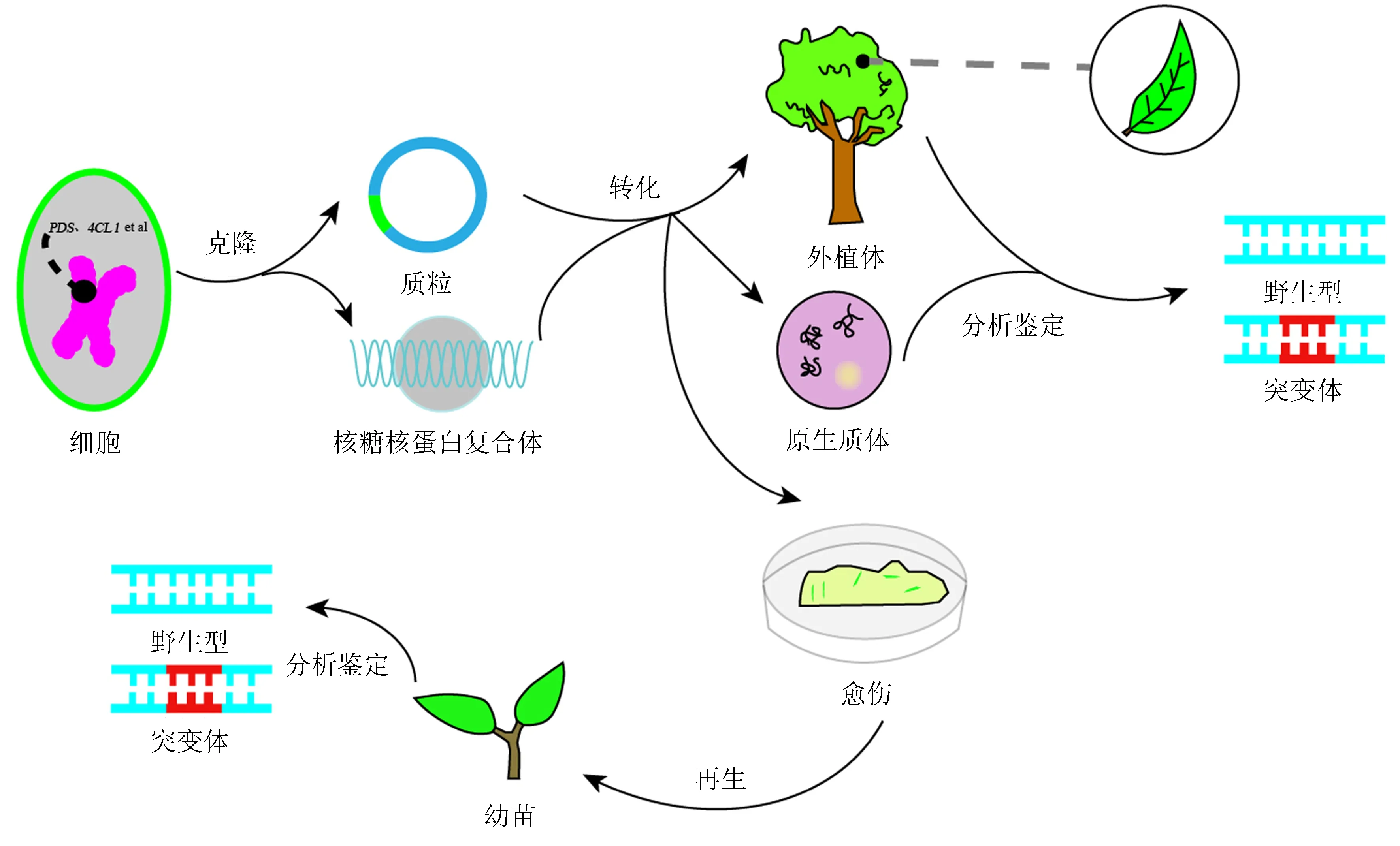
图2 CRISPR/Cas试验流程Fig.2 The flowchart of CRISPR/Cas experiment
2.2 CRISPR/Cas系统在植物中的编辑鉴定
CRISPR/Cas系统的编辑中NHEJ途径具有随机性特点,容易产生非预期目标突变.同时样本当中还混合着大量非编辑群体需要进行筛选.对多种木本植物的突变类型进行分析,结果表明插入多局限在1~2 bp,其中1 bp的插入是最常见的,且缺失范围大[43].因此,对CRISPR/Cas系统的编辑情况进行鉴定显得尤为重要.目前可应用于植物的鉴定方法主要有以下几种.
2.2.1 非测序方法 目前常用的突变鉴定方法有T7EI(T7 endonucleaseI)、Surveyor核酸酶分析、PCR-RE(PCR/restriction enzyme)、HRM(high-resolution melting assay)和ACT-PCR法等.T7EI的原理与Surveyor核酸酶分析法相似,都是利用特异性核酸酶切割异源双链错配DNA分子.Arai et al[44]在烟曲霉菌(Aspergillusfumigatus)中用Surveyor酶鉴定出cyp51A基因的单点突变株系,该方法在植物基因编辑研究中尚未见报道.PCR-RE法的原理是将目标DNA进行酶切,突变的靶序列会被富集,凝胶电泳后出现残留条带[45].Fengrui et al[46]对葡萄进行T7EI和PCR/RE检测,发现当sgRNA中GC含量为65%时,CRISPR/Cas9系统会产生最高的编辑效率.HRM法是通过将突变片段的构象和熔解曲线与野生型相比,将发生改变并且在PAGE胶上的迁移率发生变化的鉴定为突变体[47].Denbow et al用HRM法检测到了拟南芥单碱基对的插入和缺失[48].ACT-PCR利用在临界温度下退火的原理鉴定编辑样本,特异性引物包含切割位点,在突变体中无法进行有效扩增.Wang et al[49]用该方法在水稻中快速鉴定出了单突变体和多突变体.此外,还可用纯化的核糖核蛋白复合体对目标PCR产物进行切割(PCR/RNP).Gao et al[50]用PCR/RNP方法和Sanger测序成功地对六倍体小麦(TriticumaestivumL.)和水稻的突变结果进行基因型分型.
T7EI和Surveyor核酸酶分析法能够快速检测突变事件的发生,但是这两种方法无法区分野生型和纯合突变,也不能区分双等位基因突变和杂合突变体[51].PCR-RE法操作简便,检测灵敏度较高,但是受到限制性酶切位点的限制.ACT-PCR仅需一个PCR步骤便可进行凝胶电泳,确定编辑情况,可用于准确测定培养细胞的突变频率.HRM法既可以对已知的突变类型进行分析,又可以对未知的突变进行筛选,但是只能分析纯度单一的小片段DNA,无法对长片段和杂合片段进行分析鉴定.相对而言PCR/RNP法更加灵敏,无需限制酶切位点,比PCR/RE方法更适用,可以区分野生型和纯合突变、双等位基因突变与杂合突变,但是RNP的制备也较复杂.
2.2.2 测序方法 测序法包括Sanger测序和高通量测序两种.Sanger测序能够鉴定一些纯合突变,提供突变类型的详细信息,但是对多倍体基因组突变以及杂合突变的检测能力较低.Giorio et al[52]在番茄(Solanumlycopersicum)中使用Sanger测序技术检测到的编辑效率为84%,但未检测到嵌合体.Ma et al[53]考虑到杂合突变类型的测序会出现双峰现象,开发简并序列解码(degenerate sequencedecoding, DSD)方法,能够快速解码测序图中的双峰信息,但是这种方法只适用于小规模的解码.高通量二代测序(nextgenerationsequencing, NGS)技术则可以对一些嵌合突变或者大规模的突变样本进行鉴定分析[54],尤其适合基因组倍性复杂、遗传杂合度高的林木物种.基于NGS的Hi-TOM平台可对水稻中CRISPR/Cas9诱导的突变进行高通量分析,从而检测多个靶位点的突变,尤其适合对复杂的嵌合突变进行鉴定[55].Yan et al[56]通过改进MassARRAY和靶向捕获测序技术在基因组庞大复杂的玉米中实现高通量和低成本的靶标突变鉴定.高通量测序的方法虽然可以同时进行大规模的测序和分析,但费用高,耗时长[57].CRISPResso等工具的开发则在逐步缩短时间,降低成本,解决高通量测序过程中带来的难题[58].
2.2.3 荧光检测方法 荧光标签的常规应用主要是作为报告基因融合到载体T-DNA插入区来检测处理的后代是否发生CRISPR/Cas元件的插入,便于进一步筛选编辑后代[59].Yi et al[60]将GFP融合到CRISPR转化载体上,了解转化的香蕉(MusananaLour.)再生苗是否发生CRISPR/Cas元件的整合.这种方法虽然不能直接检测到编辑事件的发生,但有助于进一步筛选突变植株.Ye et al[61]在六倍体麻竹(DendrocalamuslatiflorusMunro)中将含有靶向mGFP突变位点的sgRNA质粒与含有mGFP的质粒共转化原生质体,在育孵72 h后成功观测到了绿色荧光,证明mGFP的突变位点复原,CRISPR/Cas系统可靶向编辑外源基因.该方法使得编辑事件可视化,适合原生质体转化效率较高的物种.Leblanc et al[62]通过突变连接在内源基因后的GFP来测定突变效率.此方法通过观察报告基因的表达量同样可以直观地进行突变鉴定,但是需要有稳定过表达的植株;对于缺乏有效的遗传转化体系和再生周期较长的林木物种来说不太适用.
3 CRISPR/Cas系统在林木中的应用
在林木中利用CRISPR/Cas系统通过农杆菌转化法获得稳定的编辑后代,目前该系统只应用在杨树(PopulusL.)[63]、山豆麻属植物(Parasponiaandersonii)[64]、麻疯树(Jatrophacurcas)、橡胶树(Heveabrasiliensis)以及苹果[65]、甜橙[13]等少数几个林木物种.该技术在林木中的研究应用包含以下几个方面.
3.1 技术体系的探索与建立
许多林木树种在CRISPR领域的研究尚停留在技术体系摸索阶段.Muhammad et al[66]从Cas蛋白的选择、sgRNA的设计、突变体验证、在枣椰树(PhoenixdactyliferaL.)中存在的局限性等方面论述了对椰枣树进行多重基因编辑的可行性,以期为使用该技术培育抵抗胁迫的枣椰树提供理论指导.鹅掌楸(Liriodendronchinensis)是我国特有的珍稀物种,目前其相关的CRISPR载体构建工作已经展开[67].唐雨薇等[68]针对茶树(CamelliasinensisL.)的咖啡碱合成酶编码基因(TCS)构建了CRISPR双靶点表达载体,通过优化茶树的遗传转化和组培再生体系,成功将载体导入到茶树体细胞胚中并获得插入突变[69].2015年,乔治亚大学的研究人员在毛白杨(Populustomentosa)中使用Cas9成功敲除木质素合成相关酶基因,结果表明该系统在杨树中具有较高的效率和序列特异性[70].这是CRISPR/Cas系统在木本植物中得到编辑后代的首次成功应用,自此打开了林木基因编辑的大门.
木薯(ManihotesculentaCrantz)常规的育种方法受到生命周期长、杂合度高和近亲衰退的限制[71].Odipio et al[72]开发CRISPR/Cas9技术靶向木薯MePDS基因外显子,将农杆菌介导的表达载体转化木薯胚性愈伤组织,AtU6和35S被用来构建该表达载体;编辑后代出现白化和矮化表型,并且白化植株存活不会超过3个体外繁殖周期.筛选分析发现,碱基替换比插入和缺失频率高,在T0代中纯合子突变达到了21.1%,杂合子突变达到了89.5%[72],在6~8周便可以检测突变事件的发生,可为再生周期长的树种的编辑结果提供快速的评估和优化.2018年,Lin et al[35]使用CRISPR/Cas9载体靶向绿竹的PDS基因,并进行原生质体转化,48 h后发现其转化效率仅为6.6%,这些突变均为1~13 bp的缺失或1 bp的替换;用限制性内切酶消化扩增子后检测富集的突变达到71%.该系统分别用OsU3、OsU6和35S启动子驱动.虽然没有获得再生植株,但是在原生质体中成功地对内源基因进行编辑,为林木相关基因功能鉴定提供了一种便捷的手段.这些初级阶段的工作为后续使用CRISPR/Cas系统培育具有优良生产特性的新品系奠定了基础.
3.2 生长发育相关基因的功能解析
Muhr et al[73]利用CRISPR/Cas9系统生成该团队先前鉴定的杨树候选基因BRANCHED1-1和BRANCHED2-1突变体,结果显示BRANCHED1-1突变体的芽伸长;BRANCHED2-1突变体具有极端的芽生长表型,并且在每个结节处都有两个异位叶,这对以后杨树芽伸长生长甚至是株型调控具有重要的参考意义.Christin et al敲除银灰杨(P.tremula×alba)的UGT71L1基因后,其毛状根中的水杨酸含量明显下降,表明该基因是杨树水杨酸合成途径中的关键基因[74].但是另一个基因UGT78M1并未敲除成功,表明林木sgRNA的设计要求更为严苛、精确.山豆麻属植物Parasponiaandersonii是除豆科植物外唯一可以进行固氮的木本植物.Zeijl et al对该植物与固氮有关的PanHK4、PanEIN2等候选基因进行了定点诱变.研究人员用CRISPR/Cas9系统构建二元转化表达载体,使用AtU6p驱动小核RNA,并用35S驱动拟南芥密码子优化的Cas9.使用农杆菌侵染外植体,然后将外植体与再生的愈伤分开,激发芽的形成;新生芽可在4~6周内进行无性繁殖,3个月内生成T1代小苗.基因分型显示,超过85%的转化子含Cas9并且有50%以上的发生突变.进一步试验分析表明,稳定的根癌农杆菌介导的转化与CRISPR/Cas9基因组编辑相结合可以有效地用于该物种的反向遗传分析[64].Ye et al通过农杆菌侵染愈伤组织的方式编辑麻竹,突变掉了一个响应赤霉素信号的基因GRG1,与野生型相比再生植株的节间长度明显增长,株高增大[61],这对于研究禾本科竹子的快速伸长和生长具有重要意义.
用材树种的生长发育关键在于木质部的生长发育.对桉树(Eucalyptusspp.)与木材生长的相关基因CCR1和IAA9A用毛状根转化的方式进行编辑,结果表明几乎所有的毛状根都被编辑,但等位基因的编辑效率随目标基因的不同有很大差异[75].研究发现,SNP位点越靠近PAM,越有可能造成编辑无效.这对具有高度杂合基因组的林木物种来说,其sgRNA的设计必须更加精确、特异,必须考虑到频繁出现的SNP才能实现有效的基因组编辑[70].对基因组中含有大量SNP的物种来说,其突变鉴定必须更加小心谨慎,以防受到干扰.研究同时还发现CRISPR/Cas系统在杨树中会高效生成高特异性的双等位基因突变.Luo et al[76]利用CRISPR/Cas9系统生成了毛白杨的PtoDWF4突变体,经分析发现,PtoDWF4突变体中次级细胞壁的生物合成降低,细胞层数和木质部面积都大幅度降低.该研究解析了杨树木质部发育的关键基因,为以后培育高产量木材的杨树品种打下了基础.
3.3 抗逆相关基因解析
Luo et al通过编辑ProPtrWRKY18和ProPtrWRKY35证实毛白杨中的这两个基因参与对生物胁迫的响应,突变体对真菌的抗性明显下降[77].速生杨树容易受到干旱等胁迫[78],Luo et al在2019年还敲除了毛果杨的PtrADA2b等基因,揭示了组蛋白H3K9ac参与干旱胁迫响应的关键机制[79].2017年,刘慧慧等[80]对美国白蛾(Hyphantriacunea)翅形成相关基因Wnt-1进行编辑,结果显示该基因随着胚胎发育其表达量逐渐下降,这对于解析其基因功能、降低美国白蛾的迁徙能力具有重要意义.2019年Li et al[81]对美国白蛾生殖相关基因用CRISPR/Cas9技术进行定点编辑,突变体后代表现出特异性不育.该技术虽然没有直接应用于林木自身,但对林木虫害防治具有重要的生物学意义.
柑橘是最早应用CRISPR/Cas技术的林木物种之一,其遗传改良受到物种生长缓慢、多胚和单性结实的限制[82].2019年,Jia et al[18]利用CRISPR/cpf1系统靶向了柑橘溃疡病易感基因CsLOB1并生成突变再生植株.至此,使用CRISPR/Cas技术对柑橘进行遗传改良又向前迈进了一步.上述研究通过对林木抗逆基因或与之相关的病虫害基因的解析为林业生产中防治大袋蛾(Claniavartegata)、松材线虫(Bursaphelenchusxylophilus)等林木虫害[83],培育抗逆新种提供了新思路.
3.4 非转基因育种的探究
因涉及到转基因安全问题,非转基因技术手段在水果类植物中应用较多.科学家针对葡萄易感基因MLO-7和苹果的DIPM-1、DIPM-2、DIPM-4基因进行设计,将纯化的CRISPR/Cas9核糖核蛋白直接递送进两种植物的原生质体进行定向基因组编辑.靶向深度测序以及生物学重复统计分析发现,葡萄的突变率为0.1%,而苹果的突变率是0.5%~6.9%.虽然突变频率较低,但该方法不会产生转基因残留,这为培育无外源DNA污染的新品种铺平了道路[38].
除水果作物之外,橡胶树异花授粉、童期长、缺素等容易导致生长缓慢[84],传统的育种方法难以满足生产橡胶原材料的需求.Hua et al针对橡胶树花期相关基因FT和TFL1设计sgRNA,然后通过将RNP转化原生质体的方法成功建立了橡胶树CRISPR/Cas9系统,为培育非转基因、高产橡胶新品系提供参考[85].
3.5 林木生殖相关基因研究
麻疯树种子提炼的生物油可以替代化石燃料,被认为是一种潜在的生物能源.但是现阶段麻疯树的种子产量很低,难以进行规模化生产.Xu et al报道外源细胞分裂素可以提高雌雄花的比例进而提高种子产量[86].为了探究细胞分裂素对麻疯树成花机制的调控作用,Xu et al利用CRISPR/Cas9系统突变该物种中的一个细胞分裂素合成相关基因JcCYP735A;突变体生长迟缓,其内源反式/顺式玉米素与核苷浓度显著降低,细胞分裂素含量下降.这些结果将有助于细胞分裂素代谢基因功能的进一步研究,提高麻疯树种子的产量[87].
Charrier et al[88]研究了梨(PyruscommunisL.)的PcTFL1.1基因,编辑体多为嵌合突变类型且呈现出早花表型.对该基因的研究有助于培育提前开花结实的新品种和加速梨树的快速生殖繁育.Elorriaga et al[89]对杨树的花分生组织同源基因LEAFY和两个花器官同源基因AGAMOUS进行编辑,没有发现脱靶事件,转化子达到了77.5%的突变效率.杨树多为无性生殖,其遗传力衰退严重,该研究对于研究杨树的开花结实和实生生殖意义重大.
CRISPR/Cas系统在木本模式植物杨属中的应用见表1[90-103].
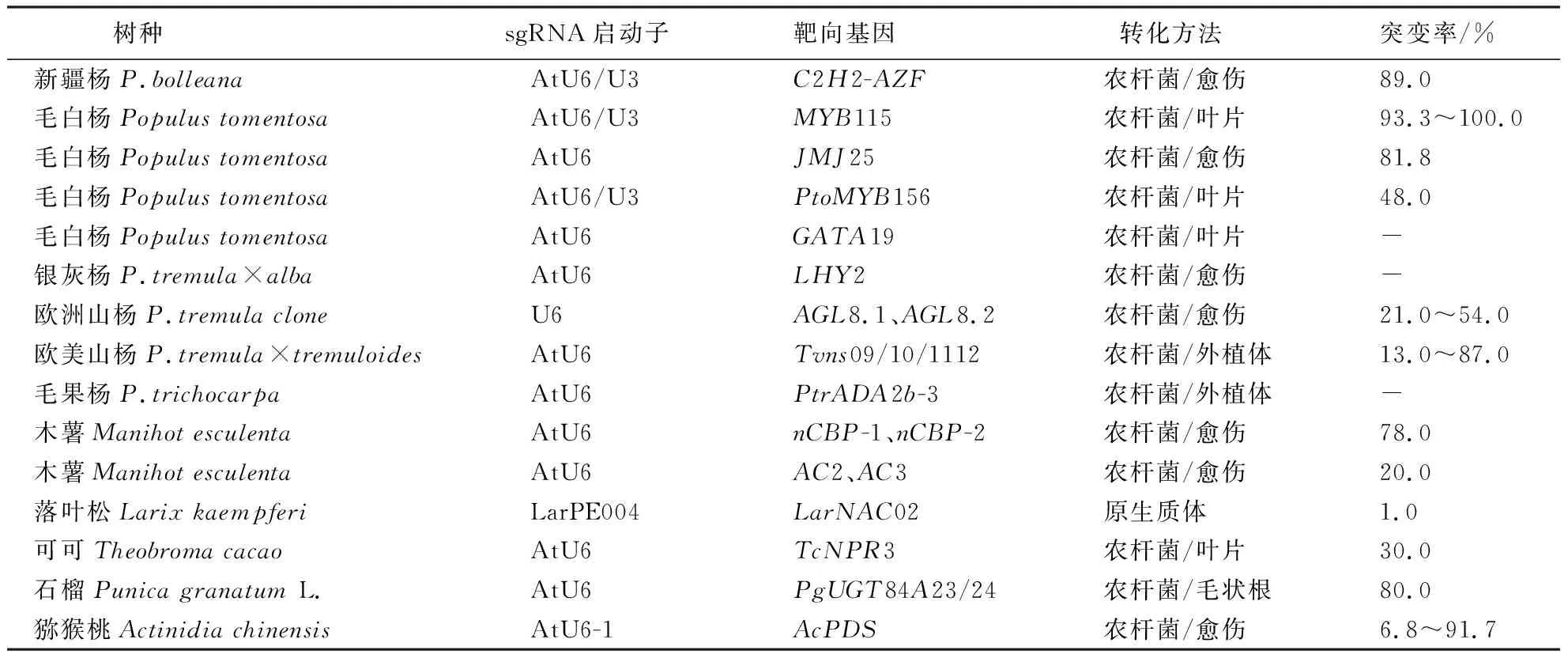
表1 CRISPR/Cas系统在杨树等林木物种中的研究与应用 Table 1 Research and application of CRISPR/Cas system in poplar and other forest species
4 展望
针对CRISPR/Cas系统的不足,科学家如今已经开发出许多CRISPR/Cas新技术.Benjamin et al设计的Cas9突变体SpRY可以靶向几乎所有的PAM序列,且在人类细胞中获得了以前无法获得的与疾病相关的遗传变异[104].Gao et al在水稻和小麦中建立的引导编辑系统(plant prime editing, PPE)能够实现12种类型的精准编辑,为植物的遗传育种提供丰富的突变类型,生成更多可供选择的优良性状[105].HDR途径、单碱基编辑器、RNA编辑和这些更加高效精准的CRISPR/Cas新技术目前只在人类医学、动物和农作物当中得到应用,在木本植物研究中应用较少或未有相关报道.这些技术将有利于林木基因序列多态性的高效开发与利用.
虽然麻竹、橡胶树等物种的原生质体实现了基因编辑,但大多数林木物种的原生质体制备、高效转化以及再生仍然是一个技术难题.而且RNP法也多依赖于原生质体的高效转化与再生.因此,开发高效的原生质体转化和再生体系对林木基因编辑新品系的开发具有重要的助推作用.同时,CRISPR/Cas系统在林业上的研究多集中在经济水果中,局限于广谱性基因的研究,对于用材和生态保护类物种的研究较少.目前仅对裸子植物中的落叶松进行相关研究[100],林木裸子物种未见相关研究报道.这与木本植物基因组测序工作相对滞后、靶位点设计分析平台收录的林木物种基因组数据不够完善、多数林业品种存在遗传转化壁垒等有关.因此建立健全开放的网络数据库,构建高通量突变体文库,加快林业良种的基因组测序,收录更多林业生产上常见品种的基因组信息,探索建立稳定高效、耗时短、成本低廉的遗传转化体系等,将会极大拓展CRISPR/Cas系统在林业科学尤其是木本良种选育中的应用范围.
在林业科学研究当中,CRISPR/Cas系统多应用于林木物种自身的基因编辑,而在与其相关的生物胁迫上的应用十分有限.当然,这涉及到昆虫学、微生物学和树木病理学等其他学科的发展.只有多学科协同发展,才能使CRISPR/Cas技术更好地服务于木本植物病虫害研究,促进林业科学发展.此外,转基因递送方式会带来外源基因的插入与表达,木本植物的世代周期一般很长,很难通过后代群体分离而剔除.这无论是对植物本身还是对生物环境都会带来不可预估的影响.在生物安全备受关注的今天,除RNP法外,更多无外源DNA污染的遗传转化方法的开发已是大势所趋.CRISPR/Cas技术的安全发展需要国家及时出台相关法律法规,对基因编辑植物进行监管.随着技术的进步,CRISPR/Cas系统必将更加安全完善,使植物遗传改良向着有利于人类社会的方向发展.
