鹅细小病毒辽宁分离株全基因测序与遗传演化分析
2021-05-15李阁锦郭维军曹祁峰周铁忠
李阁锦,郭维军,曹祁峰,周铁忠,李 冰*
(1. 锦州医科大学畜牧兽医学院,辽宁锦州121000;2. 锦州市动物疫病预防控制中心,辽宁锦州121001)
小鹅瘟又称鹅细小病毒病,由鹅细小病毒(goose parvovirus,GPV)引起4~20日龄雏鹅的一种急性或亚急性败血症,以发生渗出性肠炎为主要特征,传播快、病死率高。本病最早由方定一于1956年发现,在我国江苏扬州地区流行,并首次报道该病的流行特点和临诊特征[1]。1961年,方定一团队对该病病原进行系统研究,确定该病原是一种和新城疫、鸭病毒性肝炎无关的病毒,建议命名为小鹅瘟病毒[2]。1967年,匈牙利学者Derzsy利用13日龄鹅胚成功分离出小鹅瘟病毒[3]。1973年,Schettler 利用鹅胚成纤维细胞也成功分离到该病毒,病毒的理化特性与其他已知的禽类病毒不同,具备细小病毒的特征,建议将该病毒归类为细小病毒[4]。1974年,世界家禽学会(World's Poultry Science Association,WPSA)为纪念Derzsy 对小鹅瘟研究所做出的贡献,将该病命名为Derzsy's 病[5]。1979年,国际病毒分类委员会(The International Committee on Taxonomy of Viruses,ICTV)第3次报告将该病毒正式命名为鹅细小病毒[6]。
GPV是细小病毒科(Parvoviridae)成员,归属于细小病毒亚科(Parvovirinae)、依赖病毒属(Dependovirus)、雁形目依 赖 细 小 病 毒1 型(Anseriform dependoparvovius 1)[7]。GPV为单股线状DNA病毒,基因组全长约5 kb,编码区依次编码非结构蛋白NS1、NS2 和结构蛋白VP1、VP2、VP3[8],非编码区由回文序列(inverted terminal repeats,ITR)组成位于编码区两侧,依次为5'-ITP-VS1-VS2-VP1-VP2-VP3-ITR-3'。非结构蛋白NS1 和NS2,主要是参与病毒的转录与复制[9];结构蛋白VP1、VP2和VP3共同组成衣壳蛋白。Tullis等[10]报道,VP1蛋白在病毒感染中发挥重要作用,可以引导衣壳蛋白进入细胞核,与宿主细胞受体结合。VP3是衣壳蛋白的主要组成部分,可占衣壳蛋白总量的80%,内含GPV 主要抗原决定簇,是诱导机体产生中和抗体的主要蛋白[11]。ITR 为倒置重复序列,位于基因组的两端,头部可以折叠形成U型发夹结构,ITR主要参与病毒复制与组装的过程[12]。本研究对辽宁地区分离的鹅细小病毒进行全基因测序,并与国内外代表毒株进行序列比对分析,为辽宁地区鹅细小病毒的分子流行病学研究提供参考。
1 材料与方法
1.1 试验材料
鹅细小病毒由辽宁省锦州市疾病预防控制中心分离保存;病毒基因组DNA提取试剂盒、质粒小提试剂盒购自天根生化科技有限公司;2×Taq Master Mix(含染料)购自北京康为世纪生物科技有限公司;快速琼脂糖凝胶DNA回收购自南京维诺赞生物科技有限公司;DL2000 DNA Marker、pMD-18T Vector 购自宝生物工程有限公司;DH5α感受态细胞购自北京索莱宝科技有限公司。
1.2 试验方法
1.2.1 病毒总DNA的提取
取200 μL 细胞培养物于灭菌1.5 mL 离心管中,用病毒基因组DNA 提取试剂盒,按照使用说明书从细胞培养物中提取病毒总DNA后,置于-20 ℃保存备用。
1.2.2 引物的设计与合成
根据GenBank 中GPV 毒株DY16(登录号:MH209633)设计8对引物,对分离的病毒全基因序列进行分段扩增,引物信息详见表1,由上海生工生物工程股份有限公司合成。
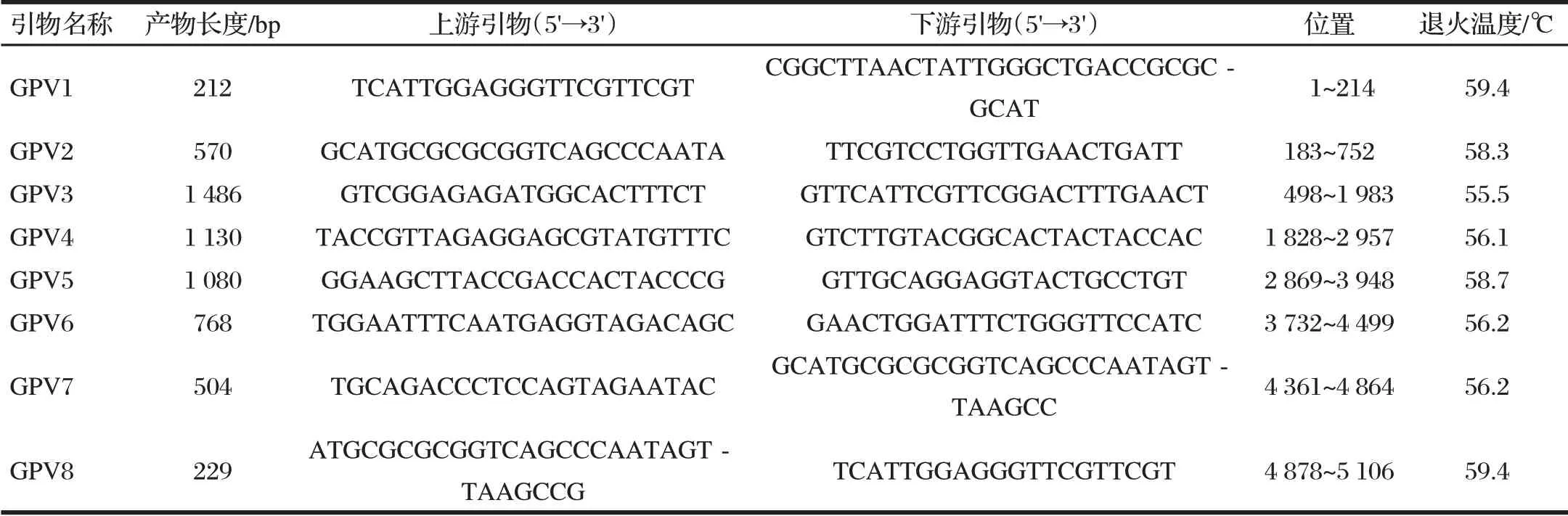
表1 全基因引物序列Tab.1 Whole gene primer sequence
1.2.3 全基因序列扩增及克隆测序
采用PCR 方法对全基因序列进行分段扩增。PCR反应体系(30 μL)如 下:2×Taq Master Mix 15 μL、ddH2O 10 μL、引物2 μL、模板DNA 3 μL,按照表1 中退火温度进行扩增,扩增产物进行胶回收,纯化后产物连接到pMD-18T 载体上并转入DH5α 大肠杆菌感受态细胞中,涂布含氨苄西林(100 μg/mL)的LB 固体培养基上进行筛选,过夜培养12~16 h,挑取单个菌落,经PCR 验证为阳性,使用质粒小提试剂盒提取质粒,放于-20 ℃保存,将所提阳性质粒送至上海生工生物科技有限公司进行测序,每个样品重复扩增及克隆测序3 次。
1.2.4 全基因序列比对分析(见表2)
测序结果经NCBI-BLAST 比对正确后,采用DNAMAN 进行各段序列拼接,获得完整的全基因序列。采用MEGA 7.0、DNAStar 7.0将辽宁分离株鹅细小病毒全基因序列与GenBank 中公布的国内外代表毒株进行序列 比对分析,并制作进化树。
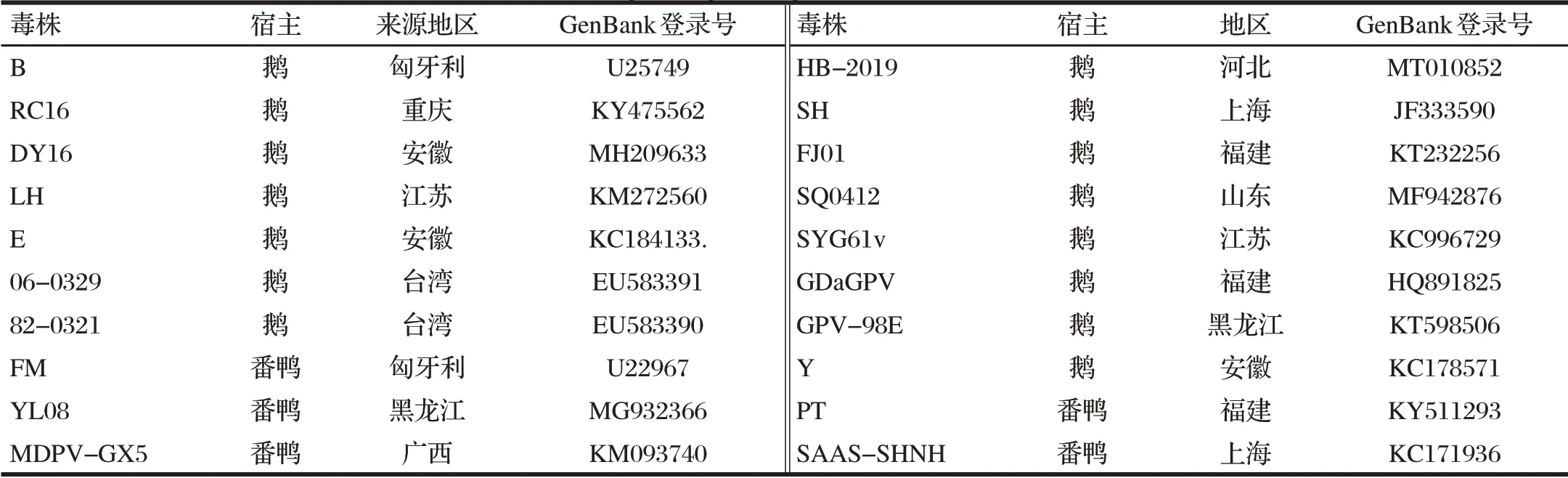
表2 全基因比对代表毒株信息Tab.2 Full gene comparison representative strain information
2 结果与分析
2.1 GPV 辽宁分离株全基因各片段PCR 扩增结果(见图1)
由图1 可知,利用PCR 方法,成功扩增出GPV 辽宁分离株全基因各片段,PCR 产物经琼脂糖凝胶电泳鉴定,目的片段与预期片段大小基本一致。
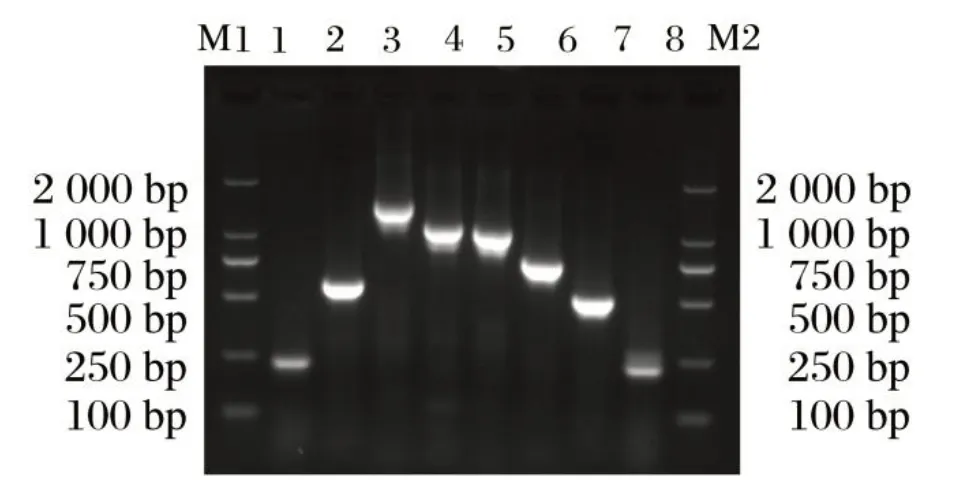
图1 GPV辽宁分离株全基因各片段PCR产物Fig.1 PCR products of all gene fragments of GPV Liaoning isolate
2.2 GPV 辽宁地区分离株全基因序列拼接与分析(见表3)
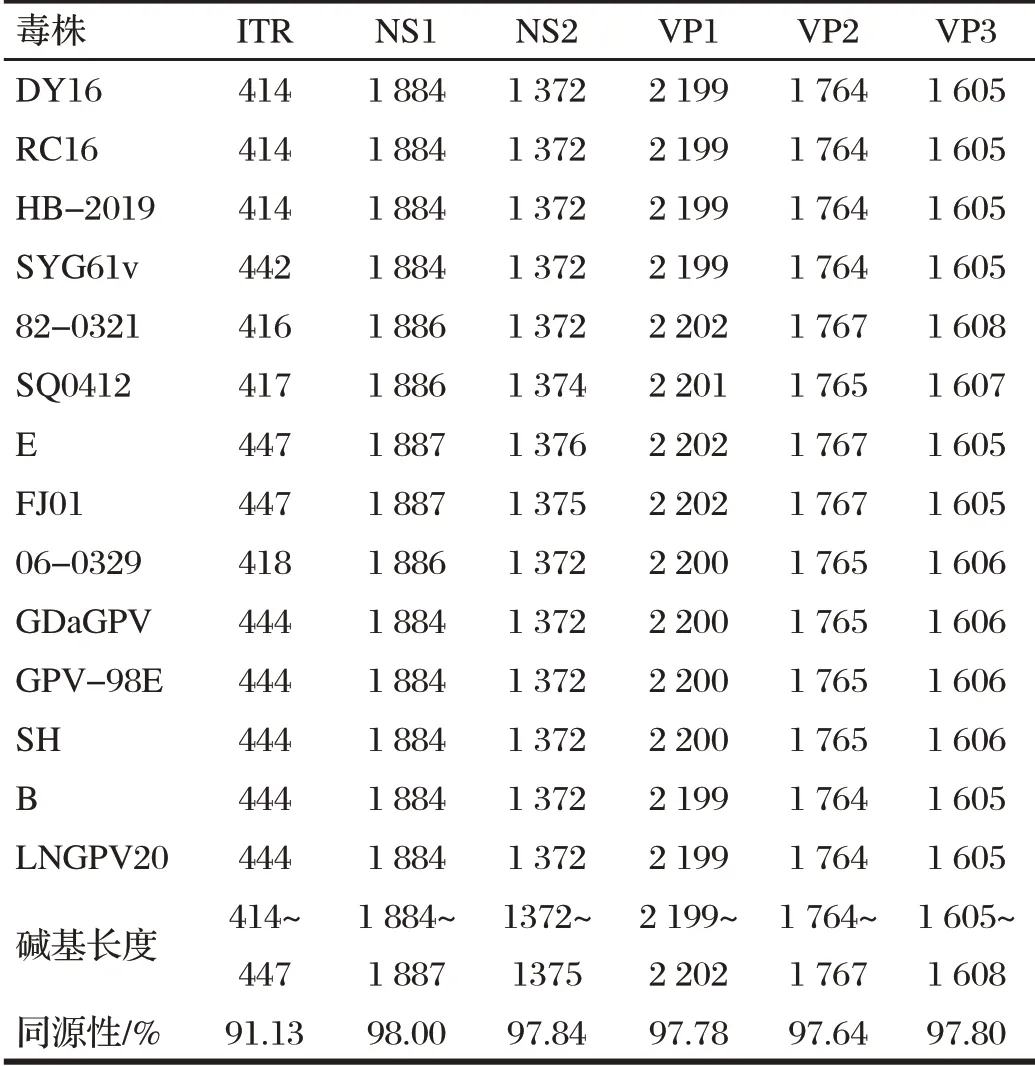
表3 LNGPV20毒株与代表毒株各段基因序列比对Tab.3 Alignment of gene sequences between LNGPV20 strain and representative strain
采用DNAMAN 软件,将测序结果进行拼接、校正,经BLAST 验证获得GPV 辽宁分离株全基因序列,命名为LNGPV20。分离毒株基因全长5106 bp,共有两个非结构蛋白NS1、NS2和3个结构蛋白VP1、VP2、VP3,ITR序列长为444 bp。由表3可知,LNGPV20毒株各段基因序列长度与欧洲标准株B 毒株各段基因序列长度一致,LNGPV20毒株的结构蛋白与非结构蛋白序列长度与DY16、RC16、HB-2019、SYG61v毒株相同,与82-0321、SQ0412、E、FJ01毒株各蛋白序列长度均存在一定差异。其中ITR 基因序列长度变化最大,序列长度在414~447 bp之间,相差33个碱基。14个毒株各段基因序列同源性为91.13%~98.00%,各基因片段核苷酸同源性较高,其中非结构蛋白NS1核苷酸同源性为98.00%,同源性最高。
2.3 基于ITR序列比对分析(见图2)
由图2可知,GPV全基因组5'端与3'端ITR序列相同,呈末端倒置重复序列。LNGPV20 毒株ITR 由444 个碱基组成,与标准毒株B 株相比ITR 的5'端缺少一个碱基C,ITR外侧405个碱基形成一个U型的发夹结构,由181个碱基对形成的“茎部”和43个碱基形成的“泡区”构成,“泡区”197~202 阴影区为SphⅠ内切酶的酶切位点。ITR 内侧有39 个碱基,不参与U 型发夹结构的形成,但全基因组5'端与3'端ITR内侧的39个碱基呈反向互补,不影响基因组形成回文结构。LNGPV20毒株与B毒株相比,在181个碱基对区域存在20个碱基的差异,但不影响互补配对。
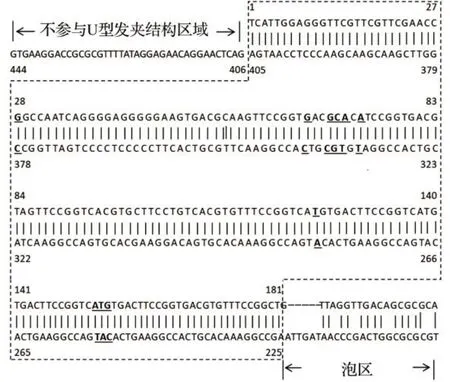
图2 LNGPV20毒株5'端ITR序列和二级结构Fig.25'ITR sequence and secondary structure of LNGPV20 strain
2.4 LNGPV20毒株全基因序列比对分析(见图3)
由图3 可知,采用DNAstar 软件Jotun Hein 方法对LNGPV20 毒株与已公布于GenBank 的14 个代表毒株进行基因序列的同源性分析,LNGPV20 毒株与14 个毒株全基因序列同源性在93.2%~98.9%之间,同源性均在90%以上。由此可见,鹅细小病毒全基因序列变化差异不大,比较保守。LNGPV20毒株与DY16、RC16毒株同源性最高,均为98.9%;与标准毒株B 株同源性为96.6%,而与GDaGPV、GPV-98E、SYG61v 疫苗毒株同源性较低,分别为93.2%、93.4%、93.5%。
2.5 基于全基因序列的系统进化分析(见图4)
由图4可知,采用MEGA7.0软件,根据LNGPV20毒株与已公布于GenBank的20个参考毒株基因序列,构建系统进化树。进化树显示鹅细小病毒的分离株与番鸭细小病毒的分离株分别处于两大遗传分支上,鹅细小病毒的疫苗株与野毒株亲缘关系较远,处于两个小分支内。LNGPV20 毒株与DY16、RC16、HB-2019、SQ0412 毒株亲缘关系较近,与标准毒株B株亲缘关系较远处于不同分支内。由此可见,国内流行的鹅细小病毒基因组变异不明显,同源性较高,亲缘关系较近。
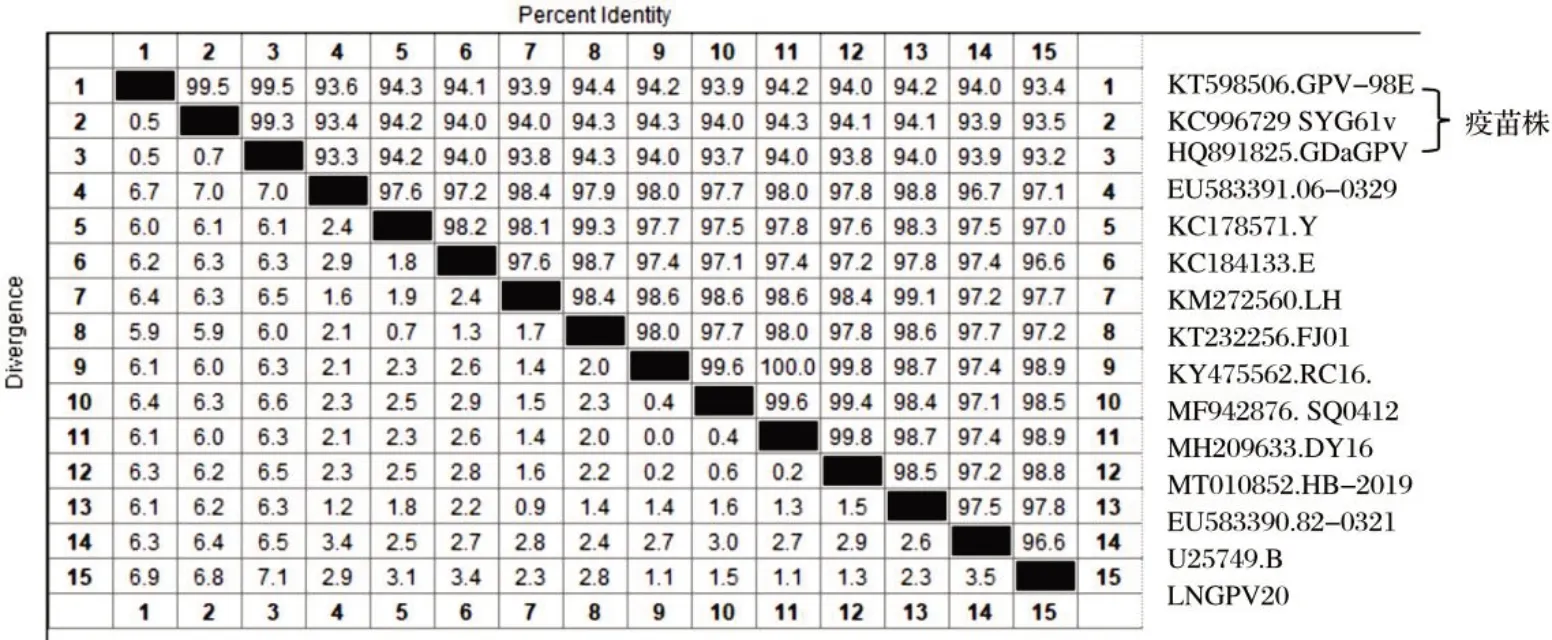
图3 LNGPV20毒株核苷酸比对分析Fig.3 Nucleotide comparison analysis of LNGPV20 strain
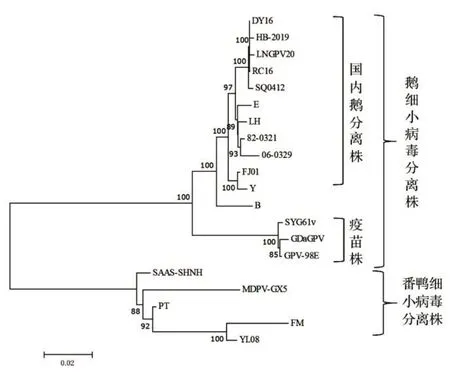
图4 20株GPV全基因进化树Fig.420 GPV whole gene evolutionary trees
3 讨论
在细小病毒科所有成员中,GPV、MDPV 与人细小病毒AAV-2 亲缘关系最近。但AAV-2 只有与腺病毒或疱疹病毒等辅助病毒共同感染时,才能进行病毒复制[11]。而GPV和MDPV不需要辅助病毒,主要通过内吞途径将衣壳蛋白带入细胞,进入细胞质,将病毒DNA 转运到细胞核中,进行DNA 复制[13]。GPV 的基因组比MDPV 的基因组短,GPV 的ITR 在414~447 之间,MDPV 的ITR 在421~457之间。由此可知,GPV、MDPV 的ITR 长度差异较大[14]。LNGPV20 毒株基因组全长5106 bp,ITR 序列长444 bp,ITR内侧有39个碱基不参与ITR的U型发夹结构的形成,而B 株ITR 内侧有38 个碱基不参与ITR 的U 型发夹结构形成,可能与LNGPV20毒株ITR的5'端缺少一个碱基C有关。有研究表明,ITR 的长度,是影响毒株毒力的关键[15],但宋娜[16]对2 株江苏省鹅细小病毒分离株,ITR 分别为416 bp 和444 bp,且毒力均较强,因此推断GPV 的毒力强弱并不能依据ITR 的长度来判断。各毒株结构蛋白与非结构蛋白同源性在97.64%~98.00%之间,相对于ITR 差异不大。近年来,学者对GPV非结构蛋白与结构蛋白抗原表位区进行定位分析[17],经郭鹭等[18]、黎明等[19]对GPV 结构蛋白、非结构蛋白进行抗原表位的精准定位,抗原表位为VP3 蛋白430~435 aa 和643~647 aa、NS1 蛋白503~509 aa,为疫苗制备、特异性抗体制备等奠定理论基础。
辽宁分离毒株与14 个毒株全基因序列同源性比对分析,与GDaGPV、GPV-98E、SYG61v 疫苗毒同源性均在93%以上,除去ITR 序列,疫苗株与国内11 个野毒株的NS、NP 蛋白同源性均在97%以上,由于疫苗株与野毒株NS、NP同源性较高,且大部分抗原决定簇集中分布于NS、NP 蛋白[20],可见国内小鹅瘟疫苗对小鹅瘟应有较高的保护率,但由于养鹅业饲养环境的差异,导致母源抗体参差不齐,大多数种鹅未进行免疫接种且呈隐性感染,虽然无发病症状,但仍可以散播病毒,导致小鹅瘟对养殖业的危害越来越严重[21]。另外,辽宁分离株与国内野毒株同源性在96.6%~98.9%之间,其中有4 个病毒株同源性在98%以上,分别源于安徽、重庆、河北、山东。由此可见,国内流行的鹅细小病毒基因组变异不明显,同源性较高,可能是近年来养鹅业迅速发展,鹅苗流通增加,交叉感染所致。
4 结论
本研究采用PCR 方法对鹅细小病毒全基因分段进行扩增,首次获得辽宁分离株全基因序列,该研究结果进一步丰富我国鹅细小病毒基因数据库,同时也为辽宁省鹅细小病毒的防控提供参考。
