RNA聚合酶II动态调控及其成像技术的研究进展
2021-05-14钱虹萍陈博林金星崔亚宁
钱虹萍 陈博 林金星 崔亚宁
(1. 北京林业大学林木分子设计育种高精尖创新中心,北京 100083;2. 北京林业大学生物科学与技术学院,北京 100083)
转录是基因表达的关键步骤,也是真核生物生长发育和生命活动中必不可少的过程。该过程是以双链DNA中的一条链为模板,以A、U、C、G四种核糖核苷酸为原料,在RNA聚合酶的催化作用下转录形成mRNA。转录过程又可以被分为转录起始、延伸、终止3个阶段,首先RNA聚合酶与基因上的启动子[1]结合诱导转录的开始,在调控因子的协助下进一步完成延伸,直至遇到终止信号完成特定基因的转录。所以,转录是将遗传信息从DNA流向RNA的过程,这对生物的多样性及生命的存在均有重要的意义。
前期的研究展示,真核生物的细胞核内的转录主要由3种RNA聚合酶介导,分别是RNA聚合酶I,RNA聚合酶II,以及RNA聚合酶III。其中RNA聚合酶I主要定位在细胞核仁,负责催化合成核糖体RNA(rRNA)中的18S rRNA、28S rRNA和5.8S rRNA;RNA聚合酶III则主要定位于细胞核基质,负责指导转运RNA(tRNA)、5S rRNA以及一些非编码RNA的合成;RNA聚合酶II也定位在细胞核基质中,负责催化核糖核酸的转录,来合成信使RNA(mRNA)及大多数snRNA、hnRNA。近期对植物体的研究发现,植物细胞核内除了上述3种RNA聚合酶外,还包含两种特异性的RNA聚合酶,即RNA聚合酶 IV和RNA聚合酶 V。RNA聚合酶IV和RNA聚合酶V能够促进非编码RNA的产生,并且这些非编码RNA通过DNA甲基化(RdDM)途径[2]对基因沉默发挥重要的调控作用。
RNAP II在转录过程中发挥的作用最大,所以关于RNAP II转录调控的研究较多,这对于揭示真核生物生命活动的分子基础具有重要的生物学意义[3]。RNAP II在进行高水平的转录调控过程中,通常需要一组转录因子(general transcription factors,GTFs), 包 括 TFIIA、TFIIB、TFIID、TFIIE、TFIIF和TFIIH来协调进行[4]。在过去的几十年里,人们针对这些转录因子的主要功能做了大量的研究,发现这些转录因子都是以一种动态结合和解离的方式来调控转录的发生[5]。那么RNAP II在转录过程中的动态变化又是怎样呢?随着单分子成像和标记技术的不断发展和应用,活细胞内单个RNAP II分子的动态研究得以实现并不断的推进。在活体胚胎干细胞内,Cho等[6]观察到RNAP II可以形成稳定存在的、或者短暂存在的clusters团簇结构。同时,孙育杰课题组[7]也在哺乳动物的活细胞核内,首次原位观察到了RNAP II clusters的动态组装和解聚过程。然而植物中关于RNAP II的活体动态研究比较少,因此本文主要对真核生物RNAP II结构和功能的研究进行综述,并重点总结动物活细胞内RNAP II的动态标记和成像技术的研究进展,以期为植物活细胞内观察RNAP II动态转录研究提供参考。
1 RNA聚合酶II的结构
RNAP II被认为是真核细胞RNA聚合酶家族中最活跃、最复杂的成员,它存在于细胞核的核质内,分子质量为550 kD,由12个亚单位组成,并分别由RPB1-RPB12基因编码[8]。这12个亚基在真核细胞中高度保守,并能分解成一个由10个亚基组成的核心酶和一个由Rpb4和Rpb7组成的二聚体。其中按照进化的保守性以及在转录过程中的作用,又可以分为3类:一类是3种RNA聚合酶共同含有的亚基Rpb5、6、8、10和12;第二类是与细菌RNA聚合酶高度保守的核心亚基Rpb1、2、3;第三类是Rpb11以及Pol II特有的并且是转录起始所必需的亚基 Rpb4、7 和 9[9]。
Rpb1是RNAP II 的最大亚基,由于其具有催化活性,因此成为RNAP II 发挥转录功能的主要执行者,它的羧基末端有一段特殊的结构域CTD(carboxyl-terminal domain),在执行转录过程中发挥关键调控作用[10]。CTD具有高度保守的一致序列(Tyr-Ser-Pro-Thr-Ser-Pro-Ser),是一段基本上无结构的多肽链,并且在不同物种中的重复次数各不相同,如酵母中为26个重复,动物和人类中为52个重复,而植物中则有39个重复[11]。CTD的这种重复特性,能够增加与之结合的蛋白数目,这对其功能的发挥产生直接的影响[12]。例如,在mRNA加工修饰的过程中,CTD结构通过与加帽酶、剪接因子以及诸多聚尾因子的结合,从而直接参与调控这些复杂的加工反应[13]。另外,CTD也会发生一系列修饰作用,譬如当外界病原菌入侵时,拟南芥RNAP II的CTD上3个氨基酸残基发生快速而短暂的磷酸化,为植物激活初级免疫应答提供了一种快速途径[14]。
2 RNA聚合酶II的功能
在真核生物中,RNAP II主要负责合成mRNA及大多数非编码RNA[15]。在转录开始时,RNAP II通过识别DNA上的核心启动子来决定转录的起始位点,同时转录因子也通过结合启动子中的顺式元件来协同激活转录的起始[16]。在转录的过程中,每一步都有转录辅激活因子的调节,也受到一些共同激活或抑制因子的调节,共同保证转录的正确延伸直到转录的结束[17]。植物中的转录过程(图1)主要包括3个阶段:转录起始、转录延伸和转录终止,具体的过程及RNAP II 在其中的作用如下。
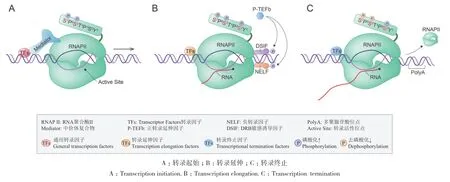
图1 RNAP II介导的转录过程Fig. 1 RNAP II mediated transcription
2.1 RNA聚合酶II与转录调控起始
转录起始是一个动态协调的过程,也是转录发生过程中最关键的一个步骤[18]。在转录因子和中介体复合物(mediator complex)[19]的协助下,未经任何修饰的RNAP II 被招募到启动子附近,形成转录预起始复合物(pre-initiation-complex,PIC)[20]。在ATP的作用下,DNA双链被打开,其中一条DNA模板链进入RNAP II的活性位点,形成开放复合物来激活转录的起始。紧接着在RNAP II酶的催化作用下,核苷三磷酸(NTPs)作为底物开始合成mRNA的前体,标志着转录起始的正常进行[21]。通过单分子荧光成像技术观测RNAP II—PIC的组装过程发现,参与组装的转录因子是通过一种短暂的、动态的方式来参与转录的起始[22]。因此,转录起始是由RNAP II和多种转录因子共同参与的动态调控过程。
在转录起始过程中,RNAP II的CTD磷酸化修饰起到了“开关”的作用。在转录初期,转录因子TFIIH使CTD的两个氨基酸残基(Ser5和Ser7)发生磷酸化修饰[23]。与动物不同的是,CDKF;1(CYCLIN-DEPENDENT KINASE F;1)是植物中特有的激酶,它可以特异性地磷酸化氨基酸残基(Ser7)[24]。其中Ser5的磷酸化状态会解除RNAP II和转录因子之间的结合,使RNAP II成功离开启动子区域,从而进入转录延伸的阶段[25]。因此,未经修饰的RNAP II主要参与转录初始阶段,而磷酸化的RNAP II主要在转录延伸阶段发挥作用。
2.2 RNA聚合酶II与转录延伸
RNAP II离开启动子并开始合成RNA时,即进入了转录延伸阶段[26]。在早期延伸过程中,RNAP II在启动子的转录起始位点(transcription start site,TSS)下游20-100 bp处会发生转录暂停。转录暂停现象既是转录过程中的限速步骤,也是发生在早期延伸和生产性延伸之间的过渡阶段。当负转录延伸因子(negative elongation factor,NELF)和DRB敏感诱导因子(DRB-sensitivity-inducing factor,DSIF)与RNAP II结合时,会引起RNAP II 的转录暂停;而当正转录延伸因子B(positive transcription elongation factor b,P-TEFb)[27]磷 酸 化 NELF 和DSIF时,负转录延伸因子转化为正转录延伸因子,就会触发RNAP II 转录的再次启动[28]。
转录延伸过程中RNAP II的CTD氨基酸残基Ser2可以被P-TEFb磷酸化,来招募其他转录延伸因子和染色质修饰因子,并且CTD的另外两个氨基酸残基Ser5和Ser7在磷酸酶的作用下会逐渐脱磷酸化。总之,在RNAP II的CTD氨基酸残基Ser2的磷酸化状态和Ser5、Ser7的去磷酸化状态的共同作用下,转录的延伸过程进入生产性延伸阶段[29]。因此,RNAP II的CTD磷酸化状态明显影响转录延伸的进程,但反过来转录延伸的过程是否也会影响CTD的磷酸化修饰,以及CTD状态的转变为什么发生在转录的特定延伸阶段,这些问题仍需进一步的研究来解释。
2.3 RNA聚合酶II与转录终止
当转录延伸遇到终止子时,RNAP II在解旋酶的作用下从DNA模板上解离,发生转录终止[30]。RNAP II介导的转录终止是一个极其复杂的过程,大多数mRNA转录终止依赖于多聚腺苷酸(Poly A)位点,并且受到RNAP II的CTD磷酸化状态的调控[31]。在转录终止时,RNAP II的CTD氨基酸残基Ser2的磷酸化水平达到最高,而另一个氨基酸残基Ser5的磷酸化水平逐渐降低。因此,RNAP II的CTD磷酸化状态的转变,会促使Poly A位点上招募到新的转录因子,导致RNAP II转录复合物的构象发生变化[32],而从RNA-DNA杂合链上解离下来,至此一次完整的转录过程发生终止。另外在转录终止时,RNAP II在磷酸酶的作用下发生去磷酸化[33],产生非磷酸化修饰的RNAP II,从而可以参与到下一轮的转录循环反应中。
3 RNA聚合酶II标记及成像技术
RNAP II的结构和功能已经被广泛研究,然而关于活细胞内是如何调控转录过程,以及它的动态特征是怎样的,这些问题目前仍不清晰。近年来,随着活体荧光标记技术和超分辨率成像技术的发展,研究人员可以实时的追踪RNAP II的动态过程,能够实现对RNAP II的高时空动态成像与观察[34]。下面对这些标记和成像技术进行介绍。
3.1 RNA聚合酶II标记技术
荧光标记技术是指将不同的荧光探针特异性地连接到目标分子上的一种技术,常用的荧光探针包括:有机染料、荧光纳米晶体和荧光蛋白等[35]。其中荧光蛋白指的是带有荧光基团结构的蛋白质,它通过对目标分子进行特异性地标记[36],是目前荧光标记中最常用的手段。绿色荧光蛋白(green fluorescent protein,GFP)[37]及其他颜色的荧光蛋白[38]由于稳定性强和亮度高的优点,受到了研究人员的青睐并实现了广泛的应用。但新型超高分辨率技术的发展,对荧光蛋白的选择产生了更高的需求,亟需更亮、更稳定的荧光蛋白作为标签。光激活(photoactivatable fluorescent proteins,PAFPs)、光转 化(photoconvertible fluorescent proteins,PCFPs)、光开关(photoswitchable fluorescent proteins,PSFPs)荧光蛋白等的出现,不仅提高了显微观察的精准性,也极大地推动了活细胞成像技术的发展[39]。
之前对转录调控的研究中,研究者通常选择荧光原位杂交技术(fluorescence in situ hybridization,FISH)[40]来进行,它能够同时识别特定的基因和转录位点并进行标记。但是这一技术需要将细胞固定,而固定后的“死细胞”获得的结果与活细胞中的结果有很大的不同,固定后的“死细胞”不能反映植物细胞感受外界信号后的真实转录情况,因而无法获取动态变化的准确信息。在这个过程中,基因编辑技术如CRISPR/Cas9(clustered regularly interspaced short palindromic repeats-CRISPR-associated protein 9)和自我标记蛋白(self-labeling proteins,SLPs)技术是目前非常有用的技术手段。
CRISPR/Cas9是一种基因靶向编辑技术,它可以将编码荧光蛋白的基因序列插入到基因组的特定位置,来实现基因转录的可视化[41]。CRISPR/Cas9技术由Cas9核酸酶和单链引导RNA(single guide RNA,sgRNA)两部分组成,Cas9核酸酶在sgRNA的指导下,可以靶向切割目的基因,再利用同源DNA修复技术完成对该目的基因的编辑和重组。研究者通过CRISPR/Cas9技术,利用光转换荧光蛋白(Dendra2)对小鼠胚胎细胞的内源性RNAP II进行特异性标记,观察到活细胞核内RNAP II团簇的存在,并且其寿命非常的短暂(约为8 s)[42],这一结果与之前标记外源性RNAP II的观察结果相一致。因此,CRISPR/Cas9是一种强大且精细的工具,通过基因工程技术将荧光蛋白敲进内源基因以使融合蛋白在生理相关水平上表达。同时由于该技术具有易于设计、载体构建简单、实施快捷和成本低的优点,在生物学领域里的多个物种中[43]都得到了广泛的应用,但CRISPR/Cas9方法在应用的过程中仍然存在脱靶[44]的问题,还需不断改进。
SLPs技 术( 如 Halo-Tag和 SNAP-Tag)[45]通过一种共价结合的方式让目标蛋白携带上荧光标签,共价键具有不可逆、稳定性强、特异性高等特点,是研究活细胞蛋白追踪的首选技术。常用的一些标签包括 HaloTag、SnapTag、CLIPtag和TMPTag,都可以通过遗传编码的方法与目标蛋白进行融合表达,并且这些标签可以催化有机染料标记的小分子底物共价连接到目标蛋白[46]。在小分子透膜染料中,janelia fluor(JF)系列的染料表现最佳[47],如JF549、JF646、PA-JF549及PA-JF646等,在近期的单分子成像领域被广泛应用。例如在一项最近的研究中,Cho[6]等分别用 JF646-Halotag和Dendra2来标记转录因子Mediator和RNAP II,双色光片显微成像显示活体细胞核内有大量聚集的双色亮点,并且通过量化分析发现,Mediator和RNAP II团簇共定位比例达到90%。另外,在植物研究应用方面,利用一种新开发的Halo-Tag可编辑技术[48],来探索转录因子和蛋白之间的相互作用,这使得许多新的转录因子被识别和研究(图2)。

图2 CRISPR/Cas9和SLPs技术标记目标分子的示意图Fig. 2 The schematic of CRISPR/Cas9 and SLPs
3.2 RNA聚合酶II成像技术
自20世纪以来,超高分辨显微技术的快速发展使光学成像有了很大突破,能将衍射极限达到200 nm以下,并且实现了三维、高速的活体成像[49],可以在单分子水平上直接观察目标蛋白的动态特征。与传统的光学显微镜相比,超高分辨率技术能够将分辨率提高近10倍,这就使亚细胞的结构细节得到前所未有的可视化[50],但不同的研究问题需要不同的成像技术作为最优的解决方案。因此,为了方便研究者技术的选择,我们概述了几种目前可用于生命科学研究的显微技术[51],并总结了不同显微技术的主要优缺点,如表1所示。
下面对这几种技术在RNAP II动态研究中的应用进行介绍。
单分子定位的显微技术,如光激活定位(photo activated localization microscopy,PALM)和随机光学 重 构(stochastic optical reconstruction microscopy,STORM)显微技术,能够实现30 nm的空间分辨率和10 μm的成像深度,设备价格也中等,并且它们成像原理基本相同,都是利用荧光分子的特性和单分子定位的精准度,通过获取多张图片进而重构出一幅超分辨图像;区别在于STORM技术是利用荧光探针等技术来标记目标蛋白[52],而PALM技术则是利用融合表达的荧光蛋白来进行标记的,因此PALM技术更适合于活细胞内的蛋白成像。在活体胚胎细胞核内,研究者就利用PALM超分辨成像技术揭示了RNAP II的动态特征,表明内源性的RNAP II是以瞬时的团簇形式存在的[42]。同样通过PALM技术,Cisse等[34]观察到了RNAP II团簇的组装过程,但对于RNAP II团簇是否发生解聚过程仍不清楚。但无论是STORM还是PALM技术,都是利用“时间换空间”的方法来提高成像分辨率,这也就限制了它们的时间分辨率,不适合应用于分子的长期动态观察。

表1 不同显微技术的主要优缺点Table 1 Major merits and demerits of different microscopic technique
受激发射损耗技术(stimulated emission depletion,STED)及其它衍生方法,它作为共聚焦显微装置的附加模式,通常是比较容易操作的一项技术。STED的原理是利用两束光源,其中激发光照射目标区域发出荧光,而损耗光则会将目标样品以外的荧光淬灭,从而达到有效荧光发光面积的最小化[53],所以STED技术将光学显微镜的分辨率提高了近10倍。另外,Wildanger等[54]通过引入3D STED,又实现了3个方向上超分辨率的提升。在最近的一项研究中,Li等[55]利用3D STED技术来实时成像、追踪和定量分析活细胞内的RNAP II分子,观察其在转录周期中的动态变化,从而为揭示RNAP II的动力学与转录调控之间的联系奠定基础。但是,STED技术也有很多缺陷,如STED技术不适用于大的样品,并且会对样品产生光毒害等,最重要的是设备的价格非常昂贵等。
光片荧光显微镜(light sheet fluorescence microscopy,LSFM)是一种适用于大型样品成像的新型显微系统[56]。LSFM虽然局限于传统的分辨率,但通过与其他超高分辨率技术的结合,如LSFM与结构光照明(structured illumination microscopy,SIM)显微技术结合而发展出的贝塞尔光束照明技术(bessel beam illumination microscopy)和晶格光片显微术(lattice Light-Sheet Microscopy,LLS),成为一种广受欢迎的成像显微技术。该显微技术采用薄片光进行侧向照明,并在垂直方向上探测成像,不仅成像速度非常快、光毒性低、信噪比高,而且可以对活体样本进行长时间的三维成像[57]。Zhao等[58]利用LSFM技术观测了哺乳动物活细胞核中RNAP II分子的动态,结果显示大多数(>70%)的转录位点来源于单个RNAP II分子,并且未检测到RNAP II分子之间发生显著的聚类反应。而在Cho等[36]研究中,通过LSFM成像显示小鼠胚胎干细胞核内出现大量RNAP II聚集的现象,进一步分析显示这些RNAP II分子有两种存在形式,分别可以形成小的、瞬时的团簇和较大的、稳定的团簇。当然,LSM也有其自身的局限性,如设备价格非常昂贵、横向分辨率较低,后期的大数据处理也比较困难等[59]。如何与其它超高分辨率显微技术更好的融合,将是LSFM未来研究的主要方向。
贝叶斯纳米显微技术(bayesian nanoscopy)就是一个将LSFM与超分辨显微技术结合的典型例子,已经发展成为一种新型快速成像方法[60]。贝叶斯算法是利用大量荧光分子的闪烁和漂白信息来还原样品的结构,通过将高斯光片照明引入到贝叶斯算法中[61],发展出一种新型的贝叶斯纳米显微技术。该技术有效地提高了显微成像的时空分辨率(50 nm的空间分辨率和4 s的时间分辨率),并大大减少了对样品的光毒性。Chen等[7]利用贝叶斯纳米显微技术的优势,观察到活细胞核中RNAP II分子的空间结构为不规则的球状几何形式,并揭示了RNAP II分子的动态组装和解聚过程。虽然贝叶斯纳米显微技术也存在局限性,比如不适合长期观察活细胞内的分子动态[57],但此方法是目前为止研究活体细胞内RNAP II分子动力学的最佳选择。
4 展望
综上所述,RNAP II的动力学、聚类反应及与转录因子之间的相互作用,对于揭示真核生物的转录调控具有重要的意义。近年来,多个实验室在活细胞荧光成像和分子标记技术的研究中取得了长足的进展,为实现在单分子水平上直接观察RNAP II的动态转录奠定了基础。例如,研究者可以选择CRISPR/Cas9或SLPs标记技术使目标分子携带荧光标签,再通过LSFM与超分辨显微技术结合的方法来实时成像和追踪目标分子,这样就使得活体细胞内的分子动态行为变得可视化。与动物细胞相比,植物的细胞存在细胞壁、叶绿体等物质极大的限制了植物细胞的活体成像,并且植物细胞也无法像动物细胞一样进行贴壁生长,这无疑对植物细胞的活体成像观察提出了更大的挑战,所以目前关于植物RNAP II的活体动态研究甚少,其响应不同生理条件下的动态调控机制也尚不清晰。因此,植物活体细胞内RNAP II动态调控转录是今后重点关注的方向,这也将会为揭示真核生物的转录机制带来更多的启示。
