陆地棉自然群体纤维强度性状的全基因组关联分析
2021-05-13贾冰马建江宋吉坤裴文锋吴嫚臧新山张金发陈全家于霁雯
贾冰,马建江,宋吉坤,,裴文锋,,3,吴嫚,臧新山,张金发,陈全家*,于霁雯,,3*
(1.新疆农业大学农学院/ 棉花教育部工程研究中心,乌鲁木齐830052;2.中国农业科学院棉花研究所/ 棉花生物学国家重点实验室,河南 安阳455000;3.中国农业科学院西部农业研究中心,新疆 昌吉831100)
棉花作为世界上最重要的经济作物之一,可以提供大量的天然纤维[1]。 陆地棉因产量高、品质较好在棉花生产中占主导地位[2]。 随着人们生活水平的不断提高, 对棉花纤维品质的要求也越来越高。纤维强度是决定棉花纤维经济价值和纺织质量的关键特性之一[3]。 纤维强度是数量性状,由多基因共同调控,同时又受环境影响,很难通过传统的育种技术加以改良[4]。 随着分子生物学及生物信息技术的快速发展,分子标记技术极大的推动了数量性状遗传基础的解析及改良。 因此,对棉花纤维强度与分子标记进行关联分析以及鉴定纤维强度相关的优异位点, 将进一步加速棉纤维强度的改良进程, 并为研究纤维强度形成的遗传基础提供理论依据。
棉花纤维的产量和质量与棉花纤维发育进程息息相关[5]。 棉纤维由胚珠表皮单个细胞分化而成,发育过程分为4 个时期:纤维起始分化、纤维伸长期、次生壁加厚期以及成熟期[6]。纤维次生壁加厚期主要是纤维素的合成及沉淀的过程,纤维强度性状主要受到该时期众多基因的调控[7]。近年来,随着分子生物技术的发展,特别是棉花全基因组测序的完成,关于棉花纤维强度关键位点、基因的挖掘及其功能的相关报道已逐渐增多。Deng 等[8]利用棉花多世代分离群体结合简单重复序列 (Simple sequence repeat,SSR)标记,鉴定到17 个与纤维强度相关的数量性状位点 (Quantitative trait locus,QTL)。 Ma 等[9]利用陆地棉重组自交系群体及回交群体,鉴定50 个与纤维强度相关的QTL。Zhang 等[10]利用陆地棉重组自交系群体,构建了全基因组高密度遗传图谱, 鉴定到33 个与纤维强度相关的稳定的QTL。 Huang 等[11]利用CottonSNP 63K 芯片和503 份陆地棉种质资源构建高密度图谱,鉴定到10个与纤维强度相关的QTL。 Ma 等[12]利用419 份陆地棉核心种质,通过重测序技术进行全基因组关联分析, 检测到533 个与纤维强度相关的显著SNP位点,并鉴定了1 个与纤维强度相关的基因。 已有的研究获得了大量的纤维强度相关的QTL 位点,但在多环境下稳定的关键位点不多,仍需进一步挖掘控制纤维强度性状的关键位点和基因。
本研究以83 份纤维强度差异显著的陆地棉为供试材料,利用63K 芯片对群体进行基因型分析,得到15 369 个SNP(Single nuclotide polymorphism,单核苷酸多态性)位点,对5 个环境及最佳线性无偏估计值(Best linear unbiased prediction,BLUP)的纤维强度进行全基因组关联分析,解析棉花纤维强度性状形成的遗传基础。
1 材料与方法
1.1 试验材料及种植方法
2014―2016 年分别在河南省安阳市安阳县(14_Ay、15_Ay、16_Ay)、2016 年 新 疆 维 吾 尔 自 治区阿拉尔市十团 (16_Ale)、2016 年海南省三亚市吉阳区(16_Sy)等5 个环境种植83 份陆地棉材料,材料信息及种植模式参考文献13、14。
1.2 表型测定与分析
在棉花成熟时, 选择棉株中部相同部位的20个棉铃,统一用皮辊轧花机轧花(SY-50)。纤维断裂比强度由农业部棉花品质检验监督检测中心的HFT9000 仪器检测完成。
利用SPSS 25.0 对83 份陆地棉材料在不同环境的纤维强度进行描述性统计分析和相关性分析,使用QTL IciMapping 4.2.0 进行广义遗传力分析,使用R 语言计算BLUP 值。
1.3 纤维强度性状QTL 定位
利用课题之前使用CottonSNP 63K 芯片对83份陆地棉材料进行基因型分析得到的15 369 个SNP 位点[13],使用TASSEL 5.0 中的广义线性模型(General linear model,GLM) 对纤维强度性状进行全基因组关联分析。 GWAS 显著位点的阈值(Bonferroni 法)P为6.51E-05[15-17],即将-lg(P)≥4.19 的位点作为与纤维强度显著关联的SNP 位点,使用R 语言绘制曼哈顿图。 本试验认为:在3个及以上不同环境检测到的同一位置的SNP 是稳定的SNP 位点。 根据每条染色体特定的LD(Linkage disequilibrium,连锁不平衡)的衰减距离[13],将稳定的SNP 位点物理位置±LD 值作为QTL 区间,重叠的区间合并为1 个QTL 区间,QTL 命名方式为:q-性状-染色体-QTL 编号[18]。
1.4 候选基因分析
将QTL 区间内的基因作为候选基因库, 根据海岛棉海7124 和陆地棉TM-1 的转录组数据库[19](https://www.ncbi.nlm.nih.gov/bioproject/PRJNA490626/)以及拟南芥同源基因功能注释,筛选与纤维强度相关基因作为候选基因。
2 结果与分析
2.1 陆地棉纤维强度表型分析
通过对5 个环境83 份陆地棉材料的纤维断裂比强度进行统计分析,发现纤维断裂比强度性状具有广泛的表型变异。 该群体纤维断裂比强度为22.50~38.50 cN·tex-1,在14_Ay、15_Ay、16_Ay 三个环境中纤维断裂比强度的平均值分别为28.94 cN·tex-1、28.70 cN·tex-1和29.58 cN·tex-1, 而在16_Sy 和16_Ale 两个环境中纤维断裂比强度的平均值分别为26.34 cN·tex-1和25.47 cN·tex-1(表1和图1)。 统计结果表明,在安阳种植的棉花材料的纤维断裂比强度3 年的分布趋于一致,与16_Sy 和16_Ale 呈现一定的差异。 在5 个环境中纤维断裂比强度的变异系数为5.55%~8.44%, 广义遗传力高达88.67%, 说明该棉花群体纤维断裂比强度变异较丰富,遗传相对稳定。 相关性分析(表2)表明,纤维断裂比强度在5 个环境中相关系数为0.6~0.76,均呈显著正相关。 对5 个环境的纤维断裂比强度表型数据进行正态分布检验, 发现除16_Ale外,其他环境的纤维断裂比强度性状基本符合正态分布,表明该群体适合进行棉花纤维断裂比强度的全基因组关联分析。 同时,83 份材料在安阳种植纤维断裂比强度较大,而在新疆和海南种植则较小,说明纤维断裂比强度受到环境的影响, 需要对单个环境进行全基因组关联分析。
2.2 全基因组关联分析及纤维断裂比强度QTL
利用TASSEL 软件的GLM(Q)模型对14_Ay、15_Ay、16_Ay、16_Sy、16_Ale 5 个环境的纤维断裂比强度及BLUP 进行全基因组关联分析(图2),分别得到39、134、14、7、16 和55 个显著相关的SNP位点,共计265 个SNP 位点。 去冗余后得到190 个SNP 位点,分布于除A13 外的其他所有染色体上,
对纤维断裂比强度的表型解释为18%~40%,其中121 个显著SNP 位点位于Dt 亚基因组,69 个显著SNP 位点位于At 亚基因组。 在3 个及以上环境中出现的稳定的SNP 位点有19 个,结合其染色体的LD 的衰减距离[13],得到9 个QTL 区间,分别位于A01、A06、D05、D08、D10、D11 和D13 等7 条染色体上 (表3), 其中4 个QTL 区间与前人定位的QTL 位置重叠,其它5 个QTL 区间未见报道。
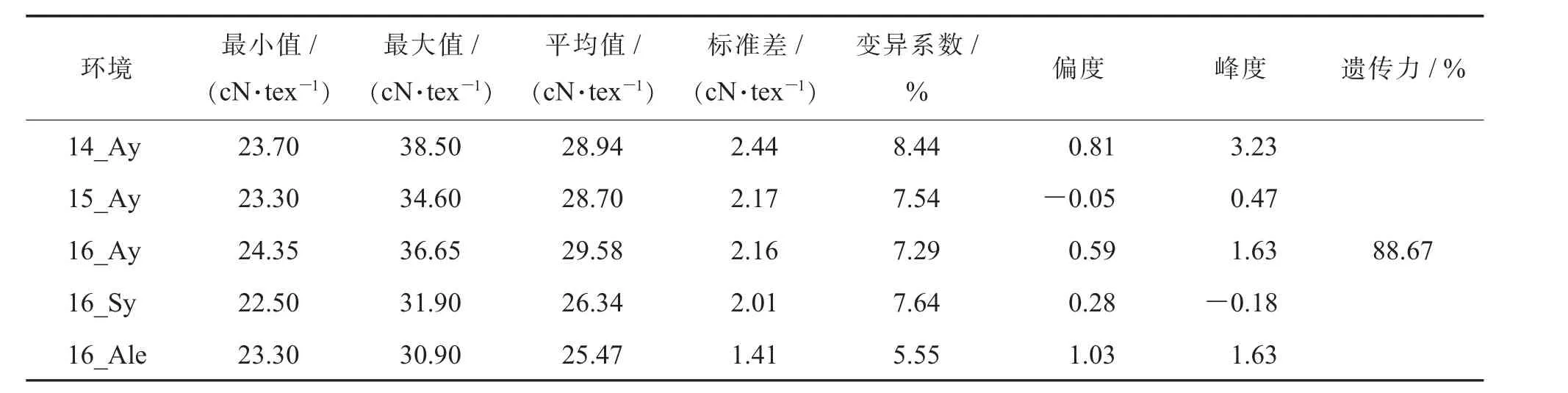
表1 陆地棉群体纤维断裂比强度表型统计

表2 五个环境群体纤维断裂比强度的相关性分析

图1 五个环境下群体纤维断裂比强度的频率分布直方图
2.3 纤维断裂比强度候选基因挖掘
纤维断裂比强度主要在纤维次生壁加厚期受纤维素合成的影响, 且海岛棉海7124 和陆地棉TM-1 的纤维断裂比强度存在显著差异, 所以筛选在20~25 DPA纤维中优势表达, 且在海7124 和TM-1 间差异表达,基因功能注释与纤维断裂比强度相关的基因作为候选基因。 9 个稳定QTL 区间包含1 177 个基因,结合海7124 和TM-1 转录组数据,鉴定到4 个与细胞壁形成或纤维素合成相关的基因(表4),推测这些基因可能影响纤维断裂比强度。Gh_D13G2032编码产物为果胶甲酯酶, 在海7124 开花后25 d 的纤维中表达量最高, 显著高于其在TM-1 同时期纤维中的表达量。Gh_A01G1734在海7124 开花后20 d 的纤维中表达量最高,显著高于其在TM-1 中的表达量。Gh_D10G0333编码纤维素合酶,Gh_D11G1626属于COBL基因家族,这2 个基因都是在20 DPA 的纤维中表达量最高,且在海7124 的表达量明显高于TM-1(图3)。

图2 陆地棉纤维断裂比强度全基因组关联分析
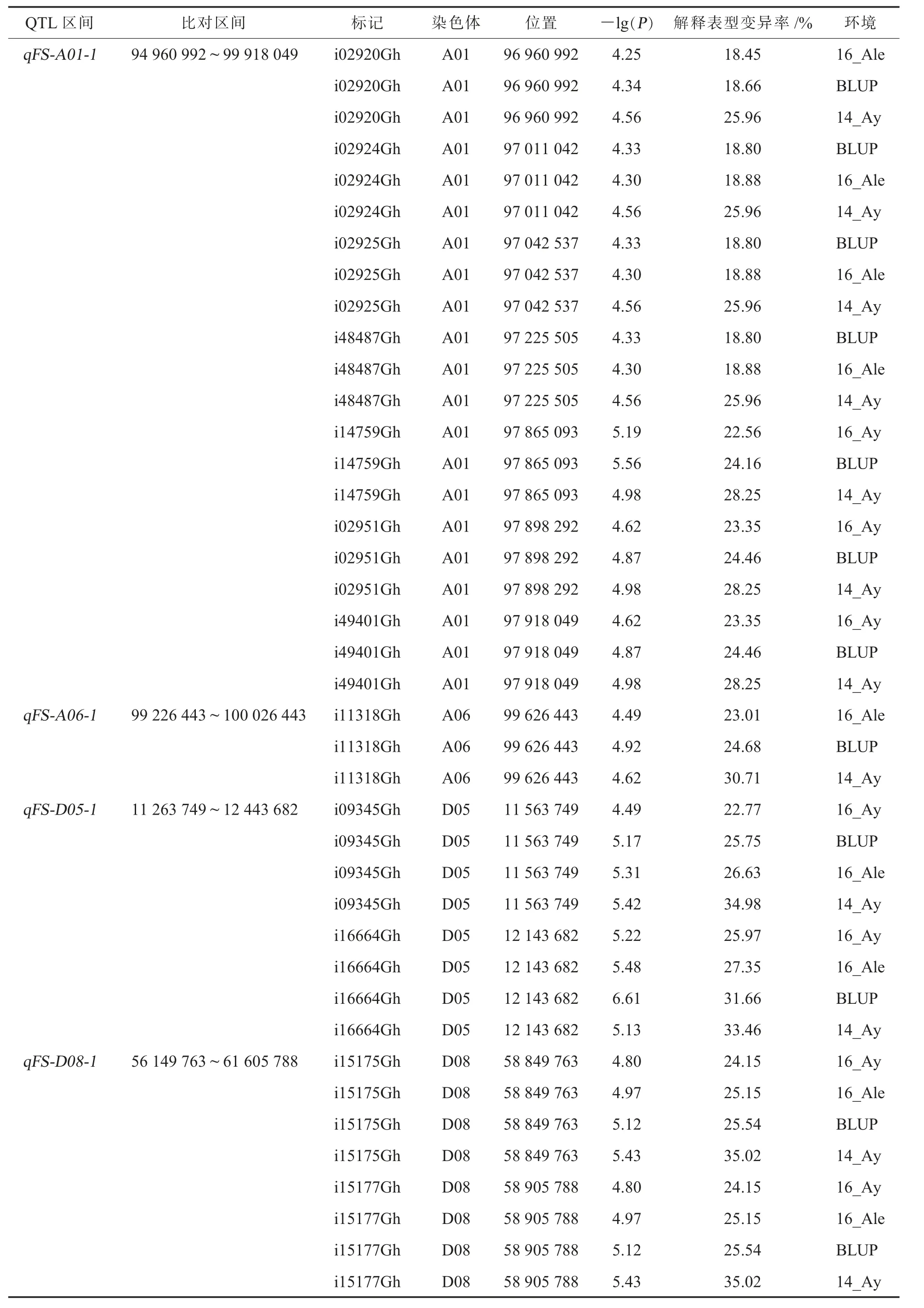
表3 纤维断裂比强度相关QTL 信息
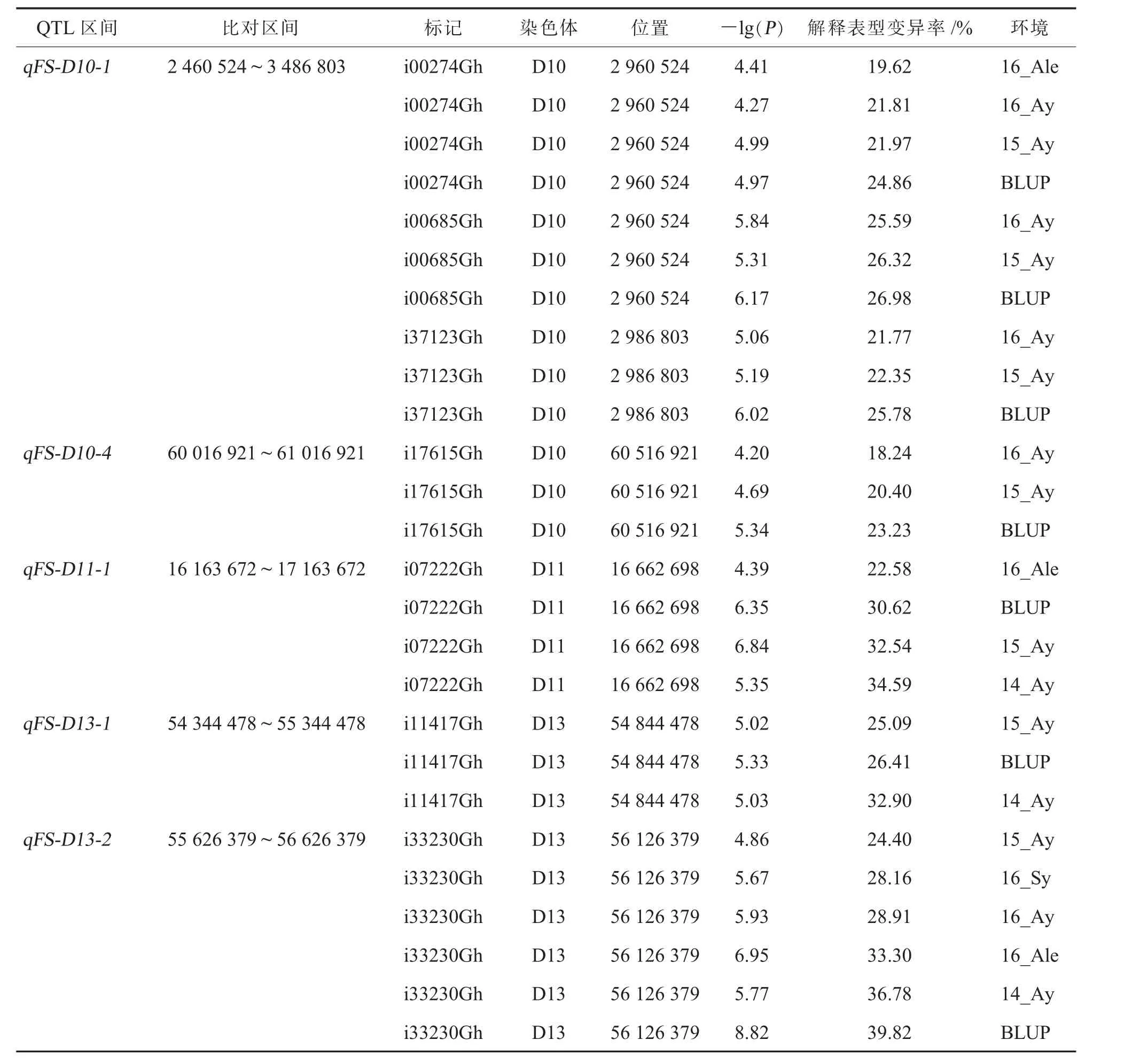
表3 (续)

表4 纤维断裂比强度相关候选基因信息

图3 海7124 和TM-1 纤维发育不同时期4 个候选基因的表达模式
3 讨论
棉花纤维品质主要看“长”、“强”、“细” ,其中“强”就是纤维断裂比强度,对棉纤维的加工和产品的质量有直接的影响。 因此,棉花纤维断裂比强度改良一直是棉花纤维品质遗传改良的热点。
本研究中用于全基因组关联分析的83 份陆地棉材料,具有丰富的表型变异。 通过对5 个环境的纤维断裂比强度表型数据及BLUP 值进行全基因组关联分析,共得到了265 个显著SNP 位点,非冗余的显著位点190 个, 其中位于Dt 亚基因组上的SNP 位点是At 亚基因组的显著SNP 位点的1.7倍。 前人研究表明Dt 亚基因组对纤维品质性状的遗传贡献比At 亚基因组更大, 本研究结果与前人的研究结果一致[19]。 另外,19 个SNP 位点出现在3个及以上环境中, 合并为9 个QTL 区间, 其中qFS-A01-1包含Sun 等[21]检测到的显著SNP 位点,qFS-D05-1与qFS-D13-2均与Wang 等[22]检测到的QTL 区间重叠,qFS-D10-1与Gu 等[23]检测到的区间重叠。 此外,本研究还发现了5 个新的稳定QTL区间,为棉花纤维断裂比强度及分子标记辅助选择育种提供参考。
在9 个QTL 区间内筛选了22 个在纤维发育次生壁加厚期优势表达、 且在海岛棉与陆地棉之间差异表达的基因, 并鉴定到4 个可能影响纤维断裂比强度的候选基因。 其中Gh_D13G2032的编码产物为果胶甲酯酶, 催化细胞壁中果胶主链发生脱甲酯化反应, 对于调节植物中的细胞膨胀非常重要。 在前人研究中,海岛棉纤维比陆地棉更细更长可能是受果胶甲酯酶含量差异的影响[24]。Gh_D11G1626作为COBL基因家族成员,编码植物特异性糖基磷脂酰肌醇锚定蛋白, 该蛋白通过影响微纤丝在细胞壁的正确排列而参与植物纤维素的合成,在纤维发育过程中影响纤维素的沉淀,进而影响棉花纤维断裂比强度[25]。Gh_A01G1734的同源基因AT5G06270显著影响拟南芥的根毛数量, 且与拟南芥的表皮毛发育密切相关[26]。Gh_D10G0333的功能注释为纤维素合酶GhCESA8, 在棉花纤维发育过程中纤维素的快速大量积累起到了增强作用[27]。
4 结论
本研究对83 份棉花材料在5 个环境的纤维断裂比强度及其BLUP 值进行全基因组关联分析,共检测到19 个与纤维断裂比强度有关的稳定的SNP位点,得到9 个QTL 区间,其中5 个是本研究新发现的。并鉴定到4 个与纤维断裂比强度有关的候选基因,为进一步挖掘与棉花纤维断裂比强度性状相关的基因提供了理论依据。
