基于新型冠状病毒SARS-CoV-2主蛋白酶结构的小分子抑制剂虚拟筛选
2021-05-08徐浩薛瑞李娟李波陈舟朴莲花常珊孔韧
徐浩 薛瑞 李娟 李波 陈舟 朴莲花 常珊 孔韧

摘 要:通过使用分子对接与虚拟筛选技术,以SARS-CoV-2主蛋白酶(Main Protease,Mpro)为靶点,利用Autodock Vina软件和Python脚本进行分子对接,结合小分子的对接评分与结合模式分析,筛选出特异性作用于主蛋白酶的小分子抑制剂。结果发现,以SARS-CoV-2主蛋白酶原配体N3为对照,Reactive blue 2等分子具有更好的对接打分与合理的结合模式,或可成为潜在的主蛋白酶小分子抑制剂,可为寻找抗新冠病毒治疗药物提供线索。
关键词:分子对接;COVID-19;虚拟筛选;主蛋白酶;小分子抑制剂
中图分类号:R914 文献标识码:A 文章编号:2095-7394(2021)02-0065-07
2019年12月,我国武汉市出现了一种传染性极强的疾病[1],其病原体被发现是一种新的冠状病毒,世界卫生组织将这种新型冠状病毒感染的肺炎命名为“COVID-19”。由于引发该肺炎的冠状病毒与引发SARS的冠状病毒具有高度亲缘性[2],因此该病毒被命名为“SARS-CoV-2”。2020年1月30日,世界卫生组织(World Health Organization)宣布该事件为国际公共卫生事件,并于3月11日报告该疫情为全球性大流行。这是由冠状病毒导致的首次全球疫情大流行。截至2020年10月20日,全球累计确诊病例已超4 000万。由于SARS-CoV-2是一种先前未在人类群体中发现的β属新型冠状病毒,因此目前没有特效治疗药物[3]。随着感染人数的逐渐上升,防控和救治已经成为全球性难题。作为一种β冠状病毒,SARS-CoV-2的遗传物质是单股正链RNA,控制冠状病毒复制的主蛋白酶Mpro在病毒生命周期中有重要作用,Mpro与小核糖核酸病毒3C蛋白酶具有相似的切割位点特异性,这一酶通常被称作3CL蛋白酶[4]。Mpro及其同源酶在病毒复制及转录过程中起到重要作用,因而Mpro受到研究者们的关注[5-7],该蛋白酶可成为抗新冠病毒药物的重要靶标。
相较2003年的SARS-CoV病毒疫情,COVID-19波及范围广、持续时间长,虽然目前疫情已经得到了较好的控制,但特异性治疗药物缺乏仍是一大问题。因此,寻找新冠病毒主蛋白酶的抑制剂对于抑制此次疫情具有重要的意义[8-9]。本文运用分子对接技术,对Mpro主蛋白酶与小分子的结合进行计算机模拟。对于筛选得到的分子,分析其结合模式,与晶体结构做对比,筛选出具有较为合理结合模式的潜在抑制剂分子,为寻找可能的抗新冠病毒药物提供线索。
1 方法与材料
1.1 复原对接
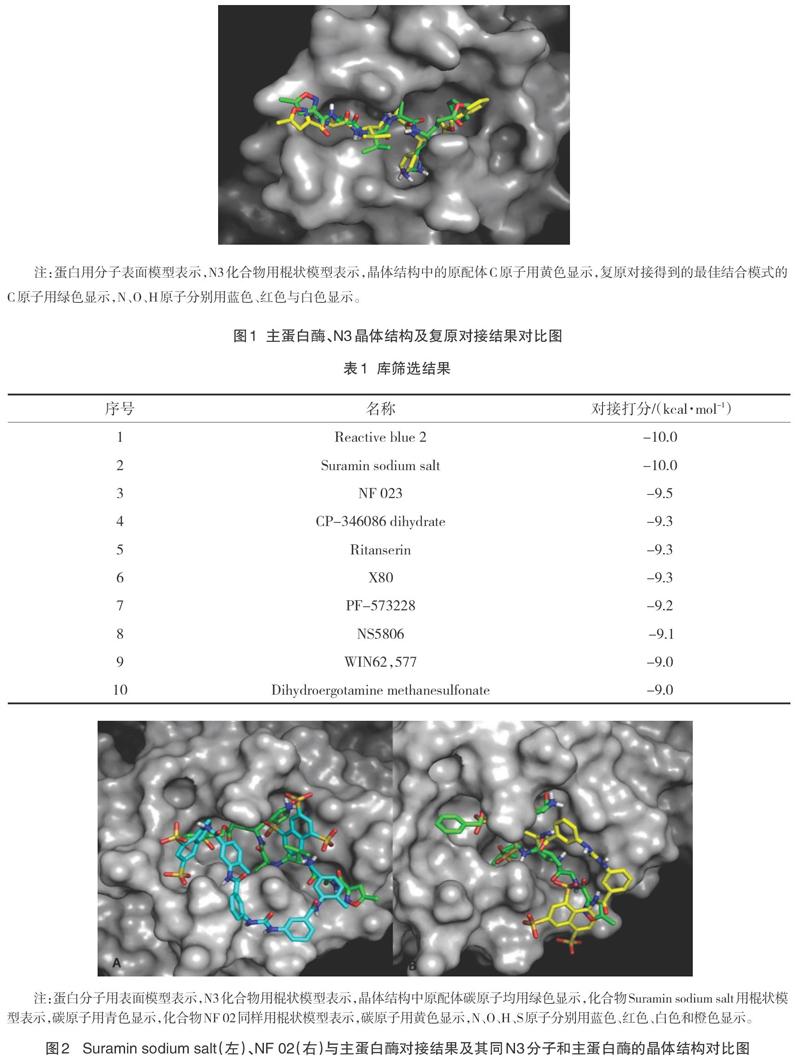
分子对接实验采用AutoDock Vina[10-11]进行,该程序采用了拉马克遗传算法,结合基于网格的能量估计。为了检验对接实验参数的可行性,对晶体结构进行了复原对接。从RCSB PDB庫中下载PDBID为6LU7的SAR-CoV-2主蛋白酶与抑制剂的复合物结构,分辨率为2.16 ?。将受体蛋白作加氢处理,配体选择可旋转柔性键,使用标准化的流程得到pdbqt文件。以晶体结构中配体的中心位置为对接实验中盒子的中心坐标(center_x = -10.85,center_y = 2.58,center_z = 68.72),定义盒子大小为30×30×30 ?,使其包围整个活性口袋区域。对小分子配体进行加氢处理,并赋予Gasteiger电荷。设置Exhaustiveness参数为12,并定义输出打分最佳的十个构像。采用PyMOL和LigPlot进行结合模式分析[12-13]。
1.2 小分子数据库建立与配体准备
筛选所需要的配体来源于LOPAC?1280-药理学活性化合物文库,合集包括1 280种小分子化合物,其中大部分为已上市的药物分子。下载所有小分子文件,采用Openbabel[14]生成小分子的三维坐标。采用AutoDock添加部分电荷信息转换文件格式,得到pdbqt格式的配体文件(批量处理利用python脚本完成)。
1.3 虚拟筛选
复原对接的结果显示原配体成功对接到靶蛋白的活性中心,表明对接参数设置合理。将靶蛋白与原配体的对接结果作为阳性对照,用python脚本SailVina调用Autodock Vina对小分子化合物库进行批量处理,通过AutoDock Vina得到每一个小分子对应的对接打分情况。本研究筛选出小分子库中打分排名前十位的化合物,并对它们与受体的相互作用及结合模式进行分析,寻找效果最佳的可能的小分子抑制剂。
2 结果与讨论
2.1 复原对接结果分析
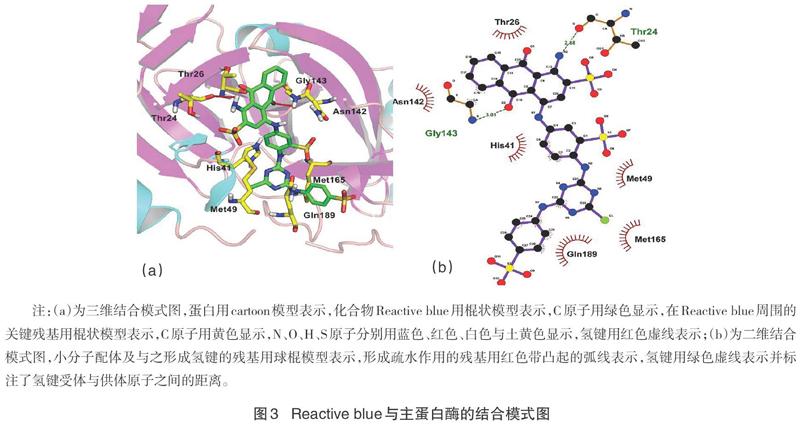
6LU7结构由SAR-CoV-2 Mpro与对应的N3抑制剂结合形成,并且表现出配体与活性点位残基的多个分子间相互作用。由文献[15]可知,在6LU7对应的晶体结构中,主蛋白酶由三个结构域组成,N3通过与主蛋白酶底物结合裂缝中残基的多种相互作用而稳定下来。其中,Cys145残基上的S与乙烯基的C发生迈克尔加成形成共价键,键长1.80 ?,在抑制蛋白活性中起到关键性作用。His163、Gly143、Glu166、Phe140等与N3分子形成多个氢键相互作用。N3分子上的Leu嵌入His41、Met49、Met165、Tyr54形成疏水分子口袋,而Ala则被Met165、Leu167的侧链包围,发生疏水相互作用。
使用6LU7晶体结构进行复原分子对接,得到的最佳打分结果为-8.40 kcal/mol。在PyMOL中观察复原对接的结合模式,并分别计算出不同构像与原配体间的RMSD[16]。二号构像与原配体最相似,且算得RMSD值为1.99 ?(<2 ?)(见图1),说明复原对接效果良好。上述结果表明,对接参数设置较为合理,复原对接获得成功。
[4] HALL D C,JI H F. A search for medications to treat COVID-19 via in silico molecular docking models of the SARS-CoV-2 spike glycoprotein and 3CL protease[J]. Travel medicine and infectious disease,2020,35:101646. doi: 10.1016/j.tmaid.2020.101646.
[5] RAJESH G,AYON C,ASHIS B,et al. Evaluation of green tea polyphenols as novel corona virus (SARS CoV-2)main protease(Mpro)inhibitors-an in silico docking and molecular dynamics simulation study[J]. Journal of biomolecular structure & dynamics,2020,760:1-13
[6] 賡迪,庞月笙,陶志,等.基于分子对接技术探讨金莲花汤抗新型冠状病毒(2019-nCoV)的潜力 [J].中国现代中药,2020,22(04):522-532.
[7] 吉米丽汗·司马依,买买提明·努尔买买提,艾尼瓦尔·吾买尔,等.基于网络药理学及分子对接探索金花清感颗粒辅助治疗新型冠状病毒肺炎(COVID-19)活性成分研究[J].中药材,2020, (05):1275-1283.
[8] 刘千勇,王晓良.新型冠状病毒(2019-nCoV)的靶向药物研究策略[J].药学学报,2020,55(02): 181-188.
[9] 张超,陈姝冰,张洁,等.浅析注册用于新冠肺炎治疗的临床试验药物[J].药学学报,2020, 55(03):355-365.
[10] TROTT O,OLSON A J. AutoDock Vina:improving the speed and accuracy of docking with a new scoring function,efficient optimization,and multithreading[J]. Journal of Computational Chemistry,2010,31(2):455-61.
[11] FORLI S,HUEY R,PIQUE M E,et al. Computational protein-ligand docking and virtual drug screening with the AutoDock suite[J]. Nature Protocols,2016,11(5):905-919.
[12] LASKOWSKI R A,SWINDELLS M B. LigPlot+:multiple ligand-protein interaction diagrams for drug discovery[J].Journal of Chemical Information and Modeling,2011,51(10):2778-2786.
[13] LEE J H,HEO J H,CHANG S O,et al. Reactive blue 2,an antagonist of rat P2Y4,increases K+ secretion in rat cochlea strial marginal cells[J]. Hearing Research,2006,219(1):66-73.
[14] O'BOYLE N M,BANCK M,JAMES C A,et al. Open Babel:An open chemical toolbox[J]. Journal of cheminformatics,2011,3:33.
[15] JIN Z,DU X,XU Y,et al. Structure of M(pro)from SARS-CoV-2 and discovery of its inhibitors[J]. Nature,2020,582(7811):289-293.
[16] BRUSCHWEILER R. Efficient RMSD measures for the comparison of two molecular ensembles.Root-mean-square deviation[J]. Proteins,2003,50(1):26-34.
[17] MARUYAMA K. Interaction of 4-carboxy-2-hydroxymuconate-6-semialdehyde dehydrogenase with Reactive Blue 2 and related dyes[J]. Journal of Biochemistry,1988,103(4):714-721.
[18] CHANDLER C E, WILDER D E,PETTINI J L, et al. CP-346086:an MTP inhibitor that lowers plasma cholesterol and triglycerides in experimental animals and in humans[J]. Journal of Lipid Research,2003,44(10):1887-1901.
[19] WIESBECK G A,WEIJERS H-G,CHICK J,et al. The effects of ritanserin on mood, sleep, vigilance, clinical impression, and social functioning in alcohol-dependent individuals. Ritanserin in Alcoholism Work Group [J]. Alcohol and Alcoholism,2000,35(4):384-389.
[20] SCOTT A K. Dihydroergotamine:a review of its use in the treatment of migraine and other headaches[J]. Clinical Neuropharmacology,1992,15(4):289-296.
[21] DOMAN T N,MCGOVERN S L,WITHERBEE B J,et al.Molecular docking and high-throughput screening for novel inhibitors of protein tyrosine phosphatas -1B[J]. Journal of Medicinal Chemistry,2002,45(11):2213-2221.
责任编辑 王继国
